È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Sedimentazione equilibrio di una membrana Piccolo Oligomer-forming Proteine: Effetto di istidina protonazione su pentamerica stabilità
In questo articolo
Riepilogo
Sedimentation equilibrium (SE) can be used to study protein-protein interactions in a physiological environment. This manuscript describes the use of this technique to determine the effect of pH on the stability of a homo-pentamer formed by the small hydrophobic (SH) protein encoded by the human syncytial respiratory virus (hRSV).
Abstract
Ultracentrifugazione analitica (AUC) può essere usato per studiare le interazioni reversibili tra macromolecole su una vasta gamma di forze di interazione e in condizioni fisiologiche. Questo rende AUC un metodo di scelta per valutare quantitativamente stechiometria e termodinamica di omo- ed etero-associazione che sono transitori e reversibili in processi biochimici. Nella modalità di sedimentazione equilibrio (SE), un equilibrio tra diffusione e sedimentazione fornisce un profilo in funzione della distanza radiale che dipende da un modello di associazione specifico. Qui, un protocollo dettagliato SE è descritto per determinare le dimensioni e monomero-monomero associazione energia di una piccola proteina di membrana oligomero utilizzando un'ultracentrifuga analitica. AUC-ES è privo di etichetta, basata solo su principi fisici, e può essere utilizzato su entrambe le proteine solubili e di membrana acqua. Un esempio è mostrato di quest'ultimo, la piccola idrofoba (SH) proteina del virus sinciziale respiratorio umano (HRSV), Un polipeptide di 65 aminoacidi con un singolo transmembrana (TM) dominio α-elica che forma canali ionici pentamerica. Dati strutturali a base di NMR mostra che la proteina SH ha due protonatable suoi residui nel suo dominio transmembrana che sono orientate ad affrontare il lume del canale. Esperimenti SE sono stati progettati per determinare come pH influenza costante di associazione e la dimensione oligomerica di proteine SH. Mentre la forma pentamerica stata conservata in tutti i casi, la costante associazione è stata ridotta a pH basso. Questi dati sono in accordo con una simile dipendenza pH osservato per l'attività del canale SH, in linea con un orientamento lumenal dei due suoi residui in proteine SH. Quest'ultima può verificare la repulsione elettrostatica e ridotto la stabilità oligomeri a basso pH. In sintesi, questo metodo è applicabile quando informazioni quantitative sottili proteina-proteina associazione cambiamenti nelle condizioni fisiologiche devono essere misurate.
Introduzione
Ultracentrifugazione analitica 1-5 è uno dei metodi più importanti per studiare le interazioni di macromolecole in condizioni fisiologiche, essendo accessibile sia interazioni deboli e forti. Il metodo è privo di etichetta e utilizza assorbimento della luce o interferenze, e anche sistemi ottici fluorescenza può essere utilizzata per accedere intervalli di concentrazione di diversi ordini di grandezza 6.
Questo metodo è particolarmente utile in quanto la maggior parte dei processi biochimici dipendono interazioni reversibili. La stechiometria e la forza di tali interazioni devono essere caratterizzate quantitativamente comprendere processi biologici, e un certo numero di metodi esistono per questo scopo 7, 8. Tuttavia, interazioni transienti sono difficili da studiare 9.
La scelta di un metodo per caratterizzare le interazioni macromolecolari dipende dalla sua natura statica o dinamica. Nel primo caso, Sedim velocità entazione (SV) viene utilizzato, dove la velocità di trasporto radiale viene misurato e complessi sono frazionata sulla base delle differenze di massa galleggiante e forma.
Al contrario, le associazioni dinamiche che sono reversibili sulla scala temporale dell'esperimento non possono essere separati fisicamente. In questo caso, auto o etero-interazioni conducono a interazioni non-covalenti sono in un equilibrio che dipende dalla concentrazione totale di proteine. Queste interazioni dinamiche possono essere studiati sia equilibrio di sedimentazione (SE) e la velocità di sedimentazione (SV) 10. Tuttavia, il primo metodo è semplice da eseguire e viene descritto qui. In SE, centrifugazione viene eseguita ad una velocità sufficientemente bassa in modo da raggiungere un equilibrio tra diffusione e sedimentazione. A questo punto, il profilo di equilibrio di un segnale ottico (UV-VIS) in funzione della distanza radiale, può essere analizzato usando modelli termodinamici pre-impostato per associazioni 11.
ve_content "> Nel presente documento, uno studio sedimentazione equilibrio è presentato di auto-associazione di una proteina di membrana virale che forma canali ionici. A causa della sua idrofobicità, l'esperimento viene eseguito in presenza di detergente, e in questo caso la densità di solvente deve essere abbinato a quello del detergente. Tuttavia, il protocollo descritto sarebbe identica nel caso di una proteina solubile in acqua, salvo che nessuna corrispondenza densità solvente sarebbe necessaria.La proteina utilizzata è codificato nel virus sinciziale respiratorio umano (HRSV), un pneumovirus avvolto nel famiglia Paramyxoviridae che causa malattie respiratorie del tratto inferiore nei bambini, anziani e delle popolazioni di tutto il mondo 12 immunocompromessi. Fino a 64 milioni di casi segnalati di infezione HRSV e 160.000 decessi si verificano ogni anno.
Il genoma HRSV trascrive 11 proteine, comprese le tre proteine di membrana F, G, e piccola idrofoba (SH). Proteina SH è coinvoltanella patogenesi dell'infezione RSV. RSV manca il gene SH (RSVΔSH) è vitale, ha causato la formazione di sincizi e crebbe così come la wild-type (WT) virus 13-16. Tuttavia, virus RSVΔSH replicato 10 volte meno efficiente rispetto al WT nel tratto respiratorio superiore 15, 16. Inoltre, il virus è stato attenuato in RSVΔSH nel topo vivo e modelli scimpanzé 13, 17.
La proteina SH è un (RSV sottogruppo A) 64 o 65 (RSV sottogruppo B) aminoacidi lungo tipo II proteina integrale di membrana che si accumula prevalentemente alle membrane del vano 18 Golgi. Proteina SH ha un unico previsto a-elica transmembrana (TM) di dominio 19, che è altamente conservata 20,21. I domini extramembrane C- e N-terminale sono orientati lumenally / extracellulare e cytoplasmically rispettivamente.
Entrambi dominio TM sintetico (residui 18-43) E lunghezza piena SH proteine hanno dimostrato di formare homopentamers in una varietà di detergenti. La forma homopentameric è responsabile per l'attività del canale in doppi strati lipidici planari 22,23. Il corretto orientamento dei monomeri TM nel doppio strato lipidico è stato determinato in primo luogo con specifico sito dicroismo infrarossi 23, che ha mostrato la sua-22 per essere in un lumenal, vicino a inter-elica, orientamento. Lo stesso orientamento dominio TM è stato confermato da studi NMR che ricostruibili la pentamerica a-elica fascio della proteina intera lunghezza in dodecylphosphocholine (DPC) micelle 22. In questo modello di 'micelle', un singolo a- dominio TM elicoidale era affiancato N-terminale da un a-elica, e C-terminale da un esteso b-tornante. I due residui protonatable di proteine SH, His-22 e His-51, si trovano nel dominio TM (lumenally orientato), e sulla punta del extramembrane C-terminale β forcella (lontano dal poro canale), rispettivamente. In un enviro bicellarnment, tuttavia, il TM α-elica si estende fino a His-51, ed entrambi i suoi residui sono accessibili al lume del canale 24. La struttura del canale adotta un imbuto architettura 22, dove la strozzatura (Ser-Cys-29 a 45) 22 è allineato con catene laterali idrofobiche (Ile-32, Ile-36, Ile-Leu-40 e 44), e Ile-36 definisce il punto più stretto nel lume del canale. His-22 si trova presso la più grande apertura di questo imbuto, che His-51 è sulla punta della piccola apertura.
Nel presente documento, centrifugazione analitica in una modalità sedimentazione equilibrio è stato usato per determinare se la sua protonazione influisce sulla stabilità della proteina pentamero SH. In questo caso, la proteina SH è stato solubilizzato in detersivo C14-betaina, che è stato utilizzato in precedenza per dimostrare che le forme della proteina SH oligomeri pentamerica 22.
Protocollo
Questo protocollo si basa sulle seguenti risorse, che devono essere di cui per ulteriori dettagli e considerazioni particolari 3, 25-28.
1. Densità corrispondenza di micelle detergenti con 2 H 2 O
Nota: La densità della soluzione tampone deve essere abbinato alla densità delle micelle detergenti. Agenti comuni densità-regolazione includono 2 H 2 O, H 2 18 O, 2 H 2 O 18, glicerolo e saccarosio 29. H 2 O 18 ha la stessa densità 2 H 2 O e può essere una scelta migliore se deuterazione di protoni scambiabili nella proteina non è desiderato. In questa procedura, la densità di 3- (N, N-dimethylmyristylammonio) -propanesulfonate (C14SB) detersivo in 50 mM Tris pH 7,3, NaCl 100 mM sarà abbinato con 2 H 2 O. Come un tentativo iniziale le seguenti concentrazioni di2 H 2 O sarà utilizzato: 10, 30, e 50% v / v.
1.1. Preparazione del campione
- Preparare le seguenti soluzioni di magazzino e filtro sterilizzare attraverso un filtro siringa da 0,2 micron: 50 mL Tris 500 mM pH 7,3 e 1 M NaCl (10X soluzione tampone); 1 ml 250 C14SB mm (soluzione detergente 50X).
- Preparare 200 ml di soluzione campione mescolando 20 microlitri 10X tampone, 4 microlitri soluzione detergente 50X, 20 microlitri 2 H 2 O (99,9%), e 156 microlitri deionizzata H 2 O. Anche preparare 200 ml di soluzione di riferimento mescolando 20 microlitri tampone 10X, 20 microlitri 2 H 2 O (99,9%), e 160 microlitri deionizzata H 2 O.
- Ripetere il punto 1.1.2 per le altre 2 H 2 O concentrazioni, cioè il 30% e il 50%, regolando gli importi 2 H 2 O e H 2 O in modo appropriato.
1.2. Assemblaggio di 6 canali AUC cellule e caricamento dei campioni nelle cellule.
Nota: Ci sono due tipi di cellule AUC seconda del metodo di caricamento del campione. Le cellule senza riempimento esterno deve essere caricato prima della sigillatura della cella, mentre esterno-riempiono le celle possono essere caricati dopo che le cellule sono sigillati. Assemblaggio di una cella AUC esterna riempimento è stato descritto in precedenza 3. In questo protocollo, il montaggio di una cella di 6 canali AUC senza riempimento esterno è descritto. La differenza principale è che ha anelli vite su entrambi i lati che devono essere serrate separatamente e non ha bisogno di tappi di alloggiamento (Fig. 1). La differenza di fasi di montaggio sono evidenziati di seguito.
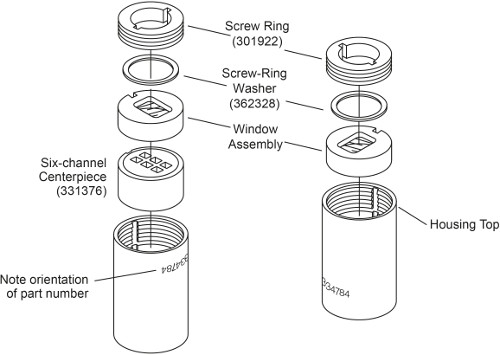
Figura 1. vista di una cella di AUC 6 canali senza riempimento esterno Esploso. Questo dato è stato modificato dalla Beckman Coulter An-50 Ti e An-60 Ti Analytical Rotor, Celle, e l'utente elev manuale.
- Preparare due finestre per ogni cella AUC con finestra zaffiro invece di finestra di quarzo (Fig. 2). Posizionare la guarnizione finestra nel supporto finestra. Piegare leggermente il rivestimento finestra e posizionarlo nel supporto finestra in modo che il gap si forma di fronte alla scanalatura titolare finestra. Posizionare la finestra di zaffiro all'interno della fodera finestra, allineando il segno con la chiavetta porta finestra.
Nota:. La finestra di quarzo è comprimibile e quindi produrrà rifrazione più luce ad alta velocità 28, 30 Pertanto per misure di interferenza sopra 30000 rpm, come in questo esperimento corrispondenza densità, sono utilizzati finestre zaffiro. Una finestra di zaffiro è più pesante finestra di quarzo e ha una "X" inciso su un lato.
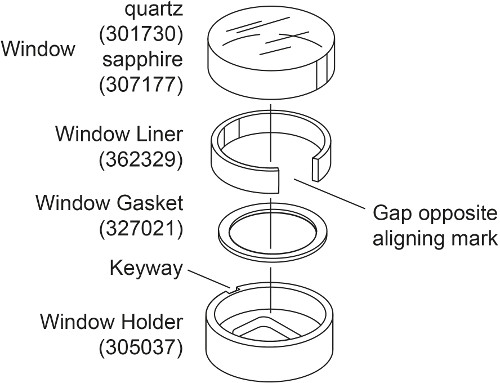
Figura 2. Esploso of l'assemblea finestra. Questo dato è stato modificato dalla Beckman Coulter An-50 Ti e An-60 Ti Analytical Rotor, Celle, e manuale utente elev
- Posizionare la custodia cella con il numero di parte capovolta. Con le chiavette allineate con la chiave alloggi, far scorrere nel corpo cellule in primo luogo un centrotavola 6-settore con il lato smussato verso il basso, seguita da una assemblea finestra con la finestra rivolta verso il basso (Fig. 1, a sinistra).
- Leggermente cappotto la rondella vite discussioni ring e ghiera con spinkote. Inserire una rondella anello vite sulla parte superiore del gruppo finestra. Installare l'anello vite nell'alloggiamento finestra con la scritta "OUT" rivolto verso l'esterno. Serrare l'anello di vite utilizzando lo strumento di allineamento delle cellule.
- Utilizzando la chiave coppia, serrare la ghiera di soli 60 pollici-libbre.
- Posizionare la cella con il numero di parte in posizione verticale e posizionato a 12-ore. Load 120 microlitri riferimento nelle righe di sinistra e di 110 campioni microlitri nelle righe giuste. Assicurarsi che ogni campione e il riferimento è abbinato correttamente.
Nota: volume del campione esatta non è critica, ma il riferimento deve avere poco più volume di campione (5-10 microlitri) in modo che il menisco campione sarà distinti. - Far scorrere con attenzione nel gruppo alloggiamento della cella una finestra con la finestra rivolta verso il basso (Fig. 1, a destra). Fare attenzione a non disturbare la cellula eccessivamente e versare il contenuto.
- Ripetere il passaggio 1.2. 3 e 1.2. 4, stringendo il secondo anello vite a 120 pollici-libbre. Invertire la cella e serrare il primo anello della vite a 120 pollici-libbre.
- Caricare le cellule nel rotore, installare il rotore nella centrifuga e installare il monocromatore secondo le istruzioni del produttore 28.
Nota: Dettagli su questo step si possono trovare anche in questo riferimento 3.
1.3. Impostazione misura interferenze
- Avviare il software di interfaccia utente per lo strumento AUC ed eseguire l'installazione e la calibrazione laser radiale per ogni cella a 3000 rpm, secondo le istruzioni del produttore, che saranno brevemente riassunti nel passaggio seguente.
- 1.3.1.1 Dopo vuoto sufficiente è stato raggiunto (<100 micron), eseguire il centrifugare a 3000 rpm. Visualizzare in anteprima il modello di interferenza nel software di interfaccia utente e regolare i parametri del laser per ottenere il massimo contrasto.
- Creare un nuovo file creato (File | Nuovo File) specificando "Equilibrio" e "interferenze" di misura. Impostare un metodo di sedimentazione di equilibrio (tasto "Method") per l'esecuzione a 45000 rpm oppure quella più alta prevista per i campioni di proteine, se superiore, con la temperatura corsa a 20 ° C e raccogliere 1 scansione ogni 15 minuti. Monitor equilibrio progressi aprendo i file di dati in HeteroAnalysis e selezionando la funzione "Match", dopo almeno 12 ore (circa durante la notte, Fig. 3).
Nota: una funzione simile è disponibile in SEDFIT (Opzioni | Caricamento Opzioni | Prova approccio alla Equilibrium) anche.

Figura 3. Risultato funzione HeteroAnalysis Match. La funzione match può essere utilizzata per monitorare i progressi equilibrio confrontando RMSD tra le scansioni successive e l'ultima scansione. Questo esempio mostra raggiungimento dell'equilibrio dopo 8 ore come indicato dai valori RMSD asintotica all'asse X.
1.4. Analisi dei dati
- Per ciascuna serie di campioni, tracciare la pendenza del profilo di distribuzione radiale contro la concentrazione di H 2 O 2.
Nota: La distribuzione sarà molto superficiale esponenziale che apapprocci linearità. Asse X intercetta corrisponde alla concentrazione di H 2 O 2 corrispondente. - Per precisare, eseguire l'esperimento in più repliche. In alternativa, ripetere l'esperimento con una gamma più ristretta di 2 H 2 O concentrazione.
2. sedimentazione equilibrio SH in micelle C14SB
2.1. Parametri della corsa
- Calcolare la densità e la viscosità del buffer, la proteina volume specifico parziale e velocità di centrifugazione utilizzando SEDNTERP. Per calcolare la densità e la viscosità del buffer, selezionare Calcola sezione "Buffer Select" e inserire i componenti tampone di conseguenza, tra cui il D 2 O concentrazione.
- 2.1.1.1 Per calcolare proteina volume specifico parziale, selezionare Calcola sezione "V-bar" e inserire la sequenza aminoacidica della proteina. Specificare la dimensione oligomerica più alto previsto in "Crea un oligomero da questo monomero: N ="campo, in questo caso N = 5. Calcolare la velocità inserendo valori nel campo RPM nella finestra principale fino σ ≈ 1; questa è una regola per garantire una buona forma esponenziale del profilo di distribuzione radiale 25.
Nota: I valori calcolati per questo esperimento è la seguente: ρ = 1,03839 g / ml, η = 1,0267 CP = 0,7569 ml / g, ω 1 = 16000 rpm. - Calcolare velocità successivi da seguire per garantire sufficiente differenza tra il profilo di distribuzione ad una velocità e il successivo 25.
Nota: questo può essere fatto anche dalla funzione "Stimare rotore equilibrio velocità funzione" in SEDFIT, che tiene conto della colonna soluzione (volume di riempimento).
2.2. Preparazione dei campioni
- Preparare 1 ml di soluzione di riferimento con 5 C14SB mM e il 32,3% 2 H 2 O, come determinato da esperimento corrispondenza densità (sezione 1), mescolando 100 microlitri di buffer 10X solution (fase 1.1.1), 20 microlitri soluzione 50X detergente (fase 1.1.1), 323 microlitri 2 H 2 O (99,9%) e 527 microlitri deionizzata H 2 O.
- Sciogliere liofilizzato, peptidi SH-HPLC purificato (espressione e purificazione descritto in precedenza 31) in opportuno solvente come metanolo o 50% v / v acetonitrile acquoso. Misurare A280 dei peptidi disciolti in un microlitro scala UV spettrofotometro / Vis e aliquota per tre campioni per dare 280 A, 12mm = 0,3, 0,5, e 0,8 (A 280, 10mm = 0,25, 0,417 e 0,67) ogni volta diluito per 130 ml. Lyophilize i campioni durante la notte e risospendere in 130 ml soluzione di riferimento (punto 2.2.1) per dare le soluzioni campione.
Nota: proteina SH può essere rilevato da UV / Vis assorbanza a 280 nm perché contiene Trp e Tyr residui. Proteine senza residui aromatici possono essere rilevati da loro tagging con un cromoforo adeguato, utilizzando un mutante Trp contenente, o utilizzando interferiscemisure SNO invece di assorbanza. - Seguire i passaggi nella sezione 1.2 per assemblare una cella AUC 6 canali con finestre di quarzo. Caricare il campione di concentrazione più alta (A 280, 12mm = 0.8) nel canale più vicino al centro del rotore e disponibilità campione concentrazione (A 280, 12mm = 0,3) più lontana dal centro del rotore.
2.3. Impostazione misure di assorbanza
- Creare un nuovo file creato (File | Nuovo File) specificando "Equilibrium" e valutazione "assorbanza". Specifica 280 nm come lunghezza d'onda del rivelatore.
- Eseguire la calibrazione radiale a 3000 rpm controllando "calibratura Radial prima prima scansione" in Opzioni di scansione, specificando la raccolta di dati a bassa risoluzione, ad esempio, con passo radiale = 0,01 centimetri, Repliche = 3 (bassa risoluzione, veloce), e l'esecuzione di una scansione singola . Dopo la scansione, deselezionare l'opzione.
- Impostare un metodo di sedimentazione di equilibrio ("Methotasto ") d per girare alla prima velocità calcolata al punto 2.1.3, a 20 ° C, e raccogliere 1 scansione ogni 30 minuti. In "Detail" reciproche cellule, specificare la raccolta di dati a bassa risoluzione come al punto 2.3.2. Monitorare i progressi equilibrio aprendo i file di dati in HeteroAnalysis e selezionando la funzione "Match", dopo almeno 18 ore (circa durante la notte, Fig. 4).
Nota: raggiungimento dell'equilibrio può richiedere tempo sostanzialmente più lunga per la prima velocità, mentre le velocità successive avranno meno tempo.
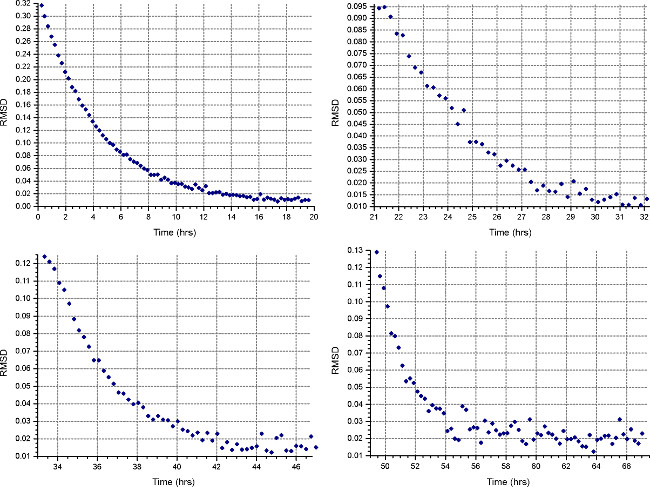
Figura 4. I risultati di funzione HeteroAnalysis Match. La prima e la seconda velocità (in alto a sinistra e destra) sembrano aver raggiunto l'equilibrio, ma è meglio aspettare qualche ora in più per essere sicuri. In confronto, il terzo e il quarto le velocità (in basso a sinistra ea destra) hanno chiaramente raggiunto l'equilibrioin un tempo più breve. Cliccate qui per vedere una versione più grande di questa figura.
- Una volta che l'equilibrio è stato raggiunto, raccogliere una singola scansione ad alta risoluzione, ad esempio con passo radiale = 0,001 centimetri, Repliche = 10 (alta risoluzione, lento).
- Dopo la scansione è completata, ripetere il punto 2.3.3 e 2.3.4 per la velocità successiva.
- Facoltativamente, quando il tempo necessario per raggiungere l'equilibrio per ogni velocità è noto (calcolata o per esperienza), impostare il metodo di scansione di sedimentazione di equilibrio a tutte le velocità calcolate nel passo 2.1.3 e per raccogliere 1 scansione dopo il tempo di equilibrio per ogni velocità. In questo caso, specificare raccolta di dati ad alta risoluzione in "dettaglio" reciproche cellule.
2.4. L'analisi dei dati in SEDFIT e SEDPHAT
Nota: Per ulteriori dettagli e considerazioni in lettore di analisi dei dati si riferisce to il seguente sito web: www.analyticalultracentrifugation.com.
- Aperte le scansioni ad alta risoluzione in SEDFIT (Dati | dati carico sedimentazione di equilibrio) e dividere i dati in 3 canali (queste corrispondono a diverse concentrazioni per ogni campione; Opzioni | Caricamento Opzioni | Salva 6 canali di dati grezzi in 3 sottoinsiemi).
- Riaprire i file di dati che appartengono allo stesso campione e la stessa concentrazione, ma diverse velocità in SEDFIT. Regolare il menisco (linea rossa verticale), fondo cella (verticale linea blu) e limiti di montaggio (linee verdi verticali), ed esportare i dati per l'utilizzo in SEDPHAT (Dati | Esportazione dati per SEDPHAT). Ingresso i parametri calcolati nel passaggio 2.1.1, così come il tipo di rotore e tipo fulcro come richiesto. Ripetere questa operazione per ogni campione e la concentrazione.
- Aprire tutti i dati dallo stesso campione (tutti concentrazioni e velocità) in SEDPHAT e compilare parametri dell'esperimento; un esempio è mostrato in Fig. 5.
Nota: quando viene aggiunto D 2 O nel buffer, Deut razione di protoni scambiabili potrebbe alterare significativamente il peso molecolare della proteina, in particolare per le proteine solubili in acqua. Le proteine di membrana, in particolare quelle di piccole dimensioni come la proteina SH, sono meno colpiti perché le regioni di membrana-embedded sono protetti dallo scambio. Per correggere questo, inserire il "buffer D frazione molecolare".
Nota: A questo punto si consiglia di salvare il set di dati modificato separatamente selezionando menu Dati | copiare tutti i dati e salvare come nuova configurazione. - Selezionare un modello e compilare parametri globali per quel modello.
Nota: Ad esempio, il modello "monomero-n-Mer Self-associazione" ei suoi parametri sono mostrati in Fig. 6.
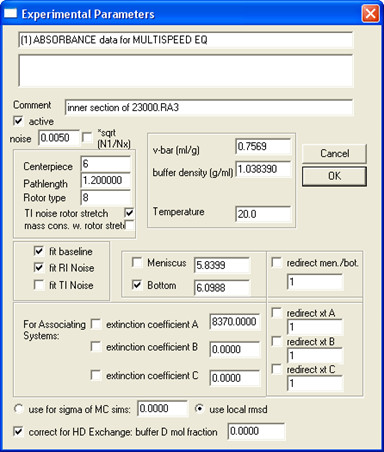
Figura 5. Un esempio su come compilare parametri sperimentali.
files / ftp_upload / 52404 / 52404fig6.jpg "/>
Figura 6. Un esempio di come compilare parametri globali per il modello monomero-n-Mer Self-Association.
- Eseguire un globale Fit selezionando il menu Fit | globale Fit e attendere che converge in forma. Annotare (o prendere uno screenshot) i risultati di montaggio, in particolare la riduzione del chi-quadrato globale e log K a valori. Estrarre altri dati, come i dati in forma e residui di montaggio dal menu Copia e display | display termodinamico informazioni.
- Torna a parametri globali e verificare M (1) in base al peso molecolare monomero e ripetere il punto 2.4.5. Annotare il peso molecolare, arredata e globale ridotta chi-quadrato.
- Ripetere il punto 2.4.3 a 2.4.6 per ogni modello da testare e confrontare la qualità adattamento di ogni modello confrontando il valore ridotto chi-quadro globale e residui di montaggio.
Nota: I piccoli e casuali residui di montaggio indica generalmente una buona misura, e il modello che si adatta meglio sarebbehanno la più piccola ridotta chi-quadrato globale. Il peso molecolare monomero arredata e il suo valore chi-quadrato non dovrebbero differire in modo sostanziale da quello del (teorica) di peso molecolare fisso. - Calcola l'intervallo di confidenza per il registro ottenuto Ka per valore selezionando prima Statistiche | Critical chi-quadrato per le proiezioni di superficie errore e inserendo l'intervallo di confidenza desiderato. Poi, vai a Statistiche | Generare 1-dimensionale proiezione in superficie di errore e deselezionare il login Ka in dialogo parametri globali per ottenere i valori di chi-quadrato per log Ka.
Nota: i lettori sono invitati a consultare le seguenti fonti (http://www.analyticalultracentrifugation.com/ sedphat / statistics.htm) per maggiori dettagli sul metodo 32 e illustrazione di questo metodo 33.
Risultati
Il profilo radiale distribuzione di C14SB micelle detergenti in Tris 50 mM, NaCl 100 mM pH 7.3 forma un esponenziali molto superficiale che può essere montato su un modello lineare (Figura 7A). La pendenza di questa distribuzione è inversamente correlato alla concentrazione di D 2 O (Figura 7B). Il punto in cui la pendenza è zero, vale a dire, l'abbinamento D 2 O concentrazione, è risultato essere 32,3%.
Vedi Fi...
Discussione
Questo documento fornisce un protocollo sperimentale per la preparazione e l'analisi di oligomerizzazione di una piccola proteina di membrana in detergente utilizzando equilibrio sedimentazione campione. Il protocollo descritto è ugualmente valido -e simpler- per proteine solubili, non è necessaria la fase corrispondente densità. Infatti, il sistema è costituito da una miscela di detergente e proteine. Per condurre studi sedimentazione, il detergente deve essere invisibile al campo gravitazionale modo che ...
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
This work has been funded by the National Research Foundation grant NRF-CRP4-2008-02 (J.T.) and Tier 1 grant RG 51/13.
Materiali
| Name | Company | Catalog Number | Comments |
| 3-(N,N-dimethylmyristylammonio)propanesulfonate | Sigma | T0807 | |
| Deuterium oxide 99.8% | Cambridge Isotope | DLM-4-99.8 | |
| An-50 Ti Rotor, Analytical, 8-Place | Beckman Coulter | 363782 | |
| An-60 Ti Rotor, Analytical, 4-Place | Beckman Coulter | 361964 | |
| Cell housing | Beckman Coulter | 334784 | |
| 12 mm six-channel centerpiece, epon charcoal-filled | Beckman Coulter | 331376 | |
| Window holder | Beckman Coulter | 305037 | |
| Window gasket | Beckman Coulter | 327021 | |
| Window liner | Beckman Coulter | 362329 | |
| Sapphire window | Beckman Coulter | 307177 | |
| Quartz window | Beckman Coulter | 301730 | |
| Screw-ring washer | Beckman Coulter | 362328 | |
| Screw ring | Beckman Coulter | 301922 | |
| Spinkote | Beckman Coulter | 306812 | |
| Torque stand assembly | Beckman Coulter | 361318 | |
| Counterbalance | Beckman Coulter | 360219 | |
| Cell alignment tool | Beckman Coulter | 362340 | |
| SEDNTERP | http://bitcwiki.sr.unh.edu/index.php/Main_Page | ||
| HeteroAnalysis | http://www.biotech.uconn.edu/auf/?i=aufftp | ||
| SEDFIT | http://www.analyticalultracentrifugation .com/sedfit.htm | ||
| SEDPHAT | http://www.analyticalultracentrifugation .com/sedphat/default.htm |
Riferimenti
- Laue, T. M., Stafford, W. F. Modern applications of analytical ultracentrifugation. Annu. Rev. Biophys. Biomol. Struct. 28, 75-100 (1999).
- Lebowitz, J., Lewis, M. S., Schuck, P. Modern analytical ultracentrifugation in protein science: A tutorial review. Protein Sci. 11, 2067-2079 (2002).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. . Assembly, Loading, and Alignment of an Analytical Ultracentrifuge Sample Cell. , e1530 (2009).
- Rivas, G., Stafford, W., Minton, A. P. Characterization of heterologous protein-protein interactions using analytical ultracentrifugation. Methods-a Companion to Methods in Enzymology. 19, 194-212 (1999).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for association and assembly the study of protein. Curr. Opin. Chem. Biol. 10, 430-436 (2006).
- MacGregor, I. K., Anderson, A. L., Laue, T. M. Fluorescence detection for the XLI analytical ultracentrifuge. Biophys. Chem. 108, 165-185 (2004).
- Phizicky, E. M., Fields, S. Protein-Protein Interactions - Methods for Detection and Analysis. Microbiol. Rev. 59, 94-123 (1995).
- Alexandrov, A. A facile method for high-throughput co-expression of protein pairs. Mol. Cell. Proteomics. 3, 934-938 (2004).
- Nooren, I. M. A., Thornton, J. M. Structural characterisation and functional significance of transient protein-protein interactions. J. Mol. Biol. 325, 991-1018 (2003).
- Ebel, C. Sedimentation velocity to characterize surfactants and solubilized membrane proteins. Methods. 54, 56-66 (2011).
- Minton, A. P. Quantitative characterization of reversible macromolecular associations via sedimentation equilibrium: an introduction. Exp. Mol. Med. 32, 1-5 (2000).
- Dowell, S. F. Respiratory syncytial virus is an important cause of community-acquired lower respiratory infection among hospitalized adults. J. Infect. Dis. 174, 456-462 (1996).
- Bukreyev, A., Whitehead, S. S., Murphy, B. R., Collins, P. L. Recombinant respiratory syncytial virus from which the entire SH gene has been deleted grows efficiently in cell culture and exhibits site-specific attenuation in the respiratory tract of the mouse. J. Virol. 71, 8973-8982 (1997).
- Fuentes, S., Tran, K. C., Luthra, P., Teng, M. N., He, B. Function of the respiratory syncytial virus small hydrophobic protein. J. Virol. 81, 8361-8366 (2007).
- Jin, H. Recombinant respiratory syncytial viruses with deletions in the NS1, NS2, SH, and M2-2 genes are attenuated in vitro and in vivo. Virology. 273, 210-218 (2000).
- Karron, R. A. Respiratory syncytial virus (RSV) SH and G proteins are not essential for viral replication in vitro: clinical evaluation and molecular characterization of a cold-passaged, attenuated RSV subgroup B. Proc. Nat. Acad. Sci. USA. 94, 13961-13966 (1997).
- Whitehead, S. S. Recombinant respiratory syncytial virus bearing a deletion of either the NS2 or SH gene is attenuated in chimpanzees. J. Virol. 73, 3438-3442 (1999).
- Rixon, H. W. The small hydrophobic (SH) protein accumulates within lipid-raft structures of the Golgi complex during respiratory syncytial virus infection. J. Gen. Virol. 85, 1153-1165 (2004).
- Collins, P. L., Mottet, G. Membrane orientation and oligomerization of the small hydrophobic protein of human respiratory syncytial virus. J. Gen. Virol. 74, 1445-1450 (1993).
- Collins, P. L., Olmsted, R. A., Johnson, P. R. The small hydrophobic protein of human respiratory syncytial virus: comparison between antigenic subgroups A and B. J. Gen. Virol. 71, 1571-1576 (1990).
- Chen, M. D., Vazquez, M., Buonocore, L., Kahn, J. S. Conservation of the respiratory syncytial virus SH gene. J. Infect. Dis. 182, 1228-1233 (2000).
- Gan, S. W. The small hydrophobic protein of the human respiratory syncytial virus forms pentameric ion channels. J. Biol. Chem. 287, 24671-24689 (2012).
- Gan, S. W., Ng, L., Xin, L., Gong, X., Torres, J. Structure and ion channel activity of the human respiratory syncytial virus (hRSV) small hydrophobic protein transmembrane domain. Protein Sci. 17, 813-820 (2008).
- Li, Y. Inhibition of the Human Respiratory Syncytial Virus Small Hydrophobic Protein and Structural variations in a bicelle environment. J. Virol. 88 (22), 11899-914 (2014).
- Burgess, N. K., Stanley, A. M., Fleming, K. G. Determination of membrane protein molecular weights and association equilibrium constants using sedimentation equilibrium and sedimentation velocity. Meth. Cell. Biol. 84, 181-211 (2008).
- Cole, J. L., Lary, J. W., Moody, T. P., Laue, T. M. Analytical Ultracentrifugation: Sedimentation Velocity and Sedimentation Equilibrium. Meth. Cell. Biol. 84, 143-179 (2008).
- Fleming, K. G. Determination of membrane protein molecular weight using sedimentation equilibrium analytical ultracentrifugation. Curr. Protoc. Prot. Sci. 53, 17.12.11-17.12.13 (2008).
- . . An-50 Ti and An-60 Ti Analytical Rotor, Cells, and Counterbalance. , (2005).
- Mayer, G. Studying membrane proteins in detergent solution by analytical ultracentrifugation: Different methods for density matching. Prog. Colloid Polym. Sci. 113, 176-181 (1999).
- Laue, T. Ch. 20.3. Current Protocols in Protein Science. 20, 20.23.21-20.23.13 (2001).
- Gan, S. W. The Small Hydrophobic Protein Of The Human Respiratory Syncytial Virus Forms Pentameric Ion Channels. J. Biol. Chem. 287, 24671-24689 (2012).
- Bevington, P. R., Robinson, D. K. . Data reduction and error analysis for the physical sciences. 336, (1969).
- Schuck, P., Radu, C. G., Ward, E. S. Sedimentation equilibrium analysis of recombinant mouse FcRn with murine IgG1. Molecular Immunology. 36, 1117-1125 (1999).
- Gan, S. W., Vararattanavech, A., Nordin, N., Eshaghi, S., Torres, J. A cost-effective method for simultaneous homo-oligomeric size determination and monodispersity conditions for membrane proteins. Anal. Biochem. 416, 100-106 (2011).
- Montserret, R. NMR structure and ion channel activity of the p7 protein from hepatitis C virus). J. Biol. Chem. 285, 31446-31461 (2010).
- Stouffer, A. L., DeGrado, W. F., Lear, J. D. Analytical Ultracentrifugation Studies of the Influenza M2 Homotetramerization Equilibrium in Detergent Solutions. Progr Colloid Polym Sci. 131, 108-115 (2006).
- Sorkin, A., von Zastrow, M. Signal transduction and endocytosis: Close encounters of many kinds. Nat. Rev. Mol. Cell Biol. 3, 600-614 (2002).
- Gan, S. W., Ng, L., Lin, X., Gong, X., Torres, J. Structure and ion channel activity of the human respiratory syncytial virus (hRSV) small hydrophobic protein transmembrane domain. Protein science : a publication of the Protein Society. 17, 813-820 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon