A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
Protocol עבור שלב מוצק סינתזה של אוליגומרים של רנ"א המכיל 2 '
In This Article
Summary
מאמר זה מספק הליך מפורט על סינתזה שלב מוצק, טיהור, אפיון של dodecamers של רנ"א שונה ב C2'- O -position. UV- מול וניתוח מעגלי דיכרואי פוטומטרי משמשים לכמת ולאפיין היבטים מבניים, כלומר , גדילים בודדים או פעמיים גדילים.
Abstract
סינתזה של שלב מוצק שימשה להשגת פולימרים קנוניים ומשתנים של חומצות גרעין, במיוחד של DNA או RNA, שהפכו אותה למתודולוגיה עממית ליישומים בתחומים שונים ולמטרות מחקר שונות. הנוהל המתואר כאן מתמקד בסינתזה, טיהור, אפיון של dodecamers של RNA 5 '- [CUA CGG AAU CAU] -3' המכיל אפס, אחד, או שני שינויים הממוקם ב C2'- O -position. בדיקות מבוססים על קבוצות 2-thiophenylmethyl, משולבים לתוך נוקליאוטידים RNA באמצעות סינתזה אורגנית סטנדרטית הציג לתוך oligonucleotides המקביל באמצעות phosphoramidites שלהם בהתאמה. דו"ח זה עושה שימוש כימית phosphoramidite באמצעות הארבעה nucleobases הקנונית (uridine (U), ציטוזין (C), guanosine (G), אדנוזין (א)), כמו גם 2-thiophenylmethyl נוקלאוטידים פונקציונליים השונים על 2'- O - עמדה; עם זאת, המתודולוגיה היא מקובלת עבור var גדולשינויי ההתפתחות שהתפתחו במשך השנים. את oligonucleotides היו מסונתז על זכוכית מבוקר הנקבוביות (CPG) תמיכה ואחריו מחשוף של שרף deprotection בתנאים סטנדרטיים, כלומר , תערובת של אמוניה מתילאמין (AMA) ואחריו מימן פלואוריד / טריאתילאמין / N-methylpyrrolidinone. Oligonucleotides המקביל היו מטוהרים באמצעות electroporesis polyacrylamide (20% denaturing) ואחריו elution, desalting, ובידוד באמצעות כרומטוגרפיה הפוכה שלב (ספטמבר, פאק, C 18 -Cumnumn). כימות ופרמטרים מבניים הוערכו באמצעות ניתוח אולטרה-סגול גלוי (UV-V) ו- Dycroism (CD), בהתאמה. דו"ח זה נועד לשמש משאב ומדריך לחוקרים מתחילים ומבקשים המעוניינים לצאת לתחום זה. הוא צפוי לשמש עבודה ב-התקדמות כמו טכנולוגיות מתודולוגיות חדשות מפותחות. תיאור המתודולוגיות והטכניקות שבתוךשל מסמך המתאים DNA / RNA סינתיסייזר (שופץ ונרכש בשנת 2013) המשתמשת כימיה phosphoramidite.
Introduction
סינתזה שלב מוצק כדי להשיג oligonucleotides של DNA / RNA הוא כלי רב עוצמה אשר שימש מספר יישומים בתחומים שונים מאז 1970s 1 , 2 , 3 באמצעות אבני הבניין phosphoramidite 4 . דוגמאות להשפעה הרחבה שלה כוללות: השפעתה על תיוג ( באמצעות תגובת כימיה בלחץ) 5 , בדיקה מבנית 6 , וטכנולוגיות אנטיסנס 7 , כמו גם הבהרה של מנגנונים ביולוגיים 8 , 9 , מקור כחומר גנטי 10 , שונה טבעי ו / או שינויים כימיים 11 , 12 , בין רבים אחרים.השינוי שאנו משתמשים כאן מייצג את הצעד הראשון במאמצינו להשיג oligonucleotides RNA המכיליםבדיקות פוטואקטיביות כדי לאפשר שליטה זמנית של המבנה והפונקציה של biopolymer חשוב זה.
סינתזה של Dodecamers RNA עם רצפים: 5 '- [CUA CG G A AU CAU] -3' / 5 '- [AUG AUU CCG UAG] -3' (עמדות קו תחתון מייצגות שילוב של שינוי C2'- O -thiophenylmethyl ) מהווה את המוקד של מחקר זה. רצפים נבחרו כדי לאפשר כימות ומדידה של גדילי רנ"א כמו גדילי יחיד, או כמו מבנים דופלקס המקביל שלהם (שום מבנים משניים אחרים לא חזו יציבות יציבה תרמודינמית). CD שימש כדי לקבוע את הפרמטרים המבניים, כלומר , היווצרות דופלקס ומעברים denaturation תרמית.
סִינתֶזָה
ההליך הכולל להשגת אלה oligonucleotides מודגם באיור 1 ו עוקב את התהליך בשלבים: אוטומטי שלב מוצק סינתיזה → DeprOtection → טיהור → כימות → אפיון. איור 2 מציג את היחידות המונומריות הנחוצות בהליך זה. הסינתזה מוצק השלב של RNA היא דומה לזה של ה- DNA בכך שהיא מבוססת על כימית phosphoramidite (איור 2, משמאל) את השימוש בקבוצות הגנה-רָפִיף בסיס האמין exocyclic nucleophilic על G, A ו- C, למשל, אצטיל, בנזואיל, phenoxyacetyl, t- בוטיל או N , N- dimethylformamide ( איור 2 , מימין). היבט נוסף שיש להביא בחשבון ב RNA, בשל נוכחות של C2'-OH הקבוצה (חסר bioxymers deoxyoligucleototide), הוא צעד נוסף שיש לשלב את ההגנה, ולאחר מכן deprotection, של עמדה זו נוקלאופילי. מבחינה זו, קבוצות הגנה מבוססות סיליקון הפכו לאסטרטגיה אטרקטיבית בשל הפוטנציאל שלהם כמו moieties biorthogonal (ספציפייםy deprotected בנוכחות פלואוריד), עם -butyldimethylsilyl טרט (TBDMS) ו triisopropylsilyloxymethyl (טום) קבוצות כמו האפשרויות הפופולריות (איור 2, ימין למטה).
בעבודה זו, הסינתזה האוטומטית בוצעה על סינתיסייזר DNA / RNA המשתמש בכימיה phosphoramidite סטנדרטי. הגדרות היצרן על המכשיר כוללות צעד דילול אוטומטי בעת שימוש בגרסאות מסחריות של phosphoramidites עבור DNA, או את האפשרות לדלל בכרכים שנקבעו על ידי המשתמש. עם זאת, החלטנו לשקול את phosphoramidite רנ"א לדלל באופן ידני בהתחשב בכך: 1) המחיר של phosphoramidites הקנונית של רנ"א גבוה (עד פי 50 פעמים יקר יותר במקרים מסוימים); 2) phosphoramidites שונה מתקבלים לעתים קרובות בכמויות קטנות; ) 3 כמות החומר המבוזבז בעת שימוש בצעד דילול אוטומטי (שנקבע על ידי היצרן) הוא גדול. בנוסף, השתמשנו: 1) תומך מוצק זמין מסחרית( למשל , CPG) המכיל נוקלאובאז מוגן לתפקד כמו 3-end; ו 2) phosphoramidites מסחריים (nucleobases הקנונית) מוגן עם קבוצת TBDMS ב C2'- O -position. רשימה מפורטת של השלבים סינתזה מסופק באיור 3 ו 1 טבלה , יחד עם תיאור נוסף והערות על צעדים שהותאמו לסינתזה RNA. יתר על כן, איור 4 ממחיש את התשואות בשלבים שנצפו עבור כל צעד לאחר בחירת האפשרות 'Trityl צג', אשר quantitates קטיון trityl שוחרר מכל צעד detritylation.
ראוי לציין כי בדרך כלל, מניסיוננו, הגורם המגביל היה קבלת phosphoramidite המכיל את השינוי הרצוי. כלומר, פיתוח מתודולוגיה סינתטית המאפשרת שילוב של שינויים באתרים נבחרים. בדוח זה, אנו מתמקדיםשילוב של נוקליאוטיד שונה שעבורו הקמנו את המתודולוגיה הסינתטית המתאימה, קבוצת C2'- O -thiophenylmethyl. קבוצה זו קטנה בגודלה ואינה משפיעה על סינתזת הפאזה המוצקה בכל צורה שהיא. מאז שילוב של קבוצה זו לתוך oligonucleotides של RNA כבר דיווחו, יחד עם פרמטרים מבניים תרמודינמיים 4 , שום היבטים של סינתזה אורגנית המובילה phosphoramidites שונה יתואר כאן.
הגנה, טיהור ואפיון
Deprotection של אמינים exocyclic וקבוצות cyanoethyl- K מתרחשת באותו צעד כמו זה של המחשוף שרף CPG. אנו להחיל את התנאים הנפוצים של חימום שרף שהתקבל בנוכחות פתרון מימי של AMA, ואחריו מחשוף של קבוצות C2'- O- silyl בנוכחות יוני פלואוריד, ולאחר מכן טיהור באמצעות ג'לאלקטרופורזה. בעוד אלה הפכו תנאים סטנדרטיים במקרים רבים, שינויים כי הם labile לתנאים בסיסיים או יונים פלואוריד עשוי לדרוש תנאים מתונים 13 , 14 , למשל , מתנול / אשלגן פחמתי (MeOH / K 2 CO 3 ), או בוטילאמין. לפיכך, קבוצה אחרת של הגנה על קבוצות על phosphoramidites המקביל הוא הכרחי. יתר על כן, בחרנו אלקטרופורזה כחלופה מועדפת כדי לטהר את oligomers deprotected בהתחשב הניסיון הקודם שלנו עם שיטה זו וחוסר מכשור אחרים. עם זאת, HPLC יכול לחילופין לשמש שיטה יעילה 15 . אפיון של oligonucleotides מטוהרים בוצע באמצעות ספקטרומטריית מסה, מטריצה בסיוע לייזר desorption / יינון זמן הטיסה (MALDI-TOF), באמצעות הליך דיווח על ידי הקבוצה שלנו 16 .
אפיון מבני יציבות מלוא של דופלקסים שהתקבלו בוצעו באמצעות תקליטור. באופן ספציפי, אנו עושים שימוש בתקליטור כדי לקבוע את מעברי denaturation תרמית של oligonucleotides שונה ושונה של RNA על ידי ביצוע הירידה אליפטיות של הלהקה ב CA. 270 ננומטר, כמו גם היעלמות של הלהקה (עם אליפטיות שלילית) עם מקסימום λ ב 210 ננומטר. השוואת ספקטרה לפני ואחרי הכלאה מסופק כדי להמחיש את ההבדלים שלהם ולספק אימות של המתודולוגיה המועסקים. השימוש בתקליטור מקובל באופן נרחב בקביעת מוטיבים מבניים בחומצות גרעין ובאמינואידים 17 , ולכן ניתן להשתמש בהם ככלי לקביעת פרמטרים מבניים ותרמודינמיים שונים 18 ; עם זאת, אין דוגמאות רבות שבו הטכניקה משמשת כדי להעריך מעברים denaturation תרמית. מקרים מסוימים כוללים קביעת יציבות תרמית על דנ"א המכיל G-quadruplexesהתחת = "xref"> 19 , 20 או בדופלקסים וסיכות של רנ"א 21 .
דוח זה מתכוון לספק את הקורא שאינו מומחה או הצופה עם סט של כלים המאפשרים התחלה חלקה של סוג זה של מחקר. זה ישמש כדי לשפר ולהשוות עם מתודולוגיות וטכניקות במעבדות מחקר אחרים המעורבים בענף זה מרגש של המדע. התוכן בדו"ח זה מוסיף לפרוטוקולים הקיימים של טכנולוגיה זו ממקורות שונים, ומעשיר ומקדם את החוויה בסיוע חזותי לכל צעד.
Protocol
1. מוצק שלב סינתזה של אוליגונוקליוטידים RNA
- הכנת פתרונות המכילים כל phosphoramidite (טבלה 1).
- לספור את מספר נוקליאוטידים ולהתאים את המשוואה n + 1 (כאשר n = מספר נוקליאוטידים) המתאים לכל בסיס ולמלא את הטבלה עם הערכים החסרים. נפח 0.15 מ"ל לכל נוקלאוטיד בריכוז של 0.1 M, מומס אצטוניטריל נטול מים.
- שקלו כל phosphoramidite לתוך בקבוקי ענבר מיובשים 10 מ"ל אמבר (ספטום למעלה אמבר 394 Amidite 13 מ"מ מזהה x 20 מ"מ OD למעלה) ומניחים מיד תחת ואקום באמצעות ייבוש המכיל סוכן ייבוש.
- ממלאים את מייבש כביסה עם ארגון יבש, להסיר את הבקבוק לדלל מיד עם כמות המקבילה של אצטוניטריל (מתקתק) לפני אבטחת על המכשיר.
הערה: הקפד להשתמש מזרקים גז הדוק ולשמור על אווירה מתקתקה בכל עת. - מוציאים את הספטה מהבקבוק ומניחים על המפרקאכין, באמצעות שינוי בבקבוק פונקציה.
הערה: תהליך זה דורש תוספת 0.15 מ"ל (ומכאן עודף נפח במשוואה n + 1 לעיל) של כל פתרון לפריים את הקו לפני סינתזה.
- הגדרת רצף סינתזה שלב מוצק באמצעות התוכנה ( איור 3 ).
- לחץ על סמל התוכנה ( לדוגמה , OligoNet 1.0.1) ובחר שם מכשיר> אישור . יצירת סינתזה חדשה על ידי קובץ> סינתזה חדשה> הזמנה .
- מילוי: תאריך; RunID; בחר מכשיר; שם רצף; רצף (5'עד 3 'סוף). תחת מחזור, להקצות את השיטה שנוצרה בעבר ( איור 3 ). בחר את הליך הסיום, ( משאיר oligonucleotide קשור שרף CPG)> ידנית.
- בחר DMT Off> (טיפול TCA בשלב האחרון כדי לקבל קבוצה 5'-OH בסוף הסינתזה) ; שמור קובץ> שמירה בשםולספק שם עבור הניסוי.
- שלח את הקובץ כדי להתחיל סינתזה הזמנה> שלח הזמנה לסינתיסייזר ולבחור עמודה 1-4 . פתח את חלון הסינתיסייזר ובחר trityl לפקח | בחר פונקציה | טריטי> לפקח על כל צעד .
- חזור על הצעדים לפי הצורך בהתאם למספר oligonucleotides להיות מסונתז בבת אחת (שים לב כי כל ההזמנות חייב להיות באותה שיטה, כלומר פעמים צימוד זהה רצף האירועים).
- התחל את הסינתיזה סינתיסייזר> התכונן להתחיל . מניחים את העמודות עם הקצה הרצוי על המיקומים המופיעים על המכשיר. על המכשיר לחץ על התחל> לא (להכנת ABI).
- לאחר הסינתזה הושלם, להסיר את הטור מן המכשיר ומניחים בקבוק תחתון עגול ואחריו ייבוש תחת לחץ מופחת של כ 0.5 h.
הערה: מומלץ לבדוק כי צבע כתום עמוק נצפתה רנדחיבורי אום (צעדים 1.2.4 - 1.2.7) כדי למנוע טעויות אפשריות מתפקוד צג trityl.
- Deprotection וטיהור של oligonucleotides RNA.
- פתח את העמודה על ידי סיבוב הכובע השחור והעברת כל (או חצי, בהתאם לצורך או המטרה) של שרף לבן לתוך צינור צנטריפוגות 1.6 מ"ל. הוסף 0.5 מ"ל של מתילאמין (40% במים) / אמוניה (40% במים) ב 1: 1.
- אבטח את הצינור צנטריפוגות עם מכסה parafilm ו, באמצעות בלוק חום, חום עד 60 מעלות צלזיוס במשך 1.5 שעות.
הערה: עצם כבד יכול להיות ממוקם על גבי הצינור על מנת להבטיח כי ריכוז אמוניה נשאר קבוע בתוך צינור התגובה. - הסר מ בלוק חום מגניב לאט לטמפרטורת החדר. בקצרה צנטריפוגה המדגם לסובב את התוכן, ולאחר מכן להעביר את supernatant לצינור צנטריפוגות חדש.
- להקפיא את דגימות ידי צינורות שקוע חנקן נוזלי (או יבש קרח / אתנול אמבטיה) ולהתרכז יובש בעוד spinnבצנטריפוגה בלחץ מופחת.
- Re- להשעות מוצקים 0.4 מ"ל של triethylamine / N-methylpyrrolidinone / טריאתילמין-טריידרופלואוריד (2: 2: 3 יחס) פתרון. מחממים עד 60 מעלות צלזיוס למשך 1.5 שעות ואחריו קירור איטי לטמפרטורת החדר.
- הוסף 0.04 מ"ל של תמיסת NaOAc (3M, pH 5.5 - מותאם עם HCl) ואחריו ערבוב עדין עם קצה פיפטה. הוסף אתנול (1 מ"ל) ו מגניב ל CA. -70 ° C (יבש קרח / אמבט אתנול) במשך 15 דקות.
- צנטריפוגה ב 15,000 סל"ד ו 4 מעלות צלזיוס למשך 10 דקות. השתמש פיפטה כדי לחלץ את aliquot ויבש את גלולה וכתוצאה מכך בלחץ מופחת.
- Re- להשעות את מוצק שהושג במאגר טוען (0.2 מ"ל, formamide aq 90%, 1 מ"מ EDTA) ומערבבים עד תערובת הומוגנית.
- טען השעיה על גבי ג'ל polyacrylamide מוכן קודם (20% denaturing, ממדים של טוב: 30 ס"מ x 1 מ"מ) 22 .
- החל זרם דרך הג'ל עד שהסמן הכחול של ברומופנול ממוקם בין 1 / 2-2 / 3למטה ג'ל (מידות ג'ל: ~ 21.5 ס"מ x 30 ס"מ).
- הסר את הג 'ל מן המעמד ולהעביר את התוכן ג' ל מן הזכוכית לעטיפת פלסטיק (מכוסה על שני הצדדים), ומניחים מעל שכבת דקה כרומטוגרפיה (TLC) צלחת מכוסה סיליקה (המכיל צבע פלואורסצנטי ב 254 ננומטר) כדי להמחיש את להקות באמצעות מנורת UV (λ מקסימום = 254 ננומטר).
- השתמש בסמן כדי להגדיר את המיקום שבו הלהקה העליונה ממוקם לחתוך אותו באמצעות סכין גילוח חדש. מקום ג'ל המכיל את RNA (ללא פלסטיק לעטוף) לתוך צינור חרוטי 50 מ"ל למחוץ חתיכות קטנות באמצעות מוט זכוכית.
- להשעות את שאריות ג 'ל לתוך aCl NaCl. פתרון (2 מ"מ, ו 1 מ"מ EDTA) ולנער את ההשעיה על 37 מעלות צלזיוס במשך 12 שעות. צנטריפוגה צינור חרוטי במשך 10 דקות.
- Desalt באמצעות מחסנית C18 הפוכה.
- הכן את המחסנית באמצעות מזרק 10 מ"ל על ידי שטיפה עם:
אצטוניטריל (10 מ"ל)
H 2 O (פעמיים, 10 מ"ל)
5 מ"מ NH4 Cl aq. פתרון (3 מ"ל)
פתרון המכיל RNA, נזהר לא לשפוך כל שיורית ג'ל. - לשטוף עם H 2 O (שלוש פעמים, 10 מ"ל).
- Elute מהעמודה באמצעות aq 60%. פתרון מתנול (3 מ"ל)
- הכן את המחסנית באמצעות מזרק 10 מ"ל על ידי שטיפה עם:
- תתרכז תחת לחץ מופחת מחדש להתמוסס במים ללא RNase (0.3 מ"ל)
- הכן פתרון לדלל (10 μL) ו 1 הפקדת μL על מכשיר UV- כלפי חוץ ( למשל , Nanodrop) כדי למדוד את UV- מול ספקטרום (200-450 ננומטר).
- השתמש בחוק של באר למברט כדי לקבוע את הריכוז של הפתרון שהתקבל:
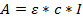
כאשר A = ספיגה שהושגה; Ε = מחושב מקדם הכחדה טוחנת; C = ריכוז, ו- l = 0.1 עבור אורך נתיב של 1 מ"מ. - חישוב מקדמי הכחדה טוחנת עבור oligonucleotides; מחושב כאן עם מחשבון מקווןהמשתמשת בתוכנת DINAMelt (http://unafold.ra.albany.edu/?q=dinamelt) 23
- קביעת ריכוז ואפיון באמצעות MALDI-TOF.
- דגימות לטעון על צלחת MALDI באמצעות קצה פיפטה טעון עם קצה C18 desalt, ואת כל נקודה oligonucleotide.
- לשטוף את קצה עם 50% אצטוניטריל (10 μL x 2). לאזן את קצה עם חומצה trifluoroacetic 0.1% (TFA, 10 μL x 2). עצה לטעון עם מדגם (בדרך כלל 100-150 pmol).
- לשטוף את קצה עם 0.1% TFA (10 μL x 2), ולאחר מכן עם מים (10 μL x 2).
- Elute המדגם לתוך פתרון המכיל את המטריצה הרצוי; השתמשנו פתרון של: 10 μL 25 mM-2,4,6-trihydroxyacetophenone מונוהידראט (THAP), 10 מ"מ ציטראט אמוניום, ופלואוריד אמוניום 300 מ"מ אצטוניטריל 50%.
- ספוט ישירות על צלחת MALDI ידי הפקדת 0.9 μL (ואחריו ייבוש האוויר) וחזרה על הנוהל לפי הצורך.
הערה: כל הספקטרה התקבלו על מצב רפלקטיבי חיובי.
- דגימות לטעון על צלחת MALDI באמצעות קצה פיפטה טעון עם קצה C18 desalt, ואת כל נקודה oligonucleotide.
2. RNA מבנה ניתוח באמצעות תקליטור
- הכנת פתרונות עבור תקליטור.
- הכן 0.25 מ"ל המכיל את RNA שונה [3 מיקרומטר], NaCl [10 מ"מ], חיץ פוספט נתרן [10 מ"מ, pH 7.2], ו MgCl 2 [5 מ"מ]. המדגם מוכן לניתוח כמו שהוא, במיוחד אם הוא מייצג מעבר unimolecular.
- אם המטרה היא לנתח מבנים דופלקס או מעברים bimolecular, להוסיף השלמת (1 שווה ערך טוחנת, אם רלוונטי) בשלב זה, או להמשיך עם השלב הבא.
- מניחים את המדגם על בלוק חום, מראש מחומם ל 90 מעלות צלזיוס, ולהכבות את החום כדי לשלוט קירור איטי לטמפרטורת החדר (בדרך כלל 2 - 4 שעות).
- רכישת ספקטרה
- הכן פתרון ריק (NaCl 10 מ"מ, פוספט נתרן 10 מ"מ pH 7.3, MgCl 2 5 מ"מ), העברת tOa מיקרו קובט (אורך 1 ס"מ אורך, 250 נפח מינימלי μL), ומניחים על עמדת מחזיק של מחליף מדגם בתוך המכשיר.
- מעבירים את המדגם המכיל RNA לתוך קובט מיקרו אחר עמדה מחליף מדגם. אם מדידת המעבר denaturation תרמית, בזהירות להוסיף מיטה של שמן מבלי לשבש את השכבה מימית ולאבטח את הכובע של קובט באמצעות חתיכת קלטת טפלון.
- פתח את מיכל החנקן כדי לספק זרימה המניעה את האוויר floater הממוקם על המכשיר כדי ca. 40. הפעל את המצנן.
- הפעל את המכשיר ופתח את התוכנה Spectra Manager icon, ולאחר מכן פתח את חלון הרכישה Spectra Measurement .
- טיהור המכשיר עם חנקן במשך 5 דקות לפני הרכישה.
- רכישת הספקטרום באמצעות הפרמטרים הבאים למדוד פרמטרים ולהתאים את הפרמטרים כדלקמן:
- תחת כללי , בחר: סרוק 200-350 ננומטר; ערוצים CD ו- HT: נתוניםהמגרש ב 0.1 ננומטר; מהירות סריקה ב 100 nm / min; רוחב פס ב 1 ננומטר; מספר הצטברות 5.
- תחת יחידת התא , בחר 20 ° C. תחת שליטה , בחר תריס נפתח ונסגר באופן אוטומטי. תחת מידע , בחר את שם, ריכוז, מפעיל.
- תחת נתונים , עיין בתיקייה הרצויה. לחץ על אישור .
- לרכוש את הספקטרה למדוד> מדידה המדגם , לזהות מיקום (ים) של cuvettes ב מחליף מדגם 1-6 בהתאם, לחץ על אישור .
- אם לוקחים מעבר denaturation תרמית:
- סגור תוכנת רכישה קובץ> צא ופתח תוכנית חדשה מדידה טמפרטורה משתנה> מדידה> פרמטרים .
- להחיל את הפרמטרים הבאים כדי להקליט מעבר תרמי, אשר יכול להיות מותאם לפי הצורך:
- תחת טמפרטורה , בחר להתחיל זמני: 4 ° C; מרווח: 0.2; TaRget: 90 ° C; שיפוע: 1 ° C / min; המתן 0 s. תחת Start / End (סיום) , בחר Start & End (תנאי התחלה) ותנאי סיום בהתאם לצורך.
- תחת כללי , בחר 3 ערוצים, CD / HT / Abs; רוחב פס: 1 ננומטר; אורך גל: 270 ננומטר (או לפי הצורך). תחת שליטה , בחר ללא. תחת מידע , בחר שם, ריכוז, מפעיל.
- תחת נתונים , עיין בתיקייה הרצויה. לחץ על אישור .
- דגימות מגניב ל 4 ° C ולחכות 5 דקות בטמפרטורה זו לפני קבלת מדידת ספקטרום.
- נתוני עבודה עבור ספקטרה.
- באמצעות הפונקציה על התוכנה, להפחית את הספקטרום ריק מרכישה היעד כדי להסביר אותות רקע הנובעים ממערכת חיץ בשימוש: ניתוח ספקטרה> קובץ> פתח .
- לאחר פתיחת קובץ ריק וקובץ ספקטרה, גרור את View 2 תחת View 1 (בצד שמאל של המסך).
- לחץ על PrOcessing> חיסור> אישור או החלפת נתונים> אישור .
- חלץ נתונים כקובץ ASCII: קובץ> יצא ובחר ASCII .
- השתמש בתוכנה כדי לשרטט את הספקטרום המתאים. החלקה של נתונים היא אופציונלית במקרים שבהם יחס אות לרעש נמוך מהצפוי. שינוי זה יכול להיות מיושם על ידי החלת אפשרות החלקה.
- נתוני עבודה עבור מעברים denaturation תרמית.
- חלץ נתונים מקובץ JASCO כקובץ ASCII.
- חלק את נקודות האליפטיות כפונקציה של הטמפרטורה. בדרך כלל, החלקה של הנתונים נחוצה בהתאם לאורך הגל הנבדק. במקרים מסוימים, השימוש באורך גל ב 210 ננומטר הציג וריאציות גדולות יותר בין מגרשים שדרשו להחליק. עם זאת, בהתאם למדגם וריכוזים, בוצעו מספר חישובים תוך שימוש בנתונים הגולמיים.
- כדי לחשב דן תרמיתAturing מעברים (T מ ' ) ערך, להשיג נגזרת ראשונה של העקום. המקסימום או המינימום היו אינדיקציה לפרמטר זה. כמו בשלב הקודם, במקרים מסוימים נדרש להחליק את הנתונים כדי לקבל את הערך המדויק ביותר.
הערה: הניסויים בוצעו בדרך כלל בשלושה עותקים, וכתוצאה מכך הממוצע הממוצע וסטיית התקן עבור המדידה.
תוצאות
הסינתזה של DNAecamers RNA המכיל אפס, אחד, או שני 2-thiophenylmethyl שינויים ב C2'- O- אופוזיציה מתואר יחד עם טיהור המקביל שלו אפיון. כמו כן, נכלל תיאור מפורט של הניתוח המבני שבוצע באמצעות התקליטור.
ארבעת גד?...
Discussion
הכוונה של כתב היד הזה היא לשמש מדריך לחוקרים בתחום, מתחילים או מומחה, כדי להשיג בהצלחה או לשפר את הסינתזה של oligonucleotides של DNA או RNA. המתודולוגיה המתוארת מתמקדת בשימוש סינתזה שלב מוצק באמצעות סינתיסייזר DNA / RNA אוטומטי באמצעות כימיה phosphoramidite סטנדרטי. הדו"ח מתאר תיאור שלב ...
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
הכנת כתב היד הזה נתמכה באמצעות קרנות הזנק מאוניברסיטת קולורדו דנבר (JMRE). AF רוצה להכיר בתמיכה מענק מחקר ופעילויות יצירתיות (RaCAS, CU Denver). מימון של משרד שירותי מחקר, אוניברסיטת קולורדו דנבר כדי לכסות את הוצאות הפרסום הוא הודה. ברצוננו להודות לחברת המעבדה גב 'קסנדרה הרברט ומר יאניק ק' דז'ו על תרומתם בחלק הווידאו.
Materials
| Name | Company | Catalog Number | Comments |
| AbsolveTM | PerkinElmer | 6NE9711 | |
| Acetonitrile 99.9%, HPLC Grade | Fisher Scientific | 75-05-8 | |
| Acetonitrile 99.9%, anhydrous for DNA sequencing | Fisher BioReagents | 75-05-8 | |
| Acrylamide, 99+% | ACROS Organics | 164850025 | |
| Ammonium chloride 98+% | Alfa Aesar | 12125-02-9 | |
| Ammonium citrate, dibasic 98% | Sigma Aldrich | 3012-65-5 | |
| Ammonium Fluoride 98.0%, ACS grade | Alfa Aesar | 12125-01-8 | |
| Ammonium hydroxide 28 - 30% in water, ACS Plus | Fisher Chemical | 1336-21-6 | |
| Ammonium persulfate | ACROS Organics | 1444 | |
| Argon-ultra high purity | Airgas | 7440-37-1 | |
| Bis-acrylamide Ultra pure | VWR-Amresco | 172 | |
| Boric Acid | Fisher Scientific | A73-1 | |
| Diethyl pyrocarbonate, 97% | ACROS Organics | A0368487 | |
| Ethanol, anhydrous, histological grade | Fisher Chemical | 64-17-5 | |
| Ethylenediamine tetraacetic acid, disodium salt dehydrate 100.2% | Fisher Chemical | 6381-92-6 | |
| Formamide | Thermo Scientific | 75-12-7 | |
| Hydrochloric acid, 36.5 - 38.0%, Certified ACS Plus | Fisher Chemical | 7647-01-0 | |
| Magnesium chloride hexahydrate, 99% | Fisher Scientific | 7786-30-3 | |
| Methanol, 99.9%, HPLC Grade | Fisher Chemical | 67-56-1 | |
| Methylamine 40% in water | Sigma Aldrich | 74-89-5 | |
| 1-Methyl-2-pyrrolidinone, andhydrous, 99.5% | Aldrich | 872-50-4 | |
| Opti-TOFTM 96 Well Insert (123 x 81 mm) | MDS SCIEX | 1020157 | |
| RNase Away | Molecular BioProducts | 7005-11 | |
| Sodium acetate, anhydrous 99.2%, Certified ACS | Fisher Chemical | 127-09-3 | |
| Sodium chloride, 100.5%, Certified ACS | Fisher Chemical | 7647-14-5 | |
| Sodium phosphate monobasic dihydrate 99.0% | Sigma | 13472-35-0 | |
| 2’,4’ Triethylamine, 99+% | Alfa Aesar | 121-44-8 | |
| TEMED | Amresco | 761 | |
| Triethylamine trihydrofluoride, 98% | Aldrich | 73602-61-6 | |
| Trifluoroacetic acid, 99% | Alfa Aesar | 76-05-1 | |
| 6’-Trihydroxyacetophenone monohydrate 98% | Sigma Aldrich | 480-66-0 | |
| Tris Base | Fisher Scientific | BP154-3 | |
| Urea | Fisher Scientific | U15-3 | |
| Reagents for the RNA synthesis: | |||
| Deblocking mix, 3% trichloroacetic acid in dichloromethane | Glen Research | 40-4140-57 | |
| Cap Mix A, THF/Pyridine/Acetic anhydride | Glen Research | 40-4110-52 | |
| Cap Mix B, 10% 1-methylimidazole in THF | Glen Research | 40-4120-52 | |
| Activator, 0.25 M 5-ethylthio-1H-tetrazole in anhydrous acetonitrile | Glen Research | 30-3140-52 | |
| Oxidizing Solution, 0.02 M iodine in THF/Pyridine/Water | Glen Research | 40-4330-52 | |
| U-RNA-CPG | Glen Research | 20-3330-xx | |
| Ac-G-RNA-CPG | Glen Research | 20-3324-xx | |
| Ac-G-CE Phosphoramidite | Glen Research | 10-3025-xx | |
| U-CE Phosphoramidite | Glen Research | 10-3030-xx | |
| Ac-C-CE Phosphoramidite | Glen Research | 10-3015-xx | |
| Bz-A-CE Phosphoramidite | Glen Research | 10-3003-xx |
References
- Matteucci, M. D., Caruthers, M. H. The synthesis of oligodeoxypyrimidines on a polymer support. Tetrahedron Lett. 21 (8), 719-722 (1980).
- Beaucage, S. L., Caruthers, M. H. Deoxynucleoside phosphoramidites-A new class of key intermediates for deoxypolynucleotides. Tetrahedron Lett. 22 (20), 1859-1862 (1981).
- Caruthers, M. H. Gene synthesis machines: DNA Chemistry and its uses. Science. 230 (4723), 281-285 (1985).
- Nguyen, J. C., et al. Synthesis, Thermal Stability, Biophysical Properties, and Molecular Modeling of Oligonucleotides of RNA Containing 2'-O-2-Thiophenylmethyl Groups. J Org Chem. 81 (19), 8947-8958 (2016).
- El-Sagheer, A. H., Brown, T. Click chemistry with DNA. Chem Soc Rev. 39, 1388-1405 (2010).
- Puffer, B., et al. 5-Fluoro pyrimidines: labels to probe DNA and RNA secondary structures by 1D 19F NMR spectroscopy. Nucleic Acids Res. 37 (22), 7728-7740 (2009).
- Wan, W. B., Seth, P. P. The medicinal chemistry of therapeutic oligonucleotides. J Med Chem. 59 (21), 9645-9667 (2016).
- Zhou, C., Greenberg, M. M. DNA Damage by histone radicals in nucleosome core particles. J Am Chem Soc. 136 (18), 6562-6565 (2014).
- Zhang, Y., et al. UV-Induced proton-coupled electron transfer in cyclic DNA miniduplexes. J Am Chem Soc. 138 (23), 7395-7401 (2016).
- Anosova, I., et al. The structural diversity of artificial genetic polymers. Nucleic Acids Res. 44 (3), 1007-1021 (2016).
- Riml, C., Micura, R. Synthesis of 5-Hydroxymethylcytidine- and 5-Hydroxymethyl-uridine-Modified RNA. Synthesis. 48, 1108-1116 (2016).
- Anderson, B. A., Hrdlicka, P. J. Merging Two Strategies for Mixed-Sequence Recognition of Double-Stranded DNA: Pseudocomplementary Invader Probes. J Org Chem. 81 (8), 3335-3346 (2016).
- Beaucage, S. L., Iyer, R. P. Synthesis of oligonucleotides by the phosphoramidite approach. Tetrahedron. 48 (12), 2223-2311 (1992).
- Gillet, L. C. J., Alzeer, J., Schärer, O. D. Site-specific incorporation of N-(deoxyguanosin-8-yl)-2-acetylaminofluorene (dG-AAF) into oligonucleotides using modified 'ultra-mild' DNA synthesis. Nucleic Acids Res. 33 (6), 1961-1969 (2005).
- Shiba, Y., et al. Chemical synthesis of a very long oligoribonucleotide with 2-cyanoethoxymethyl (CEM) as the 2'-O.-protecting group: structural identification and biological activity of a synthetic 110mer precursor-microRNA candidate. Nucleic Acids Res. 35 (10), 3287-3296 (2007).
- Choi, Y. J., Gibala, K. S., Ayele, T., Deventer, K. D., Resendiz, M. J. E. Biophysical properties, thermal stability and functional impact of 8-oxo-7,8-dihydroguanine on oligonucleotides of RNA - a study of duplex, hairpins and the aptamer for preQ1 as models. Nucleic Acids Res. 45 (4), 2099-2111 (2017).
- Ranjbar, B., Gill, P. Circular dichroism techniques: Biomolecular and nanostructural analyses - A review. Chem Biol Drug Des. 74 (2), 101-120 (2009).
- Mergny, J. -. L., Lacroix, L. Analysis of thermal melting curves. Oligonucleotides. 13 (6), 515-537 (2003).
- Jin, R., Breslauer, K. J., Jones, R. A., Gaffney, B. L. Tetraplex formation of a guanine-containing nonameric DNA fragment. Science. 250 (4980), 543-546 (1990).
- Virgilio, A., et al. 5-Hydroxymethyl-2'-deoxyuridine residues in the thrombin binding aptamer: Investigating anticoagulant activity by making a tiny chemical modification. CHEMBIOCHEM. 15 (16), 2427-2434 (2014).
- Chauca-Diaz, A. M., Choi, Y. J., Resendiz, M. J. E. Biophysical properties and thermal stability of oligonucleotides of RNA containing 7,8-dihydro-8-hydroxyadenosine. Biopolymers. 103 (3), 167-174 (2015).
- Maniatis, T., Fritsch, E. F., Sambrook, J. . Molecular cloning: a laboratory manual. 14, 173-185 (1987).
- Markham, N. R., Zuker, M. UNAFold: software for nucleic acid folding and hybridization. Methods Mol Biol. 453, 3-31 (2008).
- Breslow, R., Huang, D. -. L. Effects of metal ions including Mg2+ and lanthanides on the cleavage of ribonucleotides and RNA model compounds. Proc Natl Acad Sci U S A. 88 (10), 4080-4083 (1991).
- Blumberg, D. D. Creating a ribonuclease-free environment. Methods Enzymol. 152 (2), 20-24 (1987).
- AbouHaidar, M. G., Ivanov, I. G. Non-enzymatic RNA promoted by the combined catalytic activity of buffers and magnesium ions. Z Naturforsch C. 54 (7-8), 542-548 (1999).
- Farrell, R. E. . RNA Methodologies (4th Ed): A laboratory guide for isolation and characterization. 2, 45-80 (2010).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved