É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Protocolo para síntese de fase sólida de oligómeros de ARN contendo um 2'-
Neste Artigo
Resumo
Este artigo fornece um procedimento detalhado sobre a síntese em fase sólida, a purificação e a caracterização de dodecamers de ARN modificados na posição C2'- O . As análises fotométricas UV-vis e dicroísmo circular são usadas para quantificar e caracterizar aspectos estruturais, ou seja , de uma única linha ou de duas vertentes.
Resumo
A síntese em fase sólida tem sido utilizada para obter polímeros canônicos e modificados de ácidos nucleicos, especificamente de DNA ou RNA, o que tornou uma metodologia popular para aplicações em vários campos e para diferentes fins de pesquisa. O procedimento aqui descrito concentra-se na síntese, purificação e caracterização de dodecamers de RNA 5 '- [CUA CGG AAU CAU] -3' contendo zero, uma ou duas modificações localizadas na posição C2'- O . As sondas são baseadas em grupos 2-tiofenilmetilo, incorporados em nucleótidos de ARN através de síntese orgânica padrão e introduzidos nos oligonucleótidos correspondentes através dos seus respectivos fosforamiditos. Este relatório faz uso da química da fosforamidita através das quatro nucleobases canônicas (Uridina (U), Citosina (C), Guanosina (G), Adenosina (A)), bem como nucleotídeos funcionalizados com 2-tiofenilmetilo modificados no 2'- O- posição; No entanto, a metodologia é favorável para uma grande variedadeMuitas modificações que foram desenvolvidas ao longo dos anos. Os oligonucleótidos foram sintetizados em um suporte de vidro de poro controlado (CPG) seguido por clivagem da resina e desprotecção em condições padrão, isto é , uma mistura de amônia e metilamina (AMA) seguida de fluoreto de hidrogênio / trietilamina / N-metilpirrolidinona. Os oligonucleótidos correspondentes foram purificados por meio de eletroforese de poliacrilamida (20% de desnaturação) seguido por eluição, dessalinização e isolamento através de cromatografia de fase reversa (Sep-pak, coluna C18 ). A quantificação e os parâmetros estruturais foram avaliados através de análise fotométrica de dicroísmo circular (CD-UV) visível ultravioleta (UV-vis), respectivamente. Este relatório pretende servir como um recurso e guia para iniciantes e especialistas pesquisadores interessados em embarcar neste campo. Espera-se que sirva como um trabalho em andamento à medida que novas tecnologias e metodologias são desenvolvidas. A descrição das metodologias e técnicas dentroO documento da S corresponde a um sintetizador de DNA / RNA (remodelado e comprado em 2013) que utiliza química de fosforamidite.
Introdução
A síntese em fase sólida para obter oligonucleótidos de DNA / RNA é uma ferramenta poderosa que serviu várias aplicações em vários campos desde a década de 1970 1 , 2 , 3 usando blocos de construção de fosforamidita 4 . Exemplos de sua ampla influência incluem: seu impacto na rotulagem ( através de reações de química de clique) 5 , sondagem estrutural 6 e tecnologias anti-sentido 7 , bem como a elucidação de mecanismos biológicos 8 , 9 , fonte como material genético 10 e o estudo de Várias modificações naturais e / ou químicas 11 , 12 , entre muitos outros. A modificação que usamos aqui representa o primeiro passo em nossos esforços para obter oligonucleótidos de RNA que contenhamSondas fotoativas para permitir o controle temporal da estrutura e função deste importante biopolímero.
A síntese de dodecamers de ARN com sequências: 5 '- [CUA CG G A UA CAU] -3' / 5 '- [agosto AUU CCG UAG] -3' (posições sublinhadas representam a incorporação de uma modificação -thiophenylmethyl C2'- ó ) Constitui o foco deste estudo. As seqüências foram escolhidas para permitir a quantificação e medição de fios de ARN como fios simples, ou como suas estruturas duplex correspondentes (nenhuma outra estrutura secundária foi prevista como termodinamicamente estável). O CD foi utilizado para estabelecer os parâmetros estruturais, ou seja , a formação do duplex e as transições de desnaturação térmica.
Síntese
O procedimento geral para a obtenção destes oligonucleótidos é ilustrado na Figura 1 e segue o processo passo a passo: Síntese de fase sólida automatizada → DeprOteção → Purificação → Quantificação → Caracterização. A Figura 2 mostra as unidades monoméricas que são necessárias neste procedimento. A síntese em fase sólida do RNA é semelhante à do DNA, na medida em que se baseia na química da fosforamidite ( Figura 2 , à esquerda) e na utilização de grupos protectores base-labileis para as aminas exocíclicas nucleófilas em G, A e C, eg , Acetilo, benzoílo, fenoxiacetilo, t- butilo ou N , N- dimetilformamida ( Figura 2 , à direita). Mais um aspecto a considerar no RNA, devido à presença do grupo C2'-OH (faltando nos biopolímeros desoxioligonucleotídicos), é o passo adicional que deve ser incorporado para proteção e posterior desproteção desta posição nucleofílica. A este respeito, os grupos de proteção baseados em silício tornaram-se uma estratégia atraente devido ao seu potencial como porções biortogonais (especificamenteE desprotegido na presença de fluoreto), com os grupos terc- butildimetilsililo (TBDMS) e triisopropilsililoximetilo (TOM) como escolhas populares ( Figura 2 , inferior esquerda).
Neste trabalho, a síntese automatizada foi realizada em um sintetizador de DNA / RNA que usa química padrão de fosforamidite. As configurações do fabricante no instrumento incluem um passo de diluição automatizado ao usar as versões comerciais dos fosforamiditos para o DNA ou a opção de diluir nos volumes configurados pelo usuário. No entanto, decidimos pesar o ARN fosforamidito e diluir manualmente, dado que: 1) o preço dos fosforamiditos canônicos do RNA é maior (até 50 vezes mais caro em alguns casos); 2) os fosforamiditos modificados são frequentemente obtidos em pequenas quantidades; E 3) a quantidade de material desperdiçado após o uso de um passo de diluição automatizado (definido pelo fabricante) é grande. Além disso, utilizamos: 1) suportes sólidos comercialmente disponíveis(Por exemplo , CPG) contendo uma nucleobase protegida para funcionar como a extremidade 3 '; E 2) fosforamiditos comerciais (nucleobases canônicas) protegidos com um grupo TBDMS na posição C2'- O . A lista detalhada dos passos de síntese é fornecida na Figura 3 e Tabela 1 , juntamente com descrição e comentários adicionais para etapas que foram ajustadas para a síntese de RNA. Além disso, a Figura 4 ilustra os rendimentos passo a passo que são observados para cada passo depois de selecionar a opção 'Trityl Monitor', que quantifica o catião tritil que é liberado de cada passo de detrilação.
Vale ressaltar que tipicamente, em nossa experiência, o fator limitante foi a obtenção da fosforamidita contendo a modificação desejada. Ou seja, o desenvolvimento de uma metodologia sintética que permite a incorporação de modificações em sites selecionados. Neste relatório, nos concentramos naIncorporação de um nucleótido modificado para o qual estabelecemos a metodologia sintética correspondente, o grupo C2'- O- tiofenilmetilo. Este grupo é de tamanho pequeno e não afeta a síntese em fase sólida de qualquer maneira. Uma vez que a incorporação deste grupo em oligonucleótidos de ARN foi relatada, juntamente com os parâmetros estruturais e termodinâmicos 4 , não serão descritos aqui os aspectos de síntese orgânica que levem aos fosforamiditos modificados.
Desproteção, Purificação e Caracterização
A desprotecção das aminas exocíclicas e dos grupos ß-cianoetilo ocorre no mesmo passo que a da clivagem da resina CPG. Aplicamos as condições comumente usadas para aquecer a resina obtida na presença de uma solução aquosa de AMA, seguida por clivagem dos grupos C2'- O- sililo na presença de iões fluoreto e depois purificação através de gelEletroforese. Embora estes tenham se tornado condições padrão em muitos casos, modificações que são lábiles para condições básicas ou íons fluoreto podem exigir condições mais suaves 13 , 14 , por exemplo , metanol / carbonato de potássio (MeOH / K 2 CO 3 ) ou butilamina. Assim, é necessário um conjunto diferente de grupos de proteção sobre os fosforamidites correspondentes. Além disso, escolhemos a eletroforese como a alternativa preferida para purificar os oligómeros desprotegidos, dada a nossa experiência anterior com este método e a falta de outras instrumentações. No entanto, a HPLC pode alternativamente ser utilizada como um método eficaz 15 . A caracterização dos oligonucleótidos purificados foi realizada através de espectrometria de massa, dessorção / ionização de laser assistida por matriz - tempo de voo (MALDI-TOF), utilizando um procedimento relatado pelo nosso grupo 16 .
Caracterização estrutural e A estabilidade incorreta dos duplexs obtidos foi realizada via CD. Especificamente, utilizamos o CD para determinar as transições de desnaturação térmica de oligonucleótidos modificados e não modificados de RNA seguindo a diminuição da elipticidade da banda em aprox. 270 nm, bem como o desaparecimento da banda (com elipticidade negativa) com λ máx . A 210 nm. Uma comparação de espectros antes e após a hibridação é fornecida para ilustrar suas diferenças e fornecer a validação da metodologia empregada. O uso de CD é amplamente aceito na determinação de motivos estruturais em ácidos nucleicos e aminoácidos 17 e, portanto, pode ser empregado como uma ferramenta para determinar vários parâmetros estruturais e termodinâmicos 18 ; No entanto, não há muitos exemplos em que a técnica é utilizada para avaliar as transições de desnaturação térmica. Alguns casos incluem a determinação de estabilidades térmicas em ADN contendo quadruplexos GAss = "xref"> 19 , 20 ou em duplex e grampos de RNA 21 .
Este relatório pretende fornecer ao leitor ou espectador não especialista um conjunto de ferramentas que permitem um bom início desse tipo de pesquisa. Isso servirá para melhorar e comparar com metodologias e técnicas em outros laboratórios de pesquisa que estão envolvidos neste poderoso ramo da ciência. O conteúdo deste relatório adiciona aos protocolos existentes desta tecnologia de várias fontes, e enriquece e facilita a experiência com um auxílio visual para cada etapa.
Protocolo
1. Síntese em fase sólida de oligonucleótidos de ARN
- Preparação de soluções contendo cada fosforamidite (Tabela 1).
- Contar o número de nucleótidos e encaixar na equação n + 1 (onde n = número de nucleótidos) correspondente a cada base e preencher a tabela com os valores faltantes. Volume = 0,15 mL por nucleotídeo a uma concentração de 0,1 M, dissolvido em acetonitrilo anidro.
- Pesar cada fosforamidite em frascos âmbar de 10 mL secados no forno (Septum Top Amber 394 Amidite 13 mm ID x 20 mm OD Top) e coloque imediatamente sob vácuo usando um dessecador contendo um agente de secagem.
- Encha o dessecador com árgon seco, remova o frasco e diluir imediatamente com a quantidade correspondente de acetonitrilo (anidro) antes de fixar no instrumento.
NOTA: Certifique-se de usar seringas estanque aos gases e manter uma atmosfera anidra em todos os momentos. - Remova os septos da garrafa e coloque no mAchine, através de uma função de troca de garrafa.
NOTA: Este processo requer um aumento adicional de 0,15 mL (daí o excesso de volume na equação n + 1 acima) de cada solução para iniciar a linha antes da síntese.
- Configuração da síntese de seqüência e fase sólida usando o software ( Figura 3 ).
- Clique no ícone do software ( por exemplo , OligoNet 1.0.1) e selecione Nome do instrumento> OK . Crie uma nova síntese por Arquivo> Nova síntese> Ordem .
- Fill-in: Data; RunID; Selecione o instrumento; Nome da sequência; Sequência (fim de 5 a 3 '). Sob | ciclo, atribua o método criado anteriormente ( Figura 3 ). Escolha o procedimento final, ( deixa o oligonucleótido ligado à resina CPG)> manual.
- Selecione DMT Off> (tratamento TCA no último passo para obter um grupo 5'-OH no final da síntese) ; Salve Arquivo> Salvar comoE forneça um nome para o experimento.
- Enviar o arquivo para iniciar a síntese Ordem> Enviar pedido ao sintetizador e selecionar a coluna 1-4 . Abra a janela do sintetizador e selecione o monitor triityl | escolha a função | trity> monitor cada etapa .
- Repita as etapas conforme necessário, dependendo do número de oligonucleótidos a serem sintetizados de uma vez (observe que todas as ordens devem ter o mesmo método, ou seja , os mesmos tempos de acoplamento e a seqüência de eventos).
- Comece a síntese Sintetizador> Prepare-se para começar . Coloque as colunas com a extremidade 3 'desejada nas posições indicadas no instrumento. No instrumento, clique em Iniciar> Não (para preparação ABI).
- Uma vez que a síntese está completa, remova a coluna do instrumento e coloque em um balão de fundo redondo, seguido de secagem sob pressão reduzida durante cerca de 0,5 h.
NOTA: Recomenda-se verificar se uma cor laranja profunda é observada em randOm acoplamentos (etapas 1.2.4 - 1.2.7) para evitar possíveis erros da função do monitor trityl.
- Desprotecção e purificação de oligonucleótidos de ARN.
- Abra a coluna torcendo a tampa preta e transfira tudo (ou metade, dependendo da necessidade ou finalidade) da resina branca em um tubo de centrífuga de 1,6 mL. Adicione 0,5 mL de uma metilamina (40% na água) / amônia (40% na água) a 1: 1.
- Fixe a tampa do tubo de centrífuga com parafilme e, usando um bloco de calor, aqueça a 60 ° C por 1,5 h.
NOTA: Um objeto pesado pode ser colocado no topo do tubo para garantir que a concentração de amônia permaneça constante dentro do tubo de reação. - Retire do bloco de calor e esfrie lentamente até a temperatura ambiente. Centrifugue brevemente a amostra para girar o conteúdo, depois transfira o sobrenadante para um novo tubo de centrífuga.
- Congele as amostras submergindo tubos em nitrogênio líquido (ou banho de gelo seco / etanol) e concentre-se até a secura enquanto o spinnEm uma centrífuga sob pressão reduzida.
- Re-suspender os sólidos em 0,4 mL de uma solução de trietilamina / N-metilpirrolidinona / trimetilamina-trihidrofluoreto (proporção 2: 2: 3). Aquecer até 60 ° C durante 1,5 h, seguido de arrefecimento lento até à temperatura ambiente.
- Adicione 0,04 mL de uma solução de NaOAc (3M, pH 5,5 - ajustado com HCl) seguido de uma mistura suave com uma ponta de pipeta. Adicione etanol (1 mL) e arrefecer até ca. -70 ° C (banho de gelo seco / etanol) durante 15 min.
- Centrifugação a 15.000 rpm e 4 ° C durante 10 min. Use uma pipeta para extrair a alíquota e secar o sedimento resultante sob pressão reduzida.
- Re-suspender o sólido obtido no tampão de carga (0,2 mL, 90 mM de formamida aquosa, 1 mM de EDTA) e misturar até que a mistura seja homogênea.
- Carregar a suspensão sobre um gel de poliacrilamida previamente preparado (20% de desnaturação, dimensões do poço: 30 cm x 1 mm) 22 .
- Aplique uma corrente através do gel até que o marcador de bromofenol esteja localizado entre 1 / 2-2 / 3No gel (dimensões do gel: ~ 21,5 cm x 30 cm).
- Retire o gel do suporte e transfira o conteúdo de gel do vidro para plástico (coberto em ambos os lados) e coloque sobre uma placa de cromatografia de camada fina (TLC) coberta com sílica (contendo corante fluorescente a 254 nm) para visualizar as bandas Usando uma lâmpada UV (λ max = 254 nm).
- Use um marcador para delinear a posição onde a banda superior está localizada e cortá-la usando uma nova lâmina de barbear. Coloque gel contendo o ARN (sem plástico) em um tubo cônico de 50 mL e aplique em pequenos pedaços usando uma haste de vidro.
- Suspender os resíduos de gel em um NaCl aq. Solução (2 mM e 1 mM de EDTA) e agite a suspensão a 37 ° C durante 12 h. Centrifugue o tubo cônico por 10 min.
- Desalinhando usando um cartucho C18 de fase reversa.
- Prepare o cartucho usando uma seringa de 10 mL lavando com:
Acetonitrilo (10 mL)
H 2 O (duas vezes, 10 mL)
NH 5 mM4 Cl aq. Solução (3 mL)
Solução contendo RNA, tomando cuidado para não derramar nenhum dos resíduos de gel. - Lavar com H 2 O (três vezes, 10 mL).
- Elute da coluna usando um 60% aq. Solução de metanol (3 mL)
- Prepare o cartucho usando uma seringa de 10 mL lavando com:
- Concentre-se sob pressão reduzida e re-dissolva-se em água sem RNase (0,3 mL)
- Prepare uma solução diluída (10 μL) e deposite 1 μL no instrumento UV-vis ( por exemplo , Nanodrop) para medir o espectro UV-vis (200-450 nm).
- Use a Lei de Lambert de Beer para determinar a concentração da solução obtida:
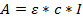
Onde A = absorvência obtida; Ε = coeficiente de extinção molar calculado; C = concentração e l = 0,1 para um comprimento de trajeto de 1 mm. - Calcular os coeficientes de extinção molar para os oligonucleótidos; Calculado aqui com uma calculadora on-lineQue usa o software DINAMelt (http://unafold.ra.albany.edu/?q=dinamelt) 23
- Determinação e caracterização de concentração via MALDI-TOF.
- Coloque amostras em uma placa MALDI usando uma ponta de pipeta carregada com uma ponta C18 para desalar e mancha cada oligonucleótido.
- Lave a ponta com 50% de acetonitrilo (10 μL x 2). Equilibre a ponta com 0,1% de ácido trifluoroacético (TFA, 10 μL x 2). Coloque a ponta com amostra (tipicamente 100-150 pmol).
- Lave a ponta com 0,1% de TFA (10 μL x 2), e depois com água (10 μL x 2).
- Eluir a amostra em uma solução contendo a matriz desejada; Usamos uma solução de: 10 μL de monohidrato de 25 mM-2,4,6-trihidroxiacetofenona (THAP), citrato de amônio 10 mM e fluoreto de amônio 300 mM em acetonitrilo a 50%.
- Coloque diretamente na placa MALDI depositando 0,9 μL (seguido de secagem ao ar) e repita o procedimento conforme necessário.
NOTA: Todos os espectros foram obtidos no modo reflector positivo.
- Coloque amostras em uma placa MALDI usando uma ponta de pipeta carregada com uma ponta C18 para desalar e mancha cada oligonucleótido.
2. Análise da Estrutura do RNA via CD
- Preparação de soluções para CD.
- Prepare 0,25 mL que continham o ARN modificado [3? M], NaCl a [10 mM], tampão de fosfato de sódio [10 mM, pH 7,2], e MgCl 2 [5 mM]. A amostra está pronta para análise tal como é, especialmente se representa uma transição unimolecular.
- Se o objetivo é analisar estruturas duplex ou transições bimoleculares, adicione o complemento (1 equivalente molar, se aplicável) neste momento ou continue com o passo abaixo.
- Coloque a amostra em um bloco de calor, aqueça previamente a 90 ° C e desligue o calor para controlar o resfriamento lento até a temperatura ambiente (tipicamente 2 a 4 h).
- Aquisição de Spectra
- Prepara-se uma solução em branco (10 mM de NaCl, fosfato de sódio 10 mM pH 7,3, MgCl 2 5 mM), transferência tO micro-cuvete (comprimento do caminho de 1 cm, volume mínimo de 250 μL) e coloque na posição de suporte do trocador de amostras dentro do instrumento.
- Transfira a amostra contendo RNA em outra micro-cuvete e posicione-se no trocador de amostras. Se medir uma transição de desnaturação térmica, adicione cuidadosamente um leito de óleo sem interromper a camada aquosa e prenda a tampa da cuvete usando um pedaço de fita de Teflon.
- Abra o tanque de nitrogênio para fornecer um fluxo que mova o flutuador de ar localizado no instrumento para ca. 40. Ligue o refrigerador.
- Ligue o instrumento e abra o ícone do software Spectra Manager e, em seguida, abra a janela de aquisição Spectra Measurement .
- Purgar o instrumento com nitrogênio durante 5 minutos antes da aquisição.
- Adquira os espectros usando os seguintes parâmetros. Medir> Parâmetros e ajustar os parâmetros da seguinte maneira:
- Em Geral , selecione: Digitalizar 200-350 nm; Canais CD e HT: dadosPasso a 0,1 nm; Velocidade de varredura a 100 nm / min; Largura da banda a 1 nm; Número de acumulações em 5.
- Sob unidade celular , escolha 20 ° C. Sob controle , selecione Obturador é aberto e fechado automaticamente. Sob informações , escolha o nome, a concentração, o operador.
- Em dados , navegue até a pasta desejada. Clique em OK .
- Adquira os espectros Medida> Medida da amostra , identifique a (s) posição (s) das cubetas no trocador de amostras 1-6 em conformidade, clique em OK .
- Se tomar uma transição de desnaturação térmica:
- Fechar software de aquisição Arquivo> Sair e abrir um novo programa Medição de temperatura variável> Medir> Parâmetros .
- Aplique os seguintes parâmetros para registrar uma transição térmica, que pode ser ajustada conforme desejado:
- Sob temperatura , selecione a temperatura de início: 4 ° C; Intervalo: 0,2; TaRget: 90 ° C; Gradiente: 1 ° C / min; Aguarde 0 s. Em Iniciar / Fim , selecione Iniciar condição e condição final como desejado.
- Em Geral , selecione 3 Canais, CD / HT / Abs; Largura de banda: 1 nm; Comprimento de onda: 270 nm (ou conforme desejado). Em Controle , selecione nenhum. Em Informações , selecione nome, concentração, operador.
- Em dados , navegue até a pasta desejada. Clique em OK .
- Arrefecer as amostras a 4 ° C e esperar 5 min a esta temperatura antes de obter uma medição de espectro.
- Trabalho de dados para espectros.
- Usando a função no software, subtrair o espectro em branco da aquisição de destino para a conta dos sinais de fundo decorrentes do sistema de buffer em uso: Spectra Analysis> File> Open .
- Uma vez que um arquivo em branco e um arquivo de espectros foram abertos, arraste View 2 em View 1 (no lado esquerdo da tela).
- Clique em PrProcessamento> subtração> OK ou troca de dados> OK .
- Extraia dados como um arquivo ASCII: Arquivo> Exportar e selecionar ASCII .
- Use o software para traçar o espectro correspondente. O brilho dos dados é opcional nos casos em que a relação sinal-ruído é menor do que o esperado. Essa transformação pode ser aplicada aplicando a opção de suavização de dados.
- Processamento de dados para transições de desnaturação térmica.
- Extraia dados do arquivo JASCO como um arquivo ASCII.
- Trace os pontos de elipticidade em função da temperatura. Normalmente, o alisamento dos dados é necessário, dependendo do comprimento de onda que está sendo examinado. Em alguns casos, o uso do comprimento de onda em 210 nm apresentou variações maiores entre os lotes que requeriam alisamento. No entanto, dependendo da amostra e das concentrações, foram realizados vários cálculos utilizando os dados brutos.
- Para calcular a cova térmicaValor de transição de atuação (T m ), obtenha uma primeira derivada da curva. Os máximos ou mínimos foram uma indicação desse parâmetro. Como no passo anterior, alguns casos requeriam o alisamento dos dados para obter o valor mais preciso.
NOTA: As experiências foram tipicamente realizadas em triplicado, o que resultou na média correspondente e desvio padrão para a medição.
Resultados
A síntese de dodecamers de RNA contendo zero, uma ou duas modificações de 2-tiofenilmetilo na posição C2'- O é descrita juntamente com a sua correspondente purificação e caracterização. Além disso, está incluída uma descrição detalhada da análise estrutural que foi realizada via CD.
As quatro cadeias de ARN (incluindo uma cadeia com uma sequência complementar) foram obtidas através de síntese em...
Discussão
A intenção deste manuscrito é servir como um guia para pesquisadores no campo, iniciante ou especialista, para alcançar com sucesso ou melhorar a síntese de oligonucleótidos de DNA ou RNA. A metodologia descrita enfoca o uso de síntese de fase sólida usando um sintetizador automatizado de DNA / RNA através de química padrão de fosforamidite. O relatório descreve uma descrição passo-a-passo da síntese, purificação e caracterização de dodecamers de RNA. Além disso, o uso de CD é utilizado para identif...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
A preparação deste manuscrito foi apoiada por meio de fundos iniciais da Universidade de Colorado Denver (JMRE). A AF gostaria de reconhecer o apoio de um prêmio de Pesquisa e Atividades Criativas (RaCAS, CU Denver). Financiamento do Escritório de Serviços de Pesquisa, Universidade de Denver Colorado para cobrir os custos de publicação é reconhecido. Gostaríamos de agradecer aos membros do laboratório, Sra Cassandra Herbert e ao Sr. Yannick K. Dzowo, por suas contribuições na seção de vídeo.
Materiais
| Name | Company | Catalog Number | Comments |
| AbsolveTM | PerkinElmer | 6NE9711 | |
| Acetonitrile 99.9%, HPLC Grade | Fisher Scientific | 75-05-8 | |
| Acetonitrile 99.9%, anhydrous for DNA sequencing | Fisher BioReagents | 75-05-8 | |
| Acrylamide, 99+% | ACROS Organics | 164850025 | |
| Ammonium chloride 98+% | Alfa Aesar | 12125-02-9 | |
| Ammonium citrate, dibasic 98% | Sigma Aldrich | 3012-65-5 | |
| Ammonium Fluoride 98.0%, ACS grade | Alfa Aesar | 12125-01-8 | |
| Ammonium hydroxide 28 - 30% in water, ACS Plus | Fisher Chemical | 1336-21-6 | |
| Ammonium persulfate | ACROS Organics | 1444 | |
| Argon-ultra high purity | Airgas | 7440-37-1 | |
| Bis-acrylamide Ultra pure | VWR-Amresco | 172 | |
| Boric Acid | Fisher Scientific | A73-1 | |
| Diethyl pyrocarbonate, 97% | ACROS Organics | A0368487 | |
| Ethanol, anhydrous, histological grade | Fisher Chemical | 64-17-5 | |
| Ethylenediamine tetraacetic acid, disodium salt dehydrate 100.2% | Fisher Chemical | 6381-92-6 | |
| Formamide | Thermo Scientific | 75-12-7 | |
| Hydrochloric acid, 36.5 - 38.0%, Certified ACS Plus | Fisher Chemical | 7647-01-0 | |
| Magnesium chloride hexahydrate, 99% | Fisher Scientific | 7786-30-3 | |
| Methanol, 99.9%, HPLC Grade | Fisher Chemical | 67-56-1 | |
| Methylamine 40% in water | Sigma Aldrich | 74-89-5 | |
| 1-Methyl-2-pyrrolidinone, andhydrous, 99.5% | Aldrich | 872-50-4 | |
| Opti-TOFTM 96 Well Insert (123 x 81 mm) | MDS SCIEX | 1020157 | |
| RNase Away | Molecular BioProducts | 7005-11 | |
| Sodium acetate, anhydrous 99.2%, Certified ACS | Fisher Chemical | 127-09-3 | |
| Sodium chloride, 100.5%, Certified ACS | Fisher Chemical | 7647-14-5 | |
| Sodium phosphate monobasic dihydrate 99.0% | Sigma | 13472-35-0 | |
| 2’,4’ Triethylamine, 99+% | Alfa Aesar | 121-44-8 | |
| TEMED | Amresco | 761 | |
| Triethylamine trihydrofluoride, 98% | Aldrich | 73602-61-6 | |
| Trifluoroacetic acid, 99% | Alfa Aesar | 76-05-1 | |
| 6’-Trihydroxyacetophenone monohydrate 98% | Sigma Aldrich | 480-66-0 | |
| Tris Base | Fisher Scientific | BP154-3 | |
| Urea | Fisher Scientific | U15-3 | |
| Reagents for the RNA synthesis: | |||
| Deblocking mix, 3% trichloroacetic acid in dichloromethane | Glen Research | 40-4140-57 | |
| Cap Mix A, THF/Pyridine/Acetic anhydride | Glen Research | 40-4110-52 | |
| Cap Mix B, 10% 1-methylimidazole in THF | Glen Research | 40-4120-52 | |
| Activator, 0.25 M 5-ethylthio-1H-tetrazole in anhydrous acetonitrile | Glen Research | 30-3140-52 | |
| Oxidizing Solution, 0.02 M iodine in THF/Pyridine/Water | Glen Research | 40-4330-52 | |
| U-RNA-CPG | Glen Research | 20-3330-xx | |
| Ac-G-RNA-CPG | Glen Research | 20-3324-xx | |
| Ac-G-CE Phosphoramidite | Glen Research | 10-3025-xx | |
| U-CE Phosphoramidite | Glen Research | 10-3030-xx | |
| Ac-C-CE Phosphoramidite | Glen Research | 10-3015-xx | |
| Bz-A-CE Phosphoramidite | Glen Research | 10-3003-xx |
Referências
- Matteucci, M. D., Caruthers, M. H. The synthesis of oligodeoxypyrimidines on a polymer support. Tetrahedron Lett. 21 (8), 719-722 (1980).
- Beaucage, S. L., Caruthers, M. H. Deoxynucleoside phosphoramidites-A new class of key intermediates for deoxypolynucleotides. Tetrahedron Lett. 22 (20), 1859-1862 (1981).
- Caruthers, M. H. Gene synthesis machines: DNA Chemistry and its uses. Science. 230 (4723), 281-285 (1985).
- Nguyen, J. C., et al. Synthesis, Thermal Stability, Biophysical Properties, and Molecular Modeling of Oligonucleotides of RNA Containing 2'-O-2-Thiophenylmethyl Groups. J Org Chem. 81 (19), 8947-8958 (2016).
- El-Sagheer, A. H., Brown, T. Click chemistry with DNA. Chem Soc Rev. 39, 1388-1405 (2010).
- Puffer, B., et al. 5-Fluoro pyrimidines: labels to probe DNA and RNA secondary structures by 1D 19F NMR spectroscopy. Nucleic Acids Res. 37 (22), 7728-7740 (2009).
- Wan, W. B., Seth, P. P. The medicinal chemistry of therapeutic oligonucleotides. J Med Chem. 59 (21), 9645-9667 (2016).
- Zhou, C., Greenberg, M. M. DNA Damage by histone radicals in nucleosome core particles. J Am Chem Soc. 136 (18), 6562-6565 (2014).
- Zhang, Y., et al. UV-Induced proton-coupled electron transfer in cyclic DNA miniduplexes. J Am Chem Soc. 138 (23), 7395-7401 (2016).
- Anosova, I., et al. The structural diversity of artificial genetic polymers. Nucleic Acids Res. 44 (3), 1007-1021 (2016).
- Riml, C., Micura, R. Synthesis of 5-Hydroxymethylcytidine- and 5-Hydroxymethyl-uridine-Modified RNA. Synthesis. 48, 1108-1116 (2016).
- Anderson, B. A., Hrdlicka, P. J. Merging Two Strategies for Mixed-Sequence Recognition of Double-Stranded DNA: Pseudocomplementary Invader Probes. J Org Chem. 81 (8), 3335-3346 (2016).
- Beaucage, S. L., Iyer, R. P. Synthesis of oligonucleotides by the phosphoramidite approach. Tetrahedron. 48 (12), 2223-2311 (1992).
- Gillet, L. C. J., Alzeer, J., Schärer, O. D. Site-specific incorporation of N-(deoxyguanosin-8-yl)-2-acetylaminofluorene (dG-AAF) into oligonucleotides using modified 'ultra-mild' DNA synthesis. Nucleic Acids Res. 33 (6), 1961-1969 (2005).
- Shiba, Y., et al. Chemical synthesis of a very long oligoribonucleotide with 2-cyanoethoxymethyl (CEM) as the 2'-O.-protecting group: structural identification and biological activity of a synthetic 110mer precursor-microRNA candidate. Nucleic Acids Res. 35 (10), 3287-3296 (2007).
- Choi, Y. J., Gibala, K. S., Ayele, T., Deventer, K. D., Resendiz, M. J. E. Biophysical properties, thermal stability and functional impact of 8-oxo-7,8-dihydroguanine on oligonucleotides of RNA - a study of duplex, hairpins and the aptamer for preQ1 as models. Nucleic Acids Res. 45 (4), 2099-2111 (2017).
- Ranjbar, B., Gill, P. Circular dichroism techniques: Biomolecular and nanostructural analyses - A review. Chem Biol Drug Des. 74 (2), 101-120 (2009).
- Mergny, J. -. L., Lacroix, L. Analysis of thermal melting curves. Oligonucleotides. 13 (6), 515-537 (2003).
- Jin, R., Breslauer, K. J., Jones, R. A., Gaffney, B. L. Tetraplex formation of a guanine-containing nonameric DNA fragment. Science. 250 (4980), 543-546 (1990).
- Virgilio, A., et al. 5-Hydroxymethyl-2'-deoxyuridine residues in the thrombin binding aptamer: Investigating anticoagulant activity by making a tiny chemical modification. CHEMBIOCHEM. 15 (16), 2427-2434 (2014).
- Chauca-Diaz, A. M., Choi, Y. J., Resendiz, M. J. E. Biophysical properties and thermal stability of oligonucleotides of RNA containing 7,8-dihydro-8-hydroxyadenosine. Biopolymers. 103 (3), 167-174 (2015).
- Maniatis, T., Fritsch, E. F., Sambrook, J. . Molecular cloning: a laboratory manual. 14, 173-185 (1987).
- Markham, N. R., Zuker, M. UNAFold: software for nucleic acid folding and hybridization. Methods Mol Biol. 453, 3-31 (2008).
- Breslow, R., Huang, D. -. L. Effects of metal ions including Mg2+ and lanthanides on the cleavage of ribonucleotides and RNA model compounds. Proc Natl Acad Sci U S A. 88 (10), 4080-4083 (1991).
- Blumberg, D. D. Creating a ribonuclease-free environment. Methods Enzymol. 152 (2), 20-24 (1987).
- AbouHaidar, M. G., Ivanov, I. G. Non-enzymatic RNA promoted by the combined catalytic activity of buffers and magnesium ions. Z Naturforsch C. 54 (7-8), 542-548 (1999).
- Farrell, R. E. . RNA Methodologies (4th Ed): A laboratory guide for isolation and characterization. 2, 45-80 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados