Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Протокол твердофазного синтеза олигомеров РНК, содержащий 2'-
В этой статье
Резюме
В этой статье представлена подробная процедура твердофазного синтеза, очистки и характеризации додекамеров РНК, модифицированных при C2'- O -позиции. Фотометрический анализ методом ультрафиолетового и кругового дихроизма используется для количественной оценки и характеристики структурных аспектов, то есть одножильных или двухжильных.
Аннотация
Твердофазный синтез был использован для получения канонических и модифицированных полимеров нуклеиновых кислот, в частности ДНК или РНК, что сделало его популярной методологией для применений в различных областях и для различных исследовательских целей. Описанная здесь процедура фокусируется на синтезе, очистке и характеристике додекамеров РНК 5 '- [CUA CGG AAU CAU] -3', содержащих нулевую, одну или две модификации, расположенные на C2'- O -позиции. Зонды основаны на 2-тиофенилметильных группах, включенных в РНК-нуклеотиды посредством стандартного органического синтеза и вводимых в соответствующие олигонуклеотиды через их соответствующие фосфорамидиты. В этом отчете используется химия фосфорамидита через четыре канонические нуклеотиды (Uridine (U), Cytosine (C), гуанозин (G), аденозин (A)), а также 2-тиофенилметилфункциональные нуклеотиды, модифицированные на 2'- O- должность; Однако, эта методология применима к большому varКоторые были разработаны на протяжении многих лет. Олигонуклеотиды синтезировали на носителе с контролируемым поровым стеклом (CPG) с последующим отщеплением из смолы и снятием защиты в стандартных условиях, то есть смесью аммиака и метиламина (АМА) с последующим фторидом водорода / триэтиламином / N-метилпирролидиноном. Соответствующие олигонуклеотиды очищали с помощью полиакриламидного электрофореза (20% денатурации) с последующим элюированием, обессоливанием и выделением с помощью хроматографии с обращенной фазой (Sep-pak, C 18- column). Количественную оценку и структурные параметры оценивали с помощью ультрафиолетового (UV-vis) и кругового дихроизма (CD) фотометрического анализа, соответственно. Этот доклад призван служить в качестве ресурса и руководства для начинающих и экспертов-исследователей, заинтересованных в походах в эту область. Ожидается, что он будет работать в процессе разработки, поскольку будут разработаны новые технологии и методологии. Описание методологий и методов вS соответствует синтезу ДНК / РНК (восстановленному и приобретенному в 2013 году), который использует химию фосфорамидита.
Введение
Твердофазный синтез для получения олигонуклеотидов ДНК / РНК является мощным инструментом, который обслуживал несколько приложений в различных областях с 1970-х годов 1 , 2 , 3 с использованием блоков 4 фосфорамидита. Примеры его широкого влияние включают в себя: его воздействие в маркировке ( с помощью химии нажмите реакции) 5, структурного зондирующее 6 и антисмысловые технологии 7, а также его выяснение биологических механизмов 8, 9, источника в качестве генетического материала 10, и изучение Различные природные и / или химические модификации 11 , 12 , среди многих других. Модификация, которую мы здесь используем, представляет собой первый шаг в наших усилиях по получению олигонуклеотидов РНК, которые содержатФотоактивные зонды для обеспечения временного контроля структуры и функции этого важного биополимера.
Синтез додекамеров РНК с последовательностями: 5 '- [CUA CG G A AU CAU] -3' / 5 '- [AUG AUU CCG UAG] -3' (подчеркнутые положения представляют собой включение C2'- O- тиофенилметильной модификации ) Составляет основное внимание в этом исследовании. Последовательности были выбраны так, чтобы обеспечить количественную оценку и измерение нитей РНК как одиночных цепей или их соответствующих дуплексных структур (никакие другие вторичные структуры не были предсказаны как термодинамически стабильные). CD был использован для установления структурных параметров, т. Е. Дуплексного образования и термических денатурационных переходов.
Синтез
Общая процедура получения этих олигонуклеотидов проиллюстрирована на рисунке 1 и следует поэтапному процессу: автоматизированный твердофазный синтез → DeprOtection → Очистка → Количественная оценка → Характеристика. На рисунке 2 показаны мономерные единицы, которые необходимы в этой процедуре. Твердофазный синтез РНК аналогичен твердофазному синтезу ДНК, поскольку он основан на химии фосфорамидитов ( рис. 2 , слева) и использовании базово-лабильных защитных групп для нуклеофильных экзоциклических аминов на G, A и C, например , Ацетил, бензоил, феноксиацетил, трет- бутил или N , N- диметилформамид ( фиг. 2 , справа). Еще один аспект, который следует учитывать в РНК из-за присутствия C2'-OH-группы (отсутствующей в дезоксиолигонуклеотидных биополимерах), является дополнительной стадией, которая должна быть включена для защиты и последующего снятия защиты с этой нуклеофильной позиции. В этом отношении защитные группы на основе кремния стали привлекательной стратегией из-за их потенциала как биортогональных фрагментов (specificallY сняли защиту в присутствии фторида), так как группы трет- бутилдиметилсилил (TBDMS) и триизопропилсилилоксиметил (TOM) являются популярными ( рис. 2 , внизу слева).
В этой работе автоматизированный синтез проводили на синтезаторе ДНК / РНК, который использует стандартную химию фосфорамидита. Настройки изготовителя на инструменте включают автоматическую стадию разбавления при использовании коммерческих версий фосфорамидитов для ДНК или возможность разбавления в объемах, заданных пользователем. Однако мы решили взвешивать РНК фосфорамидит и разбавлять вручную, учитывая, что: 1) цена канонических фосфорамидитов РНК выше (в некоторых случаях в 50 раз дороже); 2) модифицированные фосфорамидиты часто получают в небольших количествах; И 3) количество затраченного материала при использовании этапа автоматизированного разбавления (устанавливается изготовителем) является большим. Кроме того, мы использовали: 1) коммерчески доступные твердые опоры( Например , CPG), содержащую защищенную нуклеотидную основу, чтобы функционировать как 3'-конец; И 2) коммерческие фосфорамидиты (канонические нуклеотидные основания), защищенные группой TBDMS при C2'- O -позиции. Подробный перечень этапов синтеза представлен на рис. 3 и в таблице 1 вместе с дальнейшим описанием и комментариями для шагов, которые были скорректированы для синтеза РНК. Кроме того, на рисунке 4 показаны ступенчатые выходы, которые наблюдаются для каждого шага после выбора опции «Trityl Monitor», которая количественно оценивает тритильный катион, высвобождаемый с каждого этапа дебитиляции.
Стоит отметить, что, как нам показалось, лимитирующим фактором является получение фосфорамидита, содержащего требуемую модификацию. То есть, разработка синтетической методологии, которая позволяет вносить изменения в отдельные сайты. В этом отчете мы сосредоточим внимание наВведение модифицированного нуклеотида, для которого была установлена соответствующая синтетическая методика, C2'- O- тиофенилметильная группа. Эта группа мала по размеру и никоим образом не влияет на твердофазный синтез. Поскольку сообщалось о включении этой группы в олигонуклеотиды РНК, наряду со структурными и термодинамическими параметрами 4 , здесь не будут описаны аспекты органического синтеза, приводящего к модифицированным фосфорамидитам.
Снятие защиты, очистка и характеристика
Снятие защиты с экзоциклических аминов и ß-цианоэтильных групп происходит на той же стадии, что и расщепление от CPG-смолы. Мы применили общеупотребительные условия нагревания полученной смолы в присутствии водного раствора АМА с последующим расщеплением C2'- O- силильных групп в присутствии ионов фторида и затем очисткой через гельэлектрофорез. Хотя во многих случаях они стали стандартными условиями, модификации, которые являются лабильными для основных условий или ионов фторида, могут потребовать более мягкие условия 13 , 14 , например , карбонат метанола / калия (MeOH / K 2 CO 3 ) или бутиламин. Таким образом, необходим другой набор защитных групп на соответствующих фосфорамидитах. Кроме того, мы выбрали электрофорез в качестве предпочтительной альтернативы очистке снятых защитных олигомеров, учитывая наш предыдущий опыт этого метода и отсутствие других инструментов. Однако ВЭЖХ можно альтернативно использовать в качестве эффективного метода 15 . Характеристика очищенных олигонуклеотидов проводилась с помощью масс-спектрометрии, лазерной десорбции / ионизации-времени полета (МАЛДИ-ТОФ) с матричной поддержкой, с использованием описанной процедуры нашей группой 16 .
Структурная характеристика и Устойчивость полученных дуплексов была выполнена с помощью CD. В частности, мы используем CD для определения температурных денатурационных переходов модифицированных и немодифицированных олигонуклеотидов РНК, следуя уменьшению эллиптичности полосы прибл. 270 нм, а также исчезновение полосы (с отрицательной эллиптичностью) с λ max при 210 нм. Сравнение спектров до и после гибридизации предоставляется для иллюстрации их различий и обеспечения валидации применяемой методологии. Использование CD широко принимается при определении структурных мотивов в нуклеиновых кислотах и аминокислотах 17 и поэтому может быть использовано в качестве инструмента для определения различных структурных и термодинамических параметров 18 ; Однако, существует мало примеров, где метод используется для оценки тепловых денатурационных переходов. Некоторые случаи включают определение термической стабильности на ДНК, содержащей G-квадруплексыAss = "xref"> 19 , 20 или в дуплексах и шпильках РНК 21 .
Этот отчет предназначен для предоставления читателю или зрителю, не являющемуся экспертом, набором инструментов, которые позволят обеспечить плавный запуск этого типа исследований. Он будет способствовать усилению и сопоставлению с методологиями и методами в других исследовательских лабораториях, которые участвуют в этой увлекательной отрасли науки. Содержание в этом отчете добавляет к существующим протоколам этой технологии из разных источников и обогащает и облегчает опыт визуальной помощи для каждого шага.
протокол
1. Твердофазный синтез олигонуклеотидов РНК
- Получение растворов, содержащих каждый фосфорамидит (табл. 1).
- Подсчитайте количество нуклеотидов и поместите в уравнение n + 1 (где n = количество нуклеотидов), соответствующее каждой базе, и заполните таблицу отсутствующими значениями. Объем = 0,15 мл на нуклеотид в концентрации 0,1 М, растворенный в безводном ацетонитриле.
- Взвесьте каждый фосфорамидит в высушенные в печи 10 мл янтарные бутылки (Septum Top Amber 394 Amidite 13 мм ID x 20 мм OD Top) и сразу же помещайте под вакуумом, используя эксикатор, содержащий осушитель.
- Заполните эксикатор сухим аргоном, удалите бутылку и немедленно разбавьте соответствующим количеством ацетонитрила (безводного) перед закреплением на инструменте.
ПРИМЕЧАНИЕ. Обязательно используйте газонепроницаемые шприцы и всегда сохраняйте безводную атмосферу. - Удалите перегородку из бутылки и поместите на мAchine, через функцию замены бутылки.
ПРИМЕЧАНИЕ. Этот процесс требует дополнительного 0,15 мл (следовательно, избыточного объема в уравнении n + 1 выше) каждого раствора, чтобы заполнить линию до синтеза.
- Настройка последовательности и твердофазного синтеза с использованием программного обеспечения ( рисунок 3 ).
- Нажмите на значок программного обеспечения ( например , OligoNet 1.0.1) и выберите « Имя инструмента»> «ОК» . Создайте новый синтез с помощью File> New Synthesis> Order .
- Заполнение: дата; RunID; Выбрать инструмент; Имя последовательности; Последовательность (5'-to-3 'конец). В разделе | цикл назначьте ранее созданный метод ( рисунок 3 ). Выберите конечную процедуру, ( линит олигонуклеотид, связанный с смолой CPG)> руководство.
- Выберите DMT Off> (обработка TCA на последнем этапе для получения 5'-OH-группы в конце синтеза) ; Сохранить Файл> Сохранить какИ дать имя для эксперимента.
- Отправьте файл для начала синтеза. Заказ> Отправьте заказ на синтезатор и выберите столбец. 1-4 . Откройте окно синтезатора и выберите trityl monitor | выберите функцию | trity> отслеживайте каждый шаг .
- Повторите шаги по мере необходимости в зависимости от количества синтезируемых олигонуклеотидов сразу (обратите внимание, что все заказы должны иметь один и тот же метод, то есть одинаковые моменты времени и последовательность событий).
- Начните синтез синтезатора> Подготовьтесь к запуску . Поместите колонки с нужным 3'-концом в положения, указанные на инструменте. На инструменте нажмите « Пуск»> «Нет» (для подготовки ABI).
- Как только синтез завершен, удалите колонку с инструмента и поместите в круглодонную колбу с последующей сушкой при пониженном давлении в течение примерно 0,5 часа.
ПРИМЕЧАНИЕ. Рекомендуется проверить, что темно-оранжевый цвет наблюдается в randOm (шаги 1.2.4 - 1.2.7), чтобы избежать потенциальных ошибок функции trityl monitor.
- Дезактивация и очистка олигонуклеотидов РНК.
- Откройте колонку, скручивая черный колпачок и переместите все (или половину, в зависимости от потребности или цели) белой смолы в 1,6 мл центрифужную пробирку. Добавить 0,5 мл метиламина (40% в воде) / аммиака (40% в воде) при 1: 1.
- Закрепите пробку крышки центрифуги с помощью парафильма и, используя тепловой блок, нагрейте до 60 ° C в течение 1,5 часов.
ПРИМЕЧАНИЕ. Тяжелый предмет может быть помещен сверху трубки, чтобы гарантировать, что концентрация аммиака остается постоянной внутри реакционной трубы. - Удалите из теплового блока и медленно остудите до комнатной температуры. Кратко центрифугируйте образец для отжимания содержимого, затем переместите супернатант в новую пробирку для центрифугирования.
- Заморозьте образцы погружными трубами в жидком азоте (или сухом льду / этаноле) и концентрируйте досуха,В центрифуге при пониженном давлении.
- Повторно суспендируют твердые вещества в 0,4 мл раствора триэтиламин / N-метилпирролидинон / триэтиламинтригидрофторид (соотношение 2: 2: 3). Нагревают до 60 ° С в течение 1,5 ч, а затем медленно охлаждают до комнатной температуры.
- Добавить 0,04 мл раствора NaOAc (3M, pH 5,5 - с помощью HCl) с последующим осторожным перемешиванием с наконечником пипетки. Добавить этанол (1 мл) и охладить до примерно 200 мл. -70 ° C (сухой лед / этанольная ванна) в течение 15 мин.
- Центрифуга при 15000 об / мин и 4 ° С в течение 10 мин. Используйте пипетку для извлечения аликвоты и высушите полученный осадок при пониженном давлении.
- Повторно суспендируют полученное твердое вещество в загрузочном буфере (0,2 мл, 90% водный раствор формамида, 1 мМ ЭДТА) и перемешивают до однородности смеси.
- Загрузите суспензию на предварительно приготовленный полиакриламидный гель (20% денатурации, размеры лунки: 30 см x 1 мм) 22 .
- Нанесите ток через гель до тех пор, пока маркер бромфенола не будет находиться между 1 / 2-2 / 3Вниз по гелю (размеры геля: ~ 21,5 см х 30 см).
- Удалите гель с подставки и перенесите содержимое геля из стекла в пластиковую пленку (покрытую с обеих сторон) и поместите на тонкослойную хроматографическую (TLC) пластину, покрытую кремнеземом (содержащим флуоресцентный краситель при 254 нм), чтобы визуализировать полосы С использованием УФ-лампы (λ max = 254 нм).
- Используйте маркер, чтобы очертить положение, в котором находится верхняя полоса, и разрезать его с помощью нового лезвия бритвы. Поместите гель, содержащий РНК (без пластиковой обертки) в 50-миллилитровую коническую трубку и раздавите на мелкие кусочки, используя стеклянный стержень.
- Приостановить остатки геля в водном растворе NaCl. Раствора (2 мМ и 1 мМ ЭДТА) и встряхивают суспензию при 37 ° С в течение 12 часов. Центрифугируйте коническую трубку в течение 10 мин.
- Desalt с использованием картриджа C18 с обратной фазой.
- Подготовьте картридж, используя 10-мл шприц, путем промывки:
Ацетонитрил (10 мл)
H 2 O (дважды, 10 мл)
5 мМ NH4 Cl aq. Раствор (3 мл)
Раствор, содержащий РНК, заботясь о том, чтобы не налить какой-либо из остатков геля. - Промыть H 2 O (три раза, 10 мл).
- Элюат из колонки с использованием 60% вод. Метанол (3 мл)
- Подготовьте картридж, используя 10-мл шприц, путем промывки:
- Концентрируют при пониженном давлении и повторно растворяют в воде без РНКазы (0,3 мл)
- Подготовьте разбавленный раствор (10 мкл) и отложите 1 мкл на УФ-образном приборе ( например , Nanodrop) для измерения спектра УФ-видимости (200-450 нм).
- Используйте Закон Пива Ламберта, чтобы определить концентрацию полученного решения:
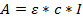
Где A = полученное поглощение; Ε = расчетный молярный коэффициент экстинкции; C = концентрация и l = 0,1 для длины пути 1 мм. - Вычислите молярные коэффициенты экстинкции для олигонуклеотидов; Рассчитывается здесь с помощью онлайн-калькулятораКоторый использует программное обеспечение DINAMelt (http://unafold.ra.albany.edu/?q=dinamelt) 23
- Определение концентрации и характеристики через MALDI-TOF.
- Загрузите образцы на пластину MALDI, используя наконечник пипетки, нагруженный кончиком C18, чтобы отслаиваться, и укажите каждый олигонуклеотид.
- Промойте наконечник 50% ацетонитрилом (10 мкл × 2). Уравновешивают наконечник 0,1% трифторуксусной кислотой (TFA, 10 мкл × 2). Загрузите наконечник с образцом (обычно 100-150 пмоль).
- Промойте наконечник 0,1% TFA (10 мкл × 2), а затем водой (10 мкл × 2).
- Элюировать образец в раствор, содержащий желаемую матрицу; Мы использовали раствор: 10 мкл моногидрата 25 мМ-2,4,6-тригидроксиацетофенона (THAP), 10 мМ цитрата аммония и 300 мМ фторида аммония в 50% ацетонитриле.
- Пятно непосредственно на пластину MALDI путем осаждения 0,9 мкл (с последующей сушкой на воздухе) и повторения процедуры по мере необходимости.
ПРИМЕЧАНИЕ. Все спектры были получены в положительном режиме рефлектора.
- Загрузите образцы на пластину MALDI, используя наконечник пипетки, нагруженный кончиком C18, чтобы отслаиваться, и укажите каждый олигонуклеотид.
2. Анализ структуры РНК с помощью CD
- Подготовка растворов для CD.
- Подготовьте 0,25 мл, содержащий модифицированную РНК [3 мкМ], NaCl [10 мМ], натрийфосфатный буфер [10 мМ, рН 7,2] и MgCl 2 [5 мМ]. Образец готов к анализу как есть, особенно если он представляет собой мономолекулярный переход.
- Если цель состоит в анализе дуплексных структур или бимолекулярных переходов, добавьте дополнение (1 молярный эквивалент, если применимо) в это время или продолжите шаг ниже.
- Поместите образец на тепловой блок, предварительно нагретый до 90 ° C и выключите тепло для управления медленным охлаждением до комнатной температуры (обычно 2-4 часа).
- Приобретение спектров
- Подготовьте чистый раствор (NaCl 10 мМ, фосфат натрия 10 мМ, pH 7,3, MgCl 2 5 мМ), переносят t(1 см длины пути, минимальный объем 250 мкл) и поместите в положение держателя смены образца в инструменте.
- Перенесите образец, содержащий РНК, в другую микро-кювету и положение в сменщике образцов. При измерении перехода термического денатурации аккуратно добавьте слой масла без нарушения водного слоя и закрепите колпачок кюветы с помощью куска тефлоновой ленты.
- Откройте азотный резервуар, чтобы обеспечить поток, который перемещает воздушный поплавок, расположенный на инструменте, до ок. 40. Включите кулер.
- Включите прибор и откройте значок программного обеспечения Spectra Manager , затем откройте окно сбора данных Spectra Measurement .
- Продуйте прибор азотом в течение 5 мин до получения.
- Приобретите спектры, используя следующие параметры Measure> Parameters и отрегулируйте параметры следующим образом:
- В разделе « Общие» выберите: Сканирование 200-350 нм; Каналы CD и HT: данныеШаг при 0,1 нм; Скорость сканирования со скоростью 100 нм / мин; Ширина полосы при 1 нм; Количество скоплений в 5.
- В ячейке ячейки выберите 20 ° C. Под управлением выберите Затвор автоматически открывается и закрывается. Под информацией выберите название, концентрацию, оператор.
- В разделе данных найдите нужную папку. Нажмите « ОК» .
- Приобретите спектры Измерение> Измерение образца , определите положение (и) кюветы в образце смены 1-6 соответственно, нажмите « ОК» .
- Если взять переход термической денатурации:
- Закрыть Программное обеспечение для сбора данных Файл> Выход и открытие новой программы Измерение переменной температуры> Измерение> Параметры .
- Примените следующие параметры для записи теплового перехода, который можно настроить по желанию:
- В разделе « Температура» выберите начальную температуру: 4 ° C; Интервал: 0,2; таRget: 90 ° C; Градиент: 1 ° С / мин; Подождать 0 с. В разделе « Начало / конец» выберите «Запустить условие и состояние окончания».
- В разделе « Общие» выберите 3 канала, CD / HT / Abs; Полоса пропускания: 1 нм; Длина волны: 270 нм (или по желанию). В разделе « Управление» выберите «Нет». В разделе « Информация» выберите имя, концентрацию, оператор.
- В разделе данных найдите нужную папку. Нажмите « ОК» .
- Прохладите образцы до 4 ° C и подождите 5 минут при этой температуре перед получением измерения спектра.
- Обработка данных для спектров.
- Используя функцию в программном обеспечении, вычтите пустой спектр из целевого захвата для учета фоновых сигналов, возникающих из используемой буферной системы: Spectra Analysis> File> Open .
- Как только пустой файл и файл спектров будут открыты, перетащите View 2 под View 1 (в левой части экрана).
- Нажмите на Pr> Вычитание> OK или обмен данными> OK .
- Извлечение данных в виде файла ASCII: Файл> Экспорт и выбор ASCII .
- Используйте программное обеспечение для построения соответствующего спектра. Сглаживание данных является необязательным в случаях, когда отношение сигнал-шум ниже ожидаемого. Это преобразование может быть применено путем применения параметра сглаживания данных.
- Обработка данных для термических денатурационных переходов.
- Извлечь данные из файла JASCO в виде файла ASCII.
- Постройте точки эллиптичности как функцию температуры. Как правило, сглаживание данных необходимо в зависимости от длины волны, которая исследуется. В некоторых случаях использование длины волны при 210 нм показало большие различия между графиками, которые требовали сглаживания. Однако в зависимости от образца и концентраций был проведен ряд расчетов с использованием исходных данных.
- Для вычисления теплового логарифма(T m ), получим первую производную кривой. Показателями этого параметра были максимальные или минимальные значения. Как и в предыдущем шаге, некоторые случаи требовали сглаживания данных для получения наиболее точного значения.
ПРИМЕЧАНИЕ. Эксперименты обычно проводились в трех экземплярах, что приводило к соответствующему среднему и стандартным отклонениям для измерения.
Результаты
Описывается синтез додекамеров РНК, содержащих нулевую, одну или две 2-тиофенилметильные модификации при C2'- O -позиции наряду с соответствующей ее очисткой и характеристикой. Кроме того, включено подробное описание структурного анализа, проведенного с помощью ...
Обсуждение
Цель этой рукописи - служить руководством для исследователей в области, начинающих или экспертов, для успешного достижения или усиления синтеза олигонуклеотидов ДНК или РНК. Описанная методика фокусируется на использовании твердофазного синтеза с использованием автоматизированног?...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Подготовка этой рукописи была поддержана через пусковые фонды из Университета Колорадо Денвер (JMRE). AF хотела бы подтвердить поддержку от конкурса исследований и творческих действий (RaCAS, CU Denver). Признается финансирование со стороны Управления исследовательских служб Университета Колорадо Денвер для покрытия расходов на публикацию. Мы хотели бы поблагодарить сотрудников лаборатории г-жу Кассандру Герберт и г-на Янника К. Дзово за их вклад в видеоролик.
Материалы
| Name | Company | Catalog Number | Comments |
| AbsolveTM | PerkinElmer | 6NE9711 | |
| Acetonitrile 99.9%, HPLC Grade | Fisher Scientific | 75-05-8 | |
| Acetonitrile 99.9%, anhydrous for DNA sequencing | Fisher BioReagents | 75-05-8 | |
| Acrylamide, 99+% | ACROS Organics | 164850025 | |
| Ammonium chloride 98+% | Alfa Aesar | 12125-02-9 | |
| Ammonium citrate, dibasic 98% | Sigma Aldrich | 3012-65-5 | |
| Ammonium Fluoride 98.0%, ACS grade | Alfa Aesar | 12125-01-8 | |
| Ammonium hydroxide 28 - 30% in water, ACS Plus | Fisher Chemical | 1336-21-6 | |
| Ammonium persulfate | ACROS Organics | 1444 | |
| Argon-ultra high purity | Airgas | 7440-37-1 | |
| Bis-acrylamide Ultra pure | VWR-Amresco | 172 | |
| Boric Acid | Fisher Scientific | A73-1 | |
| Diethyl pyrocarbonate, 97% | ACROS Organics | A0368487 | |
| Ethanol, anhydrous, histological grade | Fisher Chemical | 64-17-5 | |
| Ethylenediamine tetraacetic acid, disodium salt dehydrate 100.2% | Fisher Chemical | 6381-92-6 | |
| Formamide | Thermo Scientific | 75-12-7 | |
| Hydrochloric acid, 36.5 - 38.0%, Certified ACS Plus | Fisher Chemical | 7647-01-0 | |
| Magnesium chloride hexahydrate, 99% | Fisher Scientific | 7786-30-3 | |
| Methanol, 99.9%, HPLC Grade | Fisher Chemical | 67-56-1 | |
| Methylamine 40% in water | Sigma Aldrich | 74-89-5 | |
| 1-Methyl-2-pyrrolidinone, andhydrous, 99.5% | Aldrich | 872-50-4 | |
| Opti-TOFTM 96 Well Insert (123 x 81 mm) | MDS SCIEX | 1020157 | |
| RNase Away | Molecular BioProducts | 7005-11 | |
| Sodium acetate, anhydrous 99.2%, Certified ACS | Fisher Chemical | 127-09-3 | |
| Sodium chloride, 100.5%, Certified ACS | Fisher Chemical | 7647-14-5 | |
| Sodium phosphate monobasic dihydrate 99.0% | Sigma | 13472-35-0 | |
| 2’,4’ Triethylamine, 99+% | Alfa Aesar | 121-44-8 | |
| TEMED | Amresco | 761 | |
| Triethylamine trihydrofluoride, 98% | Aldrich | 73602-61-6 | |
| Trifluoroacetic acid, 99% | Alfa Aesar | 76-05-1 | |
| 6’-Trihydroxyacetophenone monohydrate 98% | Sigma Aldrich | 480-66-0 | |
| Tris Base | Fisher Scientific | BP154-3 | |
| Urea | Fisher Scientific | U15-3 | |
| Reagents for the RNA synthesis: | |||
| Deblocking mix, 3% trichloroacetic acid in dichloromethane | Glen Research | 40-4140-57 | |
| Cap Mix A, THF/Pyridine/Acetic anhydride | Glen Research | 40-4110-52 | |
| Cap Mix B, 10% 1-methylimidazole in THF | Glen Research | 40-4120-52 | |
| Activator, 0.25 M 5-ethylthio-1H-tetrazole in anhydrous acetonitrile | Glen Research | 30-3140-52 | |
| Oxidizing Solution, 0.02 M iodine in THF/Pyridine/Water | Glen Research | 40-4330-52 | |
| U-RNA-CPG | Glen Research | 20-3330-xx | |
| Ac-G-RNA-CPG | Glen Research | 20-3324-xx | |
| Ac-G-CE Phosphoramidite | Glen Research | 10-3025-xx | |
| U-CE Phosphoramidite | Glen Research | 10-3030-xx | |
| Ac-C-CE Phosphoramidite | Glen Research | 10-3015-xx | |
| Bz-A-CE Phosphoramidite | Glen Research | 10-3003-xx |
Ссылки
- Matteucci, M. D., Caruthers, M. H. The synthesis of oligodeoxypyrimidines on a polymer support. Tetrahedron Lett. 21 (8), 719-722 (1980).
- Beaucage, S. L., Caruthers, M. H. Deoxynucleoside phosphoramidites-A new class of key intermediates for deoxypolynucleotides. Tetrahedron Lett. 22 (20), 1859-1862 (1981).
- Caruthers, M. H. Gene synthesis machines: DNA Chemistry and its uses. Science. 230 (4723), 281-285 (1985).
- Nguyen, J. C., et al. Synthesis, Thermal Stability, Biophysical Properties, and Molecular Modeling of Oligonucleotides of RNA Containing 2'-O-2-Thiophenylmethyl Groups. J Org Chem. 81 (19), 8947-8958 (2016).
- El-Sagheer, A. H., Brown, T. Click chemistry with DNA. Chem Soc Rev. 39, 1388-1405 (2010).
- Puffer, B., et al. 5-Fluoro pyrimidines: labels to probe DNA and RNA secondary structures by 1D 19F NMR spectroscopy. Nucleic Acids Res. 37 (22), 7728-7740 (2009).
- Wan, W. B., Seth, P. P. The medicinal chemistry of therapeutic oligonucleotides. J Med Chem. 59 (21), 9645-9667 (2016).
- Zhou, C., Greenberg, M. M. DNA Damage by histone radicals in nucleosome core particles. J Am Chem Soc. 136 (18), 6562-6565 (2014).
- Zhang, Y., et al. UV-Induced proton-coupled electron transfer in cyclic DNA miniduplexes. J Am Chem Soc. 138 (23), 7395-7401 (2016).
- Anosova, I., et al. The structural diversity of artificial genetic polymers. Nucleic Acids Res. 44 (3), 1007-1021 (2016).
- Riml, C., Micura, R. Synthesis of 5-Hydroxymethylcytidine- and 5-Hydroxymethyl-uridine-Modified RNA. Synthesis. 48, 1108-1116 (2016).
- Anderson, B. A., Hrdlicka, P. J. Merging Two Strategies for Mixed-Sequence Recognition of Double-Stranded DNA: Pseudocomplementary Invader Probes. J Org Chem. 81 (8), 3335-3346 (2016).
- Beaucage, S. L., Iyer, R. P. Synthesis of oligonucleotides by the phosphoramidite approach. Tetrahedron. 48 (12), 2223-2311 (1992).
- Gillet, L. C. J., Alzeer, J., Schärer, O. D. Site-specific incorporation of N-(deoxyguanosin-8-yl)-2-acetylaminofluorene (dG-AAF) into oligonucleotides using modified 'ultra-mild' DNA synthesis. Nucleic Acids Res. 33 (6), 1961-1969 (2005).
- Shiba, Y., et al. Chemical synthesis of a very long oligoribonucleotide with 2-cyanoethoxymethyl (CEM) as the 2'-O.-protecting group: structural identification and biological activity of a synthetic 110mer precursor-microRNA candidate. Nucleic Acids Res. 35 (10), 3287-3296 (2007).
- Choi, Y. J., Gibala, K. S., Ayele, T., Deventer, K. D., Resendiz, M. J. E. Biophysical properties, thermal stability and functional impact of 8-oxo-7,8-dihydroguanine on oligonucleotides of RNA - a study of duplex, hairpins and the aptamer for preQ1 as models. Nucleic Acids Res. 45 (4), 2099-2111 (2017).
- Ranjbar, B., Gill, P. Circular dichroism techniques: Biomolecular and nanostructural analyses - A review. Chem Biol Drug Des. 74 (2), 101-120 (2009).
- Mergny, J. -. L., Lacroix, L. Analysis of thermal melting curves. Oligonucleotides. 13 (6), 515-537 (2003).
- Jin, R., Breslauer, K. J., Jones, R. A., Gaffney, B. L. Tetraplex formation of a guanine-containing nonameric DNA fragment. Science. 250 (4980), 543-546 (1990).
- Virgilio, A., et al. 5-Hydroxymethyl-2'-deoxyuridine residues in the thrombin binding aptamer: Investigating anticoagulant activity by making a tiny chemical modification. CHEMBIOCHEM. 15 (16), 2427-2434 (2014).
- Chauca-Diaz, A. M., Choi, Y. J., Resendiz, M. J. E. Biophysical properties and thermal stability of oligonucleotides of RNA containing 7,8-dihydro-8-hydroxyadenosine. Biopolymers. 103 (3), 167-174 (2015).
- Maniatis, T., Fritsch, E. F., Sambrook, J. . Molecular cloning: a laboratory manual. 14, 173-185 (1987).
- Markham, N. R., Zuker, M. UNAFold: software for nucleic acid folding and hybridization. Methods Mol Biol. 453, 3-31 (2008).
- Breslow, R., Huang, D. -. L. Effects of metal ions including Mg2+ and lanthanides on the cleavage of ribonucleotides and RNA model compounds. Proc Natl Acad Sci U S A. 88 (10), 4080-4083 (1991).
- Blumberg, D. D. Creating a ribonuclease-free environment. Methods Enzymol. 152 (2), 20-24 (1987).
- AbouHaidar, M. G., Ivanov, I. G. Non-enzymatic RNA promoted by the combined catalytic activity of buffers and magnesium ions. Z Naturforsch C. 54 (7-8), 542-548 (1999).
- Farrell, R. E. . RNA Methodologies (4th Ed): A laboratory guide for isolation and characterization. 2, 45-80 (2010).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены