È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Protocollo per la sintesi in fase solida di oligomeri di RNA contenente un 2'-
In questo articolo
Riepilogo
Questo articolo fornisce una procedura dettagliata sulla sintesi a fase solida, la purificazione e la caratterizzazione di dodecameri di RNA modificati nella posizione C2'- O . Le analisi fotometriche UV-vis e dicroismo circolare sono utilizzate per quantificare e caratterizzare gli aspetti strutturali, cioè singoli fili o doppioni.
Abstract
La sintesi a fase solida è stata utilizzata per ottenere polimeri canonici e modificati di acidi nucleici, in particolare di DNA o RNA, che lo ha reso una metodologia popolare per applicazioni in vari settori e per scopi diversi di ricerca. La procedura qui descritta si concentra sulla sintesi, la purificazione e la caratterizzazione di dodecameri di RNA 5 '- [CUA CGG AAU CAU] -3' contenenti zero, una o due modifiche situate alla posizione C2'- O . Le sonde sono basate sui gruppi 2-tiofenilmetilici, incorporati in nucleotidi RNA tramite la sintesi organica standard e introdotti nei corrispondenti oligonucleotidi attraverso i loro rispettivi fosforamiditi. Questo rapporto si avvale di fosforammidite chimica attraverso i quattro basi azotate canoniche (uridina (U), citosina (C), guanosina (G), l'adenosina (A)), nonché 2-thiophenylmethyl nucleotidi modificati funzionalizzati al 2'- O - posizione; Tuttavia, la metodologia è amenable per un grande varLe modifiche che sono state sviluppate nel corso degli anni. Gli oligonucleotidi sono stati sintetizzati su un supporto a vetrino controllato (CPG) seguito da scissione dalla resina e deprotezione in condizioni standard, vale a dire una miscela di ammoniaca e metilammina (AMA) seguita da idrogeno fluoruro / trietilammina / N-metilpirrolidinone. I corrispondenti oligonucleotidi sono stati purificati mediante elettroforesi in poliacrilammide (denaturazione del 20%) seguita da eluizione, disidratazione e isolamento mediante cromatografia a fase inversa (Sep-pak, C 18- colonna). Quantificazione e parametri strutturali sono stati valutati, rispettivamente, mediante l'analisi fotometrica a raggi ultravioletti (UV-vis) e circolare dichroismo (CD). Questo rapporto mira a servire come risorsa e guida per i principianti e per i ricercatori esperti interessati ad intraprendere questo settore. Si prevede di essere un lavoro in corso, in quanto si sviluppano nuove tecnologie e metodologie. La descrizione delle metodologie e delle tecniche in essoS corrispondono a un DNA / RNA sintetizzatore (rinnovato e acquistato nel 2013) che utilizza la chimica di fosforamidite.
Introduzione
La sintesi in fase solida per ottenere gli oligonucleotidi del DNA / RNA è un potente strumento che ha servito parecchie applicazioni in vari campi sin dagli anni '70 1 , 2 , 3 usando i blocchi di costruzione di fosforamidite 4 . Esempi della sua vasta influenza includono: il suo impatto sull'etichettatura ( tramite reazioni di chimica clicca) 5 , la sonda strutturale 6 e le tecnologie antisenso 7 , nonché la sua spiegazione dei meccanismi biologici 8 , 9 , fonte di materiale genetico 10 e lo studio di Varie modificazioni naturali e / o chimiche 11 , 12 , tra molti altri. La modifica che usiamo qui rappresenta il primo passo nei nostri sforzi per ottenere oligonucleotidi RNA che contengonoSonde fotoattive per consentire il controllo temporale della struttura e della funzione di questo importante biopolimero.
La sintesi di RNA con sequenze dodecameri: 5 '- [CUA CG G A AU CAU] -3' / 5 '- [agosto AUU CCG UAG] -3' (posizioni sottolineate rappresentano l'incorporazione di una modifica -thiophenylmethyl C2'- O ) Costituisce l'oggetto di questo studio. Le sequenze sono state scelte per consentire la quantificazione e la misurazione dei fili RNA come singoli fili, o come corrispondenti strutture duplex (nessuna altra struttura secondaria è stata predetta come stabile termodinamica). CD è stato utilizzato per stabilire i parametri strutturali, cioè la formazione di duplex e le transizioni di denaturazione termica.
Sintesi
La procedura complessiva per ottenere questi oligonucleotidi è illustrata nella figura 1 e segue il processo stepwise: Sintesi solida automatica → DeprOzione → purificazione → quantificazione → caratterizzazione. La Figura 2 mostra le unità monomeriche necessarie in questa procedura. La sintesi in fase solida di RNA è simile a quella del DNA che si basa su fosforammidito chimica (Figura 2, sinistra) e l'uso di gruppi protettivi di base-labile per le ammine esociclici nucleofile in G, A e C, ad esempio, Acetil, benzoile, fenossiacetil, t- butile o N , N- dimetilformamide ( figura 2 , a destra). Un altro aspetto da considerare in RNA, dovuto alla presenza del gruppo C2'-OH (che non presenta i biopolimeri deoxyoligonucleotidi), è la fase supplementare che deve essere incorporata per la protezione e la successiva deprotezione di questa posizione nucleofila. A questo proposito, i gruppi di protezione a base di silicio sono diventati una strategia attraente per il loro potenziale come parti biortogonali (specificamenteSi è deprotected in presenza di fluoruro), con gruppi di terz- butilidimetilsilil (TBDMS) e triisopropilsililossimetil (TOM) come scelte popolari ( Figura 2 , in basso a sinistra).
In questo lavoro, la sintesi automatizzata è stata effettuata su un sintetizzatore DNA / RNA che utilizza la chimica standard di fosforamidite. Le impostazioni del produttore sullo strumento includono una fase di diluizione automatica quando si utilizzano le versioni commerciali dei fosforamiditi per il DNA o l'opzione di diluire a volumi impostati dall'utente. Tuttavia, abbiamo deciso di pesare l'fosforamidite di RNA e diluire manualmente in quanto: 1) il prezzo delle fosforamiditi canoniche dell'RNA è maggiore (in alcuni casi fino a 50 volte più costoso); 2) i fosforamiditi modificati vengono spesso ottenuti in piccole quantità; E 3) la quantità di materiale sprecato dopo l'utilizzo di una fase di diluizione automatica (impostata dal produttore) è grande. Inoltre, abbiamo usato: 1) supporti solidi disponibili in commercio(Ad esempio , CPG) contenente una nucleobase protetta per funzionare come 3'-end; E 2) fosforamiditi commerciali (nucleobasi canonici) protetti con un gruppo TBDMS alla posizione C2'- O . L'elenco dettagliato delle fasi di sintesi è fornito in Figura 3 e Tabella 1 , insieme ad ulteriori descrizioni e commenti per i passi che sono stati regolati per la sintesi di RNA. Inoltre, la Figura 4 illustra le rese graduali osservate per ogni passo dopo aver selezionato l'opzione 'Trityl Monitor', che quantifica il tritio cation rilasciato da ogni fase di detritilazione.
Vale la pena notare che tipicamente, nella nostra esperienza, il fattore limitante stava ottenendo il fosforamidite contenente la modifica desiderata. Cioè lo sviluppo di una metodologia sintetica che consente l'inserimento di modifiche in siti selezionati. In questa relazione, ci concentriamo sulIncorporazione di un nucleotide modificato per il quale abbiamo stabilito la corrispondente metodologia sintetica, il gruppo C2'- O- tiofenilmetile. Questo gruppo è di taglia piccola e non influenza la sintesi a fase solida in alcun modo. Dal momento che è stata riportata l'incorporazione di questo gruppo in oligonucleotidi di RNA, insieme ai parametri strutturali e termodinamici 4 , non saranno descritti aspetti della sintesi organica che conduce alle modificate fosforamidite.
Deprotezione, depurazione e caratterizzazione
La deprotezione delle amine esociclici e dei gruppi ß-cianoetil si verifica nello stesso passo di quello della scissione della resina CPG. Abbiamo applicato le condizioni comunemente utilizzate per riscaldare la resina ottenuta in presenza di una soluzione acquosa di AMA, seguita dalla scissione dei gruppi C2'- O- sililici in presenza di ioni di fluoruro e quindi di purificazione mediante gelelettroforesi. Mentre questi sono diventati condizioni standard in molti casi, modifiche che sono labili a condizioni di base o ioni fluoruro può richiedere condizioni più blande 13, 14, ad esempio, carbonato di metanolo / potassio (MeOH / K 2 CO 3), o butilammina. Pertanto, è necessario un diverso insieme di gruppi protettivi sui corrispondenti fosforamiditi. Inoltre, abbiamo scelto l'elettroforesi come alternativa preferita per purificare gli oligomeri deprotected dato la nostra precedente esperienza con questo metodo e la mancanza di altre strumentazioni. Tuttavia, la HPLC può essere utilizzata alternativamente come un metodo efficace 15 . La caratterizzazione degli oligonucleotidi purificati è stata effettuata mediante spettrometria di massa, desorbimento / ionizzazione-tempo di volo (MALDI-TOF) assistita dalla matrice, utilizzando una procedura riportata dal nostro gruppo 16 .
Caratterizzazione strutturale e ther La stabilità stabile dei duplex ottenuti è stata effettuata tramite CD. In particolare, facciamo uso di CD per determinare le transizioni di denaturazione termica di oligonucleotidi modificati e non modificati di RNA seguendo la diminuzione dell'elettricità della banda a ca. 270 nm, come pure la scomparsa della banda (con ellitticità negativa) con un λ max a 210 nm. Un confronto di spettri prima e dopo l'ibridazione viene fornito per illustrare le loro differenze e fornire la convalida della metodologia impiegata. L'uso del CD è ampiamente accettato nella determinazione di motivi strutturali negli acidi nucleici e negli aminoacidi 17 e può quindi essere impiegato come strumento per determinare diversi parametri strutturali e termodinamici 18 ; Tuttavia, non esistono molti esempi in cui la tecnica viene utilizzata per valutare le transizioni di denaturazione termica. Alcuni casi includono la determinazione di stabilità termica su DNA contenente G-quadruplexesAss = "xref"> 19 , 20 o in duplex e hairpins di RNA 21 .
Questo rapporto intende fornire al lettore o al visualizzatore non esperti un insieme di strumenti che consentono un inizio regolare di questo tipo di ricerca. Sarà utile migliorare e confrontare con metodologie e tecniche in altri laboratori di ricerca che sono coinvolti in questo eccitante ramo della scienza. Il contenuto di questa relazione aggiunge i protocolli esistenti di questa tecnologia da varie fonti e arricchisce e facilita l'esperienza con un aiuto visivo per ogni passaggio.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Sintesi di fase solida di oligonucleotidi RNA
- Preparazione di soluzioni contenenti ciascuna fosforamidite (Tabella 1).
- Conte il numero di nucleotidi e rientrare nell'equazione n + 1 (dove n = numero di nucleotidi) corrispondenti a ciascuna base e riempire la tabella con i valori mancanti. Volume = 0,15 mL per nucleotide ad una concentrazione di 0,1 M, disciolto in acetonitrile anidro.
- Pesare ciascun fosforamidite in bottiglie ambrate da 10 ml essiccate a forno (Septum Top Amber 394 Amidite 13 mm ID x 20 mm OD Top) e posizionare immediatamente sotto vuoto usando un esiccatore contenente un agente essiccante.
- Riempire l'esiccatore con argon secco, rimuovere la bottiglia e diluire immediatamente con la corrispondente quantità di acetonitrile (anidra) prima di fissare lo strumento.
NOTA: Assicurarsi di utilizzare siringhe a tenuta stagna e mantenere sempre un'atmosfera anidra. - Rimuovere la septa dalla bottiglia e mettere sul mAchine, tramite una funzione di cambio di bottiglia.
NOTA: Questo processo richiede ulteriori 0.15 mL (quindi il volume in eccesso nell'equazione n + 1 sopra) di ciascuna soluzione per preparare la linea prima della sintesi.
- Impostazione della sintesi di sequenza e fase solida usando il software ( Figura 3 ).
- Fai clic sull'icona del software ( ad esempio , OligoNet 1.0.1) e seleziona Nome strumento> OK . Crea una nuova sintesi da File> Nuova sintesi> Ordine .
- Riempimento: Data; RunID; Selezionare strumento; Nome della sequenza; Sequenza (fine 5'-a-3 '). Sotto | ciclo, assegnare il metodo creato in precedenza ( Figura 3 ). Scegli la procedura finale, ( lascia oligonucleotide legata alla resina CPG)> manuale.
- Selezionare DMT Off> (trattamento TCA nell'ultimo passaggio per ottenere un gruppo 5'-OH alla fine della sintesi) ; Salvare File> Salva comeE fornire un nome per l'esperimento.
- Inviare il file per iniziare la sintesi Ordine> Invia ordine al sintetizzatore e selezionare colonna 1-4 . Apri la finestra del sintetizzatore e seleziona il monitor trityl | scegli la funzione | trity> controlla ogni passo .
- Ripetere i passaggi secondo necessità a seconda del numero di oligonucleotidi da sintetizzare contemporaneamente (notare che tutti gli ordini devono avere lo stesso metodo, cioè gli stessi tempi di accoppiamento e la sequenza di eventi).
- Iniziare la sintesi Sintetizzatore> Preparare per iniziare . Posizionare le colonne con il desiderato 3'-end sulle posizioni indicate nello strumento. Sullo strumento fare clic su Start> No (per la preparazione ABI).
- Una volta completata la sintesi, rimuovere la colonna dallo strumento e posizionarla in una tazza di fondo rotonda seguita dall'essiccamento a pressione ridotta per circa 0,5 h.
NOTA: Si raccomanda di verificare che un colore arancione profondo sia osservato in randOm (punti 1.2.4 - 1.2.7) per evitare errori potenziali dalla funzione di monitoraggio trityl.
- Deprotezione e purificazione di oligonucleotidi RNA.
- Aprire la colonna torsione del tappo nero e trasferire tutto (o metà, a seconda della necessità o della finalità) della resina bianca in un tubo da centrifuga da 1,6 ml. Aggiungere 0,5 mL di una metilammina (40% in acqua) / ammoniaca (40% in acqua) a 1: 1.
- Fissare il tappo della tubazione centrifuga con il parafilm e, usando un blocco termico, scaldare a 60 ° C per 1,5 ore.
NOTA: un oggetto pesante può essere posto sulla parte superiore del tubo per assicurare che la concentrazione di ammoniaca rimanga costante all'interno del tubo di reazione. - Rimuovere dal blocco termico e raffreddare lentamente fino a temperatura ambiente. Centrifugare brevemente il campione per far girare i contenuti, quindi trasferire il surnatante in un nuovo tubo di centrifuga.
- Fermare i campioni sommersando tubi in azoto liquido (o ghiaccio a secco / bagno di etanolo) e concentrarsi a secchezza mentre spinnIn una centrifuga a pressione ridotta.
- Riposizionare i solidi in 0,4 mL di una soluzione trietilammina / N-metilpirrolidinone / trietilammina-triidrofluoruro (rapporto 2: 2: 3). Scaldare a 60 ° C per 1,5 ore seguita da lento raffreddamento a temperatura ambiente.
- Aggiungere 0,04 ml di una soluzione NaOAc (3M, pH 5,5 - regolata con HCl) seguita da una miscelazione dolce con una punta di pipetta. Aggiungere etanolo (1 mL) e raffreddare a ca. -70 ° C (ghiaccio secco / bagno di etanolo) per 15 min.
- Centrifugare a 15.000 giri / min e 4 ° C per 10 min. Utilizzare una pipetta per estrarre l'aliquota e asciugare il pellet risultante sotto pressione ridotta.
- Ricalassare il solido ottenuto nel tampone di caricamento (0,2 mL, formamide di 90%, 1 mM EDTA) e mescolare fino a quando la miscela è omogenea.
- Sospendere la carica su un gel di poliacrilammide preparato in precedenza (denaturazione del 20%, dimensioni del pozzetto: 30 cm x 1 mm) 22 .
- Applicare una corrente attraverso il gel fino a quando il marcatore blu bromofenolo si trova tra 1 / 2-2 / 3Giù il gel (dimensioni del gel: ~ 21,5 cm x 30 cm).
- Togliere il gel dal supporto e trasferire il contenuto di gel dal vetro al involucro di plastica (coperto da entrambi i lati) e collocare su una lastra di cromatografia a strato sottile (TLC) ricoperta di silice (contenente colorante fluorescente a 254 nm) per visualizzare le bande Utilizzando una lampada UV (λ max = 254 nm).
- Utilizzare un marcatore per delineare la posizione in cui si trova la fascia superiore e tagliarla usando una nuova lama del rasoio. Posizionare il gel contenente l'RNA (senza involucro in plastica) in un tubo conico da 50 ml e schiacciare a piccoli pezzi utilizzando una barra di vetro.
- Sospendere i residui di gel in un NaCl aq. (2 mM ed 1 mM EDTA) e agitano la sospensione a 37 ° C per 12 ore. Centrifugare il tubo conico per 10 min.
- Desaltare usando una cartuccia C18 a inversione di fase.
- Preparare la cartuccia usando una siringa da 10 ml lavando con:
Acetonitrile (10 mL)
H 2 O (due volte, 10 mL)
5 mM NH4 Cl aq. Soluzione (3 mL)
Soluzione contenente RNA, facendo attenzione a non versare alcun residuo di gel. - Lavare con H 2 O (tre volte, 10 mL).
- Eluire dalla colonna usando un 60% aq. Soluzione metanolo (3 mL)
- Preparare la cartuccia usando una siringa da 10 ml lavando con:
- Concentrare sotto pressione ridotta e ristabilire in acqua libera da RNasi (0,3 mL)
- Preparare una soluzione diluita (10 μL) e depositare 1 μL sullo strumento UV ( ad es . Nanodrop) per misurare lo spettro UV-vis (200-450 nm).
- Utilizzare la legge di Birra Lambert per determinare la concentrazione della soluzione ottenuta:
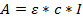
Dove A = assorbanza ottenuta; Ε = coefficiente di estinzione molare calcolato; C = concentrazione, e l = 0,1 per una lunghezza del percorso di 1 mm. - Calcolare i coefficienti di estinzione molari per gli oligonucleotidi; Calcolato qui con un calcolatore on-lineChe utilizza il software DINAMelt (http://unafold.ra.albany.edu/?q=dinamelt) 23
- Determinazione e caratterizzazione della concentrazione tramite MALDI-TOF.
- Caricare i campioni su una piastra MALDI usando una punta pipetta caricata con una punta C18 per desaltare e individuare ogni oligonucleotide.
- Lavare la punta con acetonitrile al 50% (10 μL x 2). Equilibrare la punta con l'acido trifluoroacetico 0,1% (TFA; 10 μL x 2). Caricare la punta con il campione (tipicamente 100-150 pmol).
- Lavare la punta con 0,1% TFA (10 μL x 2) e poi con acqua (10 μL x 2).
- Eluire il campione in una soluzione contenente la matrice desiderata; Abbiamo utilizzato una soluzione di: 10 μL di 25 mM-2,4,6-triidro-acetofenone monohidrato (THAP), 10 mM di ammonio citrato e 300 mM di ammonio fluoruro in acetonitrile al 50%.
- Collocare direttamente sulla piastra MALDI depositare 0,9 μL (seguito da essiccazione ad aria) e ripetere la procedura se necessario.
NOTA: Tutti gli spettri sono stati ottenuti sulla modalità positiva del riflettore.
- Caricare i campioni su una piastra MALDI usando una punta pipetta caricata con una punta C18 per desaltare e individuare ogni oligonucleotide.
2. RNA Struttura Analisi via CD
- Preparazione di soluzioni per CD.
- Preparare 0,25 mL contenenti l'RNA modificato [3 pM], NaCl [10 mM], tampone sodio fosfato [10 mM, pH 7,2], e MgCl 2 [5 mM]. Il campione è pronto per l'analisi come è, in particolare se rappresenta una transizione unimolecolare.
- Se l'obiettivo è quello di analizzare le strutture duplex o le transizioni bimolecolari, aggiungere in questo momento il complemento (1 equivalente molare, se applicabile), oppure continuare con il passo successivo.
- Posizionare il campione su un blocco termico, pre-riscaldato a 90 ° C, e spegnere il calore per controllare il raffreddamento lento a temperatura ambiente (tipicamente 2-4 h).
- Acquisizione Spectra
- Preparare una soluzione in bianco (NaCl 10 mM, fosfato di sodio 10 mM pH 7,3, MgCl 2 5 mM), trasferimento tUna micro-cuvetta (lunghezza del percorso di 1 cm, volume minimo di 250 μL) e posizionare su una posizione di supporto del cambiacampione all'interno dello strumento.
- Trasferire il campione contenente RNA in un'altra micro-cuvetta e una posizione nel cambia- re del campione. Se si misura una transizione di denaturazione termica, aggiungere con attenzione un letto di olio senza interrompere lo strato acquoso e fissare il tappo di cuvetta utilizzando un nastro di teflon.
- Aprire il serbatoio dell'azoto per fornire un flusso che sposta il flottante dell'aria situato nello strumento a ca. 40. Accendere il dispositivo di raffreddamento.
- Accendere lo strumento e aprire l'icona del software Spectra Manager , quindi aprire la finestra di acquisizione Spectra Measurement .
- Spurgare lo strumento con azoto per 5 minuti prima dell'acquisizione.
- Acquisire gli spettri utilizzando i seguenti parametri Misura> Parametri e regolare i parametri come segue:
- In Generale , selezionare: Scan 200-350 nm; Canali CD e HT: datiPitch a 0,1 nm; Velocità di scansione a 100 nm / min; Larghezza di banda a 1 nm; Numero di accumulazioni a 5.
- Sotto l'unità cellulare , scegliere 20 ° C. Sotto controllo , seleziona l'otturatore viene aperto e chiuso automaticamente. Sotto le informazioni , scegliere il nome, la concentrazione, l'operatore.
- Sotto i dati , accedere alla cartella desiderata. Fare clic su OK .
- Acquisire lo spettro Misura> Misura campione , individuare le posizioni delle cuvette nel cambia- mento del campione 1-6 , quindi fare clic su OK .
- Se prende una transizione di denaturazione termica:
- Chiudi File software di acquisizione > Chiudere e aprire un nuovo programma Misurazione della temperatura variabile> Misura> Parametri .
- Applicare i seguenti parametri per registrare una transizione termica, che può essere regolata come desiderato:
- Sotto Temperatura , selezionare temperatura di avvio: 4 ° C; Intervallo: 0,2; taR: 90 ° C; Gradiente: 1 ° C / min; Attendere 0 s. In Start / End , selezionare Avvia condizione e condizioni finali come desiderato.
- In Generale , selezionare 3 canali, CD / HT / Abs; Larghezza di banda: 1 nm; Lunghezza d'onda: 270 nm (o come desiderato). In Controllo , seleziona nessuno. In Informazioni , selezionare nome, concentrazione, operatore.
- Sotto i dati , accedere alla cartella desiderata. Fare clic su OK .
- Far raffreddare i campioni a 4 ° C e aspettare 5 min a questa temperatura prima di ottenere una misurazione dello spettro.
- Elaborazione dei dati per gli spettri.
- Utilizzando la funzione del software, sottrarre lo spettro vuoto dall'acquisizione di target per tenere conto dei segnali di sfondo derivanti dal sistema di buffer in uso: Spectra Analysis> File> Open .
- Una volta aperti un file vuoto e un file di spettri, trascinate View 2 sotto View 1 (sul lato sinistro della schermata).
- Fare clic su PrOcessing> sottrazione> OK o scambia dati> OK .
- Estrarre i dati come un file ASCII: File> Esporta e selezionare ASCII .
- Utilizzare il software per tracciare lo spettro corrispondente. Lo smorzamento dei dati è opzionale nei casi in cui il rapporto segnale / rumore è inferiore a quello previsto. Questa trasformazione può essere applicata applicando l'opzione di regolazione dei dati.
- Elaborazione dei dati per le transizioni di denaturazione termica.
- Estrarre i dati dal file JASCO come file ASCII.
- Tracciare i punti di ellitticità in funzione della temperatura. In genere, è necessario uniformare i dati a seconda della lunghezza d'onda in esame. In alcuni casi, l'utilizzo della lunghezza d'onda a 210 nm ha mostrato variazioni più grandi tra le tracce che richiedevano lo smorzamento. Tuttavia, a seconda del campione e delle concentrazioni, sono stati eseguiti diversi calcoli utilizzando i dati grezzi.
- Per calcolare la tana termicaTransizioni di atura (T m ), ottenere un primo derivato della curva. I valori massimi o minimi erano un'indicazione di questo parametro. Come nel passaggio precedente, alcuni casi hanno richiesto lo snellimento dei dati per ottenere il valore più accurato.
NOTA: Gli esperimenti sono stati tipicamente eseguiti in triplice copia, che hanno determinato la corrispondente deviazione media e standard per la misurazione.
Access restricted. Please log in or start a trial to view this content.
Risultati
Viene descritta la sintesi di dodecameri RNA contenenti zero, uno o due modificazioni 2-tiofenilmetilico alla posizione C2'- O, insieme alla sua corrispondente purificazione e caratterizzazione. Inoltre, è inclusa una descrizione dettagliata dell'analisi strutturale eseguita tramite CD.
I quattro fili di RNA (incluso uno strato con una sequenza complementare) sono stati ottenuti mediante sintesi a fase solida...
Access restricted. Please log in or start a trial to view this content.
Discussione
L'intento di questo manoscritto è quello di servire da guida ai ricercatori del campo, principianti o esperti, per raggiungere o migliorare la sintesi di oligonucleotidi di DNA o RNA. La metodologia descritta mette a fuoco sull'utilizzo di sintesi a fase solida utilizzando un sintetizzatore automatizzato DNA / RNA tramite la chimica standard di fosforamidite. Il rapporto descrive una rappresentazione passo-passo della sintesi, della purificazione e della caratterizzazione di dodecameri RNA. Inoltre, l'uso d...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno niente da rivelare.
Riconoscimenti
La preparazione di questo manoscritto è stata sostenuta attraverso fondi di start-up dell'Università del Colorado Denver (JMRE). AF desidera riconoscere il sostegno da un premio per la ricerca e la creazione di attività creative (RaCAS, CU Denver). Finanziamenti presso l'Ufficio dei servizi di ricerca, University of Colorado Denver per coprire le spese di pubblicazione è riconosciuto. Vorremmo ringraziare i membri del laboratorio, Sig.ra Cassandra Herbert e Yannick K. Dzowo per i loro contributi nella porzione video.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| AbsolveTM | PerkinElmer | 6NE9711 | |
| Acetonitrile 99.9%, HPLC Grade | Fisher Scientific | 75-05-8 | |
| Acetonitrile 99.9%, anhydrous for DNA sequencing | Fisher BioReagents | 75-05-8 | |
| Acrylamide, 99+% | ACROS Organics | 164850025 | |
| Ammonium chloride 98+% | Alfa Aesar | 12125-02-9 | |
| Ammonium citrate, dibasic 98% | Sigma Aldrich | 3012-65-5 | |
| Ammonium Fluoride 98.0%, ACS grade | Alfa Aesar | 12125-01-8 | |
| Ammonium hydroxide 28 - 30% in water, ACS Plus | Fisher Chemical | 1336-21-6 | |
| Ammonium persulfate | ACROS Organics | 1444 | |
| Argon-ultra high purity | Airgas | 7440-37-1 | |
| Bis-acrylamide Ultra pure | VWR-Amresco | 172 | |
| Boric Acid | Fisher Scientific | A73-1 | |
| Diethyl pyrocarbonate, 97% | ACROS Organics | A0368487 | |
| Ethanol, anhydrous, histological grade | Fisher Chemical | 64-17-5 | |
| Ethylenediamine tetraacetic acid, disodium salt dehydrate 100.2% | Fisher Chemical | 6381-92-6 | |
| Formamide | Thermo Scientific | 75-12-7 | |
| Hydrochloric acid, 36.5 - 38.0%, Certified ACS Plus | Fisher Chemical | 7647-01-0 | |
| Magnesium chloride hexahydrate, 99% | Fisher Scientific | 7786-30-3 | |
| Methanol, 99.9%, HPLC Grade | Fisher Chemical | 67-56-1 | |
| Methylamine 40% in water | Sigma Aldrich | 74-89-5 | |
| 1-Methyl-2-pyrrolidinone, andhydrous, 99.5% | Aldrich | 872-50-4 | |
| Opti-TOFTM 96 Well Insert (123 x 81 mm) | MDS SCIEX | 1020157 | |
| RNase Away | Molecular BioProducts | 7005-11 | |
| Sodium acetate, anhydrous 99.2%, Certified ACS | Fisher Chemical | 127-09-3 | |
| Sodium chloride, 100.5%, Certified ACS | Fisher Chemical | 7647-14-5 | |
| Sodium phosphate monobasic dihydrate 99.0% | Sigma | 13472-35-0 | |
| 2’,4’ Triethylamine, 99+% | Alfa Aesar | 121-44-8 | |
| TEMED | Amresco | 761 | |
| Triethylamine trihydrofluoride, 98% | Aldrich | 73602-61-6 | |
| Trifluoroacetic acid, 99% | Alfa Aesar | 76-05-1 | |
| 6’-Trihydroxyacetophenone monohydrate 98% | Sigma Aldrich | 480-66-0 | |
| Tris Base | Fisher Scientific | BP154-3 | |
| Urea | Fisher Scientific | U15-3 | |
| Reagents for the RNA synthesis: | |||
| Deblocking mix, 3% trichloroacetic acid in dichloromethane | Glen Research | 40-4140-57 | |
| Cap Mix A, THF/Pyridine/Acetic anhydride | Glen Research | 40-4110-52 | |
| Cap Mix B, 10% 1-methylimidazole in THF | Glen Research | 40-4120-52 | |
| Activator, 0.25 M 5-ethylthio-1H-tetrazole in anhydrous acetonitrile | Glen Research | 30-3140-52 | |
| Oxidizing Solution, 0.02 M iodine in THF/Pyridine/Water | Glen Research | 40-4330-52 | |
| U-RNA-CPG | Glen Research | 20-3330-xx | |
| Ac-G-RNA-CPG | Glen Research | 20-3324-xx | |
| Ac-G-CE Phosphoramidite | Glen Research | 10-3025-xx | |
| U-CE Phosphoramidite | Glen Research | 10-3030-xx | |
| Ac-C-CE Phosphoramidite | Glen Research | 10-3015-xx | |
| Bz-A-CE Phosphoramidite | Glen Research | 10-3003-xx |
Riferimenti
- Matteucci, M. D., Caruthers, M. H. The synthesis of oligodeoxypyrimidines on a polymer support. Tetrahedron Lett. 21 (8), 719-722 (1980).
- Beaucage, S. L., Caruthers, M. H. Deoxynucleoside phosphoramidites-A new class of key intermediates for deoxypolynucleotides. Tetrahedron Lett. 22 (20), 1859-1862 (1981).
- Caruthers, M. H. Gene synthesis machines: DNA Chemistry and its uses. Science. 230 (4723), 281-285 (1985).
- Nguyen, J. C., et al. Synthesis, Thermal Stability, Biophysical Properties, and Molecular Modeling of Oligonucleotides of RNA Containing 2'-O-2-Thiophenylmethyl Groups. J Org Chem. 81 (19), 8947-8958 (2016).
- El-Sagheer, A. H., Brown, T. Click chemistry with DNA. Chem Soc Rev. 39, 1388-1405 (2010).
- Puffer, B., et al. 5-Fluoro pyrimidines: labels to probe DNA and RNA secondary structures by 1D 19F NMR spectroscopy. Nucleic Acids Res. 37 (22), 7728-7740 (2009).
- Wan, W. B., Seth, P. P. The medicinal chemistry of therapeutic oligonucleotides. J Med Chem. 59 (21), 9645-9667 (2016).
- Zhou, C., Greenberg, M. M. DNA Damage by histone radicals in nucleosome core particles. J Am Chem Soc. 136 (18), 6562-6565 (2014).
- Zhang, Y., et al. UV-Induced proton-coupled electron transfer in cyclic DNA miniduplexes. J Am Chem Soc. 138 (23), 7395-7401 (2016).
- Anosova, I., et al. The structural diversity of artificial genetic polymers. Nucleic Acids Res. 44 (3), 1007-1021 (2016).
- Riml, C., Micura, R. Synthesis of 5-Hydroxymethylcytidine- and 5-Hydroxymethyl-uridine-Modified RNA. Synthesis. 48, 1108-1116 (2016).
- Anderson, B. A., Hrdlicka, P. J. Merging Two Strategies for Mixed-Sequence Recognition of Double-Stranded DNA: Pseudocomplementary Invader Probes. J Org Chem. 81 (8), 3335-3346 (2016).
- Beaucage, S. L., Iyer, R. P. Synthesis of oligonucleotides by the phosphoramidite approach. Tetrahedron. 48 (12), 2223-2311 (1992).
- Gillet, L. C. J., Alzeer, J., Schärer, O. D. Site-specific incorporation of N-(deoxyguanosin-8-yl)-2-acetylaminofluorene (dG-AAF) into oligonucleotides using modified 'ultra-mild' DNA synthesis. Nucleic Acids Res. 33 (6), 1961-1969 (2005).
- Shiba, Y., et al. Chemical synthesis of a very long oligoribonucleotide with 2-cyanoethoxymethyl (CEM) as the 2'-O.-protecting group: structural identification and biological activity of a synthetic 110mer precursor-microRNA candidate. Nucleic Acids Res. 35 (10), 3287-3296 (2007).
- Choi, Y. J., Gibala, K. S., Ayele, T., Deventer, K. D., Resendiz, M. J. E. Biophysical properties, thermal stability and functional impact of 8-oxo-7,8-dihydroguanine on oligonucleotides of RNA - a study of duplex, hairpins and the aptamer for preQ1 as models. Nucleic Acids Res. 45 (4), 2099-2111 (2017).
- Ranjbar, B., Gill, P. Circular dichroism techniques: Biomolecular and nanostructural analyses - A review. Chem Biol Drug Des. 74 (2), 101-120 (2009).
- Mergny, J. -L., Lacroix, L. Analysis of thermal melting curves. Oligonucleotides. 13 (6), 515-537 (2003).
- Jin, R., Breslauer, K. J., Jones, R. A., Gaffney, B. L. Tetraplex formation of a guanine-containing nonameric DNA fragment. Science. 250 (4980), 543-546 (1990).
- Virgilio, A., et al. 5-Hydroxymethyl-2'-deoxyuridine residues in the thrombin binding aptamer: Investigating anticoagulant activity by making a tiny chemical modification. CHEMBIOCHEM. 15 (16), 2427-2434 (2014).
- Chauca-Diaz, A. M., Choi, Y. J., Resendiz, M. J. E. Biophysical properties and thermal stability of oligonucleotides of RNA containing 7,8-dihydro-8-hydroxyadenosine. Biopolymers. 103 (3), 167-174 (2015).
- Maniatis, T., Fritsch, E. F., Sambrook, J. Molecular cloning: a laboratory manual. 14, Cold Spring Harbor Laboratory. Cold Spring Harbor, New York. ISBN: 0-87969-136-0 173-185 (1987).
- Markham, N. R., Zuker, M. UNAFold: software for nucleic acid folding and hybridization. Methods Mol Biol. 453, 3-31 (2008).
- Breslow, R., Huang, D. -L. Effects of metal ions including Mg2+ and lanthanides on the cleavage of ribonucleotides and RNA model compounds. Proc Natl Acad Sci U S A. 88 (10), 4080-4083 (1991).
- Blumberg, D. D. Creating a ribonuclease-free environment. Methods Enzymol. 152 (2), 20-24 (1987).
- AbouHaidar, M. G., Ivanov, I. G. Non-enzymatic RNA promoted by the combined catalytic activity of buffers and magnesium ions. Z Naturforsch C. 54 (7-8), 542-548 (1999).
- Farrell, R. E. RNA Methodologies (4th Ed): A laboratory guide for isolation and characterization. 2, Academic Press. San Diego, California. 45-80 (2010).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon