JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
2'- 포스 핀 올리고 뉴클레오타이드를 함유하는 올리고머의 고상 합성을위한 프로토콜
요약
이 기사는 C2'- O 위치에서 변형 된 RNA의 dodecamer의 고체상 합성, 정제 및 특성화에 대한 자세한 절차를 제공합니다. UV-vis 및 원 편광 이색 광도 분석을 사용하여 구조적 측면, 즉 단일 가닥 또는 이중 가닥을 정량화하고 특성을 분석합니다.
초록
고상 합성은 핵산, 특히 DNA 또는 RNA의 정식 및 변형 중합체를 수득하는데 사용되어 다양한 분야의 응용 및 상이한 연구 목적을 위해 널리 사용되는 방법론이되었다. 본원에 기술 된 절차는 C2'- O- 위치에 위치한 0 개, 1 개 또는 2 개의 변형을 함유하는 RNA 5 '- [CUA CGG AAU CAU] -3'의 도데 카머의 합성, 정제 및 특성 규명에 초점을 둔다. 프로브는 2- 티오 페닐 메틸기를 기본으로하며, 표준 유기 합성법을 통해 RNA 뉴클레오타이드에 통합되고 각각의 포스 포르 아미 다이 트를 통해 상응하는 올리고 뉴클레오타이드로 도입된다. 이 보고서는 2'- O- 아미노 말단에서 변형 된 2- 티오 페닐 메틸 작용 화 된 뉴클레오타이드뿐만 아니라 4 개의 정준 핵산 염기 (Uridine (U), Cytosine (C), Guanosine (G), Adenosine (A))를 통한 포스 포르 아미 다이 트 화학 반응을 이용한다. 위치; 그러나이 방법론은 큰 var수년에 걸쳐 개발 된 수정 사항들. 올리고 뉴클레오타이드는 표준 조건, 즉 암모니아와 메틸 아민 (AMA)과 불화 수소 / 트리 에틸 아민 / N- 메틸 피 롤리 디논의 혼합물 하에서 수지로부터 분리하고 탈 보호 한 제어 된 구멍 유리 (CPG) 지지체상에서 합성 하였다. 상응하는 올리고 뉴클레오티드는 역상 크로마토 그래피 (9 월 박, C (18) 칼 럼)을 통해 용출 탈염하고, 분리 한 다음 폴리 아크릴 아미드 전기 영동 (20 % 변성)를 통해 정제 하였다. 정량 및 구조 파라미터는 자외선 - 가시 광선 (UV-vis) 및 원 편광 이색 성 (CD) 광도 분석을 통해 각각 평가되었습니다. 이 보고서는이 분야에 착수하는 데 관심이있는 초보자 및 전문가를위한 자료 및 가이드 역할을하는 것을 목표로합니다. 새로운 기술과 방법론이 개발되면서 진행중인 작업으로 기대됩니다. 방법론과 기술에 대한 설명(phosphoramidite) 화학을 사용하는 DNA / RNA 신디사이저 (2013 년에 리퍼브되고 구매 됨)에 해당합니다.
서문
DNA / RNA의 올리고 뉴클레오타이드를 얻기위한 고상 합성은 포스 포르 아미 다이 빌딩 블록 4를 사용하는 1970 년대 1 , 2 , 3 이후 여러 분야에서 여러 분야에서 응용되고있다. 그 광범위한 영향의 예는 다음과 같습니다 : 클릭 화학 반응을 통한 라벨링에 미치는 영향 5 , 구조적 탐침 6 및 안티센스 기술 7 , 생물학적 메커니즘 8 , 9 , 유전 물질 10의 출처 및 다양한 천연 및 / 또는 화학적 변형 11 , 12를 포함 한다. 우리가 여기에서 사용하는 변형은 포함하는 RNA 올리고 뉴클레오타이드를 얻기위한 우리의 노력의 첫 번째 단계를 나타낸다.이 중요한 생체 고분자의 구조와 기능을 일시적으로 제어 할 수있는 광활성 프로브.
5 '- [CUA CG G A AU CAU] -3'/ 5 '- [AUG AUU CCG UAG] -3'(밑줄 친 위치는 C2'- O- 티오 페닐 메틸 변형의 도입을 나타낸다. )이 연구의 초점을 구성합니다. 이 서열은 RNA 가닥의 단일 가닥 또는 이들의 상응하는 이중 구조 (다른 2 차 구조는 열역학적으로 안정한 것으로 예측되지 않음)의 정량 및 측정을 가능하게하기 위해 선택되었다. CD는 구조 파라미터, 즉 이중 형성 및 열 변성 전이를 확립하는 데 사용되었습니다.
합성
이러한 올리고 뉴클레오타이드를 얻는 전반적인 절차는 그림 1 에 나와 있으며 단계별 프로세스를 따르고 있습니다 : 자동화 된 고체상 합성 → Depr→ 정제 → 정량 → 특성화. 그림 2 는이 절차에서 필요한 단량체 단위를 표시합니다. RNA의 고체상 합성 (도 2 좌측) 및 예 G, A 및 C에 친 핵성 엑소 사이 클릭 아민을위한 염기 - 불안정성 보호기의 사용이 포스 화학에 기초한다는 점에서 DNA의 것과 유사 아세틸, 벤조일, 페녹시 아세틸, t- 부틸 또는 N , N- 디메틸 포름 아미드 ( 도 2 , 오른쪽). C2'-OH 그룹 (데 옥시 올리고 뉴클레오타이드 바이오 폴리머에서 결핍 됨)의 존재로 인해 RNA에서 고려해야 할 또 하나의 측면은이 친 핵성 위치의 보호 및 후속 탈 보호에 통합되어야하는 추가 단계이다. 이와 관련하여, 실리콘 기반의 보호 그룹은 biorthogonal moieties (특정tert- 부틸 디메틸 실릴 (TBDMS) 및 트리 이소 프로필 실릴 옥시 메틸 (TOM)기를 대중적인 선택 ( 도 2 , 좌측 하단)으로 사용하여 불소 존재 하에서 탈 보호시켰다.
이 작업에서 표준화 된 phosphoramidite 화학을 사용하는 DNA / RNA 합성기에서 자동 합성을 수행했습니다. 인스트루먼트의 제조업체 설정에는 DNA 용 인산염 버전을 사용하는 경우 자동 희석 단계가 포함되며 사용자가 설정 한 양으로 희석하는 옵션이 포함됩니다. 그러나, 우리는 RNA phosphoramidite의 중량을 측정하고 수동으로 희석하기로 결정했다. 1) 표준 RNA의 가격은 더 높다 (어떤 경우에는 50 배 이상 비싸다). 2) 개질 된 포스 포르 아미 다이 트는 종종 소량으로 얻어진다; 3) 자동화 된 희석 단계 (제조자에 의해 설정 됨)를 사용할 때 낭비되는 물질의 양이 많다. 또한, 1) 상업적으로 입수 가능한 고체 지지체3 '말단으로 기능하는 보호 된 핵 염기를 포함하는 핵산 ( 예 , CPG); 및 2) C2'- O- 위치에서 TBDMS 그룹으로 보호 된 상업적 포스 포르 아미 다이 트 (표준 핵 염기). RNA 합성을 위해 조정 된 단계에 대한 자세한 설명과 설명과 함께 합성 단계의 자세한 목록이 그림 3 과 표 1 에 나와 있습니다. 또한, 그림 4 는 각각의 트리 트리 틸화 단계에서 방출 된 트리 튬 양이온을 정량화하는 'Trityl Monitor'옵션을 선택한 후 모든 단계에서 관찰되는 단계별 수율을 보여줍니다.
전형적으로, 우리의 경험에서, 제한 요인은 원하는 변형을 함유하는 포스 포르 아미 다이 트를 얻는 것이었다는 점은 주목할 가치가있다. 즉, 선택 사이트에서 수정 사항을 통합 할 수있는 합성 방법론을 개발하는 것입니다. 이 보고서에서 우리는우리가 상응하는 합성 방법론 인 C2'- O- 티오 페닐 메틸기를 확립 한 변형 된 뉴클레오타이드의 혼입. 이 그룹은 크기가 작으며 어떤 방식 으로든 고상 합성에 영향을 미치지 않습니다. 이 그룹을 RNA의 올리고 뉴클레오타이드에 혼입시키는 것이 구조적 및 열역학적 파라미터 4 와 함께보고 되었기 때문에, 변형 된 포스 포르 아미 다이 트를 유도하는 유기 합성의 어떠한 측면도 본원에서 기술되지 않을 것이다.
탈 보호, 정제 및 특성화
외래 아민 및 β- 시아 노 에틸 기의 탈 보호는 CPG- 수지로부터의 절단과 동일한 단계에서 일어난다. 우리는 AMA의 수용액의 존재 하에서 얻어진 수지를 가열하고, 플루오 라이드 이온의 존재하에 C2'- O- 실릴기를 절단하고,이어서 겔을 통해 정제하는 통상적으로 사용되는 조건을 적용 하였다전기 영동. 이들이 많은 경우에 표준 조건이되었지만, 염기성 조건이나 불소 이온에 불안정한 변형은 메탄올 / 탄산 칼륨 (MeOH / K 2 CO 3 ) 또는 부틸 아민과 같은 보다 온화한 조건 13 , 14 가 필요할 수 있습니다. 따라서, 대응하는 포스 포르 아미 다이 트상의 상이한 세트의 보호기가 필요하다. 또한, 우리는이 방법으로 이전의 경험과 다른 계기의 부족을 감안할 때 탈 보호 된 올리고머를 정제하는 데 바람직한 대안으로 전기 영동을 선택했습니다. 그러나 HPLC를 효과적인 방법으로 사용할 수도 있습니다 15 . 정제 된 올리고 뉴클레오티드의 특성 우리 그룹 (16)에 의해보고 된 절차를 사용하여, 질량 분석법, 비행의 매트릭스 보조 레이저 탈착 / 이온화 시간 (MALDI-TOF)을 통하여 수행 하였다.
구조적 특성 분석 및 얻어진 이중 가닥의 안정성은 CD 를 통해 수행되었다. 구체적으로, 우리는 CD를 이용하여, RNA의 변형 된 및 변형되지 않은 올리고 뉴클레오티드의 열 변성 전이를 결정하는데, 이는 ca에서 밴드의 타원율의 감소를 따른다. 270 nm, 그리고 210 nm에서 λ max 의 밴드 (음의 타원율)의 소멸. 하이브리드 화 전후의 스펙트럼 비교는 그들의 차이점을 설명하고 사용 된 방법론의 타당성을 입증하기 위해 제공됩니다. CD의 사용은 널리 핵산 구조적 모티프의 결정으로 인정하고 17 아미노산, 따라서 다양한 구조 및 열역학적 파라미터 (18)를 결정하는 도구로 이용 될 수있다; 그러나이 기술이 열 변성 전이를 평가하는 데 사용되는 예는 많지 않습니다. 일부 경우에는 G-quadruplexes를 포함하는 DNA에 대한 열 안정성 결정이 포함됩니다19 , 20 또는 RNA의 이중 가닥과 머리핀 21 .
이 보고서는 전문가가 아닌 독자 또는 시청자에게 이러한 유형의 조사를 원활하게 시작할 수있는 도구 세트를 제공 할 계획입니다. 이 흥미로운 과학 분야와 관련된 다른 연구소의 방법론 및 기술을 향상시키고 비교하는 데 도움이 될 것입니다. 이 보고서의 내용은 다양한 소스에서이 기술의 기존 프로토콜을 추가하고 각 단계에 대한 시각적 지원을 강화하고 경험을 촉진합니다.
프로토콜
1. RNA Oligonucleotides의 고상 합성
- 각 포스 포르 아미 다이 트를 함유 한 용액의 제조 (표 1).
- 뉴클레오타이드의 수를 세고 각 염기에 해당하는 n + 1 방정식 (n = 뉴클레오타이드의 수)에 맞추고 표에 누락 된 값을 기입하십시오. 용적 = 0.1 M 농도의 뉴클레오타이드 당 0.15 mL, 무수 아세토 니트릴에 용해.
- 오븐 건조한 10 mL 앰버 병 (Septum Top Amber 394 Amidite 13 mm ID x 20 mm OD Top)에 각 phosphoramidite를 칭량하고 건조제가 들어있는 데시 케이 터를 사용하여 진공 상태로 즉시 놓습니다.
- 건조한 아르곤으로 데시 케이 터를 채우고, 병을 꺼낸 다음, 해당 아세토 니트릴 (무수)의 양으로 즉시 희석하여 기기에 고정시킵니다.
참고 : 항상 기밀 식 주사기를 사용하고 무수 대기를 유지하십시오. - 병에서 셉터를 제거하고 m 위에 놓습니다.achine, 병 변경 기능을 통해.
참고 :이 과정은 합성하기 전에 라인을 프라임하기 위해 각 솔루션의 추가 0.15 mL (따라서 위의 n + 1 공식에서 초과 부피)가 필요합니다.
- 소프트웨어를 사용한 시퀀스 및 고체상 합성의 설정 ( 그림 3 ).
- 소프트웨어 아이콘 ( 예 : OligoNet 1.0.1)을 클릭하고 기기 이름> 확인을 선택하십시오. 파일> 새 합성> 순서 로 새 합성을 만듭니다.
- 입력 : 날짜; RunID; 악기 선택; 시퀀스 이름; 서열 (5 '에서 3'말단). 순환에서 이전에 작성된 메소드를 지정하십시오 ( 그림 3 ). 끝 절차 ( CPG 수지에 결합 된 올리고 뉴클레오타이드 잎)> 수동을 선택하십시오.
- DMT Off>를 선택하십시오 (합성 마지막 단계에서 5'-OH 그룹을 얻는 마지막 단계에서 TCA 처리) . 파일 저장> 다른 이름으로 저장실험의 이름을 제공하십시오.
- 파일을 보내어 합성을 시작하십시오. 순서> 신디사이저에 순서를 보내고 열을 선택하십시오. 1-4 . 신시사이저 창을 열고 trityl 모니터를 선택하십시오 | 기능 | trity | 모든 단계를 선택하십시오 .
- 한 번에 합성 할 올리고 뉴클레오타이드의 수에 따라 필요에 따라 단계를 반복하십시오 (모든 주문은 동일한 방법, 즉 동일한 커플 링 시간 및 일련의 사건을 가져야 함).
- 합성 시작 합성기> 시작 준비 . 측량기에 표시된 위치에 원하는 3 '끝이있는 기둥을 놓습니다. 장비에서 시작> 아니오 (ABI 준비 용)를 클릭 하십시오 .
- 합성이 완료되면, 칼럼을 장비에서 꺼내어 둥근 바닥 플라스크에 넣고 약 0.5 시간 동안 감압 건조시킵니다.
참고 : 짙은 주황색이 랜드에서 관찰되는지 확인하는 것이 좋습니다옴 커플 링 (단계 1.2.4 - 1.2.7)을 사용하여 트리 틸 모니터 기능에서 발생할 수있는 오류를 피하십시오.
- RNA oligonucleotides의 탈 보호 및 정제.
- 검정색 뚜껑을 비틀어 컬럼을 열고 흰색 레진을 1.6 mL 원심 분리 튜브로 모두 옮깁니다 (필요에 따라 또는 목적에 따라 절반). 메틸 아민 (물에서 40 %) / 암모니아 (물에서 40 %) 0.5 mL를 1 : 1로 첨가한다.
- Parafilm으로 원심 분리 튜브 캡을 고정하고 열 블록을 사용하여 1.5 시간 동안 60 ° C로 가열합니다.
참고 : 암모니아 농도가 반응 튜브 내부에서 일정하게 유지되도록 튜브 위에 무거운 물체를 올려 놓을 수 있습니다. - 가열 블록에서 꺼내 천천히 실온으로 식힌다. 샘플을 간단히 원심 분리하여 내용물을 회전시킨 다음 상징액을 새로운 원심 분리 튜브로 옮긴다.
- 액체 질소 (또는 드라이 아이스 / 에탄올 욕조)에 튜브를 담그고 샘플을 얼려서 방치하면서 건조시킵니다감압하에 원심 분리기에 넣는다.
- 0.4 mL의 triethylamine / N- methylpyrrolidinone / triethylamine-trihydrofluoride (2 : 2 : 3 비율) 용액에 고체를 재현 탁한다. 1.5 시간 동안 60 ℃로 가열 한 후 실온으로 서서히 냉각시킨다.
- 0.04 mL의 NaOAc 용액 (3M, pH 5.5 - HCl로 조정)을 첨가 한 후 피펫 팁으로 부드럽게 섞는다. 에탄올 (1 mL)을 넣고 ca. -70 ℃ (드라이 아이스 / 에탄올 욕) 15 분.
- 15,000 rpm 및 4 ° C에서 10 분간 원심 분리하십시오. 분액을 추출하고 결과 펠렛을 감압하에 건조 시키려면 피펫을 사용하십시오.
- 수득 된 고체를 로딩 완충액 (0.2 mL, 90 % 수성 포름 아미드, 1 mM EDTA)에 재현 탁시키고, 혼합물이 균질해질 때까지 혼합한다.
- 이전에 준비한 폴리 아크릴 아마이드 젤 (20 % denaturing, 잘 치수 : 30cm x 1mm) 위에 현탁액을 적재하십시오. 22 .
- 브로 모 페놀 블루 마커가 1 / 2-2 / 3 사이에있을 때까지 겔을 통해 전류를가한다.(젤 치수 : ~ 21.5 cm x 30 cm).
- 스탠드에서 젤을 꺼내고 젤 내용물을 유리에서 플라스틱 포장 (양면으로 덮음)으로 옮기고 실리카 (254 nm의 형광 염료를 포함)로 덮인 얇은 층 크로마토 그래피 (TLC) 판 위에 올려 놓아 밴드를 시각화합니다 자외선 램프 (λ max = 254 nm)를 사용하여 측정 하였다.
- 마커를 사용하여 위쪽 밴드가있는 위치를 묘사하고 새 면도날을 사용하여 자릅니다. 50 ML 원추형 튜브에 RNA가 들어있는 젤 (플라스틱 포장 없음)을 놓고 유리 막대를 사용하여 작은 조각으로 밟아.
- 겔 잔류 물을 NaCl aq에 현탁시킨다. 용액 (2 mM 및 1 mM EDTA)에 현탁시키고 37 ℃에서 12 시간 동안 현탁액을 흔든다. 10 분 동안 원추형 튜브를 원심 분리하십시오.
- 역상 C18 카트리지를 사용하여 탈염하십시오.
- 다음과 같이 세척하여 10mL 주사기를 사용하여 카트리지를 준비하십시오 :
아세토 니트릴 (10 mL)
H 2 O (2 회, 10 mL) 중
5 mM NH4 Cl aq. 용액 (3 mL)
RNA가 포함 된 용액으로 겔 잔류 물을 부어서는 안됩니다. - H 2 O로 (세 번, 10 ml)로 세척 하였다.
- 60 % aq. 메탄올 용액 (3 mL)
- 다음과 같이 세척하여 10mL 주사기를 사용하여 카트리지를 준비하십시오 :
- 감압 농축하고 RNase가없는 물 (0.3 mL)에 재용 해한다.
- 묽은 용액 (10 μL)을 준비하고 자외선 vis 스펙트럼 (200-450 nm)을 측정하기 위해 UV-vis 장비 ( 예 : Nanodrop)에 1 μL를 침전시킵니다.
- Beer Lambert의 법칙을 사용하여 얻은 용액의 농도를 결정하십시오 :
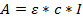
여기서 A = 얻어진 흡광도; ε = 계산 된 몰 흡광 계수; c = 농도, 1mm의 경로 길이에 대해 l = 0.1이다. - 올리고 뉴클레오타이드의 몰 흡광 계수를 계산하십시오. 온라인 계산기로 계산DINAMelt 소프트웨어 (http://unafold.ra.albany.edu/?q=dinamelt)를 사용하는 경우 23
- MALDI-TOF를 통한 농도 측정 및 특성 분석.
- 시료를 C18 팁으로 적재 된 피펫 팁을 사용하여 MALDI 플레이트에 올려 놓고 각 올리고 뉴클레오타이드를 관찰한다.
- 팁을 50 % 아세토 니트릴 (10 μL x 2)로 씻으십시오. 팁을 0.1 % 트리 플루오로 아세트산 (TFA; 10 μL × 2)로 평형화한다. tip을로드하십시오 (일반적으로 100-150 pmol).
- 팁을 0.1 % TFA (10 μL x 2)로 씻은 다음 물 (10 μL x 2)로 씻으십시오.
- 원하는 매트릭스를 포함하는 용액으로 시료를 용리십시오. 우리는 : 10 μL 25 MM - 2,4,6 - trihydroxyacetophenone 일 수화물 (THAP), 10 MM 암모늄 구연산염, 그리고 50 % 아세토 니트릴에 300 MM 암모늄 불화물의 솔루션을 사용.
- 0.9 μL을 입금하고 (공기 건조 후) 필요에 따라 절차를 반복하여 MALDI 플레이트에 직접 놓습니다.
참고 : 모든 스펙트럼은 리플렉터 포지티브 모드에서 얻습니다.
- 시료를 C18 팁으로 적재 된 피펫 팁을 사용하여 MALDI 플레이트에 올려 놓고 각 올리고 뉴클레오타이드를 관찰한다.
2. CD 를 통한 RNA 구조 분석
- CD 용 솔루션 준비.
- 수정 된 RNA [3 μM], NaCl [10 mM], 인산 나트륨 완충액 [10 mM, pH 7.2] 및 MgCl 2 [5 mM]을 함유하는 0.25 mL를 준비한다. 이 샘플은 그대로 분석을위한 준비가되어 있습니다. 특히 그것이 단 분자 변환을 나타내는 경우.
- 이중 구조 또는이 분자 전이를 분석하는 것이 목표 인 경우,이 시점에서 보체 (해당되는 경우 1 몰 당량)를 추가하거나 아래 단계를 계속 수행하십시오.
- 샘플을 90 ° C로 예열 한 열 블록에 올려 놓고 열을 꺼서 실내 온도 (일반적으로 2 ~ 4 시간)로 서서히 식히십시오.
- 스펙트럼 획득
- 빈 용액 (NaCl 10mM, 인산 나트륨 10mM, pH 7.3, MgCl 2 5mM)을 옮기고o 마이크로 큐벳 (경로 길이 1cm, 최소 용량 250 μL)을 채우고 샘플 체인저 홀더 위치에 놓습니다.
- RNA가 들어있는 시료를 다른 마이크로 큐벳으로 옮기고 시료 교환기에 넣습니다. 열 변성 전이를 측정 할 때는 수성 층을 파괴하지 않고 조심스럽게 오일 베드를 넣고 테프론 테이프를 사용하여 큐벳의 캡을 고정시킵니다.
- 질소 탱크를 열어 계기에 위치한 공기 플로터를 ca.로 이동시키는 흐름을 제공하십시오. 40. 냉각기를 켜십시오.
- 기기를 켜고 소프트웨어 Spectra Manager 아이콘을 연 다음 획득 윈도우 Spectra Measurement 를 엽니 다.
- 획득하기 전에 장비를 5 분 동안 질소로 퍼지하십시오.
- Measure> Parameters를 사용하여 스펙트럼을 획득하고 다음과 같이 매개 변수 를 조정하십시오 :
- 일반 아래에서 다음을 선택하십시오. 스캔 200-350 nm; 채널 CD 및 HT : 데이터피치 0.1nm; 100nm / 분에서 스캐닝 속도; 1nm에서의 밴드 폭; 5의 누적 횟수.
- 세포 단위 아래에서 20 ° C를 선택하십시오. 제어 하에 셔터가 자동으로 열리고 닫힙니다를 선택하십시오. 정보 아래에서 이름, 농도, 운영자를 선택하십시오.
- 데이터 아래에서 원하는 폴더를 찾습니다. 확인을 클릭하십시오.
- 측정> 샘플 측정 을 통해 샘플 체인저 1-6 에서 큐벳의 위치를 확인한 후 OK를 클릭하십시오.
- 열 변성 전이를 취하는 경우 :
- 획득 소프트웨어 닫기 파일> 종료 및 새 프로그램 가변 온도 측정> 측정> 매개 변수를 엽니 다.
- 원하는대로 조정할 수있는 열 전환을 기록하려면 다음 매개 변수를 적용하십시오.
- 온도 에서 시작 온도 를 선택하십시오 : 4 ° C; 간격 : 0.2; 고마워rget : 90 ℃; 기울기 : 1 ℃ / 분; 0 초 기다린다. Start / End 에서 Start condition & end condition을 원하는대로 선택하십시오.
- General (일반 ) 아래에서 3 Channels, CD / HT / Abs를 선택합니다. 대역폭 : 1 nm; 파장 : 270 nm (또는 원하는대로). 제어 에서 없음을 선택하십시오. 정보 에서 이름, 농도, 운영자를 선택하십시오.
- 데이터 아래에서 원하는 폴더를 찾습니다. 확인을 클릭하십시오.
- 샘플을 4 ° C로 냉각시키고 스펙트럼 측정을 얻기 전에이 온도에서 5 분 동안 기다리십시오.
- 스펙트럼에 대한 데이터 작업.
- 소프트웨어의 기능을 사용하여 대상 획득에서 공백 스펙트럼을 뺀 다음 사용중인 버퍼 시스템에서 발생하는 백그라운드 신호를 고려하십시오 : Spectra Analysis> File> Open .
- 빈 파일과 스펙트럼 파일이 열리면 뷰 1 (화면의 왼쪽)에서보기 2를 드래그하십시오.
- Pr을 클릭하십시오.ocessing> 빼기> 확인 또는 데이터 교환> 확인 .
- ASCII 파일로 데이터 추출 : 파일> 내보내기 를 선택하고 ASCII를 선택하십시오.
- 소프트웨어를 사용하여 해당 스펙트럼을 그립니다. 신호 대 잡음비가 예상보다 낮을 경우 데이터 평활화는 선택 사항입니다. 이 변환은 평활화 데이터 옵션을 적용하여 적용 할 수 있습니다.
- 열 변성 전이에 대한 데이터 작업.
- JASCO 파일의 데이터를 ASCII 파일로 추출합니다.
- 타원율 점을 온도의 함수로 플롯합니다. 일반적으로 조사되는 파장에 따라 데이터의 평활화가 필요합니다. 어떤 경우에는 210 nm에서 파장을 사용하면 평탄화가 필요한 플롯간에 큰 차이가 나타났습니다. 그러나 샘플 및 농도에 따라 여러 가지 계산이 원시 데이터를 사용하여 수행되었습니다.
- 열 덴트를 계산하려면aturation transition (T m ) 값은 곡선의 1 차 미분을 얻습니다. 최대 또는 최소값은이 매개 변수를 나타냅니다. 이전 단계 에서처럼 가장 정확한 값을 얻으려면 데이터를 부드럽게 처리해야하는 경우가있었습니다.
참고 : 실험은 일반적으로 3 회 수행되어 측정에 해당하는 평균 및 표준 편차가 발생했습니다.
결과
C2'- O- 위치에서 0, 1 또는 2 개의 2- 티오 페닐 메틸 변형을 함유하는 RNA 도데 카머의 합성은 상응하는 정제 및 특성화와 함께 기술된다. 또한 CD를 통해 수행 된 구조 분석에 대한 자세한 설명이 포함되어 있습니다.
RNA의 4 가닥 (상보적인 서열을 갖는 가닥 포함)은 고상 합성을 통해 얻어졌으며,이어서 각 올리고 뉴클?...
토론
이 원고의 취지는 DNA 또는 RNA의 올리고 뉴클레오타이드의 합성을 성공적으로 달성하거나 향상시키기위한 분야의 연구자, 초보자 또는 전문가에게 가이드 역할을하는 것입니다. 설명 된 방법론은 표준 phosphoramidite 화학을 통해 자동화 된 DNA / RNA 합성기를 사용하는 고상 합성의 사용에 중점을 둡니다. 보고서는 RNA dodecamer의 합성, 정제 및 특성 분석을 단계별로 설명합니다. 또한, CD의 사용은 2 차 ?...
공개
저자는 공개 할 것이 없습니다.
감사의 말
이 원고의 작성은 Colorado University Denver (JMRE)의 창업 자금을 통해 지원되었습니다. AF는 연구 및 창조 활동 상 (RaCAS, CU Denver)에서 지원을 인정하고자합니다. 콜로라도 주립 대학 덴버 연구 서비스 사무소 (University of Colorado Denver)에서 기부금을 충당하기 위해 자금을 지원합니다. 비디오 부분에 공헌 한 Cassandra Herbert 선생님과 Yannick K. Dzowo 선생님 께 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| AbsolveTM | PerkinElmer | 6NE9711 | |
| Acetonitrile 99.9%, HPLC Grade | Fisher Scientific | 75-05-8 | |
| Acetonitrile 99.9%, anhydrous for DNA sequencing | Fisher BioReagents | 75-05-8 | |
| Acrylamide, 99+% | ACROS Organics | 164850025 | |
| Ammonium chloride 98+% | Alfa Aesar | 12125-02-9 | |
| Ammonium citrate, dibasic 98% | Sigma Aldrich | 3012-65-5 | |
| Ammonium Fluoride 98.0%, ACS grade | Alfa Aesar | 12125-01-8 | |
| Ammonium hydroxide 28 - 30% in water, ACS Plus | Fisher Chemical | 1336-21-6 | |
| Ammonium persulfate | ACROS Organics | 1444 | |
| Argon-ultra high purity | Airgas | 7440-37-1 | |
| Bis-acrylamide Ultra pure | VWR-Amresco | 172 | |
| Boric Acid | Fisher Scientific | A73-1 | |
| Diethyl pyrocarbonate, 97% | ACROS Organics | A0368487 | |
| Ethanol, anhydrous, histological grade | Fisher Chemical | 64-17-5 | |
| Ethylenediamine tetraacetic acid, disodium salt dehydrate 100.2% | Fisher Chemical | 6381-92-6 | |
| Formamide | Thermo Scientific | 75-12-7 | |
| Hydrochloric acid, 36.5 - 38.0%, Certified ACS Plus | Fisher Chemical | 7647-01-0 | |
| Magnesium chloride hexahydrate, 99% | Fisher Scientific | 7786-30-3 | |
| Methanol, 99.9%, HPLC Grade | Fisher Chemical | 67-56-1 | |
| Methylamine 40% in water | Sigma Aldrich | 74-89-5 | |
| 1-Methyl-2-pyrrolidinone, andhydrous, 99.5% | Aldrich | 872-50-4 | |
| Opti-TOFTM 96 Well Insert (123 x 81 mm) | MDS SCIEX | 1020157 | |
| RNase Away | Molecular BioProducts | 7005-11 | |
| Sodium acetate, anhydrous 99.2%, Certified ACS | Fisher Chemical | 127-09-3 | |
| Sodium chloride, 100.5%, Certified ACS | Fisher Chemical | 7647-14-5 | |
| Sodium phosphate monobasic dihydrate 99.0% | Sigma | 13472-35-0 | |
| 2’,4’ Triethylamine, 99+% | Alfa Aesar | 121-44-8 | |
| TEMED | Amresco | 761 | |
| Triethylamine trihydrofluoride, 98% | Aldrich | 73602-61-6 | |
| Trifluoroacetic acid, 99% | Alfa Aesar | 76-05-1 | |
| 6’-Trihydroxyacetophenone monohydrate 98% | Sigma Aldrich | 480-66-0 | |
| Tris Base | Fisher Scientific | BP154-3 | |
| Urea | Fisher Scientific | U15-3 | |
| Reagents for the RNA synthesis: | |||
| Deblocking mix, 3% trichloroacetic acid in dichloromethane | Glen Research | 40-4140-57 | |
| Cap Mix A, THF/Pyridine/Acetic anhydride | Glen Research | 40-4110-52 | |
| Cap Mix B, 10% 1-methylimidazole in THF | Glen Research | 40-4120-52 | |
| Activator, 0.25 M 5-ethylthio-1H-tetrazole in anhydrous acetonitrile | Glen Research | 30-3140-52 | |
| Oxidizing Solution, 0.02 M iodine in THF/Pyridine/Water | Glen Research | 40-4330-52 | |
| U-RNA-CPG | Glen Research | 20-3330-xx | |
| Ac-G-RNA-CPG | Glen Research | 20-3324-xx | |
| Ac-G-CE Phosphoramidite | Glen Research | 10-3025-xx | |
| U-CE Phosphoramidite | Glen Research | 10-3030-xx | |
| Ac-C-CE Phosphoramidite | Glen Research | 10-3015-xx | |
| Bz-A-CE Phosphoramidite | Glen Research | 10-3003-xx |
참고문헌
- Matteucci, M. D., Caruthers, M. H. The synthesis of oligodeoxypyrimidines on a polymer support. Tetrahedron Lett. 21 (8), 719-722 (1980).
- Beaucage, S. L., Caruthers, M. H. Deoxynucleoside phosphoramidites-A new class of key intermediates for deoxypolynucleotides. Tetrahedron Lett. 22 (20), 1859-1862 (1981).
- Caruthers, M. H. Gene synthesis machines: DNA Chemistry and its uses. Science. 230 (4723), 281-285 (1985).
- Nguyen, J. C., et al. Synthesis, Thermal Stability, Biophysical Properties, and Molecular Modeling of Oligonucleotides of RNA Containing 2'-O-2-Thiophenylmethyl Groups. J Org Chem. 81 (19), 8947-8958 (2016).
- El-Sagheer, A. H., Brown, T. Click chemistry with DNA. Chem Soc Rev. 39, 1388-1405 (2010).
- Puffer, B., et al. 5-Fluoro pyrimidines: labels to probe DNA and RNA secondary structures by 1D 19F NMR spectroscopy. Nucleic Acids Res. 37 (22), 7728-7740 (2009).
- Wan, W. B., Seth, P. P. The medicinal chemistry of therapeutic oligonucleotides. J Med Chem. 59 (21), 9645-9667 (2016).
- Zhou, C., Greenberg, M. M. DNA Damage by histone radicals in nucleosome core particles. J Am Chem Soc. 136 (18), 6562-6565 (2014).
- Zhang, Y., et al. UV-Induced proton-coupled electron transfer in cyclic DNA miniduplexes. J Am Chem Soc. 138 (23), 7395-7401 (2016).
- Anosova, I., et al. The structural diversity of artificial genetic polymers. Nucleic Acids Res. 44 (3), 1007-1021 (2016).
- Riml, C., Micura, R. Synthesis of 5-Hydroxymethylcytidine- and 5-Hydroxymethyl-uridine-Modified RNA. Synthesis. 48, 1108-1116 (2016).
- Anderson, B. A., Hrdlicka, P. J. Merging Two Strategies for Mixed-Sequence Recognition of Double-Stranded DNA: Pseudocomplementary Invader Probes. J Org Chem. 81 (8), 3335-3346 (2016).
- Beaucage, S. L., Iyer, R. P. Synthesis of oligonucleotides by the phosphoramidite approach. Tetrahedron. 48 (12), 2223-2311 (1992).
- Gillet, L. C. J., Alzeer, J., Schärer, O. D. Site-specific incorporation of N-(deoxyguanosin-8-yl)-2-acetylaminofluorene (dG-AAF) into oligonucleotides using modified 'ultra-mild' DNA synthesis. Nucleic Acids Res. 33 (6), 1961-1969 (2005).
- Shiba, Y., et al. Chemical synthesis of a very long oligoribonucleotide with 2-cyanoethoxymethyl (CEM) as the 2'-O.-protecting group: structural identification and biological activity of a synthetic 110mer precursor-microRNA candidate. Nucleic Acids Res. 35 (10), 3287-3296 (2007).
- Choi, Y. J., Gibala, K. S., Ayele, T., Deventer, K. D., Resendiz, M. J. E. Biophysical properties, thermal stability and functional impact of 8-oxo-7,8-dihydroguanine on oligonucleotides of RNA - a study of duplex, hairpins and the aptamer for preQ1 as models. Nucleic Acids Res. 45 (4), 2099-2111 (2017).
- Ranjbar, B., Gill, P. Circular dichroism techniques: Biomolecular and nanostructural analyses - A review. Chem Biol Drug Des. 74 (2), 101-120 (2009).
- Mergny, J. -. L., Lacroix, L. Analysis of thermal melting curves. Oligonucleotides. 13 (6), 515-537 (2003).
- Jin, R., Breslauer, K. J., Jones, R. A., Gaffney, B. L. Tetraplex formation of a guanine-containing nonameric DNA fragment. Science. 250 (4980), 543-546 (1990).
- Virgilio, A., et al. 5-Hydroxymethyl-2'-deoxyuridine residues in the thrombin binding aptamer: Investigating anticoagulant activity by making a tiny chemical modification. CHEMBIOCHEM. 15 (16), 2427-2434 (2014).
- Chauca-Diaz, A. M., Choi, Y. J., Resendiz, M. J. E. Biophysical properties and thermal stability of oligonucleotides of RNA containing 7,8-dihydro-8-hydroxyadenosine. Biopolymers. 103 (3), 167-174 (2015).
- Maniatis, T., Fritsch, E. F., Sambrook, J. . Molecular cloning: a laboratory manual. 14, 173-185 (1987).
- Markham, N. R., Zuker, M. UNAFold: software for nucleic acid folding and hybridization. Methods Mol Biol. 453, 3-31 (2008).
- Breslow, R., Huang, D. -. L. Effects of metal ions including Mg2+ and lanthanides on the cleavage of ribonucleotides and RNA model compounds. Proc Natl Acad Sci U S A. 88 (10), 4080-4083 (1991).
- Blumberg, D. D. Creating a ribonuclease-free environment. Methods Enzymol. 152 (2), 20-24 (1987).
- AbouHaidar, M. G., Ivanov, I. G. Non-enzymatic RNA promoted by the combined catalytic activity of buffers and magnesium ions. Z Naturforsch C. 54 (7-8), 542-548 (1999).
- Farrell, R. E. . RNA Methodologies (4th Ed): A laboratory guide for isolation and characterization. 2, 45-80 (2010).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유