Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Protokoll für die Festphasensynthese von Oligomeren der RNA mit einem 2'-
In diesem Artikel
Zusammenfassung
Dieser Artikel liefert ein detailliertes Verfahren zur Festphasensynthese, -reinigung und -charakterisierung von an der C2'- O- Position modifizierten Dodecamern von RNA. UV-vis- und zirkuläre Dichroismus-photometrische Analysen dienen zur Quantifizierung und Charakterisierung von Strukturaspekten, dh Einzelsträngen oder Doppelsträngen.
Zusammenfassung
Die Festphasensynthese wurde verwendet, um kanonische und modifizierte Polymere von Nukleinsäuren, insbesondere von DNA oder RNA, zu erhalten, die es zu einer populären Methodik für Anwendungen in verschiedenen Bereichen und für verschiedene Forschungszwecke gemacht hat. Das hier beschriebene Verfahren konzentriert sich auf die Synthese, Reinigung und Charakterisierung von Dodecamern von RNA 5 '- [CUA CGG AAU CAU] -3' mit null, einer oder zwei Modifikationen, die sich an der C2'- O- Position befinden. Die Sonden basieren auf 2-Thiophenylmethylgruppen, die über Standard-organische Synthese in RNA-Nukleotide eingebaut und über ihre jeweiligen Phosphoramidite in die entsprechenden Oligonukleotide eingeführt werden. Dieser Bericht nutzt die Phosphoramidit-Chemie über die vier kanonischen Nukleobasen (Uridin (U), Cytosin (C), Guanosin (G), Adenosin (A)) sowie 2-Thiophenylmethyl-funktionalisierte Nukleotide, modifiziert am 2'- O- Position; Allerdings ist die Methodik für eine große varDie im Laufe der Jahre entwickelt wurden. Die Oligonukleotide wurden auf einem kontrollierten Porenglas (CPG) -Träger, gefolgt von einer Spaltung aus dem Harz und einer Entschützung unter Standardbedingungen, dh einem Gemisch aus Ammoniak und Methylamin (AMA), gefolgt von Fluorwasserstoff / Triethylamin / N-Methylpyrrolidinon, synthetisiert. Die entsprechenden Oligonukleotide wurden über Polyacrylamid-Elektrophorese (20% Denaturierung) gereinigt, gefolgt von Elution, Entsalzung und Isolierung über Umkehrphasenchromatographie (Sep-pak, C 18- Säule). Quantifizierungs- und Strukturparameter wurden mittels Ultraviolett-sichtbare (UV-vis) bzw. zirkuläre Dichroismus (CD) photometrische Analyse beurteilt. Dieser Bericht zielt darauf ab, als Ressource und Führer für Anfänger und Experten Forscher interessiert, um in diesem Bereich zu dienen dienen. Es wird erwartet, dass es als ein work-in-progress dienen, wie neue Technologien und Methoden entwickelt werden. Die Beschreibung der Methoden und Techniken innerhalb von ThiS Dokument entspricht einem DNA / RNA Synthesizer (renoviert und im Jahr 2013 gekauft), der Phosphoramidit Chemie verwendet.
Einleitung
Die Festphasensynthese zur Gewinnung von Oligonukleotiden von DNA / RNA ist ein leistungsfähiges Werkzeug, das seit den 1970er Jahren 1 , 2 , 3 mit Hilfe von Phosphoramidit-Bausteinen 4 verschiedene Anwendungen in verschiedenen Bereichen bedient hat. Beispiele für seinen breiten Einfluss sind: ihre Auswirkungen auf die Etikettierung ( über Klick-Chemie-Reaktionen) 5 , strukturelle Sondierung 6 und Antisense-Technologien 7 sowie die Aufklärung der biologischen Mechanismen 8 , 9 , Quelle als genetisches Material 10 und die Untersuchung von Verschiedene natürliche und / oder chemische Modifikationen 11 , 12 , unter vielen anderen. Die Modifikation, die wir hier verwenden, stellt den ersten Schritt in unseren Bemühungen dar, RNA-Oligonukleotide zu erhalten, die enthaltenPhotoaktive Sonden, um die zeitliche Kontrolle der Struktur und Funktion dieses wichtigen Biopolymers zu ermöglichen.
Die Synthese von RNA-Dodekameraden mit Sequenzen: 5 '- [CUA CG G A AU CAU] -3' / 5 '- [AUG AUU CCG UAG] -3' (unterstrichene Positionen stellen den Einbau einer C2'- O- Thiophenylmethyl-Modifikation dar ) Steht im Mittelpunkt dieser Studie. Die Sequenzen wurden ausgewählt, um die Quantifizierung und Messung von RNA-Strängen als Einzelstränge oder als entsprechende Duplexstrukturen zu ermöglichen (keine anderen Sekundärstrukturen wurden als thermodynamisch stabil vorausgesagt). CD wurde verwendet, um die strukturellen Parameter, dh Duplexbildung und thermische Denaturierungsübergänge, zu etablieren.
Synthese
Das Gesamtverfahren zur Gewinnung dieser Oligonukleotide ist in Fig. 1 dargestellt und folgt dem schrittweisen Verfahren: automatisierte Festphasensynthese → DeprOtection → Reinigung → Quantifizierung → Charakterisierung. Abbildung 2 zeigt die monomeren Einheiten, die in diesem Verfahren notwendig sind. Die Festphasen - Synthese von RNA ähnlich ist zu der DNA, dass sie basiert auf dem Phosphoramidit - Chemie (Figur 2, links) und die Verwendung von basenlabilen Schutzgruppen für die nukleophile exocyclischen Amine an G, A und C, beispielsweise Acetyl, Benzoyl, Phenoxyacetyl, t- Butyl oder N , N- Dimethylformamid ( Abbildung 2 , rechts). Ein weiterer Aspekt, der in der RNA zu berücksichtigen ist, ist aufgrund des Vorhandenseins der C2'-OH-Gruppe (fehlt in den Desoxyoligonukleotid-Biopolymeren) der zusätzliche Schritt, der für den Schutz und die anschließende Entschützung dieser nukleophilen Position einbezogen werden muss. In dieser Hinsicht sind Silizium-basierte Schutzgruppen aufgrund ihres Potenzials als biorthogonale Reste zu einer attraktiven Strategie geworden (spezifischY in Gegenwart von Fluorid entschützt), wobei die tert- Butyldimethylsilyl- (TBDMS) und Triisopropylsilyloxymethyl (TOM) -Gruppen als beliebte Entscheidungen ( Abbildung 2 , unten links).
In dieser Arbeit wurde die automatisierte Synthese auf einem DNA / RNA-Synthesizer durchgeführt, der die Standardphosphoramidit-Chemie verwendet. Die Herstellereinstellungen auf dem Instrument beinhalten einen automatisierten Verdünnungsschritt bei Verwendung der kommerziellen Versionen der Phosphoramidite für DNA oder die Möglichkeit, die vom Benutzer eingestellten Volumina zu verdünnen. Allerdings haben wir beschlossen, den RNA-Phosphoramidit zu wiegen und manuell zu verdünnen, wobei: 1) der Preis der kanonischen Phosphoramidite der RNA höher ist (bis zu 50-mal teurer in einigen Fällen); 2) die modifizierten Phosphoramidite werden oft in geringen Mengen erhalten; Und 3) die Menge des verschwendeten Materials bei Verwendung eines automatisierten Verdünnungsschrittes (vom Hersteller festgelegt) ist groß. Darüber hinaus verwendeten wir: 1) handelsübliche feste Träger(Z. B. CPG), die eine geschützte Nukleobase enthält, um als das 3'-Ende zu funktionieren; Und 2) kommerzielle Phosphoramidite (kanonische Nukleobasen), die mit einer TBDMS-Gruppe an der C2'- O- Position geschützt sind. Die detaillierte Liste der Syntheseschritte ist in Abbildung 3 und Tabelle 1 zusammen mit einer weiteren Beschreibung und Kommentaren für Schritte, die für die RNA-Synthese angepasst wurden, bereitgestellt. Weiterhin veranschaulicht Fig. 4 die schrittweisen Ausbeuten, die für jeden Schritt nach Auswahl der Option "Trityl-Monitor" beobachtet werden, die das aus jedem Detritylierungsschritt freigesetzte Trityl-Kation quantifiziert.
Es ist bemerkenswert, dass typischerweise nach unserer Erfahrung der limitierende Faktor das Phosphoramidit, das die gewünschte Modifikation enthielt, erhielt. Das heißt, die Entwicklung einer synthetischen Methodik, die den Einbau von Modifikationen an ausgewählten Standorten ermöglicht. In diesem Bericht konzentrieren wir uns auf dieEinarbeitung eines modifizierten Nukleotids, für das wir die entsprechende Synthesemethode, die C2'- O- Thiophenylmethylgruppe, etabliert haben. Diese Gruppe ist klein und beeinflusst die Festphasensynthese in keiner Weise. Da der Einbau dieser Gruppe in Oligonukleotide von RNA beschrieben wurde, werden zusammen mit den strukturellen und thermodynamischen Parametern 4 keine Aspekte der organischen Synthese, die zu den modifizierten Phosphoramiditen führt, beschrieben.
Entschützung, Reinigung und Charakterisierung
Die Entschützung der exocyclischen Amine und ß-Cyanoethylgruppen erfolgt im gleichen Schritt wie die der Spaltung aus dem CPG-Harz. Wir nutzten die üblicherweise verwendeten Bedingungen des Erhitzens des erhaltenen Harzes in Gegenwart einer wäßrigen AMA-Lösung, gefolgt von einer Spaltung der C2'- O- Silylgruppen in Gegenwart von Fluoridionen und anschließender Reinigung über GelElektrophorese Während diese in vielen Fällen zu Standardbedingungen geworden sind, können Modifikationen, die gegenüber basischen Bedingungen oder Fluoridionen labil sind, mildere Bedingungen 13 , 14 , z. B. Methanol / Kaliumcarbonat (MeOH / K 2 CO 3 ) oder Butylamin erfordern. So ist ein anderer Satz von Schutzgruppen an den entsprechenden Phosphoramiditen notwendig. Darüber hinaus wählten wir die Elektrophorese als bevorzugte Alternative zur Reinigung der entschützten Oligomere aufgrund unserer bisherigen Erfahrung mit dieser Methode und dem Mangel an anderen Instrumenten. Jedoch kann HPLC alternativ als eine effektive Methode 15 verwendet werden. Die Charakterisierung der gereinigten Oligonukleotide erfolgte mittels Massenspektrometrie, matrixunterstützter Laserdesorption / Ionisationszeit des Fluges (MALDI-TOF) unter Verwendung eines gemeldeten Verfahrens durch unsere Gruppe 16 .
Strukturelle Charakterisierung und ther Die Stabilität der erhaltenen Duplexe erfolgte über CD. Speziell verwenden wir CD, um die thermischen Denaturierungsübergänge von modifizierten und unmodifizierten Oligonukleotiden von RNA zu bestimmen, indem man die Abnahme der Elliptizität des Bandes bei ca. 270 nm sowie das Verschwinden des Bandes (mit negativer Elliptizität) mit einem λ max bei 210 nm. Ein Spektralvergleich vor und nach der Hybridisierung wird zur Veranschaulichung ihrer Unterschiede und zur Validierung der eingesetzten Methodik gegeben. Die Verwendung von CD wird bei der Bestimmung von Strukturmotiven in Nukleinsäuren und Aminosäuren 17 weithin akzeptiert und kann daher als Werkzeug zur Bestimmung verschiedener struktureller und thermodynamischer Parameter verwendet werden 18 ; Allerdings gibt es nicht viele Beispiele, wo die Technik verwendet wird, um thermische Denaturierungsübergänge zu bewerten. In einigen Fällen handelt es sich um die Bestimmung von thermischen Stabilitäten auf DNA, die G-Quadruplexe enthältAss = "xref"> 19 , 20 oder in Duplexen und Haarnadeln der RNA 21 .
Dieser Bericht beabsichtigt, den nicht-fachkundigen Leser oder Betrachter mit einer Reihe von Werkzeugen zur Verfügung zu stellen, die einen reibungslosen Start in diese Art von Forschung ermöglichen. Es wird dazu dienen, mit Methoden und Techniken in anderen Forschungslaboratorien, die in diesem spannenden Zweig der Wissenschaft beteiligt sind, zu verbessern und zu vergleichen. Der Inhalt in diesem Bericht ergänzt die vorhandenen Protokolle dieser Technologie aus verschiedenen Quellen und bereichert und erleichtert die Erfahrung mit einer visuellen Hilfe für jeden Schritt.
Protokoll
1. Festphasensynthese von RNA-Oligonukleotiden
- Herstellung von Lösungen, die jeden Phosphoramidit enthalten (Tabelle 1).
- Zähle die Anzahl der Nukleotide und passt in die n + 1-Gleichung (wobei n = Anzahl der Nukleotide) jeder Base entspricht und die Tabelle mit den fehlenden Werten ausfüllen. Volumen = 0,15 ml pro Nukleotid bei einer Konzentration von 0,1 M, gelöst in wasserfreiem Acetonitril.
- Wiegen Sie jeden Phosphoramidit in ofengetrocknete 10 ml Bernsteinflaschen (Septum Top Amber 394 Amidite 13 mm ID x 20 mm OD Top) und legen Sie sofort unter Vakuum mit einem Exsikkator, der ein Trockenmittel enthält.
- Füllen Sie den Exsikkator mit trockenem Argon, entfernen Sie die Flasche und verdünnen Sie sofort mit der entsprechenden Menge Acetonitril (wasserfrei), bevor Sie auf dem Instrument sichern.
HINWEIS: Achten Sie darauf, gasdichte Spritzen zu verwenden und zu jeder Zeit eine wasserfreie Atmosphäre zu halten. - Septa aus der Flasche nehmen und auf die m legenAchine, über eine Flaschenwechselfunktion.
HINWEIS: Dieses Verfahren erfordert zusätzliche 0,15 ml (also das überschüssige Volumen in der n + 1-Gleichung oben) jeder Lösung, um die Linie vor der Synthese zu prime.
- Aufbau der Sequenz- und Festphasensynthese mit der Software ( Abbildung 3 ).
- Klicken Sie auf das Software-Symbol ( zB OligoNet 1.0.1) und wählen Sie Instrumentname> OK . Erstellen Sie eine neue Synthese nach Datei> Neue Synthese> Bestellung .
- Fill-In: Datum; RunID; Instrument auswählen; Sequenzname; Sequenz (5'-to-3'-Ende). Unter "Zyklus" die zuvor erstellte Methode zuordnen ( Abbildung 3 ). Wählen Sie das Ende Verfahren, ( verlässt Oligonukleotid an das CPG-Harz gebunden)> Handbuch.
- Wählen Sie DMT Off> (TCA-Behandlung im letzten Schritt, um eine 5'-OH-Gruppe am Ende der Synthese zu erhalten) ; Speichern Sie Datei> Speichern unterUnd geben Sie einen Namen für das Experiment.
- Senden Sie die Datei, um die Synthese zu beginnen. Bestellen> Sendung an den Synthesizer senden und die Spalte auswählen 1-4 Öffnen Sie das Synthesizer-Fenster und wählen Sie trityl monitor | select function | trity> überwachen jeden Schritt .
- Wiederholen Sie die notwendigen Schritte, je nach Anzahl der zu synthetisierenden Oligonukleotide (beachten Sie, dass alle Aufträge die gleiche Methode haben müssen, dh die gleichen Kopplungszeiten und die Reihenfolge der Ereignisse).
- Beginne die Synthese Synthesizer> Vorbereitung zu starten . Legen Sie die Säulen mit den gewünschten 3'-Enden auf die am Instrument angegebenen Positionen. Klicken Sie auf dem Instrument auf Start> Nein (für ABI Vorbereitung).
- Sobald die Synthese abgeschlossen ist, entfernen Sie die Säule aus dem Instrument und legen Sie sie in einen Rundkolben, gefolgt von einem Trocknen unter vermindertem Druck für ca. 0,5 h.
HINWEIS: Es wird empfohlen, zu überprüfen, ob eine tiefe orange Farbe in rand beobachtet wirdOm-Kupplungen (Schritte 1.2.4 - 1.2.7), um mögliche Fehler aus der Trityl-Monitor-Funktion zu vermeiden.
- Entschützung und Reinigung von RNA-Oligonukleotiden.
- Öffnen Sie die Säule, indem Sie die schwarze Kappe verdrehen und alle (oder die Hälfte je nach Bedarf oder Zweck) des weißen Harzes in ein 1,6 mL Zentrifugenröhrchen überführen. Füge 0,5 ml eines Methylamins (40% in Wasser) / Ammoniak (40% in Wasser) bei 1: 1 zu.
- Sichern Sie die Zentrifugenröhrchenkappe mit Parafilm und erwärmen Sie mit einem Wärmeblock 1,5 h auf 60 ° C.
HINWEIS: Ein schweres Objekt kann auf das Rohr gelegt werden, um sicherzustellen, dass die Ammoniakkonzentration im Reaktionsrohr konstant bleibt. - Vom Heizblock entfernen und langsam auf Raumtemperatur abkühlen lassen. Die Probe sorgfältig zentrifugieren, um den Inhalt zu spinnen, dann den Überstand in ein neues Zentrifugenröhrchen überführen.
- Einfrieren der Proben durch Eintauchen von Röhrchen in flüssigem Stickstoff (oder Trockeneis / Ethanol-Bad) und konzentrieren sich zur Trockene während SpinnIn einer Zentrifuge unter vermindertem Druck.
- Die Suspensionen in 0,4 ml einer Triethylamin / N-Methylpyrrolidinon / Triethylamin-Trihydrofluoridlösung (2: 2: 3-Verhältnis) erneut suspendieren. Auf 60 ° C für 1,5 h erhitzen, gefolgt von langsamer Abkühlung auf Raumtemperatur.
- Fügen Sie 0,04 ml einer NaOAc-Lösung (3M, pH 5,5 - eingestellt mit HCl) hinzu, gefolgt von sanftem Mischen mit einer Pipettenspitze. Ethanol (1 ml) zugeben und auf ca. -70 ° C (Trockeneis / Ethanol-Bad) für 15 min.
- Zentrifugieren bei 15.000 U / min und 4 ° C für 10 min. Verwenden Sie eine Pipette, um das Aliquot zu extrahieren und das erhaltene Pellet unter vermindertem Druck zu trocknen.
- Den erhaltenen Feststoff im Beladungspuffer (0,2 ml, 90% iges wässriges Formamid, 1 mM EDTA) erneut suspendieren und mischen, bis das Gemisch homogen ist.
- Beladung auf ein zuvor hergestelltes Polyacrylamidgel (20% Denaturierung, Wellungsmaße: 30 cm x 1 mm) 22 .
- Tragen Sie einen Strom durch das Gel, bis der Bromphenol-Blau-Marker zwischen 1 / 2-2 / 3 liegtDas Gel (Gelmaße: ~ 21,5 cm x 30 cm).
- Entfernen Sie das Gel vom Ständer und übertragen Sie den Gelgehalt vom Glas auf die Plastikfolie (beidseitig abgedeckt) und legen Sie eine Dünnschichtchromatographie (TLC) mit Siliciumdioxid (mit Fluoreszenzfarbstoff bei 254 nm) auf, um die Bänder zu visualisieren Mit einer UV-Lampe (λ max = 254 nm).
- Verwenden Sie eine Markierung, um die Position, wo das obere Band befindet, abzugrenzen und schneiden Sie es mit einem neuen Rasierklinge. Legen Sie das Gel, das die RNA (ohne Plastikfolie) enthält, in ein 50-ml-konisches Röhrchen und zerkleinert mit einem Glasstab zu kleinen Stücken.
- Suspendieren der Gelreste in eine NaCl-aq. Lösung (2 mM und 1 mM EDTA) und schüttelte die Suspension bei 37 ° C für 12 h. Das Kegelrohr für 10 min zentrifugieren.
- Mit einer Umkehrphasen-C18-Patrone entsorgen.
- Vorbereitung der Patrone mit einer 10-ml-Spritze durch Waschen mit:
Acetonitril (10 ml)
H 2 O (zweimal 10 ml)
5 mM NH4 Cl aq. Lösung (3 ml)
Lösung, die RNA enthält, wobei darauf geachtet wird, keine der Gelreste zu gießen. - Waschen mit H 2 O (dreimal, 10 mL).
- Eluieren Sie aus der Säule mit einem 60% aq. Methanollösung (3 ml)
- Vorbereitung der Patrone mit einer 10-ml-Spritze durch Waschen mit:
- Unter vermindertem Druck konzentrieren und in RNase-freiem Wasser (0,3 ml) wieder auflösen
- Eine verdünnte Lösung (10 μl) vorbereiten und 1 μl auf das UV-Vis-Instrument ( zB Nanodrop) abgeben, um das UV-Vis-Spektrum (200-450 nm) zu messen.
- Verwenden Sie das Bier-Lambert-Gesetz, um die Konzentration der erhaltenen Lösung zu bestimmen:
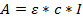
Wo A = erhaltene Extinktion; Ε = berechneter molarer Extinktionskoeffizient; C = Konzentration und l = 0,1 für eine Weglänge von 1 mm. - Berechnen Sie die molaren Extinktionskoeffizienten für die Oligonukleotide; Hier mit einem Online-Rechner berechnetDas verwendet DINAMelt Software (http://unafold.ra.albany.edu/?q=dinamelt) 23
- Konzentrationsbestimmung und Charakterisierung über MALDI-TOF.
- Legen Sie Proben auf eine MALDI-Platte mit einer Pipettenspitze, die mit einer C18-Spitze beladen ist, um zu entsalzen und jedes Oligonukleotid zu lokalisieren.
- Die Spitze mit 50% Acetonitril (10 μl x 2) waschen. Die Spitze mit 0,1% Trifluoressigsäure (TFA, 10 μl x 2) äquilibrieren. Lastspitze mit Probe (typischerweise 100-150 pmol).
- Die Spitze mit 0,1% TFA (10 μl x 2) und dann mit Wasser (10 μL x 2) waschen.
- Eluieren Sie die Probe in eine Lösung, die die gewünschte Matrix enthält; Wir verwendeten eine Lösung von: 10 μl 25 mM-2,4,6-Trihydroxyacetophenon-Monohydrat (THAP), 10 mM Ammoniumcitrat und 300 mM Ammoniumfluorid in 50% Acetonitril.
- Spot direkt auf die MALDI-Platte durch Abscheiden von 0,9 μl (gefolgt von Lufttrocknung) und Wiederholen des Verfahrens nach Bedarf.
HINWEIS: Alle Spektren wurden auf dem Reflektor-Positiv-Modus erhalten.
- Legen Sie Proben auf eine MALDI-Platte mit einer Pipettenspitze, die mit einer C18-Spitze beladen ist, um zu entsalzen und jedes Oligonukleotid zu lokalisieren.
2. RNA Strukturanalyse über CD
- Vorbereitung von Lösungen für CD.
- 0,25 ml, enthaltend die modifizierte RNA [3 μM], NaCl [10 mM], Natriumphosphatpuffer [10 mM, pH 7,2] und MgCl 2 [5 mM]. Die Probe ist bereit für die Analyse, wie es ist, besonders wenn sie einen unimolekularen Übergang darstellt.
- Wenn es darum geht, Duplexstrukturen oder bimolekulare Übergänge zu analysieren, fügen Sie zu diesem Zeitpunkt Komplement (1 Moläquivalent, falls zutreffend) hinzu oder fahren Sie mit dem folgenden Schritt fort.
- Legen Sie die Probe auf einen Hitzeblock, der auf 90 ° C vorgeheizt ist, und schalten Sie die Hitze ab, um die langsame Abkühlung auf Raumtemperatur (typischerweise 2 - 4 h) zu kontrollieren.
- Spectra-Akquisition
- Eine Blindlösung (NaCl 10 mM, Natriumphosphat 10 mM pH 7,3, MgCl 2 5 mM), Transfer tEine Mikroküvette (1 cm Pfadlänge, 250 μl Mindestvolumen) und auf eine Halteposition des Probenwechslers innerhalb des Gerätes stellen.
- Übertragen Sie die Probe, die RNA enthält, in eine andere Mikroküvette und Position in Probenwechsler. Wenn Sie einen thermischen Denaturierungsübergang messen, fügen Sie vorsichtig ein Ölbett hinzu, ohne die wässrige Schicht zu stören und die Kappe der Küvette mit einem Teflonband zu sichern.
- Öffnen Sie den Stickstofftank, um einen Durchfluss zu erzielen, der den an dem Gerät befindlichen Luftschwimmer nach ca. 40. Kühler einschalten.
- Schalten Sie das Instrument ein und öffnen Sie das Software- Spectra Manager- Symbol und öffnen Sie dann das Erfassungsfenster Spectra Measurement .
- Spülen Sie das Instrument mit Stickstoff für 5 Minuten vor dem Erwerb.
- Erfassen Sie die Spektren mit den folgenden Parametern Messen Sie> Parameter und stellen Sie die Parameter wie folgt ein:
- Wählen Sie unter Allgemein : Scan 200-350 nm; Kanäle CD und HT: DatenTonhöhe bei 0,1 nm; Abtastgeschwindigkeit bei 100 nm / min; Bandbreite bei 1 nm; Anzahl der Akkumulationen bei 5.
- Unter Zelleneinheit wählen Sie 20 ° C. Unter Kontrolle wählen Sie Shutter wird automatisch geöffnet und geschlossen. Unter Information wählen Sie den Namen, die Konzentration, den Betreiber.
- Klicken Sie unter Daten auf den gewünschten Ordner. Klicken Sie auf OK .
- Erfassen Sie die Spektren Maßnahme> Probenmessung, identifizieren Sie die Position (en) der Küvetten im Probenwechsler 1-6 entsprechend, klicken Sie auf OK .
- Bei einem thermischen Denaturierungsübergang:
- Schließen Akquisitionssoftware Datei> Beenden und Öffnen eines neuen Programms Variable Temperaturmessung> Messen> Parameter .
- Tragen Sie die folgenden Parameter auf, um einen thermischen Übergang aufzuzeichnen, der wie gewünscht eingestellt werden kann:
- Unter Temperatur wählen Sie Starttemp .: 4 ° C; Intervall: 0,2; TaRget: 90 ° C; Gradienten: 1 ° C / min; Warten 0 s. Wählen Sie unter Start / Ende die Option Startbedingung & Endbedingung wie gewünscht aus.
- Wählen Sie unter Allgemein 3 Kanäle, CD / HT / Abs; Bandbreite: 1 nm; Wellenlänge: 270 nm (oder nach Wunsch). Wählen Sie unter Steuerung keine. Unter Information wählen Sie Name, Konzentration, Operator.
- Klicken Sie unter Daten auf den gewünschten Ordner. Klicken Sie auf OK .
- Kühlen Sie die Proben auf 4 ° C und warten Sie 5 min bei dieser Temperatur, bevor Sie eine Spektralmessung erhalten.
- Datenaufarbeitung für Spektren.
- Verwenden Sie die Funktion auf der Software, subtrahieren Sie das Blindspektrum von der Zielerfassung, um Hintergrundsignale zu berücksichtigen, die sich aus dem verwendeten Puffersystem ergeben: Spectra Analysis> File> Open .
- Sobald eine leere Datei und eine Spektren-Datei geöffnet wurden, ziehen Sie Ansicht 2 unter Ansicht 1 (auf der linken Seite des Bildschirms).
- Klicken Sie auf PrOcessing> subtraktion> OK oder tausche Daten> OK .
- Extrahieren von Daten als ASCII-Datei: Datei> Exportieren und ASCII auswählen.
- Verwenden Sie Software, um das entsprechende Spektrum zu zeichnen. Das Glätten von Daten ist optional, wenn das Signal-Rausch-Verhältnis niedriger ist als erwartet. Diese Transformation kann durch Anwenden der Glättungsdatenoption angewendet werden.
- Datenaufarbeitung für thermische Denaturierungsübergänge.
- Extrahieren Sie Daten aus der JASCO-Datei als ASCII-Datei.
- Zeichnen Sie die Elliptizität Punkte als Funktion der Temperatur. Typischerweise ist eine Glättung der Daten abhängig von der zu untersuchenden Wellenlänge erforderlich. In einigen Fällen zeigte die Verwendung der Wellenlänge bei 210 nm größere Variationen zwischen den Plots, die eine Glättung erforderten. Je nach Probe und Konzentrationen wurde jedoch eine Anzahl von Berechnungen unter Verwendung der Rohdaten durchgeführt.
- Zur Berechnung der thermischen HöhleAturationsübergänge (T m ), erhalten eine erste Ableitung der Kurve. Die Maxima oder Minima waren ein Hinweis auf diesen Parameter. Wie im vorherigen Schritt, einige Fälle erforderten Glättung der Daten, um den genauesten Wert zu erhalten.
HINWEIS: Experimente wurden typischerweise dreifach durchgeführt, was zu der entsprechenden Durchschnitts- und Standardabweichung für die Messung führte.
Ergebnisse
Die Synthese von RNA-Dodekameraden, die null, ein oder zwei 2-Thiophenylmethyl-Modifikationen an der C2'- O- Position enthalten, wird zusammen mit ihrer entsprechenden Reinigung und Charakterisierung beschrieben. Weiterhin ist eine detaillierte Beschreibung der Strukturanalyse, die über CD durchgeführt wurde, enthalten.
Die vier Stränge der RNA (einschließlich eines Strangs mit einer komplementären Sequenz) w...
Diskussion
Die Absicht dieses Manuskripts dient als Leitfaden für Forscher auf dem Gebiet, Anfänger oder Experte, um die Synthese von Oligonukleotiden von DNA oder RNA erfolgreich zu erreichen oder zu verstärken. Die beschriebene Methodik konzentriert sich auf die Verwendung der Festphasensynthese unter Verwendung eines automatisierten DNA / RNA-Synthesizers über Standard-Phosphoramidit-Chemie. Der Bericht beschreibt eine schrittweise Darstellung der Synthese, Reinigung und Charakterisierung von RNA-Dodecamern. Darüber hinaus...
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Die Vorbereitung dieses Manuskripts wurde über Start-up-Fonds von der University of Colorado Denver (JMRE) unterstützt. AF möchte die Unterstützung von einem Research and Creative Activities Award (RaCAS, CU Denver) anerkennen. Die Finanzierung durch das Amt für Forschungsdienste, Universität von Colorado Denver zur Deckung der Veröffentlichungsgebühren wird anerkannt. Wir danken den Labormitgliedern Frau Cassandra Herbert und Herrn Yannick K. Dzowo für ihre Beiträge im Videobereich.
Materialien
| Name | Company | Catalog Number | Comments |
| AbsolveTM | PerkinElmer | 6NE9711 | |
| Acetonitrile 99.9%, HPLC Grade | Fisher Scientific | 75-05-8 | |
| Acetonitrile 99.9%, anhydrous for DNA sequencing | Fisher BioReagents | 75-05-8 | |
| Acrylamide, 99+% | ACROS Organics | 164850025 | |
| Ammonium chloride 98+% | Alfa Aesar | 12125-02-9 | |
| Ammonium citrate, dibasic 98% | Sigma Aldrich | 3012-65-5 | |
| Ammonium Fluoride 98.0%, ACS grade | Alfa Aesar | 12125-01-8 | |
| Ammonium hydroxide 28 - 30% in water, ACS Plus | Fisher Chemical | 1336-21-6 | |
| Ammonium persulfate | ACROS Organics | 1444 | |
| Argon-ultra high purity | Airgas | 7440-37-1 | |
| Bis-acrylamide Ultra pure | VWR-Amresco | 172 | |
| Boric Acid | Fisher Scientific | A73-1 | |
| Diethyl pyrocarbonate, 97% | ACROS Organics | A0368487 | |
| Ethanol, anhydrous, histological grade | Fisher Chemical | 64-17-5 | |
| Ethylenediamine tetraacetic acid, disodium salt dehydrate 100.2% | Fisher Chemical | 6381-92-6 | |
| Formamide | Thermo Scientific | 75-12-7 | |
| Hydrochloric acid, 36.5 - 38.0%, Certified ACS Plus | Fisher Chemical | 7647-01-0 | |
| Magnesium chloride hexahydrate, 99% | Fisher Scientific | 7786-30-3 | |
| Methanol, 99.9%, HPLC Grade | Fisher Chemical | 67-56-1 | |
| Methylamine 40% in water | Sigma Aldrich | 74-89-5 | |
| 1-Methyl-2-pyrrolidinone, andhydrous, 99.5% | Aldrich | 872-50-4 | |
| Opti-TOFTM 96 Well Insert (123 x 81 mm) | MDS SCIEX | 1020157 | |
| RNase Away | Molecular BioProducts | 7005-11 | |
| Sodium acetate, anhydrous 99.2%, Certified ACS | Fisher Chemical | 127-09-3 | |
| Sodium chloride, 100.5%, Certified ACS | Fisher Chemical | 7647-14-5 | |
| Sodium phosphate monobasic dihydrate 99.0% | Sigma | 13472-35-0 | |
| 2’,4’ Triethylamine, 99+% | Alfa Aesar | 121-44-8 | |
| TEMED | Amresco | 761 | |
| Triethylamine trihydrofluoride, 98% | Aldrich | 73602-61-6 | |
| Trifluoroacetic acid, 99% | Alfa Aesar | 76-05-1 | |
| 6’-Trihydroxyacetophenone monohydrate 98% | Sigma Aldrich | 480-66-0 | |
| Tris Base | Fisher Scientific | BP154-3 | |
| Urea | Fisher Scientific | U15-3 | |
| Reagents for the RNA synthesis: | |||
| Deblocking mix, 3% trichloroacetic acid in dichloromethane | Glen Research | 40-4140-57 | |
| Cap Mix A, THF/Pyridine/Acetic anhydride | Glen Research | 40-4110-52 | |
| Cap Mix B, 10% 1-methylimidazole in THF | Glen Research | 40-4120-52 | |
| Activator, 0.25 M 5-ethylthio-1H-tetrazole in anhydrous acetonitrile | Glen Research | 30-3140-52 | |
| Oxidizing Solution, 0.02 M iodine in THF/Pyridine/Water | Glen Research | 40-4330-52 | |
| U-RNA-CPG | Glen Research | 20-3330-xx | |
| Ac-G-RNA-CPG | Glen Research | 20-3324-xx | |
| Ac-G-CE Phosphoramidite | Glen Research | 10-3025-xx | |
| U-CE Phosphoramidite | Glen Research | 10-3030-xx | |
| Ac-C-CE Phosphoramidite | Glen Research | 10-3015-xx | |
| Bz-A-CE Phosphoramidite | Glen Research | 10-3003-xx |
Referenzen
- Matteucci, M. D., Caruthers, M. H. The synthesis of oligodeoxypyrimidines on a polymer support. Tetrahedron Lett. 21 (8), 719-722 (1980).
- Beaucage, S. L., Caruthers, M. H. Deoxynucleoside phosphoramidites-A new class of key intermediates for deoxypolynucleotides. Tetrahedron Lett. 22 (20), 1859-1862 (1981).
- Caruthers, M. H. Gene synthesis machines: DNA Chemistry and its uses. Science. 230 (4723), 281-285 (1985).
- Nguyen, J. C., et al. Synthesis, Thermal Stability, Biophysical Properties, and Molecular Modeling of Oligonucleotides of RNA Containing 2'-O-2-Thiophenylmethyl Groups. J Org Chem. 81 (19), 8947-8958 (2016).
- El-Sagheer, A. H., Brown, T. Click chemistry with DNA. Chem Soc Rev. 39, 1388-1405 (2010).
- Puffer, B., et al. 5-Fluoro pyrimidines: labels to probe DNA and RNA secondary structures by 1D 19F NMR spectroscopy. Nucleic Acids Res. 37 (22), 7728-7740 (2009).
- Wan, W. B., Seth, P. P. The medicinal chemistry of therapeutic oligonucleotides. J Med Chem. 59 (21), 9645-9667 (2016).
- Zhou, C., Greenberg, M. M. DNA Damage by histone radicals in nucleosome core particles. J Am Chem Soc. 136 (18), 6562-6565 (2014).
- Zhang, Y., et al. UV-Induced proton-coupled electron transfer in cyclic DNA miniduplexes. J Am Chem Soc. 138 (23), 7395-7401 (2016).
- Anosova, I., et al. The structural diversity of artificial genetic polymers. Nucleic Acids Res. 44 (3), 1007-1021 (2016).
- Riml, C., Micura, R. Synthesis of 5-Hydroxymethylcytidine- and 5-Hydroxymethyl-uridine-Modified RNA. Synthesis. 48, 1108-1116 (2016).
- Anderson, B. A., Hrdlicka, P. J. Merging Two Strategies for Mixed-Sequence Recognition of Double-Stranded DNA: Pseudocomplementary Invader Probes. J Org Chem. 81 (8), 3335-3346 (2016).
- Beaucage, S. L., Iyer, R. P. Synthesis of oligonucleotides by the phosphoramidite approach. Tetrahedron. 48 (12), 2223-2311 (1992).
- Gillet, L. C. J., Alzeer, J., Schärer, O. D. Site-specific incorporation of N-(deoxyguanosin-8-yl)-2-acetylaminofluorene (dG-AAF) into oligonucleotides using modified 'ultra-mild' DNA synthesis. Nucleic Acids Res. 33 (6), 1961-1969 (2005).
- Shiba, Y., et al. Chemical synthesis of a very long oligoribonucleotide with 2-cyanoethoxymethyl (CEM) as the 2'-O.-protecting group: structural identification and biological activity of a synthetic 110mer precursor-microRNA candidate. Nucleic Acids Res. 35 (10), 3287-3296 (2007).
- Choi, Y. J., Gibala, K. S., Ayele, T., Deventer, K. D., Resendiz, M. J. E. Biophysical properties, thermal stability and functional impact of 8-oxo-7,8-dihydroguanine on oligonucleotides of RNA - a study of duplex, hairpins and the aptamer for preQ1 as models. Nucleic Acids Res. 45 (4), 2099-2111 (2017).
- Ranjbar, B., Gill, P. Circular dichroism techniques: Biomolecular and nanostructural analyses - A review. Chem Biol Drug Des. 74 (2), 101-120 (2009).
- Mergny, J. -. L., Lacroix, L. Analysis of thermal melting curves. Oligonucleotides. 13 (6), 515-537 (2003).
- Jin, R., Breslauer, K. J., Jones, R. A., Gaffney, B. L. Tetraplex formation of a guanine-containing nonameric DNA fragment. Science. 250 (4980), 543-546 (1990).
- Virgilio, A., et al. 5-Hydroxymethyl-2'-deoxyuridine residues in the thrombin binding aptamer: Investigating anticoagulant activity by making a tiny chemical modification. CHEMBIOCHEM. 15 (16), 2427-2434 (2014).
- Chauca-Diaz, A. M., Choi, Y. J., Resendiz, M. J. E. Biophysical properties and thermal stability of oligonucleotides of RNA containing 7,8-dihydro-8-hydroxyadenosine. Biopolymers. 103 (3), 167-174 (2015).
- Maniatis, T., Fritsch, E. F., Sambrook, J. . Molecular cloning: a laboratory manual. 14, 173-185 (1987).
- Markham, N. R., Zuker, M. UNAFold: software for nucleic acid folding and hybridization. Methods Mol Biol. 453, 3-31 (2008).
- Breslow, R., Huang, D. -. L. Effects of metal ions including Mg2+ and lanthanides on the cleavage of ribonucleotides and RNA model compounds. Proc Natl Acad Sci U S A. 88 (10), 4080-4083 (1991).
- Blumberg, D. D. Creating a ribonuclease-free environment. Methods Enzymol. 152 (2), 20-24 (1987).
- AbouHaidar, M. G., Ivanov, I. G. Non-enzymatic RNA promoted by the combined catalytic activity of buffers and magnesium ions. Z Naturforsch C. 54 (7-8), 542-548 (1999).
- Farrell, R. E. . RNA Methodologies (4th Ed): A laboratory guide for isolation and characterization. 2, 45-80 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten