A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
בידוד ואפיון המיקרוביוטה הטבעית של מודל נמטודה Caenorhabditis elegans
In This Article
Summary
Caenorhabditis elegans הוא אחד ממיני המודל העיקריים בביולוגיה, אך כמעט כל המחקר מתבצע בהיעדר מיקרובים הקשורים אליו באופן טבעי. השיטות המתוארות כאן יסייעו לשפר את הבנתנו את מגוון המיקרובים הקשורים כבסיס למחקר פונקציונלי עתידי של C. elegans.
Abstract
הנמטודה Caenorhabditis elegans אינטראקציה עם מגוון גדול של מיקרואורגניזמים בטבע. באופן כללי, C. elegans נמצא בדרך כלל בחומר צמחי רקוב, במיוחד פירות רקובים כמו תפוחים או על ערימות קומפוסט. הוא קשור גם למארחים חסרי חוליות מסוימים כגון שבלולים ועץ. בתי גידול אלה עשירים במיקרובים, המשמשים כמזון ל-C. elegans ואשר יכולים גם ליישב בהתמדה את מעי הנמטודה. נכון להיום, המגוון והעקביות המדויקים של המיקרוביוטה המקומית C. elegans על פני בתי גידול ומיקומים גיאוגרפיים אינם מובנים במלואם. במאמר זה נתאר גישה מתאימה לבידוד C. elegans מהטבע ולאפיון המיקרוביוטה של התולעים. נמטודות ניתן לבודד בקלות מחומר קומפוסט, תפוחים נרקבים, שבלולים, או למשוך על ידי הנחת תפוחים על ערימות קומפוסט. זמן הפריים טיים למציאת C. elegans בחצי הכדור הצפוני הוא מספטמבר עד נובמבר. ניתן לשטוף תולעים מחומר המצע שנאסף על ידי טבילת המצע בתמיסת חיץ, ולאחר מכן איסוף נמטודות והעברתן למדיום גידול נמטודות או מאגר PCR לניתוח לאחר מכן. אנו ממחישים עוד יותר כיצד ניתן להשתמש בדגימות כדי לבודד ולטהר את המיקרואורגניזמים הקשורים לתולעים ולעבד תולעים עבור ניתוח RNA ריבוזומלי 16S של הרכב קהילת המיקרוביוטה. באופן כללי, השיטות המתוארות עשויות לעורר מחקר חדש על אפיון המיקרוביוטה של C. elegans על פני בתי גידול ומיקומים גיאוגרפיים, ובכך לסייע בהשגת הבנה מקיפה של המגוון והיציבות של המיקרוביוטה של הנמטודה כבסיס למחקר פונקציונלי עתידי.
Introduction
בטבע, C. elegans נמצא בדרך כלל בחומר צמחי רקוב, במיוחד פירות רקובים כמו תפוחים או על ערימות קומפוסט1. הוא קשור גם לפונדקאים חסרי חוליות מסוימים כמו שבלולים ועץ 2,3. בתי גידול אלה עשירים במיקרובים, אשר לא רק משמשים מזון לתולעת, אלא עשויים גם ליצור קשרים יציבים איתה. מידע על מגוון המיקרואורגניזמים הקשורים באופן טבעי פורסם רק בשנת 2016 4,5,6. מאז, מחקרים אלה ורק כמה מחקרים עדכניים יותר גילו כי C. elegans קשורה למגוון של חיידקים ופטריות, בדרך כלל כולל חיידקים מהסוג Pseudomonas, Enterobacter, Ochrobactrum, Erwinia, Comamonas, Gluconobacter, ועודכמה 6,7,8. מספר חיידקים קשורים יכולים ליישב ביציבות את מעי התולעת, אם כי לא כולם 6,9,10,11,12. הם עשויים להיות בעלי חשיבות מרכזית להבנתנו את הביולוגיה של C. elegans מכיוון שהם יכולים לספק תזונה, להגן מפני פתוגנים ואולי גורמי עקה אחרים, ולהשפיע על תכונות מרכזיות בהיסטוריית החיים כגון קצב הרבייה, התפתחות או תגובות התנהגותיות.
לדוגמה, מבודדים הקשורים באופן טבעי של הסוגים Pseudomonas, Ochrobactrum, וגם Enterobacter או Gluconobacter יכול להגן על התולעת מפני זיהום פתוגן והרג בדרכים שונות 5,6,11,13,14. בידוד ספציפי של הסוג Comamonas משפיע על התגובה התזונתית של נמטודה, התפתחות, תוחלת חיים ופוריות15,16,17. חיידקי פרובידנסיה מייצרים את הטיראמין הנוירומודולטורי ובכך מווסתים את פעילות מערכת העצבים המארחת ואת התגובות ההתנהגותיות הנובעות מכך18. קבוצה של חיידקים שונים הקשורים באופן טבעי הודגמו כמשפיעים על קצב גידול האוכלוסייה, הפוריות והתגובות ההתנהגותיות 5,6,9,11,19.
נכון להיום, המגוון והעקביות המדויקים של המיקרוביוטה המקומית C. elegans על פני בתי גידול ומיקומים גיאוגרפיים אינם מובנים במלואם, וקשרים נוספים בין התולעת למיקרובים מסביבתה עדיין נחשפים. מספר מחקרים קודמים השתמשו בזני חיידקים שבודדו מסביבת קרקע כלשהי, מבתי גידול טבעיים של C. elegans, או מניסויים במזוקוסמוס (כלומר, סביבות מבוססות מעבדה המשחזרות בתי גידול טבעיים) עם זני מעבדה של C. elegans 4,5,20. אף על פי שמחקרים אלה השיגו תובנות חדשות על השפעתם של מיקרובים על תכונות נמטודות ספציפיות (למשל, מטבוליזם של נמטודות21), הרלוונטיות של אינטראקציות אלה לביולוגיה של C. elegans בטבע אינה ברורה. לכן, כתב יד זה מתאר את השיטות לבודד ישירות C. elegans מהטבע ולבודד ולאחר מכן לאפיין את המיקרובים הקשורים באופן טבעי הן מתולעים בודדות והן מקבוצות של תולעים. השיטות המתוארות הן גרסה מעודכנת ומשופרת של ההליכים ששימשו בעבר לבידוד ואפיון של C. elegans טבעיים והמיקרוביוטה המקורית שלה 2,6,7. בהתחשב בכך ש- C. elegans נמצא באופן נרחב בחומר צמחי מתפרק ברחבי העולם (במיוחד בפירות נרקבים, באזורים ממוזגים ובסתיו)1,2,22,23,24,25, פרוטוקול זה יכול להיות מיושם על ידי כל מעבדה בכל פעם שיש עניין בהתייחסות ל- C. elegans תכונות למיקרובים הקשורים באופן טבעי ולכן הקשר רלוונטי יותר באופן טבעי. זה האחרון הוא חיוני להבנה מלאה של הביולוגיה של הנמטודה מכיוון שידוע ממגוון של מערכות מארחות אחרות שהמיקרוביוטה הקשורה יכולה להשפיע על מאפייני היסטוריית חיים מגוונים26, היבט שכיום מוזנח במידה רבה בריבוי המחקרים של C. elegans כמעט בכל תחומי מדעי החיים.
Protocol
1. הכנת מאגרים ומדיה
- הכן את מאגר ה-S על-ידי הוספת 5.85 גרם של NaCl, 1.123 גרם של K 2 HPO 4, 5.926 גרם של KH 2 PO4, ו-1 L של H2O שעבר דה-יוניזציה לבקבוקון ואוטוקלאב למשך 20 דקות ב-121 °C.
- הכינו מדיום צמיג על ידי הוספת S-buffer המכיל 1.2% (w/v) הידרוקסימתיל צלולוז (החומר הגורם לצמיגות המדיום), 5 מ"ג/מ"ל כולסטרול, 1 מ"מ MgSO4, 1 mM CaCl2 ואצטון 0.1% (v/v). Autoclave ומערבבים את המדיום צמיג עד שהוא הומוגני לחלוטין.
הערה: פעולה זו עשויה להימשך מספר שעות. כמו כן, S-buffer ניתן להכין ישירות ולהוסיף למדיום צמיג ללא עיקור מוקדם. - הכן את מאגר M9 על-ידי הוספת 3 גרם של KH 2 PO 4, 6 גרם של NA 2 HPO4·2 H 2 O, 5 גרם של NaCl, ו-1 L של H2O שעבר דה-יוניזציה לבקבוק של 1ליטר. Autoclave את התמיסה, ולאחר קירור, להוסיף 1 מ"ל של 1 M MgSO 4 (123.24 גרם של MgSO4·7H 2 O ב 500 מ"ל של H2O deionized, מסנן מעוקר).
- הכן 10% (v/v) פתרון מלאי Triton X-100 על ידי ערבוב 5 מ"ל של Triton X-100 עם 45 מ"ל של מאגר M9. סננו ועקרו את התמיסה באמצעות מסנן 0.2 מיקרומטר.
- הכן את מאגר M9 עם Triton X-100 (M9-T) על-ידי הוספת 2.5 מ"ל מתוך 10% (v/v) פתרון המניות של Triton X-100 ל-1 ליטר של מאגר M9 לאחר autoclaving כדי להשיג 0.025% (v/v) M9-T.
- הכינו 30% (v/v) גליצרול ב-S-buffer על ידי ערבוב של 15 מ"ל של 100% גליצרול סטרילי ו-35 מ"ל של S-buffer סטרילי בצינור של 50 מ"ל.
2. הכנת דגימות סביבתיות (איור 1)
- אספו דגימות סביבתיות כמו קומפוסט או פירות רקובים והניחו כל דגימה בשקית ניילון בודדת, בצינור או במיכל נקי אחר.
הערה: על מנת למשוך נמטודות, ניתן להניח תפוחים על קומפוסט מספר שבועות לפני הדגימה. - פזרו חתיכות מהדגימה הסביבתית באופן שווה בצלחת פטרי ריקה, סטרילית, בקוטר 9 ס"מ.
הערה: דגימות עם רמות גבוהות יותר של דעיכה נוטות יותר להכיל C. elegans. לחלופין, ניתן להשתמש בצלחת פטרי מלאה במדיום אגר נטול פפטון (PFM) כדי להגביר את הניגודיות. - לכסות את הדגימה בזהירות עם כ 20 מ"ל של מדיום צמיג סטרילי.
הערה: נמטודות צפות אל פני השטח תוך 1-2 שעות. המדיום הצמיג מאט את תנועת התולעים ומקל על דגימתן. ניתן להשתמש במאגר M9 סטרילי כחלופה, עם זאת, תנועת התולעת תהיה מהירה יותר.
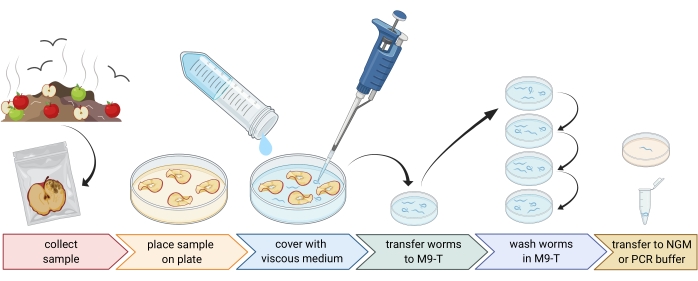
איור 1: בידוד נמטודות ממצעים. דגימות המצע מונחות בצלחות פטרי ריקות ומכוסות במדיום צמיג כדי לשטוף נמטודות. נמטודות מועברות ל-M9-T ונשטפות שוב ושוב כדי להסיר חיידקים מבחוץ. נמטודות בודדות יכולות לשמש לבידוד דנ"א, בידוד של חיידקים קשורים, או מונחות על צלחות אגר לאוכלוסיות תולעי תרבית. איור שנוצר עם BioRender.com. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
3. בידוד של נמטודות Caenorhabditis (איור 1)
- חפש נמטודות Caenorhabditis באמצעות מיקרוסקופ מנתח, בהתאם להנחיות שהוצגו בפרק WormBook על הבידוד של C. elegans ונמטודות קשורות על ידי Barrière ו Félix27.
הערה: Caenorhabditis hermaphrodites / נקבות יש מעיים של צבע חום בהיר (תחת תאורה משודרת). יתר על כן, לתאי המעיים יש גרעיני תאים גדולים, הנראים כנקודות לבנות. לעומת זאת, למיני נמטודות אחרים יש לעתים קרובות מעיים חומים כהים והם עשויים להראות אסימטריה אנטרופוסטרית בעוצמת הפיגמנט ובדרך כלל ללא גרעינים נראים לעין. הפות של הרמפרודיטים/נקבות Caenorhabditis בוגרות נמצא במרכז החיה. לוקליזציה זו של הפות לא תמיד מוצגת על ידי טקסה נמטודות אחרות. נמטודות Caenorhabditis הן בעלות שתי נורות לוע, ששתיהן נראות לעין עם הגדלה מספקת ותאורה מועברת ומנוגדות לטקסות נמטודות רבות אחרות. לנמטודות Caenorhabditis יש זנב מחודד, בעוד שלכמה נמטודות אחרות יש זנב עגול. - לאסוף את נמטודות Caenorhabditis תחת מיקרוסקופ מנתח בנוזל קטן ככל האפשר באמצעות פיפטה 20-100 μL. העבירו את הנמטודות שנאספו ישירות ל-1-3 מ"ל של M9-T סטרילי בצלחת פטרי סטרילית בקוטר 3 ס"מ לשטיפת התולעים כדי להסיר מיקרובים שאינם מחוברים (שלב 3.3) או לחילופין, העבירו תולעים בודדות לצלחת עם מדיום גידול נמטודות (NGM) כדי לבסס אוכלוסיית תולעים (שלב 3.4).
- שטפו את הנמטודות כדי להסיר חיידקים מהחלק החיצוני של הנמטודה.
הערה: פרוטוקול זה מעשיר את חיידק המעיים, אך אינו מבטל לחלוטין את היצמדות החיידקים לציפורן התולעת.- דגירה של הנמטודות במשך 10-15 דקות ב-M9-T.
- מוזגים את הנמטודות בנוזל קטן ככל האפשר לתוך 1-3 מ"ל של M9-T טרי בצלחת פטרי סטרילית חדשה בגודל 3 ס"מ.
- חזור על הדגירה והעבר את הנמטודות ל- M9-T טרי פעמיים נוספות.
הערה: ניתן לבצע את השלבים הבאים עם תולעים בודדות או עם אוכלוסיות תולעים. התולעים יכולות כעת לשמש לאפיון של C. elegans ומיקרואורגניזמים קשורים, כמו גם לבידוד של חיידקים (שלבים 4 ו -5). לחלופין, ניתן להעביר אותם בנפרד לצלחות NGM כדי לבסס אוכלוסיית תולעים (שלב 3.4).
- כדי להשיג אוכלוסיית תולעים, פיפטה נמטודה בודדת לצלחת NGM.
הערה: רק נמטודות הרמפרודיטיות כגון C. elegans או C . briggsae מסוגלות לייצר צאצאים מתולעים בודדות. עם זאת, תולעים בודדות של מינים אחרים עדיין עשויות לייצר צאצאים אם הם כבר הזדווגו.- נמטודות מבודדות באופן טבעי מכילות בדרך כלל חיידקים במעיים שלהן, שאותם הן משילות על צלחות ה-NGM, שם החיידקים האלה גדלים וזמינים כמזון ל-C. elegans. אין להוסיף אורגניזמים נפרדים של מזון כמו אורגניזם המזון הסטנדרטי במעבדה, זן E. coli OP50.
הערה: גידול תולעים על צלחות משפיע על הרכב קהילת החיידקים הקשורים, אך ההרכב עדיין דומה לזה של C. elegans הטבעי מבודד 6,7. - אפשרו לנמטודות להתרבות עד 10 ימים בטמפרטורה המתאימה (למשל, 15-20 מעלות צלזיוס במקומות ממוזגים). הקפיאו את הנמטודות האלה (שלב 3.5) או השתמשו בהן לאפיון של C. elegans ומיקרובים קשורים, כמו גם לבידוד של מיקרובים (שלבים 4 ו-5).
- נמטודות מבודדות באופן טבעי מכילות בדרך כלל חיידקים במעיים שלהן, שאותם הן משילות על צלחות ה-NGM, שם החיידקים האלה גדלים וזמינים כמזון ל-C. elegans. אין להוסיף אורגניזמים נפרדים של מזון כמו אורגניזם המזון הסטנדרטי במעבדה, זן E. coli OP50.
- הקפאת נמטודות לאחסון לטווח ארוך
- השאירו את הנמטודות על צלחות עד שחיידקי המזון נעלמו, ויש בעיקר שלבי זחל קטנים על הצלחות. לשטוף את התולעים מן הצלחות ב 1.5 מ"ל של S-buffer.
- ערבבו היטב 500 μL של S-buffer המכיל תולעת עם 500 μL של 30% (v/v) גליצרול ב-S-buffer בצינור סטרילי של 2 מ"ל. הקפיאו את הצינורות מיד בטמפרטורה של -80 מעלות צלזיוס לאחסון לטווח ארוך, אחרת הגליצרול עלול להזיק לנמטודות.
4. הכנת התולעים לזיהוי מולקולרי של C. elegans ומיקרובים
- לזיהוי בלתי משוחד של מיקרובים הקשורים לנמטודה, הכינו צלחת בגודל 96 בארות עם שלושה חרוזים סטריליים בקוטר 1 מ"מ, 19.5 מיקרוגרם PCR ו-0.5 מיקרוליטר פרוטאינאז K (20 מ"ג/מ"ל) לכל באר. פיפטה בודדת, נמטודה שטופה לכל באר, העברת כמה שפחות נוזלים.
- ניתן להשתמש גם באוכלוסיות תולעים. לשם כך, לשטוף את התולעים מן הצלחות עם M9-buffer ולהעביר ~ 300 μL של תולעת המכילה M9-buffer ל 2 מ"ל צינורות עם 10-15 חרוזים.
- מפרקים את הנמטודות באמצעות הומוגנייזר חרוזים (למשל, מכות חרוזים במשך 3 דקות ב-30 הרץ). צנטריפוגה של הצלחת או הצינורות לזמן קצר כדי להביא את הנוזל לתחתית (למשל, במשך 10 שניות ב 8000 x גרם בטמפרטורת החדר [RT]).
- זיהוי של C. elegans
- לבודד את הדנ"א של נמטודות בודדות על ידי חימום הדגימות במחזור PCR למשך שעה אחת ב-50 מעלות צלזיוס ו-15 דקות ב-95 מעלות צלזיוס. לבודד את הדנ"א של אוכלוסיות תולעים בכל שיטת בידוד שתבחרו (פרוטוקולים לדוגמה של שיטות בידוד שונות באמצעות ערכות מסחריות 7,9). הקפיאו את הדנ"א בטמפרטורה של -20 מעלות צלזיוס לאחסון לטווח ארוך.
- לזיהוי C. elegans, השתמש בדנ"א ובזוג הפריימרים nlp30-F (טבלה 1, 5'-ACACATACAACTGATCACTCA-3') ו-nlp30-R (טבלה 1, 5'-TACTTTCCCCCGTATC-3') ב-PCR, בהתאם להוראות של ספק Taq לפי בחירה.
- השתמש בתנאי ה-PCR הבאים: שלב דנטורציה ראשוני ב-95°C למשך 2 דקות, ואחריו 35 מחזורים של 95°C למשך 45 שניות, 55 °C למשך 30 שניות, 72°C למשך דקה אחת, ושלב התארכות סופי ב-72°C למשך 5 דקות.
- אפיון החיידקים הקשורים לנמטודה באמצעות ריצוף אמפליקון 16S של אזור V3-V4, באמצעות הדנ"א המבודד.
- הכן ספריית 16S עם פריימרים 16S לבחירה ופעל לפי פרוטוקול ערכת הכנת הספרייה. אפשרות אחת היא להשתמש בפריימרים 341F (טבלה 1, 5'-CCTACGGGNGGCWGCAG-3') ו-806R (טבלה 1, 5'-GACTACHVGGGTATCTAATCC-3') המכסים את אזור V3-V4 של הגן 16S rRNA, מה שמביא לרצפים שניתן לסווג עם מסדי נתונים סטנדרטיים ברזולוציה טובה7.
הערה: כמות הדנ"א של הקלט היא קריטית בשלב זה. דנ"א שנרכש מתולעים בודדות הולך להיות הרבה פחות מזה המתקבל מאוכלוסיות תולעים. עבור תולעים בודדות, ייתכן שיהיה צורך להגדיל את כמות הדנ"א הקלט או להגדיל את כמות מחזורי ה-PCR7. - ניתן לרצף ספריות על פלטפורמת רצף באמצעות ערכת רצף מתאימה.
הערה: כאן, פלטפורמת MiSeq משמשת עם ערכת ריאגנטים מתאימה של MiSeq. הריאגנטים משתפרים כל הזמן ויש לבחור אותם בסטנדרטים החדשים ביותר.
- הכן ספריית 16S עם פריימרים 16S לבחירה ופעל לפי פרוטוקול ערכת הכנת הספרייה. אפשרות אחת היא להשתמש בפריימרים 341F (טבלה 1, 5'-CCTACGGGNGGCWGCAG-3') ו-806R (טבלה 1, 5'-GACTACHVGGGTATCTAATCC-3') המכסים את אזור V3-V4 של הגן 16S rRNA, מה שמביא לרצפים שניתן לסווג עם מסדי נתונים סטנדרטיים ברזולוציה טובה7.
5. בידוד וטיפוח של חיידקים הקשורים לנמטודות (איור 2)
- כדי לבודד חיידקים, להכין צלחת 96 באר עם שלושה חרוזים סטריליים 1 מ"מ, 20 μL של M9-buffer לכל באר, פיפטה בודד, נמטודה שטף לכל באר, העברת נוזל קטן ככל האפשר.
הערה: לחלופין, ניתן לשטוף אוכלוסיות תולעים מהצלחות עם מאגר M9 ו~ 300 μL של חיץ M9 המכיל תולעת ניתן להעביר לצינורות 2 מ"ל עם 10-15 חרוזים. בכל המקרים, התולעים צריכות להגיע מתרבית שזוהתה כ- C. elegans באמצעות שלבים 4.1-4.4. - מפרקים את הנמטודות באמצעות הומוגנייזר חרוזים (למשל, מכות חרוזים במשך 3 דקות ב-30 הרץ). צנטריפוגה של הצלחת או הצינורות לזמן קצר כדי להביא את הנוזל לתחתית (למשל, עבור 10 שניות ב 8000 x גרם ב RT).
הערה: השימוש בשיטה זו הוביל לבידוד של טקסה חיידקית דומה לאלה שהתגלו על ידי ריצוף אמפליקון rDNA 16S7, מה שמרמז על כך שהכאת החרוזים המתוארת משאירה את רוב תאי החיידקים ללא פגע. - אוספים את הסופרנטנט, מדללים אותו באופן סדרתי בשעה 1:10, ולוחים עד 100 μL על צלחות אגר בקוטר 9 ס"מ.
- כדי להבטיח שניתן יהיה לטפח את רוב החיידקים, השתמשו במגוון מדיות חלופיות עם הרכבים תזונתיים שונים, כולל אגר סויה טריפטיקאז מדולל (TSA, דילול 1:10), אגר מקונקי, אגר גלוקוז סבורו, אגר דקסטרוז תפוחי אדמה, או אגר דקסטרוז פפטון שמרים.
- דגירה של הלוחות בתנאי הטמפרטורה הממוצעת של מיקום הדגימה (למשל, טמפרטורות בין 15-20 מעלות צלזיוס עבור מקומות ממוזגים) במשך 24-48 שעות.
- השתמשו בטכניקה הסטנדרטית של שלושה פסים ולולאה סטרילית כדי להשיג תרביות חיידקים טהורות (איור 2).
- בחרו מושבה בודדת מצלחת באמצעות לולאה סטרילית או קיסם ופזרו אותה על צלחת אגר חדשה המכילה את אותו מדיום אגר המשמש לטיהור. הקפידו להשתמש רק בכ-1/3 מהצלחת.
- השתמש בלולאה סטרילית חדשה או עקר לולאה לשימוש חוזר וגרור אותה דרך הפס הראשון כדי ליצור פס שני על 1/3 נוסף מאותו לוח.
- חזור על שלב זה על-ידי גרירת לולאה סטרילית דרך הפס השני.
- לדגור את הצלחת באותם תנאי גידול המשמשים לבידוד. טכניקה זו חייבת לגרום למושבות בודדות לגדול באזור הפס השלישי.
הערה: ייתכן שיהיה צורך לחזור על שלב הטיהור מספר פעמים מכיוון שמבודדים טבעיים נוטים ליצור ביופילמים ו/או אגרגטים.
- לגדל את המושבות הטהורות במדיום נוזלי (מאותו סוג כמו מדיום האגר) תוך שימוש באותה טמפרטורה וזמן גדילה כמו לעיל (שלב 5.4)
- הכן מלאי חיידקים על ידי הוספת 300 μL של תרבית החיידקים ל-200 μL של 86% (v/v) גליצרול (במדיום הגידול המתאים, למשל, TSB) ופיפטה למעלה ולמטה כדי לערבב כראוי. לחלופין, הכינו מלאי DMSO על ידי ערבוב של 50 μL של תרבית חיידקים עם 50 μL של 7% (v/v) DMSO. הקפאה בטמפרטורה של -80°C לאחסון לטווח ארוך.
- אפיון חיידקים באמצעות ריצוף של גן ה-RNA הריבוזומלי המלא 16S
- חלצו את הדנ"א החיידקי מתרביות נוזליות טהורות בטכניקה מתאימה (למשל, ערכת מיצוי DNA; מניסיון, פרוטוקול מיצוי מבוסס CTAB עובד טוב מאוד22).
- הגבר את הגן 16S rRNA באמצעות הפריימרים 27F (טבלה 1, 5'-GAGAGTTTTTCCTGGCTCG-3') ו- 1495R (טבלה 1, 5'-CTACGGCTACCTTGTTACGA -3')28 ותנאי ה-PCR הבאים: 95 °C, 2 דקות, 22x (95 °C, 30 שניות; 55 °C, 30 שניות; 72 °C, 100 שניות), ותקופת הארכה סופית ב- 72 °C, 5 דקות.
- על מנת לרכוש את הרצפים המלאים, השתמש בנוסף בשני פריימרים פנימיים לריצוף, כגון 701F (טבלה 1, 5'-GTGTAGCGGTGAAATGCG-3') ו- 785R (טבלה 1, 5'-GGATTAGATACCTGGTAGTCC-3')6.
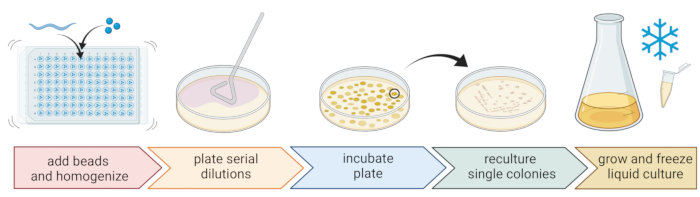
איור 2: זיהוי מינים ובידודם של חיידקים בודדים. נמטודות בודדות מתפרקות באמצעות הומוגנייזר חרוזים, והדנ"א מבודד לקביעת מינים באמצעות PCR או ריצוף. לחלופין, חומר הנמטודה המתפרק מדולל באופן סדרתי ומצופה על לוחות בינוניים לצמיחה. לוחות דוגרים עד להופעת מושבות חיידקים, ומושבות בודדות מפוספסות ללוחות חדשים כדי להשיג תרביות טהורות. מושבות בודדות של תרביות טהורות משמשות לגידול תרביות חיידקים נוזליות להכנת מלאי חיידקים לאחסון לטווח ארוך ב -80 מעלות צלזיוס. איור שנוצר עם BioRender.com. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
תוצאות
הנמטודה C. elegans נמצאת לעתים קרובות בפירות מתפרקים, כגון תפוחים, וגם בדגימות קומפוסט. בצפון גרמניה, C. elegans כמו גם מינים מולדים (במיוחד C. remanei אך גם C. briggsae) נמצאים בעיקר מספטמבר עד2 בנובמבר. הנמטודות נמצאות לרוב בחומר צמחי מתפרק, במיוחד פירות נרקבים כמו תפוחים או ?...
Discussion
הנמטודה Caenorhabditis elegans היא אחד מאורגניזמי המודל הנחקרים ביותר במחקר ביולוגי. הוא הוצג על ידי סידני ברנר בשנות ה -60, במקור להבנת ההתפתחות והתפקוד של מערכת העצבים29. מאז, C. elegans הפך למודל רב עוצמה לחקר תהליכים בסיסיים בכל הדיסציפלינות הביולוגיות, כולל ביולוגיה התנהגותית, נ...
Disclosures
אנו מצהירים כי אין לנו ניגוד עניינים.
Acknowledgements
אנו מכירים בתמיכה כספית מקרן המדע הגרמנית (פרויקטים A1.1 ו- A1.2 של מרכז המחקר השיתופי 1182 על מקורם ותפקודם של מטאאורגניזמים). אנו מודים לחברי מעבדת שולנבורג על עצתם ותמיכתם.
Materials
| Name | Company | Catalog Number | Comments |
| COMSOL | COMSOL | multiphysics simulation software |
References
- Schulenburg, H., Félix, M. -. A. The natural biotic environment of Caenorhabditis elegans. Genetics. 206 (1), 55-86 (2017).
- Petersen, C., Dirksen, P., Prahl, S., Strathmann, E. A., Schulenburg, H. The prevalence of Caenorhabditis elegans across 1.5 years in selected North German locations: the importance of substrate type, abiotic parameters, and Caenorhabditis competitors. BMC Ecology. 14 (1), 4 (2014).
- Petersen, C., et al. Travelling at a slug's pace: possible invertebrate vectors of Caenorhabditis nematodes. BMC Ecology. 15 (1), 19 (2015).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. -. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences. 113 (27), 3941-3949 (2016).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14 (1), 38 (2016).
- Johnke, J., Dirksen, P., Schulenburg, H. Community assembly of the native C. elegans microbiome is influenced by time, substrate and individual bacterial taxa. Environmental Microbiology. 22 (4), 1265-1279 (2020).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485 (2017).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3 Genes|Genomes|Genetics. 10 (9), 3025-3039 (2020).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2019).
- Kissoyan, K. A. B., et al. Exploring effects of C. elegans protective natural microbiota on host physiology. Frontiers in Cellular and Infection Microbiology. 12, 775728 (2022).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10 (1), 604 (2019).
- Kissoyan, K. A. B., et al. Natural C. elegans microbiota protects against infection via production of a cyclic lipopeptide of the viscosin group. Current Biology. 29 (6), 1030-1037 (2019).
- Watson, E., MacNeil, L. T., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Integration of metabolic and gene regulatory networks modulates the C. elegans dietary response. Cell. 153 (1), 253-266 (2013).
- Watson, E., et al. Interspecies systems biology uncovers metabolites affecting C. elegans gene expression and life history traits. Cell. 156 (4), 759-770 (2014).
- MacNeil, L. T., Watson, E., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Diet-induced developmental acceleration independent of TOR and insulin in C. elegans. Cell. 153 (1), 240-252 (2013).
- O'Donnell, M. P., Fox, B. W., Chao, P. -. H., Schroeder, F. C., Sengupta, P. A neurotransmitter produced by gut bacteria modulates host sensory behaviour. Nature. 583 (7816), 415-420 (2020).
- Snoek, B. L., et al. A multi-parent recombinant inbred line population of C. elegans allows identification of novel QTLs for complex life history traits. BMC Biology. 17 (1), 24 (2019).
- Avery, L., Shtonda, B. B. Food transport in the C. elegans pharynx. Journal of Experimental Biology. 206 (14), 2441-2457 (2003).
- Zhang, J., et al. A delicate balance between bacterial iron and reactive oxygen species supports optimal C. elegans development. Cell Host & Microbe. 26 (3), 400-411 (2019).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Félix, M. -. A., Duveau, F. Population dynamics and habitat sharing of natural populations of Caenorhabditis elegans and C. briggsae. BMC Biology. 10 (1), 59 (2012).
- Barrière, A., Félix, M. -. A. Temporal dynamics and linkage disequilibrium in natural Caenorhabditis elegans populations. Genetics. 176 (2), 999-1011 (2007).
- Dolgin, E. S., Félix, M. -. A., Cutter, A. D. Hakuna Nematoda: genetic and phenotypic diversity in African isolates of Caenorhabditis elegans and C. briggsae. Heredity. 100 (3), 304-315 (2008).
- Douglas, A. E. Simple animal models for microbiome research. Nature Reviews Microbiology. 17 (12), 764-775 (2019).
- Barrière, A., Félix, M. -. A. Isolation of C. elegans and related nematodes. WormBook. , 1-19 (2014).
- Weisburg, W. G., Barns, S. M., Pelletier, D. A., Lane, D. J. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 173 (2), 697-703 (1991).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77 (1), 71-94 (1974).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. Molecular Ecology. 31 (8), 2327-2347 (2022).
- Haber, M. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: Evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2004).
- Watson, E., et al. Metabolic network rewiring of propionate flux compensates vitamin B12 deficiency in C. elegans. eLife. 5, 17670 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved