Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Выделение и характеристика естественной микробиоты модельной нематоды Caenorhabditis elegans
В этой статье
Резюме
Caenorhabditis elegans является одним из основных модельных видов в биологии, но почти все исследования проводятся в отсутствие его естественно связанных микробов. Методы, описанные здесь, помогут улучшить наше понимание разнообразия ассоциированных микробов в качестве основы для будущих функциональных исследований C. elegans .
Аннотация
Нематода Caenorhabditis elegans взаимодействует с большим разнообразием микроорганизмов в природе. В целом, C. elegans обычно встречается в гнилом растительном материале, особенно в гнилых плодах, таких как яблоки или на компостных кучах. Он также связан с определенными беспозвоночными хозяевами, такими как слизни и мокрицы. Эти места обитания богаты микробами, которые служат пищей для C. elegans и которые также могут постоянно колонизировать кишечник нематоды. На сегодняшний день точное разнообразие и консистенция местной микробиоты C. elegans в разных местах обитания и географических местоположениях до конца не изучены. Здесь мы опишем подходящий подход для выделения C. elegans от природы и характеристики микробиоты червей. Нематод можно легко изолировать из компостного материала, гниющих яблок, слизней или привлечь, поместив яблоки на компостные кучи. Лучшее время для поиска C. elegans в Северном полушарии — с сентября по ноябрь. Черви могут быть вымыты из собранного материала субстрата путем погружения субстрата в буферный раствор с последующим сбором нематод и их переносом на среду роста нематод или буфер ПЦР для последующего анализа. Далее мы иллюстрируем, как образцы могут быть использованы для выделения и очистки микроорганизмов, связанных с червями, и для обработки червей для анализа рибосомной РНК 16S состава сообщества микробиоты. В целом, описанные методы могут стимулировать новые исследования по характеристике микробиоты C. elegans в местах обитания и географических местоположениях, тем самым помогая получить всестороннее понимание разнообразия и стабильности микробиоты нематоды в качестве основы для будущих функциональных исследований.
Введение
В природе C. elegans обычно встречается в гнилом растительном веществе, особенно в гнилых фруктах, таких как яблоки или на компостных кучах1. Он также связан с определенными беспозвоночными хозяевами, такими как слизни и мокрицы 2,3. Эти места обитания богаты микробами, которые не только служат пищей для червя, но и могут образовывать с ним устойчивые ассоциации. Информация о разнообразии естественно ассоциированных микроорганизмов была опубликована только в 2016 году 4,5,6. С тех пор эти и только несколько более поздних исследований показали, что C. elegans связан с различными бактериями и грибами, чаще всего включая бактерии рода Pseudomonas, Enterobacter, Ochrobactrum, Erwinia, Comamonas, Gluconobacter и некоторые другие 6,7,8. Несколько ассоциированных бактерий могут стабильно колонизировать кишечник червя, хотя не все 6,9,10,11,12. Они, вероятно, будут иметь ключевое значение для нашего понимания биологии C. elegans, потому что они могут обеспечить питание, защитить от патогенов и, возможно, других стрессоров и влиять на основные черты истории жизни, такие как репродуктивная скорость, развитие или поведенческие реакции.
Например, естественно ассоциированные изоляты родов Pseudomonas, Ochrobactrum, а также Enterobacter или Gluconobacter могут защищать червя от заражения патогенами и убивать различными способами 5,6,11,13,14. Специфический изолят рода Comamonas влияет на диетическую реакцию нематод, развитие, продолжительность жизни и фертильность 15,16,17. Бактерии Providencia продуцируют нейромодулятор тирамин и тем самым модулируют активность нервной системы хозяина и результирующие поведенческие реакции18. Было продемонстрировано, что набор различных естественно ассоциированных бактерий влияет на темпы роста населения, фертильность и поведенческие реакции 5,6,9,11,19.
На сегодняшний день точное разнообразие и консистенция местной микробиоты C. elegans в местах обитания и географических местоположениях не полностью поняты, и дальнейшие ассоциации между червем и микробами из его окружающей среды еще предстоит раскрыть. В нескольких предыдущих исследованиях использовались бактериальные штаммы, выделенные из некоторой почвенной среды, естественных мест обитания C. elegans или из экспериментов с мезокосмом (то есть лабораторных сред, которые воссоздают естественную среду обитания) с лабораторными штаммами C. elegans 4,5,20. Несмотря на то, что эти исследования дали новое представление о влиянии микробов на конкретные черты нематод (например, метаболизм нематод21), актуальность этих взаимодействий для биологии C. elegans в природе неясна. Поэтому в данной рукописи описываются методы непосредственного выделения C. elegans от природы и выделения и последующей характеристики естественно связанных микробов как от отдельных червей, так и от групп червей. Описанные способы представляют собой обновленный и улучшенный вариант процедур, применявшихся ранее для выделения и характеристики природного C. elegans и его нативной микробиоты 2,6,7. Учитывая, что C. elegans широко встречается в разлагающемся растительном веществе по всему земному шару (особенно в гниющих фруктах, умеренных регионах и осенью)1,2,22,23,24,25, этот протокол может применяться любой лабораторией всякий раз, когда есть интерес к связи C. elegans черты к естественно связанным микробам и, следовательно, более естественно релевантный контекст. Последнее имеет решающее значение для полного понимания биологии нематоды, потому что из множества других систем-хозяев известно, что связанная микробиота может влиять на различные характеристики истории жизни26, аспект, который в настоящее время в значительной степени игнорируется во множестве исследований C. elegans почти во всех дисциплинах наук о жизни.
протокол
1. Подготовка буферов и сред
- Получают S-буфер, добавляя 5,85 г NaCl, 1,123 гK2HPO4, 5,926 г KH2PO4 и 1 л деионизированногоH2O в колбу и автоклав в течение 20 мин при 121 °C.
- Готовят вязкую среду, добавляя S-буфер, содержащий 1,2% (мас./об.) гидроксиметилцеллюлозы (вещество, вызывающее вязкость среды), 5 мг/мл холестерина, 1 мМ МгСО4, 1 мМCaCl2 и 0,1% (v/v) ацетона. Автоклав и перемешайте вязкую среду до тех пор, пока она не станет полностью однородной.
ПРИМЕЧАНИЕ: Это может занять несколько часов. Кроме того, S-буфер может быть непосредственно приготовлен и добавлен в вязкую среду без предварительной стерилизации. - Готовят M9-буфер, добавляя 3 г KH2PO4, 6 г NA2HPO4·2H2O, 5 г NaCl и 1 л деионизированногоH2O в колбу объемом 1 л. Автоклавируют раствор, а после охлаждения добавляют 1 мл 1 М МгСО4 (123,24 г МгСО4·7Н2Ов 500 мл деионизированногоН2О, фильтр стерилизуют).
- Готовят 10% (v/v) раствор Triton X-100, смешивая 5 мл Triton X-100 с 45 мл M9-буфера. Фильтруют-стерилизуют раствор с помощью фильтра 0,2 мкм.
- Получают M9-буфер с Triton X-100 (M9-T) путем добавления 2,5 мл 10% (v/v) запасного раствора Triton X-100 в 1 л M9-буфера после автоклавирования для получения 0,025% (v/v) M9-T.
- Готовят 30% (v/v) глицерина в S-буфере, смешивая 15 мл стерильного 100% глицерина и 35 мл стерильного S-буфера в пробирке объемом 50 мл.
2. Подготовка проб окружающей среды (рисунок 1)
- Соберите образцы окружающей среды, такие как компост или гнилые фрукты, и поместите каждый образец в отдельный пластиковый пакет, трубку или другой чистый контейнер.
ПРИМЕЧАНИЕ: Чтобы привлечь нематод, яблоки можно поместить на компост за несколько недель до отбора проб. - Равномерно распределите кусочки образца окружающей среды в пустую, стерильную, 9 см чашку Петри.
ПРИМЕЧАНИЕ: Образцы с более высокими уровнями распада с большей вероятностью содержат C. elegans. Опционально, чашка Петри, наполненная безпептидной агаровой средой (PFM), может быть использована для повышения контрастности. - Тщательно накройте образец приблизительно 20 мл стерильной вязкой среды.
ПРИМЕЧАНИЕ: Нематоды всплывают на поверхность в течение 1-2 ч. Вязкая среда замедляет движение червей и облегчает их отбор проб. В качестве альтернативы можно использовать стерильный M9-буфер, однако движение червя будет происходить быстрее.
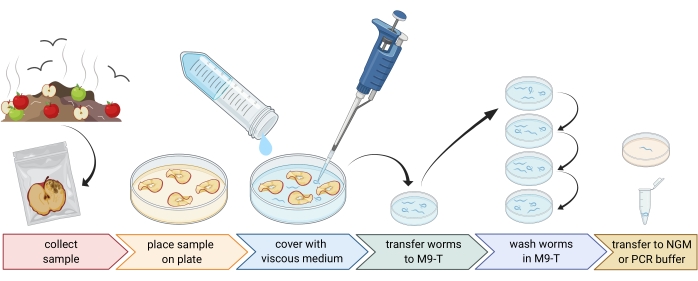
Рисунок 1: Выделение нематод из субстратов. Образцы субстрата помещают в пустые чашки Петри и покрывают вязкой средой для вымывания нематод. Нематод переносят в М9-Т и многократно промывают для удаления бактерий извне. Отдельные нематоды могут быть использованы для выделения ДНК, выделения ассоциированных бактерий или помещены на агаровые пластины для культивирования популяций червей. Рисунок, созданный с помощью BioRender.com. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
3. Выделение нематод Caenorhabditis (рисунок 1)
- Поиск нематод Caenorhabditis с помощью рассекающего микроскопа в соответствии с рекомендациями, представленными в главе WormBook об изоляции C. elegans и родственных нематод Barrière и Félix27.
ПРИМЕЧАНИЕ: Caenorhabditis hermaphrodites/ самки имеют кишечник светло-коричневого цвета (при передаваемом освещении). Более того, клетки кишечника имеют большие клеточные ядра, которые видны в виде белых точек. Напротив, другие виды нематод часто имеют темно-коричневый кишечник и могут проявлять переднезаднюю асимметрию в интенсивности пигмента и, как правило, отсутствие видимых ядер. Вульва взрослых гермафродитов Caenorhabditis / самок находится в середине животного. Эта локализация вульвы не всегда проявляется у других таксонов нематод. Нематоды Caenorhabditis обладают двумя глоточными луковицами, которые видны с достаточным увеличением и передаваемым освещением и контрастом со многими другими таксонами нематод. Caenorhabditis nematodes имеют заостренный хвост, тогда как некоторые другие нематоды имеют круглый хвост. - Соберите нематод Caenorhabditis под рассекающим микроскопом в как можно меньше жидкости, используя пипетку 20-100 мкл. Перенесите собранные нематоды непосредственно в 1-3 мл стерильного M9-T в стерильную 3 см чашку Петри для мытья червей для удаления неприсоединенных микробов (стадия 3.3) или, альтернативно, переложите отдельных червей на пластину со средой роста нематод (NGM) для создания популяции червей (этап 3.4).
- Вымойте нематод, чтобы удалить микробы с внешней стороны нематоды.
ПРИМЕЧАНИЕ: Этот протокол обогащает кишечные бактерии, но не полностью устраняет бактерии, прилипшие к кутикуле червя.- Инкубировать нематод в течение 10-15 мин в М9-Т.
- Пипетку нематод в как можно меньшем количестве жидкости в 1-3 мл свежего М9-Т в новой стерильной чашке Петри 3 см.
- Повторите инкубацию и переведите нематод на свежий М9-Т еще два раза.
ПРИМЕЧАНИЕ: Следующие шаги могут быть выполнены как с отдельными червями, так и с популяциями червей. Черви теперь могут быть использованы для характеристики C. elegans и связанных с ними микробов, а также для выделения бактерий (шаги 4 и 5). Альтернативно, они могут быть перенесены индивидуально на пластины NGM для создания популяции червей (шаг 3.4).
- Чтобы получить популяцию червей, пипетку отдельной нематоды на пластину NGM.
ПРИМЕЧАНИЕ: Только гермафродитные нематоды, такие как C. elegans или C. briggsae , способны производить потомство от одиночных червей. Тем не менее, одиночные черви других видов все еще могут производить потомство, если они уже были спарены.- Естественно изолированные нематоды обычно содержат бактерии в кишечнике, которые они проливают на пластины NGM, где эти бактерии растут и доступны в качестве пищи для C. elegans. Не добавляйте отдельные пищевые организмы, такие как стандартный лабораторный пищевой организм, штамм E. coli OP50.
ПРИМЕЧАНИЕ: Выращивание червей на пластинах влияет на состав ассоциированного бактериального сообщества, но состав по-прежнему сопоставим с составом природного C. elegans изолирует 6,7. - Дайте нематодам размножаться до 10 дней при соответствующей температуре (например, 15-20 ° C для умеренных мест). Заморозьте этих нематод (шаг 3.5) или используйте их для характеристики C. elegans и связанных с ними микробов, а также для выделения микробов (шаги 4 и 5).
- Естественно изолированные нематоды обычно содержат бактерии в кишечнике, которые они проливают на пластины NGM, где эти бактерии растут и доступны в качестве пищи для C. elegans. Не добавляйте отдельные пищевые организмы, такие как стандартный лабораторный пищевой организм, штамм E. coli OP50.
- Замораживание нематод для длительного хранения
- Оставляйте нематод на тарелках до тех пор, пока пищевые бактерии не исчезнут, и на пластинах в основном есть небольшие личиночные стадии. Смойте червей с пластин в 1,5 мл S-буфера.
- Тщательно смешайте 500 мкл червесодержащего S-буфера с 500 мкл 30% (v/v) глицерина в S-буфере в стерильной пробирке объемом 2 мл. Быстро заморозьте трубки при -80 °C для длительного хранения, иначе глицерин может нанести вред нематодам.
4. Подготовка червей к молекулярной идентификации C. elegans и микробов
- Для объективной идентификации нематод-ассоциированных микробов подготовьте 96-луночную пластину с тремя стерильными шариками по 1 мм, 19,5 мкл ПЦР-буфера и 0,5 мкл протеиназы К (20 мг/мл) на лунку. Пипетка индивидуальная, промытая нематода к каждой лунке, передавая как можно меньше жидкости.
- Популяции червей также могут быть использованы. Для этого промыть червей с пластин с М9-буфером и перенести ~300 мкл червесодержащего М9-буфера в 2 мл пробирки по 10-15 шариков.
- Разбейте нематод с помощью гомогенизатора бусин (например, бисерное взбивание в течение 3 мин при 30 Гц). Ненадолго центрифугируйте пластину или трубки, чтобы доставить жидкость на дно (например, в течение 10 с при 8000 х г при комнатной температуре [RT]).
- Идентификация C. elegans
- Выделяют ДНК отдельных нематод путем нагревания образцов в цикле ПЦР в течение 1 ч при 50 °C и 15 мин при 95 °C. Изолируйте ДНК популяций червей с помощью любого выбранного метода изоляции (примеры протоколов различных методов изоляции с использованием коммерческих наборов 7,9). Заморозьте ДНК при -20 °C для длительного хранения.
- Для идентификации C. elegans используйте ДНК и праймерную пару nlp30-F (таблица 1, 5'-ACACATACAACTGATCACTCA-3') и nlp30-R (таблица 1, 5'-TACTTTCCCCATCCGTATC-3') в ПЦР, следуя инструкциям поставщика Taq по выбору.
- Используйте следующие условия ПЦР: начальная стадия денатурации при 95 °C в течение 2 мин, затем 35 циклов 95 °C в течение 45 с, 55 °C в течение 30 с, 72 °C в течение 1 мин и конечная стадия удлинения при 72 °C в течение 5 мин. C. elegans производит ПЦР-продукт 154 bp.
- Охарактеризовать нематод-ассоциированные бактерии с помощью секвенирования 16S ампликонов области V3-V4, используя изолированную ДНК.
- Подготовьте библиотеку 16S с выбранными букварями 16S и следуйте протоколу комплекта подготовки библиотеки. Одним из вариантов является использование праймеров 341F (Таблица 1, 5'-CCTACGGGNGGCWGCAG-3') и 806R (Таблица 1, 5'-GACTACHVGGGTATCTAATCC-3'), охватывающих область V3-V4 гена 16S рРНК, что приводит к последовательностям, которые могут быть классифицированы со стандартными базами данных с хорошим разрешением7.
ПРИМЕЧАНИЕ: Количество входной ДНК имеет решающее значение на этом этапе. ДНК, полученной от одиночных червей, будет намного меньше, чем у популяций червей. Для одиночных червей может потребоваться увеличить количество входной ДНК или увеличить количество циклов ПЦР7. - Библиотеки могут быть секвенированы на платформе виртуализации с использованием подходящего комплекта секвенирования.
ПРИМЕЧАНИЕ: Здесь используется платформа MiSeq с подходящим набором реагентов MiSeq. Реагенты постоянно совершенствуются и должны быть подобраны по новейшим стандартам.
- Подготовьте библиотеку 16S с выбранными букварями 16S и следуйте протоколу комплекта подготовки библиотеки. Одним из вариантов является использование праймеров 341F (Таблица 1, 5'-CCTACGGGNGGCWGCAG-3') и 806R (Таблица 1, 5'-GACTACHVGGGTATCTAATCC-3'), охватывающих область V3-V4 гена 16S рРНК, что приводит к последовательностям, которые могут быть классифицированы со стандартными базами данных с хорошим разрешением7.
5. Выделение и культивирование нематод-ассоциированных бактерий (Рисунок 2)
- Чтобы изолировать бактерии, подготовьте 96-луночную пластину с тремя стерильными шариками 1 мм, 20 мкл M9-буфера на лунку и пипетку индивидуальной, промытой нематодой к каждой лунке, передавая как можно меньше жидкости.
ПРИМЕЧАНИЕ: Альтернативно, популяции червей могут быть смыты с пластин с M9-буфером и ~300 мкл червесодержащего M9-буфера могут быть перенесены в трубки 2 мл с 10-15 шариками. Во всех случаях черви должны происходить из культуры, которая была идентифицирована как C. elegans на шагах 4.1-4.4. - Разбейте нематод с помощью гомогенизатора бусин (например, бисерное взбивание в течение 3 мин при 30 Гц). Ненадолго центрифугируйте пластину или трубки, чтобы доставить жидкость на дно (например, в течение 10 с при 8000 х г при RT).
ПРИМЕЧАНИЕ: Использование этого метода привело к выделению бактериальных таксонов, аналогичных тем, которые были выявлены при секвенировании ампликона 16S рДНК7, предполагая, что описанное биение шариков оставляет большинство бактериальных клеток нетронутыми. - Соберите супернатант, последовательно разбавьте его в 1:10 и нанесите до 100 мкл на агаровую пластину размером 9 см.
- Чтобы обеспечить возможность культивирования большинства бактерий, используйте различные альтернативные среды с различными питательными составами, включая разбавленный триптиказный соевый агар (TSA, разведение 1: 10), агар Макконки, агар глюкозы Sabouraud, агар декстрозы картофеля или дрожжевой пептон декстрозы агар.
- Инкубируйте плиты в средних температурных условиях места отбора проб (например, при температуре между 15-20 °C для умеренных мест) в течение 24-48 ч.
- Используйте стандартную трехполосную технику и стерильную петлю для получения чистых бактериальных культур (рисунок 2).
- Выберите одну колонию из тарелки, используя стерильную петлю или зубочистку, и выложите ее на новую агаровую пластину, содержащую ту же агаровую среду, которая используется для очистки. Убедитесь, что используете только примерно 1/3 пластины.
- Либо используйте новую стерильную петлю, либо стерилизуйте многоразовую петлю и проведите ее через первую полосу, чтобы создать вторую полосу на другой 1/3 той же пластины.
- Повторите этот шаг, протащив стерильную петлю через вторую полосу.
- Инкубируйте пластину в тех же условиях роста, которые используются для изоляции. Этот метод должен привести к тому, что одиночные колонии растут в области третьей полосы.
ПРИМЕЧАНИЕ: Возможно, потребуется повторить стадию очистки несколько раз, поскольку природные изоляты имеют тенденцию образовывать биопленки и/или агрегаты.
- Выращивайте чистые колонии в жидкой среде (того же типа, что и агаровая среда), используя ту же температуру и время роста, что и выше (шаг 5.4)
- Подготовьте запасы бактерий, добавив 300 мкл бактериальной культуры к 200 мкл 86% (v/v) глицерина (в соответствующей питательной среде, например, TSB) и пипетку вверх и вниз для правильного смешивания. Альтернативно, получают запасы ДМСО путем смешивания 50 мкл бактериальной культуры с 50 мкл 7% (v/v) DMSO. Заморозить при -80 °C для длительного хранения.
- Характеристика бактерий с использованием секвенирования полного гена рибосомальной РНК 16S
- Экстрагируйте бактериальную ДНК из чистых жидких культур с использованием подходящей техники (например, набора для экстракции ДНК; по опыту, протокол экстракции на основе CTAB работает очень хорошо22).
- Амплифицируют ген 16S рРНК с помощью праймеров 27F (Таблица 1, 5'-GAGAGTTTGATCCTGGCTCAG-3') и 1495R (Таблица 1, 5'-CTACGGCTACCTTGTTACGA -3')28 и следующих условий ПЦР: 95 °C, 2 мин, 22x (95 °C, 30 с; 55 °C, 30 с; 72 °C, 100 с) и окончательного периода расширения при 72 °C, 5 мин.
- Чтобы получить полные последовательности, дополнительно используйте два внутренних праймера секвенирования, такие как 701F (Таблица 1, 5'-GTGTAGCGGTGAAATGCG-3') и 785R (Таблица 1, 5'-GGATTAGATACCCTGGTAGTCC-3')6.
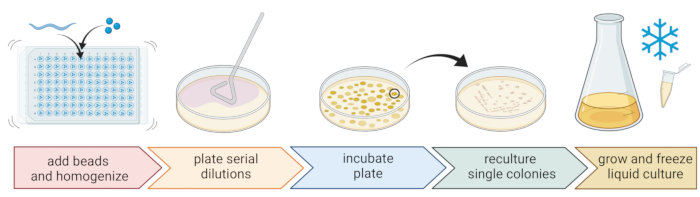
Рисунок 2: Видовая идентификация и выделение отдельных бактерий. Отдельные нематоды разбиваются с помощью гомогенизатора бусин, а ДНК выделяется для определения видов с помощью ПЦР или секвенирования. В качестве альтернативы, разрушенный нематодный материал последовательно разбавляют и наносят на пластины среды роста. Пластины инкубируют до тех пор, пока не появятся бактериальные колонии, а отдельные колонии переносятся на новые пластины для получения чистых культур. Одиночные колонии чистых культур используют для выращивания жидких бактериальных культур для подготовки бактериальных запасов к длительному хранению при -80 °C. Рисунок, созданный с помощью BioRender.com. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Результаты
Нематода C. elegans часто встречается в разлагающихся фруктах, таких как яблоки, а также в образцах компоста. В Северной Германии C. elegans, а также родственные виды (особенно C. remanei, а также C. briggsae) в основном встречаются с сентября по2 ноября. Нематоды чаще всего в...
Обсуждение
Нематода Caenorhabditis elegans является одним из наиболее интенсивно изучаемых модельных организмов в биологических исследованиях. Он был введен Сиднеем Бреннером в 1960-х годах, первоначально для понимания развития и функции нервной системы29. С тех пор C. elegans стал мощной м?...
Раскрытие информации
Мы заявляем, что у нас нет конфликта интересов.
Благодарности
Мы признаем финансовую поддержку со стороны Немецкого научного фонда (проекты A1.1 и A1.2 Центра совместных исследований 1182 по происхождению и функциям метаорганизмов). Мы благодарим членов лаборатории Шуленбурга за их советы и поддержку.
Материалы
| Name | Company | Catalog Number | Comments |
| COMSOL | COMSOL | multiphysics simulation software |
Ссылки
- Schulenburg, H., Félix, M. -. A. The natural biotic environment of Caenorhabditis elegans. Genetics. 206 (1), 55-86 (2017).
- Petersen, C., Dirksen, P., Prahl, S., Strathmann, E. A., Schulenburg, H. The prevalence of Caenorhabditis elegans across 1.5 years in selected North German locations: the importance of substrate type, abiotic parameters, and Caenorhabditis competitors. BMC Ecology. 14 (1), 4 (2014).
- Petersen, C., et al. Travelling at a slug's pace: possible invertebrate vectors of Caenorhabditis nematodes. BMC Ecology. 15 (1), 19 (2015).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. -. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences. 113 (27), 3941-3949 (2016).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14 (1), 38 (2016).
- Johnke, J., Dirksen, P., Schulenburg, H. Community assembly of the native C. elegans microbiome is influenced by time, substrate and individual bacterial taxa. Environmental Microbiology. 22 (4), 1265-1279 (2020).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485 (2017).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3 Genes|Genomes|Genetics. 10 (9), 3025-3039 (2020).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2019).
- Kissoyan, K. A. B., et al. Exploring effects of C. elegans protective natural microbiota on host physiology. Frontiers in Cellular and Infection Microbiology. 12, 775728 (2022).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10 (1), 604 (2019).
- Kissoyan, K. A. B., et al. Natural C. elegans microbiota protects against infection via production of a cyclic lipopeptide of the viscosin group. Current Biology. 29 (6), 1030-1037 (2019).
- Watson, E., MacNeil, L. T., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Integration of metabolic and gene regulatory networks modulates the C. elegans dietary response. Cell. 153 (1), 253-266 (2013).
- Watson, E., et al. Interspecies systems biology uncovers metabolites affecting C. elegans gene expression and life history traits. Cell. 156 (4), 759-770 (2014).
- MacNeil, L. T., Watson, E., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Diet-induced developmental acceleration independent of TOR and insulin in C. elegans. Cell. 153 (1), 240-252 (2013).
- O'Donnell, M. P., Fox, B. W., Chao, P. -. H., Schroeder, F. C., Sengupta, P. A neurotransmitter produced by gut bacteria modulates host sensory behaviour. Nature. 583 (7816), 415-420 (2020).
- Snoek, B. L., et al. A multi-parent recombinant inbred line population of C. elegans allows identification of novel QTLs for complex life history traits. BMC Biology. 17 (1), 24 (2019).
- Avery, L., Shtonda, B. B. Food transport in the C. elegans pharynx. Journal of Experimental Biology. 206 (14), 2441-2457 (2003).
- Zhang, J., et al. A delicate balance between bacterial iron and reactive oxygen species supports optimal C. elegans development. Cell Host & Microbe. 26 (3), 400-411 (2019).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Félix, M. -. A., Duveau, F. Population dynamics and habitat sharing of natural populations of Caenorhabditis elegans and C. briggsae. BMC Biology. 10 (1), 59 (2012).
- Barrière, A., Félix, M. -. A. Temporal dynamics and linkage disequilibrium in natural Caenorhabditis elegans populations. Genetics. 176 (2), 999-1011 (2007).
- Dolgin, E. S., Félix, M. -. A., Cutter, A. D. Hakuna Nematoda: genetic and phenotypic diversity in African isolates of Caenorhabditis elegans and C. briggsae. Heredity. 100 (3), 304-315 (2008).
- Douglas, A. E. Simple animal models for microbiome research. Nature Reviews Microbiology. 17 (12), 764-775 (2019).
- Barrière, A., Félix, M. -. A. Isolation of C. elegans and related nematodes. WormBook. , 1-19 (2014).
- Weisburg, W. G., Barns, S. M., Pelletier, D. A., Lane, D. J. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 173 (2), 697-703 (1991).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77 (1), 71-94 (1974).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. Molecular Ecology. 31 (8), 2327-2347 (2022).
- Haber, M. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: Evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2004).
- Watson, E., et al. Metabolic network rewiring of propionate flux compensates vitamin B12 deficiency in C. elegans. eLife. 5, 17670 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены