Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Isolement et caractérisation du microbiote naturel du nématode modèle Caenorhabditis elegans
Dans cet article
Résumé
Caenorhabditis elegans est l’une des principales espèces modèles en biologie, mais presque toutes les recherches sont effectuées en l’absence de ses microbes naturellement associés. Les méthodes décrites ici aideront à améliorer notre compréhension de la diversité des microbes associés comme base pour les futures recherches fonctionnelles sur C. elegans.
Résumé
Le nématode Caenorhabditis elegans interagit avec une grande diversité de micro-organismes dans la nature. En général, C. elegans se trouve couramment dans la matière végétale pourrie, en particulier les fruits pourris comme les pommes ou sur les tas de compost. Il est également associé à certains hôtes invertébrés tels que les limaces et les cloportes. Ces habitats sont riches en microbes, qui servent de nourriture à C. elegans et qui peuvent également coloniser de manière persistante l’intestin des nématodes. À ce jour, la diversité et la cohérence exactes du microbiote indigène de C. elegans dans les habitats et les emplacements géographiques ne sont pas entièrement comprises. Nous décrivons ici une approche appropriée pour isoler C. elegans de la nature et caractériser le microbiote des vers. Les nématodes peuvent être facilement isolés des matières compostables, des pommes en décomposition, des limaces ou attirés en plaçant des pommes sur des tas de compost. Le moment idéal pour trouver C. elegans dans l’hémisphère nord est de septembre à novembre. Les vers peuvent être éliminés du substrat collecté en immergeant le substrat dans une solution tampon, puis en collectant des nématodes et en les transférant sur un milieu de croissance des nématodes ou un tampon PCR pour une analyse ultérieure. Nous illustrons en outre comment les échantillons peuvent être utilisés pour isoler et purifier les micro-organismes associés aux vers et pour traiter les vers pour l’analyse de l’ARN ribosomique 16S de la composition de la communauté du microbiote. Dans l’ensemble, les méthodes décrites pourraient stimuler de nouvelles recherches sur la caractérisation du microbiote de C. elegans dans les habitats et les emplacements géographiques, aidant ainsi à obtenir une compréhension globale de la diversité et de la stabilité du microbiote du nématode comme base pour de futures recherches fonctionnelles.
Introduction
Dans la nature, C. elegans se trouve couramment dans la matière végétale pourrie, en particulier les fruits pourris comme les pommes ou sur les tas de compost1. Il est également associé à certains hôtes invertébrés tels que les limaces et les cloportes 2,3. Ces habitats sont riches en microbes, qui non seulement servent de nourriture au ver, mais peuvent également former des associations stables avec lui. Les informations sur la diversité des micro-organismes naturellement associés n’ont été publiées qu’en 2016 4,5,6. Depuis lors, ces études et seulement quelques études plus récentes ont révélé que C. elegans est associé à une variété de bactéries et de champignons, y compris le plus souvent des bactéries du genre Pseudomonas, Enterobacter, Ochrobactrum, Erwinia, Comamonas, Gluconobacter, et plusieurs autres 6,7,8. Plusieurs bactéries associées peuvent coloniser de manière stable l’intestin du ver, mais pas toutes 6,9,10,11,12. Ils sont susceptibles d’être d’une importance clé pour notre compréhension de la biologie de C. elegans, car ils peuvent fournir de la nutrition, protéger contre les agents pathogènes et éventuellement d’autres facteurs de stress, et affecter les traits centraux de l’histoire de vie tels que le taux de reproduction, le développement ou les réponses comportementales.
À titre d’exemple, les isolats naturellement associés des genres Pseudomonas, Ochrobactrum, ainsi que Enterobacter ou Gluconobacter peuvent protéger le ver contre l’infection pathogène et tuer de manière distincte 5,6,11,13,14. Un isolat spécifique du genre Comamonas influence la réponse alimentaire, le développement, la durée de vie et la fertilité des nématodes15,16,17. Les bactéries de Providencia produisent le neuromodulateur tyramine et modulent ainsi l’activité du système nerveux de l’hôte et les réponses comportementales résultantes18. Il a été démontré qu’un ensemble de différentes bactéries naturellement associées affectait le taux de croissance de la population, la fertilité et les réponses comportementales 5,6,9,11,19.
À ce jour, la diversité et la cohérence exactes du microbiote indigène de C. elegans dans les habitats et les emplacements géographiques ne sont pas entièrement comprises, et d’autres associations entre le ver et les microbes de son environnement restent à découvrir. Plusieurs études antérieures ont utilisé des souches bactériennes isolées dans certains environnements pédologiques, des habitats naturels de C. elegans ou des expériences de mésocosme (c.-à-d. des environnements de laboratoire qui recréent des habitats naturels) avec des souches de laboratoire 4,5,20 de C. elegans. Même si ces études ont permis d’obtenir de nouvelles connaissances sur l’influence des microbes sur des caractéristiques spécifiques des nématodes (p. ex. métabolisme des nématodes21), la pertinence de ces interactions pour la biologie de C. elegans dans la nature n’est pas claire. Par conséquent, ce manuscrit décrit les méthodes permettant d’isoler directement C. elegans de la nature et d’isoler et de caractériser par la suite les microbes naturellement associés à la fois des vers uniques et des groupes de vers. Les méthodes décrites sont une version mise à jour et améliorée des procédures utilisées précédemment pour l’isolement et la caractérisation de C. elegans naturel et de son microbiote natif 2,6,7. Étant donné que C. elegans est largement présent dans la matière végétale en décomposition à travers le monde (en particulier dans les fruits pourris, les régions tempérées et en automne)1,2,22,23,24,25, ce protocole peut être appliqué par n’importe quel laboratoire chaque fois qu’il y a un intérêt à relier C. elegans des microbes naturellement associés et donc un contexte plus naturellement pertinent. Ce dernier est essentiel pour une compréhension complète de la biologie du nématode, car il est connu par une diversité d’autres systèmes hôtes que le microbiote associé peut affecter diverses caractéristiques du cycle biologique26, un aspect qui est actuellement largement négligé dans la multitude d’études de C. elegans dans presque toutes les disciplines des sciences de la vie.
Protocole
1. Préparation des tampons et des milieux
- Préparer le tampon S en ajoutant 5,85 g de NaCl, 1,123 g de K2HPO4, 5,926 g de KH2PO4 et 1 L deH2Odésionisé dans une fiole et un autoclave pendant 20 min à 121°C.
- Préparer un milieu visqueux en ajoutant du tampon S contenant 1,2 % (p/v) d’hydroxyméthylcellulose (la substance causant la viscosité du milieu), 5 mg/mL de cholestérol, 1 mM de MgSO4, 1 mM de CaCl2 et 0,1 % (v/v) d’acétone. Autoclaver et agiter le milieu visqueux jusqu’à ce qu’il soit complètement homogène.
REMARQUE: Cela peut prendre plusieurs heures. En outre, le tampon S peut être directement préparé et ajouté au milieu visqueux sans stérilisation préalable. - Préparer le tampon M9 en ajoutant 3 g de KH 2PO4, 6 g de NA2HPO4·2H2O, 5 g de NaCl et 1 L deH2Odésionisé dans une fiole de 1 L. Autoclaver la solution, et après refroidissement, ajouter 1 mL de 1 M MgSO4 (123,24 g deMgSO4·7H2O dans 500 mL deH2Odésionisé, filtre stérilisé).
- Préparer la solution mère de Triton X-100 à 10 % (v/v) en mélangeant 5 mL de Triton X-100 avec 45 mL de tampon M9. Filtrer-stériliser la solution à l’aide d’un filtre de 0,2 μm.
- Préparer le tampon M9 avec Triton X-100 (M9-T) en ajoutant 2,5 ml de la solution mère Triton X-100 à 10 % (v/v) à 1 L de tampon M9 après autoclavage pour obtenir 0,025 % (v/v) M9-T.
- Préparer 30 % (v/v) de glycérol dans du tampon S en mélangeant 15 mL de glycérol stérile à 100 % et 35 mL de tampon S stérile dans un tube de 50 mL.
2. Préparation d’échantillons environnementaux (figure 1)
- Prélevez des échantillons environnementaux comme du compost ou des fruits pourris et placez chaque échantillon dans un sac en plastique, un tube ou un autre contenant propre.
REMARQUE : Afin d’attirer les nématodes, les pommes peuvent être placées sur du compost plusieurs semaines avant l’échantillonnage. - Répartir uniformément les morceaux de l’échantillon environnemental dans une boîte de Petri vide stérile de 9 cm.
REMARQUE : Les échantillons présentant des niveaux plus élevés de désintégration sont plus susceptibles de contenir C. elegans. En option, une boîte de Petri remplie d’un milieu gélosé sans peptone (PFM) peut être utilisée pour augmenter le contraste. - Couvrir soigneusement l’échantillon avec environ 20 mL de milieu visqueux stérile.
NOTE: Les nématodes flottent à la surface en 1-2 heures. Le milieu visqueux ralentit le mouvement des vers et facilite leur échantillonnage. Un tampon M9 stérile peut être utilisé comme alternative, mais le mouvement des vers sera plus rapide.
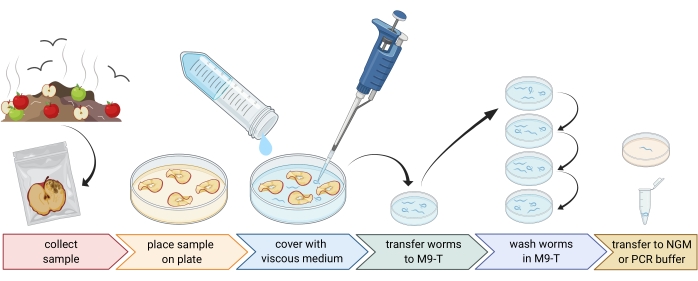
Figure 1 : Isolement des nématodes des substrats. Les échantillons de substrat sont placés dans des boîtes de Petri vides et recouverts d’un milieu visqueux pour éliminer les nématodes. Les nématodes sont transférés à M9-T et lavés à plusieurs reprises pour éliminer les bactéries de l’extérieur. Les nématodes individuels peuvent être utilisés pour isoler l’ADN, isoler les bactéries associées ou être placés sur des plaques de gélose pour cultiver des populations de vers. Figure créée avec BioRender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Isolement des nématodes Caenorhabditis (figure 1)
- Recherche de nématodes Caenorhabditis à l’aide d’un microscope à dissection, en suivant les lignes directrices présentées dans le chapitre WormBook sur l’isolement de C. elegans et des nématodes apparentés par Barrière et Félix27.
NOTE: Caenorhabditis hermaphrodites/femelles ont un intestin de couleur brun clair (sous éclairage transmis). De plus, les cellules intestinales ont de grands noyaux cellulaires, qui sont visibles sous forme de points blancs. En revanche, d’autres espèces de nématodes ont souvent un intestin brun foncé et peuvent présenter une asymétrie antéropostérieure dans l’intensité pigmentaire et généralement pas de noyaux visibles. La vulve des Caenorhabditis hermaphrodites/femelles adultes se trouve au milieu de l’animal. Cette localisation de la vulve n’est pas toujours démontrée par les autres taxons de nématodes. Les nématodes Caenorhabditis possèdent deux bulbes pharyngés, qui sont tous deux visibles avec un grossissement suffisant et un éclairage transmis et contrastent avec de nombreux autres taxons de nématodes. Les nématodes Caenorhabditis ont une queue pointue, tandis que d’autres nématodes ont une queue ronde. - Recueillir les nématodes Caenorhabditis au microscope à dissection dans le moins de liquide possible à l’aide d’une pipette de 20 à 100 μL. Transférer les nématodes prélevés directement dans 1 à 3 mL de M9-T stérile dans une boîte de Petri stérile de 3 cm pour laver les vers afin d’éliminer les microbes non attachés (étape 3.3) ou, alternativement, transférer les vers individuels sur une plaque avec un milieu de croissance des nématodes (NGM) pour établir une population de vers (étape 3.4).
- Lavez les nématodes pour éliminer les microbes de l’extérieur du nématode.
REMARQUE: Ce protocole enrichit les bactéries intestinales mais n’élimine pas complètement les bactéries qui collent à la cuticule du ver.- Incuber les nématodes pendant 10-15 min dans M9-T.
- Pipeter les nématodes dans le moins de liquide possible dans 1-3 mL de M9-T frais dans une nouvelle boîte de Pétri stérile de 3 cm.
- Répétez l’incubation et transférez les nématodes sur M9-T frais deux fois de plus.
REMARQUE : les étapes suivantes peuvent être effectuées avec des vers individuels ou avec des populations de vers. Les vers peuvent maintenant être utilisés pour la caractérisation de C. elegans et des microbes associés, ainsi que pour l’isolement des bactéries (étapes 4 et 5). Alternativement, ils peuvent être transférés individuellement sur des plaques NGM pour établir une population de vers (étape 3.4).
- Pour obtenir une population de vers, pipeter un nématode individuel sur une plaque NGM.
NOTE: Seuls les nématodes hermaphrodites tels que C. elegans ou C . briggsae sont capables de produire une progéniture à partir de vers uniques. Cependant, les vers uniques d’autres espèces peuvent encore produire une progéniture s’ils étaient déjà accouplés.- Les nématodes isolés naturellement contiennent généralement des bactéries dans leur intestin, qu’ils excrètent sur les plaques NGM, où ces bactéries se développent et sont disponibles comme nourriture pour C. elegans. N’ajoutez pas d’organismes alimentaires distincts comme l’organisme alimentaire de laboratoire standard, la souche OP50 d’E. coli.
NOTE: Les vers d’élevage sur les plaques influencent la composition de la communauté bactérienne associée, mais la composition est encore comparable à celle des isolats naturels de C. elegans 6,7. - Laisser les nématodes proliférer jusqu’à 10 jours à la température appropriée (p. ex. 15 à 20 °C pour les endroits tempérés). Congeler ces nématodes (étape 3.5) ou les utiliser pour la caractérisation de C. elegans et des microbes associés ainsi que pour l’isolement des microbes (étapes 4 et 5).
- Les nématodes isolés naturellement contiennent généralement des bactéries dans leur intestin, qu’ils excrètent sur les plaques NGM, où ces bactéries se développent et sont disponibles comme nourriture pour C. elegans. N’ajoutez pas d’organismes alimentaires distincts comme l’organisme alimentaire de laboratoire standard, la souche OP50 d’E. coli.
- Congélation des nématodes pour un stockage à long terme
- Laissez les nématodes dans les assiettes jusqu’à ce que les bactéries alimentaires aient disparu et qu’il y ait principalement de petits stades larvaires dans les assiettes. Laver les vers des plaques dans 1,5 mL de tampon S.
- Bien mélanger 500 μL de tampon S contenant des vers avec 500 μL de glycérol à 30 % (v/v) dans du tampon S dans un tube stérile de 2 mL. Congelez rapidement les tubes à -80 °C pour un stockage à long terme, sinon le glycérol pourrait nuire aux nématodes.
4. Préparation des vers pour l’identification moléculaire de C. elegans et des microbes
- Pour une identification impartiale des microbes associés aux nématodes, préparer une plaque de 96 puits avec trois billes stériles de 1 mm, 19,5 μL de tampon PCR et 0,5 μL de protéinase K (20 mg/mL) par puits. Pipeter un nématode lavé individuel dans chaque puits, en transférant le moins de liquide possible.
- Les populations de vers peuvent également être utilisées. Pour cela, lavez les vers des plaques avec un tampon M9 et transférez ~300 μL de tampon M9 contenant des vers à des tubes de 2 mL avec 10-15 billes.
- Briser les nématodes à l’aide d’un homogénéisateur de billes (p. ex., battre les perles pendant 3 minutes à 30 Hz). Centrifuger brièvement la plaque ou les tubes pour amener le liquide au fond (par exemple, pendant 10 s à 8000 x g à température ambiante [RT]).
- Identification de C. elegans
- Isoler l’ADN de nématodes individuels en chauffant les échantillons dans un cycleur de PCR pendant 1 h à 50 °C et 15 min à 95 °C. Isoler l’ADN des populations de vers en utilisant n’importe quelle méthode d’isolement de votre choix (exemples de protocoles de différentes méthodes d’isolement utilisant des kits commerciaux 7,9). Congeler l’ADN à -20 °C pour un stockage à long terme.
- Pour l’identification de C. elegans, utiliser l’ADN et la paire d’amorces nlp30-F (tableau 1, 5'-ACACATACAACTGATCACTCA-3') et nlp30-R (tableau 1, 5'-TACTTTCCCCATCCGTATC-3') dans une PCR, en suivant les instructions d’un fournisseur Taq de choix.
- Utilisez les conditions de PCR suivantes : étape initiale de dénaturation à 95 °C pendant 2 min, suivie de 35 cycles de 95 °C pendant 45 s, 55 °C pendant 30 s, 72 °C pendant 1 min, et une étape finale d’allongement à 72 °C pendant 5 min. C. elegans produit un produit PCR de 154 pb.
- Caractériser les bactéries associées aux nématodes par séquençage d’amplicon 16S de la région V3-V4, en utilisant l’ADN isolé.
- Préparez une bibliothèque 16S avec les amorces 16S de votre choix et suivez le protocole du kit de préparation de bibliothèque. Une option consiste à utiliser les amorces 341F (Tableau 1, 5'-CCTACGGGNGGCWGCAG-3') et 806R (Tableau 1, 5'-GACTACHVGGGTATCTAATCC-3') couvrant la région V3-V4 du gène de l’ARNr 16S, ce qui donne des séquences qui peuvent être classées avec des bases de données standard avec une bonne résolution7.
REMARQUE: La quantité d’ADN d’entrée est critique dans cette étape. L’ADN acquis à partir de vers uniques sera beaucoup moins important que celui obtenu à partir de populations de vers. Pour les vers simples, il peut être nécessaire d’augmenter la quantité d’ADN entrant ou d’augmenter la quantité de cycles de PCR7. - Les bibliothèques peuvent être séquencées sur une plate-forme de séquençage à l’aide d’un kit de séquençage approprié.
REMARQUE: Ici, la plate-forme MiSeq est utilisée avec un kit de réactif MiSeq approprié. Les réactifs sont constamment améliorés et doivent être choisis selon les normes les plus récentes.
- Préparez une bibliothèque 16S avec les amorces 16S de votre choix et suivez le protocole du kit de préparation de bibliothèque. Une option consiste à utiliser les amorces 341F (Tableau 1, 5'-CCTACGGGNGGCWGCAG-3') et 806R (Tableau 1, 5'-GACTACHVGGGTATCTAATCC-3') couvrant la région V3-V4 du gène de l’ARNr 16S, ce qui donne des séquences qui peuvent être classées avec des bases de données standard avec une bonne résolution7.
5. Isolement et culture de bactéries associées aux nématodes (figure 2)
- Pour isoler les bactéries, préparez une plaque de 96 puits avec trois billes stériles de 1 mm, 20 μL de tampon M9 par puits, et pipeter un nématode lavé individuel dans chaque puits, en transférant le moins de liquide possible.
REMARQUE: Alternativement, les populations de vers peuvent être lavées des plaques avec un tampon M9 et ~300 μL de tampon M9 contenant des vers peuvent être transférés dans des tubes de 2 ml avec 10-15 billes. Dans tous les cas, les vers doivent provenir d’une culture qui a été identifiée comme étant C. elegans en utilisant les étapes 4.1-4.4. - Briser les nématodes à l’aide d’un homogénéisateur de billes (p. ex., battre les perles pendant 3 minutes à 30 Hz). Centrifuger brièvement la plaque ou les tubes pour amener le liquide au fond (par exemple, pendant 10 s à 8000 x g à TA).
NOTE: L’utilisation de cette méthode a conduit à l’isolement de taxons bactériens similaires à ceux révélés par le séquençage de l’amplicon de l’ADNr16S 7, ce qui suggère que le battement de billes décrit laisse la plupart des cellules bactériennes intactes. - Recueillir le surnageant, le diluer en série à 1:10 et plaquer jusqu’à 100 μL sur des plaques de gélose de 9 cm.
- Pour vous assurer que la plupart des bactéries peuvent être cultivées, utilisez une variété de milieux alternatifs avec différentes compositions nutritives, y compris la gélose trypticase de soja diluée (TSA, dilution 1:10), la gélose MacConkey, la gélose au glucose Sabouraud, la gélose au dextrose de pomme de terre ou la gélose peptone de levure dextrose.
- Incuber les plaques aux conditions de température moyenne du lieu d’échantillonnage (par exemple, des températures comprises entre 15 et 20 °C pour les endroits tempérés) pendant 24 à 48 h.
- Utilisez la technique standard à trois stries et une boucle stérile pour obtenir des cultures bactériennes pures (Figure 2).
- Prélever une seule colonie dans une assiette à l’aide d’une boucle stérile ou d’un cure-dent et la glisser sur une nouvelle plaque de gélose contenant le même milieu gélose que celui utilisé pour la purification. Assurez-vous de n’utiliser qu’environ 1/3 de la plaque.
- Utilisez une nouvelle boucle stérile ou stérilisez une boucle réutilisable et faites-la glisser à travers la première traînée pour créer une deuxième traînée sur un autre 1/3 de la même plaque.
- Répétez cette étape en faisant glisser une boucle stérile à travers la deuxième strie.
- Incuber la plaque dans les mêmes conditions de croissance que celles utilisées pour l’isolement. Cette technique doit entraîner la croissance de colonies uniques dans la zone de la troisième traînée.
REMARQUE: Il peut être nécessaire de répéter l’étape de purification plusieurs fois car les isolats naturels ont tendance à former des biofilms et / ou des agrégats.
- Cultiver les colonies pures dans un milieu liquide (du même type que le milieu gélosé) en utilisant la même température et le même temps de croissance que ci-dessus (étape 5.4)
- Préparer les stocks de bactéries en ajoutant 300 μL de la culture bactérienne à 200 μL de glycérol à 86 % (v/v) (dans le milieu de croissance respectif, p. ex. BST) et pipeter de haut en bas pour bien mélanger. Vous pouvez également préparer des stocks de DMSO en mélangeant 50 μL de culture bactérienne avec 50 μL de DMSO à 7 % (v/v). Congeler à -80 °C pour un stockage à long terme.
- Caractérisation des bactéries par séquençage du gène complet de l’ARN ribosomique 16S
- Extraire l’ADN bactérien de cultures liquides pures en utilisant une technique appropriée (par exemple, un kit d’extraction d’ADN; par expérience, un protocole d’extraction basé sur CTAB fonctionne très bien22).
- Amplifier le gène de l’ARNr 16S à l’aide des amorces 27F (Tableau 1, 5'-GAGAGTTTGATCCTGGCTCAG-3') et 1495R (Tableau 1, 5'-CTACGGCTACCTTGTTACGA -3')28 et des conditions de PCR suivantes : 95 °C, 2 min, 22x (95 °C, 30 s; 55 °C, 30 s; 72 °C, 100 s) et une période de prolongation finale à 72 °C, 5 min.
- Afin d’acquérir les séquences complètes, utilisez en outre deux amorces de séquençage internes, telles que 701F (Tableau 1, 5'-GTGTAGCGGTGAAATGCG-3') et 785R (Tableau 1, 5'-GGATTAGATACCCTGGTAGTCC-3')6.
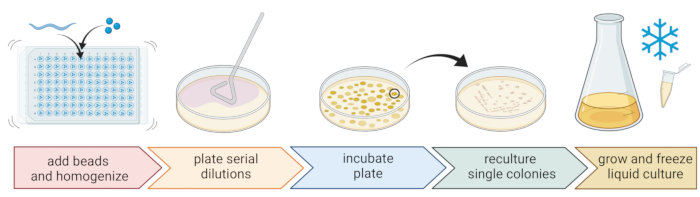
Figure 2 : Identification des espèces et isolement des bactéries individuelles. Les nématodes individuels sont brisés à l’aide d’un homogénéisateur de billes, et l’ADN est isolé pour la détermination de l’espèce par PCR ou séquençage. Alternativement, le nématode brisé est dilué en série et plaqué sur des plaques de milieu de croissance. Les plaques sont incubées jusqu’à l’apparition de colonies bactériennes, et les colonies individuelles sont striées sur de nouvelles plaques pour obtenir des cultures pures. Des colonies uniques de cultures pures sont utilisées pour cultiver des cultures bactériennes liquides pour la préparation de stocks bactériens pour un stockage à long terme à -80 ° C. Figure créée avec BioRender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
Le nématode C. elegans est fréquemment trouvé dans les fruits en décomposition, tels que les pommes, ainsi que dans les échantillons de compost. Dans le nord de l’Allemagne, C. elegans ainsi que des espèces congénériques (en particulier C. remanei mais aussi C. briggsae) se trouvent principalement de septembre au2 novembre. Les nématodes se trouvent le plus souvent dans la matière végétale en décomposition, en particulier les fruits pourris tels q...
Discussion
Le nématode Caenorhabditis elegans est l’un des organismes modèles les plus étudiés en recherche biologique. Il a été introduit par Sydney Brenner dans les années 1960, à l’origine pour comprendre le développement et la fonction du système nerveux29. Depuis lors, C. elegans est devenu un modèle puissant pour l’étude des processus fondamentaux dans toutes les disciplines biologiques, y compris la biologie comportementale, la neurobiologie, le vieillissement, la b...
Déclarations de divulgation
Nous déclarons que nous n’avons aucun conflit d’intérêts.
Remerciements
Nous reconnaissons le soutien financier de la Fondation allemande pour la science (projets A1.1 et A1.2 du Centre de recherche collaborative 1182 sur l’origine et la fonction des métaorganismes). Nous remercions les membres du laboratoire de Schulenburg pour leurs conseils et leur soutien.
matériels
| Name | Company | Catalog Number | Comments |
| COMSOL | COMSOL | multiphysics simulation software |
Références
- Schulenburg, H., Félix, M. -. A. The natural biotic environment of Caenorhabditis elegans. Genetics. 206 (1), 55-86 (2017).
- Petersen, C., Dirksen, P., Prahl, S., Strathmann, E. A., Schulenburg, H. The prevalence of Caenorhabditis elegans across 1.5 years in selected North German locations: the importance of substrate type, abiotic parameters, and Caenorhabditis competitors. BMC Ecology. 14 (1), 4 (2014).
- Petersen, C., et al. Travelling at a slug's pace: possible invertebrate vectors of Caenorhabditis nematodes. BMC Ecology. 15 (1), 19 (2015).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. -. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences. 113 (27), 3941-3949 (2016).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14 (1), 38 (2016).
- Johnke, J., Dirksen, P., Schulenburg, H. Community assembly of the native C. elegans microbiome is influenced by time, substrate and individual bacterial taxa. Environmental Microbiology. 22 (4), 1265-1279 (2020).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485 (2017).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3 Genes|Genomes|Genetics. 10 (9), 3025-3039 (2020).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2019).
- Kissoyan, K. A. B., et al. Exploring effects of C. elegans protective natural microbiota on host physiology. Frontiers in Cellular and Infection Microbiology. 12, 775728 (2022).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10 (1), 604 (2019).
- Kissoyan, K. A. B., et al. Natural C. elegans microbiota protects against infection via production of a cyclic lipopeptide of the viscosin group. Current Biology. 29 (6), 1030-1037 (2019).
- Watson, E., MacNeil, L. T., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Integration of metabolic and gene regulatory networks modulates the C. elegans dietary response. Cell. 153 (1), 253-266 (2013).
- Watson, E., et al. Interspecies systems biology uncovers metabolites affecting C. elegans gene expression and life history traits. Cell. 156 (4), 759-770 (2014).
- MacNeil, L. T., Watson, E., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Diet-induced developmental acceleration independent of TOR and insulin in C. elegans. Cell. 153 (1), 240-252 (2013).
- O'Donnell, M. P., Fox, B. W., Chao, P. -. H., Schroeder, F. C., Sengupta, P. A neurotransmitter produced by gut bacteria modulates host sensory behaviour. Nature. 583 (7816), 415-420 (2020).
- Snoek, B. L., et al. A multi-parent recombinant inbred line population of C. elegans allows identification of novel QTLs for complex life history traits. BMC Biology. 17 (1), 24 (2019).
- Avery, L., Shtonda, B. B. Food transport in the C. elegans pharynx. Journal of Experimental Biology. 206 (14), 2441-2457 (2003).
- Zhang, J., et al. A delicate balance between bacterial iron and reactive oxygen species supports optimal C. elegans development. Cell Host & Microbe. 26 (3), 400-411 (2019).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Félix, M. -. A., Duveau, F. Population dynamics and habitat sharing of natural populations of Caenorhabditis elegans and C. briggsae. BMC Biology. 10 (1), 59 (2012).
- Barrière, A., Félix, M. -. A. Temporal dynamics and linkage disequilibrium in natural Caenorhabditis elegans populations. Genetics. 176 (2), 999-1011 (2007).
- Dolgin, E. S., Félix, M. -. A., Cutter, A. D. Hakuna Nematoda: genetic and phenotypic diversity in African isolates of Caenorhabditis elegans and C. briggsae. Heredity. 100 (3), 304-315 (2008).
- Douglas, A. E. Simple animal models for microbiome research. Nature Reviews Microbiology. 17 (12), 764-775 (2019).
- Barrière, A., Félix, M. -. A. Isolation of C. elegans and related nematodes. WormBook. , 1-19 (2014).
- Weisburg, W. G., Barns, S. M., Pelletier, D. A., Lane, D. J. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 173 (2), 697-703 (1991).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77 (1), 71-94 (1974).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. Molecular Ecology. 31 (8), 2327-2347 (2022).
- Haber, M. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: Evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2004).
- Watson, E., et al. Metabolic network rewiring of propionate flux compensates vitamin B12 deficiency in C. elegans. eLife. 5, 17670 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon