Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Isolierung und Charakterisierung der natürlichen Mikrobiota des Modellnematoden Caenorhabditis elegans
In diesem Artikel
Zusammenfassung
Caenorhabditis elegans ist eine der wichtigsten Modellarten in der Biologie, aber fast alle Forschungen werden in Abwesenheit ihrer natürlich assoziierten Mikroben durchgeführt. Die hier beschriebenen Methoden werden dazu beitragen, die Vielfalt assoziierter Mikroben als Grundlage für zukünftige funktionelle C. elegans-Forschung besser zu verstehen.
Zusammenfassung
Der Fadenwurm Caenorhabditis elegans interagiert mit einer großen Vielfalt von Mikroorganismen in der Natur. Im Allgemeinen wird C. elegans häufig in faulem Pflanzenmaterial gefunden, insbesondere in faulen Früchten wie Äpfeln oder auf Komposthaufen. Es ist auch mit bestimmten wirbellosen Wirten wie Schnecken und Asseln verbunden. Diese Lebensräume sind reich an Mikroben, die als Nahrung für C. elegans dienen und auch den Nematodendarm persistent besiedeln können. Bis heute ist die genaue Vielfalt und Konsistenz der einheimischen C. elegans-Mikrobiota über Lebensräume und geografische Standorte hinweg nicht vollständig verstanden. Hier beschreiben wir einen geeigneten Ansatz, um C. elegans aus der Natur zu isolieren und die Mikrobiota von Würmern zu charakterisieren. Nematoden können leicht aus Kompostmaterial, verrottenden Äpfeln, Schnecken isoliert oder durch das Platzieren von Äpfeln auf Komposthaufen angezogen werden. Die Hauptzeit für den Fund von C. elegans in der nördlichen Hemisphäre ist von September bis November. Würmer können aus gesammeltem Substratmaterial ausgewaschen werden, indem das Substrat in Pufferlösung getaucht wird, gefolgt von der Sammlung von Nematoden und deren Transfer auf Nematodenwachstumsmedium oder PCR-Puffer zur anschließenden Analyse. Wir veranschaulichen weiterhin, wie die Proben verwendet werden können, um die wurmassoziierten Mikroorganismen zu isolieren und zu reinigen und Würmer für die 16S ribosomale RNA-Analyse der Zusammensetzung der Mikrobiota-Gemeinschaft zu verarbeiten. Insgesamt können die beschriebenen Methoden neue Forschungen zur Charakterisierung der C. elegans-Mikrobiota über Lebensräume und geografische Standorte hinweg anregen und so zu einem umfassenden Verständnis der Vielfalt und Stabilität der Mikrobiota des Fadenwurms als Grundlage für zukünftige funktionelle Forschung beitragen.
Einleitung
In der Natur wird C. elegans häufig in faulem Pflanzenmaterial gefunden, insbesondere in faulen Früchten wie Äpfeln oder auf Komposthaufen1. Es ist auch mit bestimmten wirbellosen Wirten wie Schnecken und Asseln assoziiert 2,3. Diese Lebensräume sind reich an Mikroben, die nicht nur als Nahrung für den Wurm dienen, sondern auch stabile Assoziationen mit ihm bilden können. Informationen zur Vielfalt natürlich assoziierter Mikroorganismen wurden erst 2016 veröffentlicht 4,5,6. Seitdem haben diese und nur wenige neuere Studien gezeigt, dass C. elegans mit einer Vielzahl von Bakterien und Pilzen assoziiert ist, am häufigsten einschließlich Bakterien der Gattung Pseudomonas, Enterobacter, Ochrobactrum, Erwinia, Comamonas, Gluconobacter und einige andere 6,7,8. Mehrere assoziierte Bakterien können den Wurmdarm stabil besiedeln, wenn auch nicht alle 6,9,10,11,12. Sie sind wahrscheinlich von zentraler Bedeutung für unser Verständnis der Biologie von C. elegans, da sie Nahrung liefern, vor Krankheitserregern und möglicherweise anderen Stressoren schützen und zentrale Merkmale der Lebensgeschichte wie Fortpflanzungsrate, Entwicklung oder Verhaltensreaktionen beeinflussen können.
Zum Beispiel können natürlich assoziierte Isolate der Gattungen Pseudomonas, Ochrobactrum und auch Enterobacter oder Gluconobacter den Wurm auf unterschiedliche Weise vor Erregerinfektion und -abtötung schützen 5,6,11,13,14. Ein spezifisches Isolat der Gattung Comamonas beeinflusst die Nahrungsreaktion, Entwicklung, Lebensdauer und Fruchtbarkeit von Nematoden15,16,17. Providencia-Bakterien produzieren den Neuromodulator Tyramin und modulieren dadurch die Aktivität des Nervensystems des Wirts und die daraus resultierenden Verhaltensreaktionen18. Es wurde gezeigt, dass eine Reihe verschiedener natürlich assoziierter Bakterien die Wachstumsrate, die Fruchtbarkeit und die Verhaltensreaktionen der Bevölkerung beeinflusst 5,6,9,11,19.
Bis heute ist die genaue Vielfalt und Konsistenz der einheimischen C. elegans-Mikrobiota über Lebensräume und geografische Standorte hinweg nicht vollständig verstanden, und weitere Assoziationen zwischen dem Wurm und Mikroben aus seiner Umgebung müssen noch aufgedeckt werden. Mehrere frühere Studien verwendeten Bakterienstämme, die aus einer Bodenumgebung, natürlichen C. elegans-Lebensräumen oder aus Mesokosmenexperimenten (dh Laborumgebungen, die natürliche Lebensräume nachbilden) mit C. elegans-Laborstämmen 4,5,20 isoliert wurden. Obwohl diese Studien neue Erkenntnisse über den Einfluss von Mikroben auf bestimmte Nematodenmerkmale (z.B. Nematodenstoffwechsel21) lieferten, ist die Relevanz dieser Interaktionen für die Biologie von C. elegans in der Natur unklar. Daher beschreibt dieses Manuskript die Methoden, C. elegans direkt aus der Natur zu isolieren und die natürlich assoziierten Mikroben sowohl aus einzelnen Würmern als auch aus Gruppen von Würmern zu isolieren und anschließend zu charakterisieren. Die beschriebenen Methoden sind eine aktualisierte und verbesserte Version der bisher verwendeten Verfahren zur Isolierung und Charakterisierung von natürlichem C. elegans und seiner nativen Mikrobiota 2,6,7. In Anbetracht der Tatsache, dass C. elegans bei der Zersetzung von Pflanzenmaterial auf der ganzen Welt weit verbreitet ist (insbesondere in verrottenden Früchten, gemäßigten Regionen und im Herbst)1,2,22,23,24,25, kann dieses Protokoll von jedem Labor angewendet werden, wann immer Interesse an einer Beziehung zu C. elegans besteht. Merkmale zu natürlich assoziierten Mikroben und damit zu einem natürlich relevanteren Kontext. Letzteres ist entscheidend für ein vollständiges Verständnis der Biologie des Fadenwurms, da aus einer Vielzahl anderer Wirtssysteme bekannt ist, dass die assoziierte Mikrobiota verschiedene Merkmale der Lebensgeschichte beeinflussen kann26, ein Aspekt, der derzeit in der Vielzahl von C. elegans-Studien in fast allen lebenswissenschaftlichen Disziplinen weitgehend vernachlässigt wird.
Protokoll
1. Vorbereitung von Puffern und Medien
- Der S-Puffer wird durch Zugabe von 5,85 g NaCl, 1,123 g K2HPO4, 5,926 g KH2PO4 und 1 l deionisiertemH2Oin einen Kolben und Autoklaven für 20 min bei 121 °C hergestellt.
- Bereiten Sie ein viskoses Medium durch Zugabe von S-Puffer vor, der 1,2% (w/v) Hydroxymethylcellulose (die Substanz, die die Viskosität des Mediums verursacht), 5 mg/ml Cholesterin, 1 mMMgSO4, 1 mM CaCl2 und 0,1% (v/v) Aceton enthält. Autoklavieren und rühren Sie das viskose Medium, bis es vollständig homogen ist.
HINWEIS: Dies kann mehrere Stunden dauern. Auch S-Puffer kann direkt hergestellt und dem viskosen Medium ohne vorherige Sterilisation zugesetzt werden. - Der M9-Puffer wird durch Zugabe von 3 g KH 2 PO 4, 6 g NA2 HPO4·2 H2O, 5 g NaCl und1 L deionisiertemH2Oin einen 1 L Kolben hergestellt. Autoklavieren Sie die Lösung und fügen Sie nach dem Abkühlen 1 ml 1 M MgSO4 hinzu (123,24 gMgSO4·7H2Oin 500 mL deionisiertemH2O, Filter sterilisiert).
- Bereiten Sie 10% (v/v) Triton X-100 Stammlösung vor, indem Sie 5 mL Triton X-100 mit 45 mL M9-Puffer mischen. Filtersterilisieren Sie die Lösung mit einem 0,2-μm-Filter.
- M9-Puffer mit Triton X-100 (M9-T) vorbereiten, indem 2,5 ml der 10%igen (v/v) Triton X-100 Stammlösung zu 1 l M9-Puffer nach dem Autoklavieren gegeben werden, um 0,025% (v/v) M9-T zu erhalten.
- Bereiten Sie 30% (v/v) Glycerin in S-Puffer vor, indem Sie 15 mL steriles 100% Glycerin und 35 ml steriles S-Puffer in einem 50 ml Röhrchen mischen.
2. Vorbereitung von Umweltproben (Abbildung 1)
- Sammeln Sie Umweltproben wie Kompost oder faule Früchte und legen Sie jede Probe in einen einzelnen Plastikbeutel, eine Tube oder einen anderen sauberen Behälter.
HINWEIS: Um Nematoden anzulocken, können Äpfel einige Wochen vor der Probenahme auf Kompost gelegt werden. - Teile der Umweltprobe gleichmäßig in einer leeren, sterilen 9 cm langen Petrischale verteilen.
HINWEIS: Proben mit höherem Zerfallsgrad enthalten eher C. elegans. Optional kann eine Petrischale verwendet werden, die mit peptonfreiem Agarmedium (PFM) gefüllt ist, um den Kontrast zu erhöhen. - Decken Sie die Probe vorsichtig mit ca. 20 ml sterilem viskosem Medium ab.
HINWEIS: Nematoden schweben innerhalb von 1-2 h an die Oberfläche. Das viskose Medium verlangsamt die Bewegung der Würmer und erleichtert die Probenahme. Alternativ kann ein steriler M9-Puffer verwendet werden, die Wurmbewegung ist jedoch schneller.
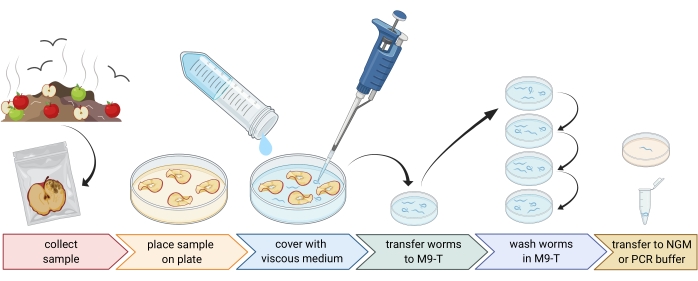
Abbildung 1: Isolierung von Nematoden aus Substraten. Substratproben werden in leere Petrischalen gegeben und mit viskosem Medium bedeckt, um Nematoden auszuspülen. Nematoden werden auf M9-T übertragen und wiederholt gewaschen, um Bakterien von außen zu entfernen. Einzelne Nematoden können zur DNA-Isolierung, Isolierung assoziierter Bakterien oder auf Agarplatten platziert werden, um Wurmpopulationen zu kultivieren. Figur erstellt mit BioRender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
3. Isolierung von Caenorhabditis-Nematoden (Abbildung 1)
- Suchen Sie mit einem Seziermikroskop nach Caenorhabditis-Nematoden , gemäß den Richtlinien im WormBook-Kapitel über die Isolierung von C. elegans und verwandten Nematoden von Barrière und Félix27.
HINWEIS: Caenorhabditis hermaphrodites / Weibchen haben einen Darm von hellbrauner Farbe (unter Durchlicht). Darüber hinaus haben die Darmzellen große Zellkerne, die als weiße Punkte sichtbar sind. Im Gegensatz dazu haben andere Nematodenarten oft einen dunkelbraunen Darm und können anteroposteriore Asymmetrie in der Pigmentintensität und normalerweise keine sichtbaren Kerne aufweisen. Die Vulva erwachsener Caenorhabditis Hermaphroditen/Weibchen befindet sich in der Mitte des Tieres. Diese Vulvalokalisation wird nicht immer von anderen Nematodentaxa gezeigt. Caenorhabditis-Nematoden besitzen zwei Rachenkolben, die beide mit ausreichender Vergrößerung und Durchlicht sichtbar sind und im Gegensatz zu vielen anderen Nematodentaxa stehen. Caenorhabditis-Nematoden haben einen spitzen Schwanz, während einige andere Nematoden einen runden Schwanz haben. - Sammeln Sie die Caenorhabditis-Nematoden unter einem Seziermikroskop in möglichst wenig Flüssigkeit mit einer 20-100 μL Pipette. Die gesammelten Nematoden werden direkt in 1-3 ml steriles M9-T in eine sterile 3 cm Petrischale überführt, um die Würmer zu waschen, um nicht anhaftende Mikroben zu entfernen (Schritt 3.3), oder alternativ einzelne Würmer auf eine Platte mit Nematodenwachstumsmedium (NGM) übertragen, um eine Wurmpopulation zu etablieren (Schritt 3.4).
- Waschen Sie die Nematoden, um Mikroben von der Außenseite des Nematoden zu entfernen.
HINWEIS: Dieses Protokoll bereichert Darmbakterien, eliminiert jedoch nicht vollständig Bakterien, die an der Wurmkutikula haften.- Inkubieren Sie die Nematoden für 10-15 min in M9-T.
- Pipettieren Sie die Nematoden in so wenig Flüssigkeit wie möglich in 1-3 mL frisches M9-T in einer neuen sterilen 3 cm Petrischale.
- Wiederholen Sie die Inkubation und übertragen Sie die Nematoden noch zwei weitere Male auf frisches M9-T.
HINWEIS: Die folgenden Schritte können entweder mit einzelnen Würmern oder mit Wurmpopulationen durchgeführt werden. Die Würmer können nun zur Charakterisierung von C. elegans und assoziierten Mikroben sowie zur Isolierung von Bakterien (Schritte 4 und 5) verwendet werden. Alternativ können sie einzeln auf NGM-Platten übertragen werden, um eine Wurmpopulation zu etablieren (Schritt 3.4).
- Um eine Wurmpopulation zu erhalten, pipetten Sie einen einzelnen Nematoden auf eine NGM-Platte.
HINWEIS: Nur zwittrige Nematoden wie C. elegans oder C . briggsae sind in der Lage, Nachkommen aus einzelnen Würmern zu zeugen. Einzelne Würmer anderer Arten können jedoch noch Nachkommen produzieren, wenn sie bereits gedeckt wurden.- Natürlich isolierte Nematoden enthalten normalerweise Bakterien in ihrem Darm, die sie auf die NGM-Platten abgeben, wo diese Bakterien wachsen und als Nahrung für C. elegans zur Verfügung stehen. Fügen Sie keine separaten Lebensmittelorganismen wie den Standard-Laborlebensmittelorganismus E. coli-Stamm OP50 hinzu.
HINWEIS: Die Aufzucht von Würmern auf Platten beeinflusst die Zusammensetzung der assoziierten Bakteriengemeinschaft, dennoch ist die Zusammensetzung vergleichbar mit der der natürlichen C. elegans-Isolate 6,7. - Lassen Sie die Nematoden bis zu 10 Tage bei der entsprechenden Temperatur (z. B. 15-20 °C für gemäßigte Standorte) vermehren. Frieren Sie diese Nematoden ein (Schritt 3.5) oder verwenden Sie sie für die Charakterisierung von C. elegans und assoziierten Mikroben sowie die Isolierung von Mikroben (Schritte 4 und 5).
- Natürlich isolierte Nematoden enthalten normalerweise Bakterien in ihrem Darm, die sie auf die NGM-Platten abgeben, wo diese Bakterien wachsen und als Nahrung für C. elegans zur Verfügung stehen. Fügen Sie keine separaten Lebensmittelorganismen wie den Standard-Laborlebensmittelorganismus E. coli-Stamm OP50 hinzu.
- Einfrieren von Nematoden für die Langzeitlagerung
- Lassen Sie die Nematoden auf Tellern, bis die Nahrungsbakterien verschwunden sind, und es gibt hauptsächlich kleine Larvenstadien auf den Tellern. Waschen Sie die Würmer in 1,5 ml S-Puffer von den Platten.
- Mischen Sie 500 μL wurmhaltigen S-Puffer gründlich mit 500 μL 30% (v/v) Glycerin in S-Puffer in einem sterilen 2 ml Röhrchen. Gefrieren Sie die Röhrchen umgehend bei -80 °C zur Langzeitlagerung, da sonst das Glycerin die Nematoden schädigen kann.
4. Vorbereitung der Würmer zur molekularen Identifizierung von C. elegans und Mikroben
- Für die unvoreingenommene Identifizierung von Nematoden-assoziierten Mikroben ist eine 96-Well-Platte mit drei sterilen 1-mm-Perlen, 19,5 μL PCR-Puffer und 0,5 μL Proteinase K (20 mg/ml) pro Vertiefung vorzubereiten. Pipettieren Sie einen einzelnen, gewaschenen Fadenwurm in jeden Brunnen und übertragen Sie so wenig Flüssigkeit wie möglich.
- Wurmpopulationen können ebenfalls verwendet werden. Dazu waschen Sie die Würmer von den Platten mit M9-Puffer und geben ~300 μL wurmhaltigen M9-Puffer in 2 mL Röhrchen mit 10-15 Perlen.
- Zerlegen Sie die Nematoden mit einem Perlenhomogenisator (z. B. Perlenschlagen für 3 min bei 30 Hz). Zentrifugieren Sie die Platte oder die Röhrchen kurz, um die Flüssigkeit auf den Boden zu bringen (z. B. für 10 s bei 8000 x g bei Raumtemperatur [RT]).
- Identifizierung von C. elegans
- Isolieren Sie die DNA einzelner Nematoden, indem Sie die Proben in einem PCR-Cycler für 1 h bei 50 °C und 15 min bei 95 °C erhitzen. Isolieren Sie die DNA von Wurmpopulationen mit einer beliebigen Isolationsmethode Ihrer Wahl (Beispielprotokolle verschiedener Isolationsmethoden mit kommerziellen Kits 7,9). Die DNA zur Langzeitlagerung bei -20 °C einfrieren.
- Für die Identifizierung von C. elegans verwenden Sie die DNA und das Primerpaar nlp30-F (Tabelle 1, 5'-ACACATACAACTGATCACTCA-3') und nlp30-R (Tabelle 1, 5'-TACTTTCCCCATCCGTATC-3') in einer PCR gemäß den Anweisungen eines Taq-Lieferanten Ihrer Wahl.
- Verwenden Sie die folgenden PCR-Bedingungen: erster Denaturierungsschritt bei 95 °C für 2 min, gefolgt von 35 Zyklen von 95 °C für 45 s, 55 °C für 30 s, 72 °C für 1 min und einem letzten Dehnungsschritt bei 72 °C für 5 min. elegans erzeugt ein 154 bp PCR-Produkt.
- Charakterisierung der Nematoden-assoziierten Bakterien mittels 16S-Amplikonsequenzierung der V3-V4-Region unter Verwendung der isolierten DNA.
- Bereiten Sie eine 16S-Bibliothek mit den 16S-Primern Ihrer Wahl vor und befolgen Sie das Protokoll des Bibliotheksvorbereitungskits. Eine Möglichkeit ist die Verwendung der Primer 341F (Tabelle 1, 5'-CCTACGGGNGGCWGCAG-3') und 806R (Tabelle 1, 5'-GACTACHVGGGTATCTAATCC-3'), die die V3-V4-Region des 16S-rRNA-Gens abdecken, was zu Sequenzen führt, die mit Standarddatenbanken mit guter Auflösung klassifiziert werden können7.
HINWEIS: Die Menge der Eingabe-DNA ist in diesem Schritt kritisch. DNA, die von einzelnen Würmern gewonnen wird, wird viel geringer sein als die von Wurmpopulationen. Bei einzelnen Würmern kann es notwendig sein, die Menge der Eingangs-DNA zu erhöhen oder die Anzahl der PCR-Zyklen zu erhöhen7. - Bibliotheken können auf einer Sequenzierungsplattform mit einem geeigneten Sequenzierungskit sequenziert werden.
HINWEIS: Hier wird die MiSeq-Plattform mit einem geeigneten MiSeq-Reagenzien-Kit verwendet. Die Reagenzien werden ständig verbessert und sollten nach den neuesten Standards ausgewählt werden.
- Bereiten Sie eine 16S-Bibliothek mit den 16S-Primern Ihrer Wahl vor und befolgen Sie das Protokoll des Bibliotheksvorbereitungskits. Eine Möglichkeit ist die Verwendung der Primer 341F (Tabelle 1, 5'-CCTACGGGNGGCWGCAG-3') und 806R (Tabelle 1, 5'-GACTACHVGGGTATCTAATCC-3'), die die V3-V4-Region des 16S-rRNA-Gens abdecken, was zu Sequenzen führt, die mit Standarddatenbanken mit guter Auflösung klassifiziert werden können7.
5. Isolierung und Kultivierung von Nematoden-assoziierten Bakterien (Abbildung 2)
- Um Bakterien zu isolieren, bereiten Sie eine 96-Well-Platte mit drei sterilen 1-mm-Perlen, 20 μL M9-Puffer pro Vertiefung vor und pipettieren Sie einen einzelnen, gewaschenen Nematoden in jede Vertiefung, wobei so wenig Flüssigkeit wie möglich übertragen wird.
HINWEIS: Alternativ können Wurmpopulationen mit M9-Puffer von den Platten gewaschen und ~300 μL wurmhaltiger M9-Puffer in 2 mL Röhrchen mit 10-15 Perlen überführt werden. In allen Fällen sollten die Würmer aus einer Kultur stammen, die anhand der Schritte 4.1-4.4 als C. elegans identifiziert wurde. - Zerlegen Sie die Nematoden mit einem Perlenhomogenisator (z. B. Perlenschlagen für 3 min bei 30 Hz). Zentrifugieren Sie die Platte oder die Röhrchen kurz, um die Flüssigkeit auf den Boden zu bringen (z. B. für 10 s bei 8000 x g RT).
HINWEIS: Die Verwendung dieser Methode führte zur Isolierung bakterieller Taxa, ähnlich denen, die durch die 16S rDNA-Amplikonsequenzierung7 aufgedeckt wurden, was darauf hindeutet, dass die beschriebene Perlenschlagung die meisten Bakterienzellen intakt lässt. - Sammeln Sie den Überstand, verdünnen Sie ihn seriell bei 1:10 und platen Sie bis zu 100 μL auf 9 cm Agarplatten.
- Um sicherzustellen, dass die meisten Bakterien kultiviert werden können, verwenden Sie eine Vielzahl alternativer Medien mit unterschiedlichen Nährstoffzusammensetzungen, einschließlich verdünntem Trypticase-Sojaagar (TSA, 1:10 Verdünnung), MacConkey-Agar, Sabouraud Glucose-Agar, Kartoffeldextrose-Agar oder Hefe-Pepton-Dextrose-Agar.
- Inkubieren Sie die Platten bei den durchschnittlichen Temperaturbedingungen des Probenahmeortes (z. B. Temperaturen zwischen 15-20 °C für gemäßigte Standorte) für 24-48 h.
- Verwenden Sie die Standard-Drei-Streifen-Technik und eine sterile Schleife, um reine Bakterienkulturen zu erhalten (Abbildung 2).
- Nehmen Sie eine einzelne Kolonie von einer Platte mit einer sterilen Schlaufe oder einem Zahnstocher und streifen Sie sie auf eine neue Agarplatte, die das gleiche Agarmedium enthält, das für die Reinigung verwendet wird. Stellen Sie sicher, dass Sie nur etwa 1/3 der Platte verwenden.
- Verwenden Sie entweder eine neue sterile Schlaufe oder sterilisieren Sie eine wiederverwendbare Schlaufe und ziehen Sie sie durch den ersten Streifen, um einen zweiten Streifen auf einem weiteren 1/3 derselben Platte zu erzeugen.
- Wiederholen Sie diesen Schritt, indem Sie eine sterile Schleife durch den zweiten Streifen ziehen.
- Inkubieren Sie die Platte unter den gleichen Wachstumsbedingungen, die für die Isolierung verwendet werden. Diese Technik muss dazu führen, dass einzelne Kolonien im Bereich des dritten Streifens wachsen.
HINWEIS: Es kann notwendig sein, den Reinigungsschritt mehrmals zu wiederholen, da natürliche Isolate dazu neigen, Biofilme und / oder Aggregate zu bilden.
- Züchten Sie die reinen Kolonien in einem flüssigen Medium (vom gleichen Typ wie das Agarmedium) mit der gleichen Temperatur und Wachstumszeit wie oben (Schritt 5.4)
- Bereiten Sie Bakterienbrühen vor, indem Sie 300 μL der Bakterienkultur zu 200 μL 86% (v/v) Glycerin (im jeweiligen Wachstumsmedium, z. B. TSB) geben und nach oben und unten pipettieren, um richtig zu mischen. Alternativ können DMSO-Brühen hergestellt werden, indem 50 μL Bakterienkultur mit 50 μL 7% (v/v) DMSO gemischt werden. Zur Langzeitlagerung bei -80 °C einfrieren.
- Charakterisierung von Bakterien durch Sequenzierung des vollständigen 16S ribosomalen RNA-Gens
- Extrahieren Sie die bakterielle DNA aus reinen flüssigen Kulturen mit einer geeigneten Technik (z. B. einem DNA-Extraktionskit; erfahrungsgemäß funktioniert ein CTAB-basiertes Extraktionsprotokoll sehr gut22).
- Amplifizieren Sie das 16S rRNA-Gen mit den Primern 27F (Tabelle 1, 5'-GAGAGTTTGATCCTGGCTCAG-3') und 1495R (Tabelle 1, 5'-CTACGGCTACCTTTACGA -3')28 und den folgenden PCR-Bedingungen: 95 °C, 2 min, 22x (95 °C, 30 s; 55 °C, 30 s; 72 °C, 100 s) und einer letzten Verlängerungsperiode bei 72 °C, 5 min.
- Um die vollständigen Sequenzen zu erfassen, verwenden Sie zusätzlich zwei interne Sequenzierungsprimer, wie 701F (Tabelle 1, 5'-GTGTAGCGTGAAATGCG-3') und 785R (Tabelle 1, 5'-GGATTAGATACCCTGGTAGTCC-3')6.
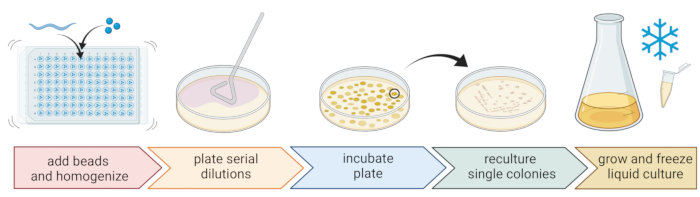
Abbildung 2: Artenbestimmung und Isolierung einzelner Bakterien. Einzelne Nematoden werden mit einem Perlenhomogenisator aufgebrochen und DNA zur Artbestimmung mittels PCR oder Sequenzierung isoliert. Alternativ wird das aufgebrochene Nematodenmaterial seriell verdünnt und auf Wachstumsmediumplatten plattiert. Platten werden inkubiert, bis Bakterienkolonien erscheinen, und einzelne Kolonien werden zu neuen Platten gestreift, um Reinkulturen zu erhalten. Aus einzelnen Kolonien der Reinkulturen werden flüssige Bakterienkulturen zur Vorbereitung von Bakterienbeständen für die Langzeitlagerung bei -80 °C gezüchtet. Figur erstellt mit BioRender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ergebnisse
Der Fadenwurm C. elegans findet sich häufig in zersetzenden Früchten wie Äpfeln und auch Kompostproben. In Norddeutschland kommen C. elegans sowie kongenerische Arten (insbesondere C. remanei, aber auch C. briggsae) hauptsächlich von September bis2. November vor. Die Nematoden werden am häufigsten in zersetzendem Pflanzenmaterial gefunden, insbesondere in verrottenden Früchten wie Äpfeln oder Birnen, aber auch in Kompost, insbesondere in Material, das ein...
Diskussion
Der Fadenwurm Caenorhabditis elegans ist einer der am intensivsten untersuchten Modellorganismen in der biologischen Forschung. Es wurde von Sydney Brenner in den 1960er Jahren eingeführt, ursprünglich zum Verständnis der Entwicklung und der Funktion des Nervensystems29. Seitdem ist C. elegans zu einem leistungsfähigen Modell für die Untersuchung grundlegender Prozesse in allen biologischen Disziplinen geworden, einschließlich Verhaltensbiologie, Neurobiologie, Altern, Evol...
Offenlegungen
Wir erklären, dass wir keinen Interessenkonflikt haben.
Danksagungen
Wir danken für die finanzielle Unterstützung durch die Deutsche Forschungsgemeinschaft (Projekte A1.1 und A1.2 des Sonderforschungsbereichs 1182 "Entstehung und Funktion von Metaorganismen"). Wir danken den Mitgliedern des Schulenburg-Labors für ihre Beratung und Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| COMSOL | COMSOL | multiphysics simulation software |
Referenzen
- Schulenburg, H., Félix, M. -. A. The natural biotic environment of Caenorhabditis elegans. Genetics. 206 (1), 55-86 (2017).
- Petersen, C., Dirksen, P., Prahl, S., Strathmann, E. A., Schulenburg, H. The prevalence of Caenorhabditis elegans across 1.5 years in selected North German locations: the importance of substrate type, abiotic parameters, and Caenorhabditis competitors. BMC Ecology. 14 (1), 4 (2014).
- Petersen, C., et al. Travelling at a slug's pace: possible invertebrate vectors of Caenorhabditis nematodes. BMC Ecology. 15 (1), 19 (2015).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. -. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences. 113 (27), 3941-3949 (2016).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14 (1), 38 (2016).
- Johnke, J., Dirksen, P., Schulenburg, H. Community assembly of the native C. elegans microbiome is influenced by time, substrate and individual bacterial taxa. Environmental Microbiology. 22 (4), 1265-1279 (2020).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485 (2017).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3 Genes|Genomes|Genetics. 10 (9), 3025-3039 (2020).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2019).
- Kissoyan, K. A. B., et al. Exploring effects of C. elegans protective natural microbiota on host physiology. Frontiers in Cellular and Infection Microbiology. 12, 775728 (2022).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10 (1), 604 (2019).
- Kissoyan, K. A. B., et al. Natural C. elegans microbiota protects against infection via production of a cyclic lipopeptide of the viscosin group. Current Biology. 29 (6), 1030-1037 (2019).
- Watson, E., MacNeil, L. T., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Integration of metabolic and gene regulatory networks modulates the C. elegans dietary response. Cell. 153 (1), 253-266 (2013).
- Watson, E., et al. Interspecies systems biology uncovers metabolites affecting C. elegans gene expression and life history traits. Cell. 156 (4), 759-770 (2014).
- MacNeil, L. T., Watson, E., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Diet-induced developmental acceleration independent of TOR and insulin in C. elegans. Cell. 153 (1), 240-252 (2013).
- O'Donnell, M. P., Fox, B. W., Chao, P. -. H., Schroeder, F. C., Sengupta, P. A neurotransmitter produced by gut bacteria modulates host sensory behaviour. Nature. 583 (7816), 415-420 (2020).
- Snoek, B. L., et al. A multi-parent recombinant inbred line population of C. elegans allows identification of novel QTLs for complex life history traits. BMC Biology. 17 (1), 24 (2019).
- Avery, L., Shtonda, B. B. Food transport in the C. elegans pharynx. Journal of Experimental Biology. 206 (14), 2441-2457 (2003).
- Zhang, J., et al. A delicate balance between bacterial iron and reactive oxygen species supports optimal C. elegans development. Cell Host & Microbe. 26 (3), 400-411 (2019).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Félix, M. -. A., Duveau, F. Population dynamics and habitat sharing of natural populations of Caenorhabditis elegans and C. briggsae. BMC Biology. 10 (1), 59 (2012).
- Barrière, A., Félix, M. -. A. Temporal dynamics and linkage disequilibrium in natural Caenorhabditis elegans populations. Genetics. 176 (2), 999-1011 (2007).
- Dolgin, E. S., Félix, M. -. A., Cutter, A. D. Hakuna Nematoda: genetic and phenotypic diversity in African isolates of Caenorhabditis elegans and C. briggsae. Heredity. 100 (3), 304-315 (2008).
- Douglas, A. E. Simple animal models for microbiome research. Nature Reviews Microbiology. 17 (12), 764-775 (2019).
- Barrière, A., Félix, M. -. A. Isolation of C. elegans and related nematodes. WormBook. , 1-19 (2014).
- Weisburg, W. G., Barns, S. M., Pelletier, D. A., Lane, D. J. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 173 (2), 697-703 (1991).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77 (1), 71-94 (1974).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. Molecular Ecology. 31 (8), 2327-2347 (2022).
- Haber, M. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: Evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2004).
- Watson, E., et al. Metabolic network rewiring of propionate flux compensates vitamin B12 deficiency in C. elegans. eLife. 5, 17670 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten