Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Model Nematod Caenorhabditis elegans'ın Doğal Mikrobiyotasının İzolasyonu ve Karakterizasyonu
Bu Makalede
Özet
Caenorhabditis elegans , biyolojideki ana model türlerden biridir, ancak neredeyse tüm araştırmalar doğal olarak ilişkili mikroplarının yokluğunda gerçekleştirilir. Burada açıklanan yöntemler, gelecekteki fonksiyonel C. elegans araştırmalarının temeli olarak ilişkili mikropların çeşitliliği hakkındaki anlayışımızı geliştirmeye yardımcı olacaktır.
Özet
Nematod Caenorhabditis elegans, doğada çok çeşitli mikroorganizmalarla etkileşime girer. Genel olarak, C. elegans genellikle çürümüş bitki maddesinde, özellikle elma gibi çürümüş meyvelerde veya kompost yığınlarında bulunur. Ayrıca sümüklü böcek ve woodlice gibi bazı omurgasız konakçılarla da ilişkilidir. Bu habitatlar, C. elegans için yiyecek görevi gören ve aynı zamanda nematod bağırsağını ısrarla kolonize edebilen mikroplar bakımından zengindir. Bugüne kadar, yerli C. elegans mikrobiyotasının habitatlar ve coğrafi konumlar arasındaki tam çeşitliliği ve tutarlılığı tam olarak anlaşılamamıştır. Burada, C. elegans'ı doğadan izole etmek ve solucanların mikrobiyotasını karakterize etmek için uygun bir yaklaşım açıklıyoruz. Nematodlar kompost malzemesinden, çürüyen elmalardan, sümüklü böceklerden kolayca izole edilebilir veya elmaları kompost yığınlarına yerleştirerek çekilebilir. Kuzey Yarımküre'de C. elegans'ı bulmak için en uygun zaman eylül ayından kasım ayına kadardır. Solucanlar, substratı tampon çözeltisine batırarak toplanan substrat malzemesinden yıkanabilir, ardından nematodların toplanması ve sonraki analizler için nematod büyüme ortamına veya PCR tamponuna aktarılması ile takip edilebilir. Ayrıca, örneklerin solucanla ilişkili mikroorganizmaları izole etmek ve saflaştırmak ve mikrobiyota topluluğu kompozisyonunun 16S ribozomal RNA analizi için solucanları işlemek için nasıl kullanılabileceğini gösteriyoruz. Genel olarak, açıklanan yöntemler, C. elegans mikrobiyotasının habitatlar ve coğrafi konumlar arasında karakterizasyonu üzerine yeni araştırmaları teşvik edebilir, böylece gelecekteki fonksiyonel araştırmalar için bir temel olarak nematodun mikrobiyotasının çeşitliliği ve stabilitesi hakkında kapsamlı bir anlayış elde edilmesine yardımcı olabilir.
Giriş
Doğada, C. elegans genellikle çürümüş bitki maddesinde, özellikle elma gibi çürümüş meyvelerde veya kompost yığınlarındabulunur 1. Ayrıca sümüklü böcek ve woodlice 2,3 gibi bazı omurgasız konakçılarla da ilişkilidir. Bu habitatlar, sadece solucan için yiyecek olarak hizmet etmekle kalmayıp, aynı zamanda onunla istikrarlı ilişkiler kurabilen mikroplar bakımından zengindir. Doğal olarak ilişkili mikroorganizmaların çeşitliliği ile ilgili bilgiler sadece 2016 yılında yayınlanmıştır 4,5,6. O zamandan beri, bu ve sadece birkaç yeni çalışma, C. elegans'ın, en yaygın olarak Pseudomonas, Enterobacter, Ochrobactrum, Erwinia, Comamonas, Gluconobacter ve diğerleri 6,7,8 cinsinin bakterilerini içeren çeşitli bakteri ve mantarlarla ilişkili olduğunu ortaya koymuştur. Birkaç ilişkili bakteri,solucan bağırsağını istikrarlı bir şekilde kolonize edebilir, ancak hepsi 6,9,10,11,12 değildir. C. elegans biyolojisini anlamamız için kilit öneme sahip olmaları muhtemeldir, çünkü beslenme sağlayabilir, patojenlere ve muhtemelen diğer stresörlere karşı koruyabilir ve üreme hızı, gelişim veya davranışsal tepkiler gibi merkezi yaşam öyküsü özelliklerini etkileyebilirler.
Örnek olarak, Pseudomonas, Ochrobactrum ve ayrıca Enterobacter veya Gluconobacter cinslerinin doğal olarak ilişkili izolatları, solucanı patojen enfeksiyonundan koruyabilir ve 5,6,11,13,14 farklı şekillerde öldürebilir. Comamonas cinsinin spesifik bir izolatı nematod diyet tepkisini, gelişimini, ömrünü ve doğurganlığı etkiler15,16,17. Providencia bakterileri nöromodülatör tiramin üretir ve böylece konakçı sinir sistemi aktivitesini ve bunun sonucunda ortaya çıkan davranışsal tepkileri modüle eder18. Doğal olarak ilişkili bir dizi farklı bakterinin nüfus artış hızını, doğurganlığı ve davranışsal tepkileri etkilediği gösterilmiştir 5,6,9,11,19.
Bugüne kadar, yerli C. elegans mikrobiyotasının habitatlar ve coğrafi konumlar arasındaki tam çeşitliliği ve tutarlılığı tam olarak anlaşılamamıştır ve solucan ile çevresinden mikroplar arasındaki diğer ilişkiler ortaya çıkarılmaya devam etmektedir. Önceki birkaç çalışmada, bazı toprak ortamlarından, doğal C. elegans habitatlarından veya mezokozmos deneylerinden (yani, doğal yaşam alanlarını yeniden yaratan laboratuvar tabanlı ortamlar) izole edilen bakteri suşları C. elegans laboratuvar suşları 4,5,20 ile kullanılmıştır. Bu çalışmalar, mikropların spesifik nematod özellikleri (örneğin, nematod metabolizması21) üzerindeki etkisine dair yeni bilgiler edinmiş olsa da, bu etkileşimlerin C. elegans biyolojisi ile doğadaki ilgisi belirsizdir. Bu nedenle, bu el yazması C. elegans'ı doğadan doğrudan izole etme ve doğal olarak ilişkili mikropları hem tek solucanlardan hem de solucan gruplarından izole etme ve daha sonra karakterize etme yöntemlerini açıklamaktadır. Açıklanan yöntemler, daha önce doğal C. elegans ve doğal mikrobiyotası 2,6,7'nin izolasyonu ve karakterizasyonu için kullanılan prosedürlerin güncellenmiş ve geliştirilmiş bir versiyonudur. C. elegans'ın dünya çapında bitki maddesinin ayrışmasında yaygın olarak bulunduğu göz önüne alındığında (özellikle çürüyen meyvelerde, ılıman bölgelerde ve sonbaharda)1,2,22,23,24,25, bu protokol, C. elegans ile ilgili ilgiye ilgi duyulduğunda herhangi bir laboratuvar tarafından uygulanabilir. doğal olarak ilişkili mikroplara ve dolayısıyla daha doğal olarak alakalı bir bağlama sahip olma özellikleri. İkincisi, nematodun biyolojisinin tam olarak anlaşılması için çok önemlidir, çünkü diğer konakçı sistemlerin çeşitliliğinden, ilişkili mikrobiyotanın çeşitli yaşam tarihi özelliklerini etkileyebileceği bilinmektedir26, şu anda neredeyse tüm yaşam bilimleri disiplinlerinde çok sayıda C. elegans çalışmasında büyük ölçüde ihmal edilen bir yön.
Protokol
1. Tamponların ve ortamların hazırlanması
- 121 ° C'de 20 dakika boyunca bir şişeye ve otoklavlara 5,85 g NaCl, 1,123g K2HPO 4, 5,926 g KH 2 PO4 ve 1 L deiyonize H2O ekleyerek S-tamponunu hazırlayın.
- % 1.2 (w / v) hidroksimetilselüloz (ortamın viskozitesine neden olan madde), 5 mg / mL kolesterol, 1 mM MgSO4, 1 mM CaCl2 ve% 0.1 (v / v) aseton içeren S-tamponu ekleyerek viskoz bir ortam hazırlayın. Otoklav yapın ve viskoz ortamı tamamen homojen olana kadar karıştırın.
NOT: Bu işlem birkaç saat sürebilir. Ayrıca, S-tamponu önceden sterilizasyon yapılmadan doğrudan hazırlanabilir ve viskoz ortama eklenebilir. - 1 L şişeye 3 g KH 2 PO 4, 6 g NA 2 HPO4 · 2 H2O, 5 g NaCl ve 1 L deiyonize H2O ekleyerek M9 tamponunu hazırlayın. Çözeltiyi otoklav edin ve soğuduktan sonra, 1 mL'lik 1 M MgSO 4 ekleyin (500 mL deiyonize H2 O'da123.24g MgSO4 · 7H2O, filtre sterilize).
- 5 mL Triton X-100'ü 45 mL M9 tamponuyla karıştırarak %10 (v/v) Triton X-100 stok çözeltisi hazırlayın. 0,2 μm'lik bir filtre kullanarak çözeltiyi filtreleyerek sterilize edin.
- %0,025 (v/v) M9-T elde etmek için otoklavlamadan sonra 1 L M9 tamponuna %10 (v/v) Triton X-100 stok çözeltisinin 2,5 mL'sini ekleyerek Triton X-100 (M9-T) ile M9 tamponunu hazırlayın.
- 50 mL'lik bir tüpte 15 mL steril% 100 gliserol ve 35 mL steril S-tamponu karıştırarak S-tamponunda% 30 (v / v) gliserol hazırlayın.
2. Çevre örneklerinin hazırlanması (Şekil 1)
- Kompost veya çürük meyveler gibi çevresel örnekleri toplayın ve her numuneyi ayrı bir plastik torbaya, tüpe veya başka bir temiz kaba yerleştirin.
NOT: Nematodları çekmek için, elmalar örneklemeden birkaç hafta önce kompost üzerine yerleştirilebilir. - Çevresel numunenin parçalarını boş, steril, 9 cm'lik bir Petri kabına eşit şekilde yayın.
NOT: Daha yüksek bozunma seviyelerine sahip numunelerin C. elegans içerme olasılığı daha yüksektir. İsteğe bağlı olarak, kontrastı artırmak için pepton içermeyen agar ortamı (PFM) ile doldurulmuş bir Petri kabı kullanılabilir. - Numuneyi yaklaşık 20 mL steril viskoz ortam ile dikkatlice örtün.
NOT: Nematodlar 1-2 saat içinde yüzeye çıkar. Viskoz ortam, solucanların hareketini yavaşlatır ve örneklemeyi kolaylaştırır. Alternatif olarak steril M9-tampon kullanılabilir, ancak solucan hareketi daha hızlı olacaktır.
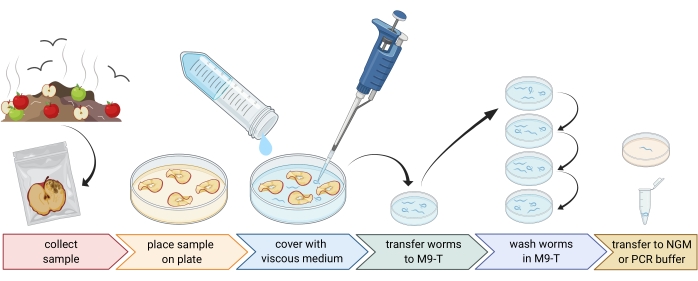
Şekil 1: Nematodların substratlardan izolasyonu. Substrat numuneleri boş Petri kaplarına yerleştirilir ve nematodları temizlemek için viskoz ortamla kaplanır. Nematodlar M9-T'ye aktarılır ve bakterileri dışarıdan uzaklaştırmak için tekrar tekrar yıkanır. Bireysel nematodlar DNA izolasyonu, ilişkili bakterilerin izolasyonu için kullanılabilir veya kültür solucanı popülasyonlarına agar plakalarına yerleştirilebilir. BioRender.com ile oluşturulan figür. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
3. Caenorhabditis nematodlarının izolasyonu (Şekil 1)
- Barrière ve Félix27 tarafından C. elegans ve ilgili nematodların izolasyonu ile ilgili WormBook bölümünde sunulan yönergeleri izleyerek, diseksiyon mikroskobu kullanarak Caenorhabditis nematodlarını araştırın.
NOT: Caenorhabditis hermafroditleri / dişileri açık kahverengi renkte bir bağırsağa sahiptir (iletilen aydınlatma altında). Dahası, bağırsak hücreleri, beyaz noktalar olarak görülebilen büyük hücre çekirdeklerine sahiptir. Buna karşılık, diğer nematod türleri genellikle koyu kahverengi bir bağırsağa sahiptir ve pigment yoğunluğunda anteroposterior asimetri gösterebilir ve genellikle görünür çekirdekler göstermeyebilir. Yetişkin Caenorhabditis hermafroditlerinin / dişilerinin vulvası hayvanın ortasında bulunur. Bu vulva lokalizasyonu her zaman diğer nematod taksonları tarafından gösterilmez. Caenorhabditis nematodları, hem yeterli büyütme hem de iletilen aydınlatma ve diğer birçok nematod taksonu ile kontrast ile görülebilen iki faringeal ampule sahiptir. Caenorhabditis nematodları sivri bir kuyruğa sahipken, diğer bazı nematodların yuvarlak bir kuyruğu vardır. - Caenorhabditis nematodlarını diseksiyon mikroskobu altında 20-100 μL'lik bir pipet kullanarak mümkün olduğunca az sıvı içinde toplayın. Toplanan nematodları, bağlanmamış mikropları çıkarmak için solucanları yıkamak için steril bir 3 cm Petri kabında doğrudan 1-3 mL steril M9-T'ye aktarın (adım 3.3) veya alternatif olarak, bir solucan popülasyonu oluşturmak için bireysel solucanları nematod büyüme ortamı (NGM) içeren bir tabağa aktarın (adım 3.4).
- Nematodun dışından mikropları çıkarmak için nematodları yıkayın.
NOT: Bu protokol bağırsak bakterilerini zenginleştirir, ancak solucan kütikülüne yapışan bakterileri tamamen ortadan kaldırmaz.- Nematodları M9-T'de 10-15 dakika boyunca inkübe edin.
- Nematodları mümkün olduğunca az sıvı içinde pipetle 1-3 mL taze M9-T içine yeni steril 3 cm'lik Petri kabında yerleştirin.
- Kuluçkayı tekrarlayın ve nematodları iki kez daha taze M9-T'ye aktarın.
NOT: Aşağıdaki adımlar tek tek solucanlarla veya solucan popülasyonlarıyla yapılabilir. Solucanlar artık C. elegans ve ilişkili mikropların karakterizasyonu ve bakterilerin izolasyonu için kullanılabilir (adım 4 ve 5). Alternatif olarak, bir solucan popülasyonu oluşturmak için ayrı ayrı NGM plakalarına aktarılabilirler (adım 3.4).
- Bir solucan popülasyonu elde etmek için, bireysel bir nematodu bir NGM plakasına pipetleyin.
NOT: Sadece C. elegans veya C. briggsae gibi hermafroditik nematodlar tek solucanlardan yavru üretebilir. Bununla birlikte, diğer türlerin tek solucanları, zaten çiftleşmiş olsalar bile hala yavru üretebilirler.- Doğal olarak izole edilmiş nematodlar genellikle bağırsaklarında bakteri içerir ve bu bakterilerin büyüdüğü ve C. elegans için yiyecek olarak bulunduğu NGM plakalarına dökülürler. Standart laboratuvar gıda organizması E. coli suşu OP50 gibi ayrı gıda organizmaları eklemeyin.
NOT: Solucanların plakalar üzerinde yetiştirilmesi, ilişkili bakteri topluluğunun bileşimini etkiler, ancak bileşim hala doğal C. elegans izolatları 6,7 ile karşılaştırılabilir. - Nematodların uygun sıcaklıkta 10 güne kadar çoğalmasına izin verin (örneğin, ılıman yerler için 15-20 ° C). Bu nematodları dondurun (adım 3.5) veya C. elegans ve ilişkili mikropların karakterizasyonu ve mikropların izolasyonu için kullanın (adım 4 ve 5).
- Doğal olarak izole edilmiş nematodlar genellikle bağırsaklarında bakteri içerir ve bu bakterilerin büyüdüğü ve C. elegans için yiyecek olarak bulunduğu NGM plakalarına dökülürler. Standart laboratuvar gıda organizması E. coli suşu OP50 gibi ayrı gıda organizmaları eklemeyin.
- Uzun süreli depolama için dondurucu nematodlar
- Gıda bakterileri kaybolana kadar nematodları tabaklarda bırakın ve plakalarda çoğunlukla küçük larva aşamaları vardır. Solucanları plakalardan 1,5 mL S-tamponunda yıkayın.
- 500 μL solucan içeren S-tamponunu, steril 2 mL'lik bir tüp içinde S-tamponunda 500 μL% 30 (v / v) gliserol ile iyice karıştırın. Uzun süreli depolama için tüpleri derhal -80 ° C'de dondurun, aksi takdirde gliserol nematodlara zarar verebilir.
4. C. elegans ve mikropların moleküler tanımlanması için solucanların hazırlanması
- Nematodla ilişkili mikropların tarafsız bir şekilde tanımlanması için, üç steril 1 mm boncuk, 19.5 μL PCR tamponu ve kuyucuk başına 0.5 μL Proteinaz K (20 mg / mL) içeren 96 delikli bir plaka hazırlayın. Pipet bir birey, yıkanmış nematodun her bir oyuğa mümkün olduğunca az sıvı aktarmasıdır.
- Solucan popülasyonları da kullanılabilir. Bunun için, solucanları plakalardan M9 tamponu ile yıkayın ve ~ 300 μL solucan içeren M9 tamponunu 10-15 boncuklu 2 mL tüplere aktarın.
- Bir boncuk homojenizatörü kullanarak nematodları parçalayın (örneğin, 30 Hz'de 3 dakika boyunca boncuk çırpma). Sıvıyı tabana ulaştırmak için plakayı veya tüpleri kısaca santrifüj edin (örneğin, oda sıcaklığında [RT] 8000 x g'de 10 s için).
- C. elegans'ın tanımlanması
- Numuneleri bir PCR siklerindeki 50 ° C'de 1 saat ve 95 ° C'de 15 dakika ısıtarak bireysel nematodların DNA'sını izole edin. Solucan popülasyonlarının DNA'sını, tercih edilen herhangi bir izolasyon yöntemini kullanarak izole edin (ticari kitleri kullanan farklı izolasyon yöntemlerinin örnek protokolleri 7,9). Uzun süreli depolama için DNA'yı -20 ° C'de dondurun.
- C. elegans'ın tanımlanması için, DNA'yı ve nlp30-F primer çiftini (Tablo 1, 5'-ACACATACAACTGATCACTCA-3') ve nlp30-R (Tablo 1, 5'-TACTTTCCCCATCCGTATC-3') bir PCR'de, tercih edilen bir Taq tedarikçisinin talimatlarını izleyerek kullanın.
- Aşağıdaki PCR koşullarını kullanın: 2 dakika boyunca 95 ° C'de ilk denatürasyon adımı, ardından 45 s için 95 ° C'lik 35 döngü, 30 s için 55 ° C, 1 dakika için 72 ° C ve 5 dakika boyunca 72 ° C'de son bir uzama adımı.
- İzole DNA'yı kullanarak V3-V4 bölgesinin 16S amplikon dizilimi yoluyla nematod ile ilişkili bakterileri karakterize edin.
- Tercih edilen 16S astarlarla bir 16S kütüphanesi hazırlayın ve kütüphane hazırlama kiti protokolünü takip edin. Bir seçenek, 16S rRNA geninin V3-V4 bölgesini kapsayan 341F (Tablo 1, 5'-CCTACGGGNGGCWGCAG-3') ve 806R (Tablo 1, 5'-GACTACHVGGGTATCTAATCC-3') primerlerini kullanmaktır, bu da iyi çözünürlük7 olan standart veritabanlarıyla sınıflandırılabilen dizilerle sonuçlanır.
NOT: Bu adımda girdi DNA'sının miktarı kritik öneme sahiptir. Tek solucanlardan elde edilen DNA, solucan popülasyonlarından elde edilenden çok daha az olacaktır. Tek solucanlar için, girdi DNA miktarını artırmak veya PCR döngülerinin miktarını artırmakgerekebilir 7. - Kütüphaneler, uygun bir sıralama kiti kullanılarak bir sıralama platformunda sıralanabilir.
NOT: Burada, MiSeq platformu uygun bir MiSeq Reaktif Kiti ile birlikte kullanılır. Reaktifler sürekli olarak geliştirilir ve en yeni standartlara göre seçilmelidir.
- Tercih edilen 16S astarlarla bir 16S kütüphanesi hazırlayın ve kütüphane hazırlama kiti protokolünü takip edin. Bir seçenek, 16S rRNA geninin V3-V4 bölgesini kapsayan 341F (Tablo 1, 5'-CCTACGGGNGGCWGCAG-3') ve 806R (Tablo 1, 5'-GACTACHVGGGTATCTAATCC-3') primerlerini kullanmaktır, bu da iyi çözünürlük7 olan standart veritabanlarıyla sınıflandırılabilen dizilerle sonuçlanır.
5. Nematodla ilişkili bakterilerin izolasyonu ve yetiştirilmesi (Şekil 2)
- Bakterileri izole etmek için, üç steril 1 mm boncuk, kuyu başına 20 μL M9 tamponu içeren 96 delikli bir plaka hazırlayın ve her bir kuyucuğa ayrı bir yıkanmış nematod pipetin, mümkün olduğunca az sıvı aktarın.
NOT: Alternatif olarak, solucan popülasyonları plakalardan M9 tamponu ile yıkanabilir ve ~ 300 μL solucan içeren M9 tamponu, 10-15 boncuklu 2 mL tüplere aktarılabilir. Her durumda, solucanlar 4.1-4.4 adımları kullanılarak C. elegans olarak tanımlanan bir kültürden gelmelidir. - Bir boncuk homojenizatörü kullanarak nematodları parçalayın (örneğin, 30 Hz'de 3 dakika boyunca boncuk çırpma). Sıvıyı tabana almak için plakayı veya tüpleri kısaca santrifüj edin (örneğin, RT'de 8000 x g'de 10 s için).
NOT: Bu yöntemin kullanımı, 16S rDNA amplikon dizilimi7 tarafından ortaya çıkarılanlara benzer bakteri taksonlarının izolasyonuna yol açmıştır, bu da tarif edilen boncuk çırpmanın çoğu bakteri hücresini sağlam bıraktığını düşündürmektedir. - Süper natantı toplayın, 1:10'da seri olarak seyreltin ve 9 cm agar plakalarına 100 μL'ye kadar tabaklayın.
- Çoğu bakterinin yetiştirilebilmesini sağlamak için, seyreltilmiş triptikaz soya agar (TSA, 1:10 seyreltme), MacConkey agar, Sabouraud glikoz agar, patates dekstroz agar veya maya pepton dekstroz agar dahil olmak üzere farklı besin bileşimlerine sahip çeşitli alternatif ortamlar kullanın.
- Plakaları, numune alma yerinin ortalama sıcaklık koşullarında (örneğin, ılıman yerler için 15-20 °C arasındaki sıcaklıklar) 24-48 saat boyunca inkübe edin.
- Saf bakteri kültürleri elde etmek için standart üç çizgili tekniği ve steril bir döngüyü kullanın (Şekil 2).
- Steril bir halka veya kürdan kullanarak bir plakadan tek bir koloni seçin ve saflaştırma için kullanılanla aynı agar ortamını içeren yeni bir agar plakasına çizin. Plakanın sadece kabaca 1 / 3'ünü kullandığınızdan emin olun.
- Ya yeni bir steril döngü kullanın ya da yeniden kullanılabilir bir döngüyü sterilize edin ve aynı plakanın başka bir 1 / 3'ünde ikinci bir çizgi oluşturmak için ilk çizgi boyunca sürükleyin.
- Steril bir döngüyü ikinci çizgi boyunca sürükleyerek bu adımı tekrarlayın.
- Plakayı izolasyon için kullanılan aynı büyüme koşullarında inkübe edin. Bu teknik, üçüncü çizgi alanında büyüyen tek kolonilerle sonuçlanmalıdır.
NOT: Doğal izolatlar biyofilmler ve / veya agregalar oluşturma eğiliminde olduğundan saflaştırma adımını birkaç kez tekrarlamak gerekebilir.
- Saf kolonileri, yukarıdakiyle aynı sıcaklığı ve büyüme süresini kullanarak sıvı bir ortamda (agar ortamı ile aynı tipte) büyütün (adım 5.4)
- Bakteri kültürünün 300 μL'sini 200 μL% 86 (v / v) gliserol (ilgili büyüme ortamında, örneğin TSB) ve pipete ekleyerek bakteri stoklarını hazırlayın. Alternatif olarak, 50 μL bakteri kültürünü 50 μL% 7 (v / v) DMSO ile karıştırarak DMSO stoklarını hazırlayın. Uzun süreli depolama için -80 °C'de dondurun.
- Tam 16S ribozomal RNA geninin dizilimini kullanarak bakterileri karakterize etme
- Bakteriyel DNA'yı uygun bir teknik kullanarak saf sıvı kültürlerden çıkarın (örneğin, bir DNA ekstraksiyon kiti; deneyimlere göre, CTAB tabanlı bir ekstraksiyon protokolü çok iyi çalışır22).
- 27F (Tablo 1, 5'-GAGAGTTTGATCCTGGCTCAG-3') ve 1495R (Tablo 1, 5'-CTACGGCTCTTTGTTACGA -3')28 primerlerini ve aşağıdaki PCR koşullarını kullanarak 16S rRNA genini yükseltin: 95 °C, 2 dakika, 22x (95 °C, 30 s; 55 °C, 30 s; 72 °C, 100 s) ve 72 °C, 5 dk'da son uzatma periyodu.
- Tam dizileri elde etmek için, ayrıca 701F (Tablo 1, 5'-GTGTAGCGTGAAATGCG-3') ve 785R (Tablo 1, 5'-GGATTAGATACCCTGGTAGTCC-3')6 gibi iki dahili sıralama astarı kullanın.
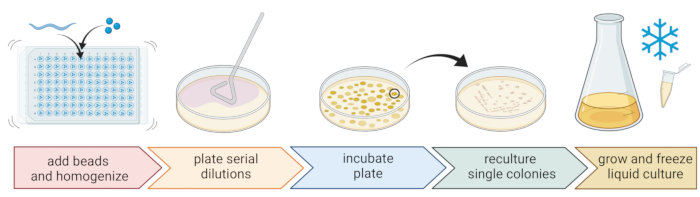
Şekil 2: Türlerin tanımlanması ve bireysel bakterilerin izolasyonu. Bireysel nematodlar bir boncuk homojenizatörü kullanılarak parçalanır ve DNA, PCR veya dizileme yoluyla tür tayini için izole edilir. Alternatif olarak, parçalanmış nematod malzemesi seri olarak seyreltilir ve büyüme ortamı plakaları üzerine kaplanır. Bakteri kolonileri ortaya çıkana kadar plakalar inkübe edilir ve saf kültürler elde etmek için tek koloniler yeni plakalara dizilir. Saf kültürlerin tek kolonileri, -80 ° C'de uzun süreli depolama için bakteri stoklarının hazırlanması için sıvı bakteri kültürlerinin yetiştirilmesinde kullanılır. BioRender.com ile oluşturulan figür. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Sonuçlar
Nematod C. elegans sıklıkla elma gibi meyvelerin ayrışmasında ve ayrıca kompost örneklerinde bulunur. Kuzey Almanya'da, C. elegans ve konjenerik türler (özellikle C. remanei ve aynı zamanda C. briggsae) çoğunlukla Eylülayından 2 Kasım'a kadar bulunur. Nematodlar en yaygın olarak bitki maddesinin, özellikle elma veya armut gibi çürüyen meyvelerin ayrışmasında ve ayrıca kompostta, özellikle de yüksek derecede ayrışma gösteren malzemele...
Tartışmalar
Nematod Caenorhabditis elegans, biyolojik araştırmalarda en yoğun çalışılan model organizmalardan biridir. Sydney Brenner tarafından 1960'larda, başlangıçta sinir sisteminin gelişimini ve işlevini anlamak için tanıtıldı29. O zamandan beri, C. elegans davranışsal biyoloji, nörobiyoloji, yaşlanma, evrimsel biyoloji, hücre biyolojisi, gelişim biyolojisi ve immünoloji dahil olmak üzere tüm biyolojik disiplinlerdeki temel süreçleri incelemek için güçlü ...
Açıklamalar
Çıkar çatışmamız olmadığını beyan ederiz.
Teşekkürler
Alman Bilim Vakfı'nın mali desteğini kabul ediyoruz (Metaorganizmaların Kökeni ve İşlevi üzerine İşbirlikçi Araştırma Merkezi 1182'nin A1.1 ve A1.2 projeleri). Schulenburg laboratuvarı üyelerine tavsiye ve destekleri için teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| COMSOL | COMSOL | multiphysics simulation software |
Referanslar
- Schulenburg, H., Félix, M. -. A. The natural biotic environment of Caenorhabditis elegans. Genetics. 206 (1), 55-86 (2017).
- Petersen, C., Dirksen, P., Prahl, S., Strathmann, E. A., Schulenburg, H. The prevalence of Caenorhabditis elegans across 1.5 years in selected North German locations: the importance of substrate type, abiotic parameters, and Caenorhabditis competitors. BMC Ecology. 14 (1), 4 (2014).
- Petersen, C., et al. Travelling at a slug's pace: possible invertebrate vectors of Caenorhabditis nematodes. BMC Ecology. 15 (1), 19 (2015).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. -. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences. 113 (27), 3941-3949 (2016).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14 (1), 38 (2016).
- Johnke, J., Dirksen, P., Schulenburg, H. Community assembly of the native C. elegans microbiome is influenced by time, substrate and individual bacterial taxa. Environmental Microbiology. 22 (4), 1265-1279 (2020).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485 (2017).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3 Genes|Genomes|Genetics. 10 (9), 3025-3039 (2020).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2019).
- Kissoyan, K. A. B., et al. Exploring effects of C. elegans protective natural microbiota on host physiology. Frontiers in Cellular and Infection Microbiology. 12, 775728 (2022).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10 (1), 604 (2019).
- Kissoyan, K. A. B., et al. Natural C. elegans microbiota protects against infection via production of a cyclic lipopeptide of the viscosin group. Current Biology. 29 (6), 1030-1037 (2019).
- Watson, E., MacNeil, L. T., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Integration of metabolic and gene regulatory networks modulates the C. elegans dietary response. Cell. 153 (1), 253-266 (2013).
- Watson, E., et al. Interspecies systems biology uncovers metabolites affecting C. elegans gene expression and life history traits. Cell. 156 (4), 759-770 (2014).
- MacNeil, L. T., Watson, E., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Diet-induced developmental acceleration independent of TOR and insulin in C. elegans. Cell. 153 (1), 240-252 (2013).
- O'Donnell, M. P., Fox, B. W., Chao, P. -. H., Schroeder, F. C., Sengupta, P. A neurotransmitter produced by gut bacteria modulates host sensory behaviour. Nature. 583 (7816), 415-420 (2020).
- Snoek, B. L., et al. A multi-parent recombinant inbred line population of C. elegans allows identification of novel QTLs for complex life history traits. BMC Biology. 17 (1), 24 (2019).
- Avery, L., Shtonda, B. B. Food transport in the C. elegans pharynx. Journal of Experimental Biology. 206 (14), 2441-2457 (2003).
- Zhang, J., et al. A delicate balance between bacterial iron and reactive oxygen species supports optimal C. elegans development. Cell Host & Microbe. 26 (3), 400-411 (2019).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Félix, M. -. A., Duveau, F. Population dynamics and habitat sharing of natural populations of Caenorhabditis elegans and C. briggsae. BMC Biology. 10 (1), 59 (2012).
- Barrière, A., Félix, M. -. A. Temporal dynamics and linkage disequilibrium in natural Caenorhabditis elegans populations. Genetics. 176 (2), 999-1011 (2007).
- Dolgin, E. S., Félix, M. -. A., Cutter, A. D. Hakuna Nematoda: genetic and phenotypic diversity in African isolates of Caenorhabditis elegans and C. briggsae. Heredity. 100 (3), 304-315 (2008).
- Douglas, A. E. Simple animal models for microbiome research. Nature Reviews Microbiology. 17 (12), 764-775 (2019).
- Barrière, A., Félix, M. -. A. Isolation of C. elegans and related nematodes. WormBook. , 1-19 (2014).
- Weisburg, W. G., Barns, S. M., Pelletier, D. A., Lane, D. J. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 173 (2), 697-703 (1991).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77 (1), 71-94 (1974).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. Molecular Ecology. 31 (8), 2327-2347 (2022).
- Haber, M. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: Evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2004).
- Watson, E., et al. Metabolic network rewiring of propionate flux compensates vitamin B12 deficiency in C. elegans. eLife. 5, 17670 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır