É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Isolamento e Caracterização da Microbiota Natural do Nematoide Modelo Caenorhabditis elegans
Neste Artigo
Resumo
Caenorhabditis elegans é uma das principais espécies modelo em biologia, mas quase todas as pesquisas são realizadas na ausência de seus micróbios naturalmente associados. Os métodos descritos aqui ajudarão a melhorar nossa compreensão da diversidade de micróbios associados como base para futuras pesquisas funcionais de C. elegans.
Resumo
O nematoide Caenorhabditis elegans interage com uma grande diversidade de microrganismos na natureza. Em geral, C. elegans é comumente encontrado em matéria vegetal podre, especialmente frutas podres como maçãs ou em pilhas de compostagem. Também está associada a certos hospedeiros invertebrados, como lesmas e piolhos. Esses habitats são ricos em micróbios, que servem de alimento para C. elegans e que também podem colonizar persistentemente o intestino dos nematoides. Até o momento, a diversidade exata e a consistência da microbiota nativa de C. elegans em habitats e localizações geográficas não são totalmente compreendidas. Aqui, descrevemos uma abordagem adequada para isolar C. elegans da natureza e caracterizar a microbiota de vermes. Os nematoides podem ser facilmente isolados do material de compostagem, maçãs podres, lesmas ou atraídos pela colocação de maçãs em pilhas de compostagem. O horário nobre para encontrar C. elegans no Hemisfério Norte é de setembro a novembro. Os vermes podem ser lavados do material de substrato coletado por imersão do substrato em solução tampão, seguida pela coleta de nematoides e sua transferência para o meio de crescimento de nematoides ou tampão de PCR para análise subsequente. Ilustramos ainda como as amostras podem ser usadas para isolar e purificar os microrganismos associados a vermes e para processar vermes para análise de RNA ribossômico 16S da composição da comunidade de microbiota. No geral, os métodos descritos podem estimular novas pesquisas sobre a caracterização da microbiota de C. elegans em habitats e localizações geográficas, ajudando assim a obter uma compreensão abrangente da diversidade e estabilidade da microbiota do nematoide como base para futuras pesquisas funcionais.
Introdução
Na natureza, C. elegans é comumente encontrada em matéria vegetal podre, especialmente frutas podres como maçãs ou em pilhas de composto1. Também está associada a certos hospedeiros invertebrados, como lesmas e piolhos 2,3. Esses habitats são ricos em micróbios, que não só servem de alimento para o verme, mas também podem formar associações estáveis com ele. As informações sobre a diversidade de microrganismos naturalmente associados só foram publicadas em 2016 4,5,6. Desde então, esses e apenas alguns estudos mais recentes revelaram que C. elegans está associada a uma variedade de bactérias e fungos, mais comumente incluindo bactérias do gênero Pseudomonas, Enterobacter, Ochrobactrum, Erwinia, Comamonas, Gluconobacter e várias outras 6,7,8. Várias bactérias associadas podem colonizar de forma estável o intestino do verme, embora nem todas 6,9,10,11,12. É provável que eles sejam de importância fundamental para a nossa compreensão da biologia de C. elegans porque podem fornecer nutrição, proteger contra patógenos e possivelmente outros estressores e afetar características centrais da história de vida, como taxa reprodutiva, desenvolvimento ou respostas comportamentais.
Como exemplo, isolados naturalmente associados dos gêneros Pseudomonas, Ochrobactrum, e também Enterobacter ou Gluconobacter podem proteger o verme da infecção por patógenos e matar de maneiras distintas 5,6,11,13,14. Um isolado específico do gênero Comamonas influencia a resposta dietética, o desenvolvimento, a expectativa de vida e a fertilidade dos nematoides15,16,17. As bactérias de Providencia produzem o neuromodulador tiramina e, assim, modulam a atividade do sistema nervoso do hospedeiro e as respostas comportamentais resultantes18. Um conjunto de diferentes bactérias naturalmente associadas demonstrou afetar a taxa de crescimento populacional, a fertilidade e as respostas comportamentais 5,6,9,11,19.
Até o momento, a diversidade exata e a consistência da microbiota nativa de C. elegans em habitats e localizações geográficas não são totalmente compreendidas, e outras associações entre o verme e os micróbios de seu ambiente ainda precisam ser descobertas. Vários estudos anteriores utilizaram cepas bacterianas isoladas de algum ambiente de solo, habitats naturais de C. elegans ou de experimentos de mesocosmo (ou seja, ambientes baseados em laboratório que recriam habitats naturais) com cepas de laboratório de C. elegans 4,5,20. Embora esses estudos tenham obtido novos insights sobre a influência de micróbios em características específicas de nematoides (por exemplo, metabolismo de nematoides21), a relevância dessas interações para a biologia de C. elegans na natureza não é clara. Portanto, este manuscrito descreve os métodos para isolar diretamente C. elegans da natureza e para isolar e, posteriormente, caracterizar os micróbios naturalmente associados de ambos os vermes únicos e grupos de vermes. Os métodos descritos são uma versão atualizada e aprimorada dos procedimentos utilizados anteriormente para o isolamento e caracterização de C. elegans natural e sua microbiota nativa 2,6,7. Considerando que C. elegans é amplamente encontrada em matéria vegetal em decomposição em todo o mundo (especialmente em frutos em decomposição, regiões temperadas e no outono)1,22,22,23,24,25, esse protocolo pode ser aplicado por qualquer laboratório sempre que houver interesse em relacionar C. elegans características de micróbios naturalmente associados e, portanto, um contexto mais naturalmente relevante. Este último é fundamental para uma compreensão completa da biologia do nematoide, porque é sabido a partir de uma diversidade de outros sistemas hospedeiros que a microbiota associada pode afetar diversas características da história de vida26, um aspecto que atualmente é amplamente negligenciado na infinidade de estudos de C. elegans em quase todas as disciplinas de ciências da vida.
Protocolo
1. Preparação de buffers e meios
- Preparar o tampão S adicionando 5,85 g de NaCl, 1,123 g de K 2 HPO 4, 5,926 g de KH 2 PO4 e 1 L de H 2O deionizado a um balão e a uma autoclave durante 20 minutos a 121 °C.
- Prepare um meio viscoso adicionando tampão S contendo 1,2% (p/v) de hidroximetilcelulose (a substância causadora da viscosidade do meio), 5 mg/mL de colesterol, 1 mM de MgSO4, 1 mM de CaCl2 e 0,1% (v/v) de acetona. Autoclave e mexa o meio viscoso até que fique completamente homogêneo.
Observação : isso pode levar várias horas. Além disso, o tampão S pode ser preparado diretamente e adicionado ao meio viscoso sem esterilização prévia. - Preparar o tampão M9 adicionando 3 g de KH 2 PO 4, 6 g de NA 2 HPO4·2 H 2 O, 5 g de NaCl e 1 L de H2O deionizado a um balão de 1 L. Autoclave a solução e, após resfriamento, adicione 1 mL de 1 M MgSO 4 (123,24 g de MgSO4·7H 2 O em 500 mL de H2O deionizado, filtro esterilizado).
- Preparar a solução de reserva Triton X-100 a 10% (v/v) misturando 5 ml de Triton X-100 com 45 ml de tampão M9. Filtrar-esterilizar a solução utilizando um filtro de 0,2 μm.
- Preparar o tampão M9 com Triton X-100 (M9-T) adicionando 2,5 mL da solução-mãe Triton X-100 a 10% (v/v) a 1 L de tampão M9 após autoclave para obter M9-T a 0,025% (v/v).
- Prepare 30% (v/v) de glicerol em tampão S misturando 15 mL de glicerol estéril a 100% e 35 mL de tampão S estéril em um tubo de 50 mL.
2. Preparação de amostras ambientais (Figura 1)
- Colete amostras ambientais, como composto ou frutas podres, e coloque cada amostra em um saco plástico, tubo ou outro recipiente limpo individual.
NOTA: Para atrair nematoides, as maçãs podem ser colocadas em composto várias semanas antes da amostragem. - Espalhe pedaços da amostra ambiental uniformemente em uma placa de Petri vazia, estéril e de 9 cm.
NOTA: Amostras com níveis mais elevados de decaimento são mais propensas a conter C. elegans. Opcionalmente, uma placa de Petri preenchida com meio de ágar livre de peptona (PFM) pode ser usada para aumentar o contraste. - Cobrir cuidadosamente a amostra com aproximadamente 20 ml de meio viscoso estéril.
NOTA: Os nemátodos flutuam para a superfície dentro de 1-2 h. O meio viscoso retarda o movimento dos vermes e facilita a amostragem. O buffer M9 estéril pode ser usado como uma alternativa, no entanto, o movimento do worm será mais rápido.
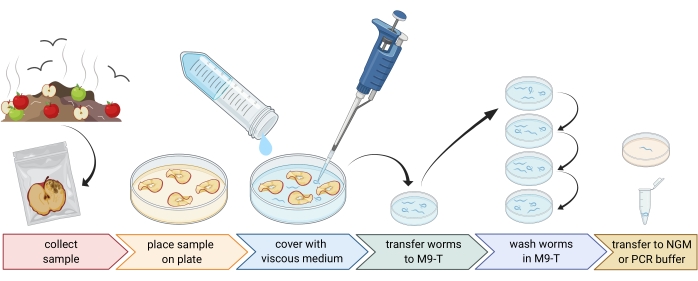
Figura 1: Isolamento de nematoides de substratos. As amostras de substrato são colocadas em placas de Petri vazias e cobertas com meio viscoso para eliminar os nematoides. Os nematoides são transferidos para M9-T e repetidamente lavados para remover bactérias do exterior. Nematoides individuais podem ser usados para isolamento de DNA, isolamento de bactérias associadas ou colocados em placas de ágar para cultivar populações de vermes. Figura criada com BioRender.com. Por favor, clique aqui para ver uma versão maior desta figura.
3. Isolamento dos nematoides de Caenorhabditis (Figura 1)
- Pesquisa de nematoides de Caenorhabditis utilizando microscópio dissecante, seguindo as diretrizes apresentadas no capítulo do WormBook sobre o isolamento de C. elegans e nematoides relacionados por Barrière e Félix27.
NOTA: Caenorhabditis hermafroditas/fêmeas têm um intestino de cor castanha clara (sob iluminação transmitida). Além disso, as células intestinais têm grandes núcleos celulares, que são visíveis como pontos brancos. Por outro lado, outras espécies de nematoides geralmente têm um intestino marrom escuro e podem mostrar assimetria anteroposterior na intensidade do pigmento e geralmente sem núcleos visíveis. A vulva de Caenorhabditis hermafroditas/fêmeas adultas é encontrada no meio do animal. Esta localização da vulva nem sempre é mostrada por outros táxons de nematoides. Os nematoides de Caenorhabditis possuem dois bulbos faríngeos, que são visíveis com ampliação suficiente e iluminação e contraste transmitidos com muitos outros táxons de nematoides. Os nematoides de Caenorhabditis têm uma cauda pontiaguda, enquanto alguns outros nematoides têm uma cauda redonda. - Recolher os nemátodos de Caenorhabditis sob um microscópio de dissecação com o mínimo de líquido possível utilizando uma pipeta de 20-100 μL. Transfira os nematoides coletados diretamente para 1-3 mL de M9-T estéril em uma placa de Petri estéril de 3 cm para lavar os vermes para remover micróbios não ligados (etapa 3.3) ou, alternativamente, transferir vermes individuais para uma placa com meio de crescimento de nematoides (NGM) para estabelecer uma população de vermes (etapa 3.4).
- Lave os nematoides para remover micróbios do lado de fora do nematoide.
NOTA: Este protocolo enriquece para as bactérias intestinais, mas não elimina completamente as bactérias que aderem à cutícula do verme.- Incubar os nematoides por 10-15 min em M9-T.
- Pipetar os nematoides com o mínimo de líquido possível em 1-3 mL de M9-T fresco em uma nova placa de Petri estéril de 3 cm.
- Repita a incubação e transfira os nematoides para M9-T fresco mais duas vezes.
Observação : as etapas a seguir podem ser feitas com worms individuais ou com populações de worms. Os vermes podem agora ser utilizados para a caracterização de C. elegans e micróbios associados, bem como para o isolamento de bactérias (passos 4 e 5). Alternativamente, eles podem ser transferidos individualmente para placas de NGM para estabelecer uma população de vermes (etapa 3.4).
- Para obter uma população de vermes, pipete um nematoide individual para uma placa NGM.
NOTA: Somente nematoides hermafroditas, como C. elegans ou C. briggsae, são capazes de produzir descendentes de vermes únicos. No entanto, vermes solteiros de outras espécies ainda podem produzir descendentes se já foram acasalados.- Nematoides naturalmente isolados geralmente contêm bactérias em seu intestino, que eles derramam nas placas de NGM, onde essas bactérias crescem e estão disponíveis como alimento para C. elegans. Não adicione organismos alimentares separados como o organismo alimentar de laboratório padrão, a estirpe OP50 de E. coli.
NOTA: A criação de vermes em placas influencia a composição da comunidade bacteriana associada, mas a composição ainda é comparável à dos isolados naturais de C. elegans 6,7. - Permita que os nematoides se proliferem por até 10 dias na temperatura apropriada (por exemplo, 15-20 °C para locais temperados). Congelar esses nematoides (etapa 3.5) ou usá-los para a caracterização de C. elegans e micróbios associados, bem como o isolamento de micróbios (etapas 4 e 5).
- Nematoides naturalmente isolados geralmente contêm bactérias em seu intestino, que eles derramam nas placas de NGM, onde essas bactérias crescem e estão disponíveis como alimento para C. elegans. Não adicione organismos alimentares separados como o organismo alimentar de laboratório padrão, a estirpe OP50 de E. coli.
- Nematoides congelados para armazenamento a longo prazo
- Deixe os nematoides em pratos até que as bactérias alimentares tenham desaparecido, e haja principalmente pequenos estágios larvais nos pratos. Lave os vermes das placas em 1,5 mL de tampão S.
- Misture bem 500 μL de tampão S contendo vermes com 500 μL de glicerol a 30% (v/v) em tampão S em um tubo estéril de 2 mL. Congele os tubos prontamente a -80 °C para armazenamento a longo prazo, caso contrário, o glicerol pode prejudicar os nemátodos.
4. Preparação dos vermes para identificação molecular de C. elegans e micróbios
- Para identificação imparcial de micróbios associados a nematoides, prepare uma placa de 96 poços com três esferas estéreis de 1 mm, 19,5 μL de tampão PCR e 0,5 μL de proteinase K (20 mg/mL) por poço. Pipetar um nematoide individual, lavado para cada poço, transferindo o mínimo de líquido possível.
- Populações de vermes também podem ser usadas. Para isso, lave os vermes das placas com tampão M9 e transfira ~300 μL de tampão M9 contendo vermes para tubos de 2 mL com 10-15 contas.
- Quebre os nematoides usando um homogeneizador de contas (por exemplo, batida de contas por 3 minutos a 30 Hz). Centrifugar a placa ou os tubos brevemente para levar o líquido ao fundo (por exemplo, por exemplo, por 10 s a 8000 x g à temperatura ambiente [RT]).
- Identificação de C. elegans
- Isolar o ADN de nemátodos individuais aquecendo as amostras num ciclador de PCR durante 1 h a 50 °C e 15 min a 95 °C. Isolar o DNA de populações de vermes usando qualquer método de isolamento de escolha (protocolos de exemplo de diferentes métodos de isolamento usando kits comerciais 7,9). Congelar o ADN a -20 °C para armazenamento a longo prazo.
- Para a identificação de C. elegans , utilizar o ADN e o par de iniciadores nlp30-F (Tabela 1, 5'-ACACATACAACTGATCACTCA-3') e nlp30-R (Quadro 1, 5'-TACTTTCCCCATCCGTATC-3') numa PCR, seguindo as instruções de um fornecedor de Taq de eleição.
- Use as seguintes condições de PCR: etapa inicial de desnaturação a 95 °C por 2 min, seguida por 35 ciclos de 95 °C por 45 s, 55 °C por 30 s, 72 °C por 1 min e uma etapa final de alongamento a 72 °C por 5 min. C. elegans produz um produto de PCR de 154 pb.
- Caracterizar as bactérias associadas a nematoides via sequenciamento amplicon 16S da região V3-V4, utilizando o DNA isolado.
- Prepare uma biblioteca 16S com os primers 16S de escolha e siga o protocolo do kit de preparação da biblioteca. Uma opção é utilizar os primers 341F (Tabela 1, 5'-CCTACGGGNGGCWGCAG-3') e 806R (Tabela 1, 5'-GACTACHVGGGTATCTAATCC-3') cobrindo a região V3-V4 do gene 16S rRNA, o que resulta em sequências que podem ser classificadas com bancos de dados padrão com boa resolução7.
NOTA: A quantidade de DNA de entrada é crítica nesta etapa. O DNA adquirido de vermes individuais será muito menor do que o obtido de populações de vermes. Para vermes solteiros, pode ser necessário aumentar a quantidade de DNA de entrada ou aumentar a quantidade de ciclos de PCR7. - As bibliotecas podem ser sequenciadas em uma plataforma de sequenciamento usando um kit de sequenciamento adequado.
NOTA: Aqui, a plataforma MiSeq é usada com um Kit de Reagente MiSeq adequado. Os reagentes são melhorados constantemente e devem ser escolhidos para os mais novos padrões.
- Prepare uma biblioteca 16S com os primers 16S de escolha e siga o protocolo do kit de preparação da biblioteca. Uma opção é utilizar os primers 341F (Tabela 1, 5'-CCTACGGGNGGCWGCAG-3') e 806R (Tabela 1, 5'-GACTACHVGGGTATCTAATCC-3') cobrindo a região V3-V4 do gene 16S rRNA, o que resulta em sequências que podem ser classificadas com bancos de dados padrão com boa resolução7.
5. Isolamento e cultivo de bactérias associadas a nematoides (Figura 2)
- Para isolar as bactérias, prepare uma placa de 96 poços com três contas estéreis de 1 mm, 20 μL de tampão M9 por poço e pipete um nematoide lavado individual para cada poço, transferindo o mínimo de líquido possível.
NOTA: Alternativamente, as populações de vermes podem ser lavadas das placas com tampão M9 e ~300 μL de tampão M9 contendo vermes podem ser transferidos para tubos de 2 mL com 10-15 contas. Em todos os casos, os vermes devem vir de uma cultura que foi identificada como C. elegans usando as etapas 4.1-4.4. - Quebre os nematoides usando um homogeneizador de contas (por exemplo, batida de contas por 3 minutos a 30 Hz). Centrifugar a placa ou os tubos brevemente para levar o líquido ao fundo (por exemplo, por exemplo, por 10 s a 8000 x g em RT).
NOTA: O uso deste método levou ao isolamento de táxons bacterianos semelhantes aos revelados pelo sequenciamento7 do 16S rDNA amplicon, sugerindo que o batimento de contas descrito deixa a maioria das células bacterianas intactas. - Recolher o sobrenadante, diluí-lo em série à 1:10 e colocar em placa até 100 μL em placas de ágar de 9 cm.
- Para garantir que a maioria das bactérias possa ser cultivada, use uma variedade de meios alternativos com diferentes composições de nutrientes, incluindo ágar soja tripticase diluído (TSA, diluição 1:10), ágar MacConkey, ágar glicose Sabouraud, ágar batata-dextrose ou ágar peptona dextrose levedura.
- Incubar as placas nas condições de temperatura média do local de amostragem (por exemplo, temperaturas entre 15-20 °C para locais temperados) por 24-48 h.
- Utilizar a técnica padrão de três estrias e uma alça estéril para obter culturas bacterianas puras (Figura 2).
- Escolha uma única colônia de uma placa usando um laço estéril ou palito de dente e coloque-a em uma nova placa de ágar contendo o mesmo meio de ágar usado para purificação. Certifique-se de usar apenas cerca de 1/3 da placa.
- Use um novo laço estéril ou esterilize um laço reutilizável e arraste-o pela primeira raia para criar uma segunda sequência em outro 1/3 da mesma placa.
- Repita esta etapa arrastando um laço estéril pela segunda faixa.
- Incubar a placa nas mesmas condições de crescimento utilizadas para o isolamento. Esta técnica deve resultar em colônias únicas crescendo na área da terceira faixa.
NOTA: Pode ser necessário repetir a etapa de purificação várias vezes, pois os isolados naturais tendem a formar biofilmes e/ou agregados.
- Cultivar as colônias puras em um meio líquido (do mesmo tipo que o meio ágar) usando a mesma temperatura e tempo de crescimento acima (etapa 5.4)
- Prepare os estoques de bactérias adicionando 300 μL da cultura bacteriana a 200 μL de 86% (v/v) de glicerol (no respectivo meio de crescimento, por exemplo, TSB) e pipeta para cima e para baixo para misturar adequadamente. Alternativamente, prepare os estoques de DMSO misturando 50 μL de cultura bacteriana com 50 μL de 7% (v/v) de DMSO. Congelar a -80 °C para armazenamento a longo prazo.
- Caracterização de bactérias usando o sequenciamento do gene completo do RNA ribossômico 16S
- Extraia o DNA bacteriano de culturas líquidas puras usando uma técnica adequada (por exemplo, um kit de extração de DNA; por experiência, um protocolo de extração baseado em CTAB funciona muito bem22).
- Amplificar o gene 16S rRNA utilizando os primers 27F (Tabela 1, 5'-GAGAGTTTGATCCTGGCTCAG-3') e 1495R (Tabela 1, 5'-CTACGGCTACCTTGTTACGA -3')28 e as seguintes condições de PCR: 95 °C, 2 min, 22x (95 °C, 30 s; 55 °C, 30 s; 72 °C, 100 s), e um período de extensão final a 72 °C, 5 min.
- Para adquirir as sequências completas, use adicionalmente dois primers de sequenciamento internos, como 701F (Tabela 1, 5'-GTGTAGCGGTGAAATGCG-3') e 785R (Tabela 1, 5'-GGATTAGATACCCTGGTAGTCC-3')6.
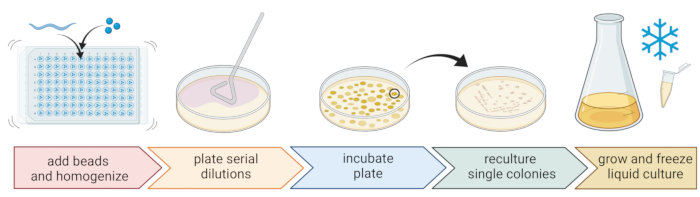
Figura 2: Identificação de espécies e isolamento de bactérias individuais. Nematoides individuais são quebrados usando um homogeneizador de contas, e o DNA é isolado para determinação de espécies via PCR ou sequenciamento. Alternativamente, o material de nematoide quebrado é diluído em série e chapeado em placas de meio de crescimento. As placas são incubadas até que as colônias bacterianas apareçam, e as colônias únicas são riscadas em novas placas para obter culturas puras. Colônias únicas das culturas puras são usadas para cultivar culturas bacterianas líquidas para a preparação de estoques bacterianos para armazenamento a longo prazo a -80 °C. Figura criada com BioRender.com. Por favor, clique aqui para ver uma versão maior desta figura.
Resultados
O nematoide C. elegans é frequentemente encontrado em frutas em decomposição, como maçãs, e também amostras de compostagem. No norte da Alemanha, C. elegans, bem como espécies congenéricas (particularmente C. remanei, mas também C. briggsae) são encontradas principalmente de setembro a2 de novembro. Os nematoides são mais comumente encontrados em matéria vegetal em decomposição, especialmente frutas podres, como maçãs ou peras, e também composto...
Discussão
O nematoide Caenorhabditis elegans é um dos organismos modelo mais intensamente estudados em pesquisas biológicas. Foi introduzido por Sydney Brenner na década de 1960, originalmente para entender o desenvolvimento e a função do sistema nervoso29. Desde então, C. elegans tornou-se um modelo poderoso para estudar processos fundamentais em todas as disciplinas biológicas, incluindo biologia comportamental, neurobiologia, envelhecimento, biologia evolutiva, biologia celular, ...
Divulgações
Declaramos que não temos conflito de interesses.
Agradecimentos
Reconhecemos o apoio financeiro da Fundação Alemã de Ciência (projetos A1.1 e A1.2 do Centro de Pesquisa Colaborativa 1182 sobre a Origem e Função dos Metaorganismos). Agradecemos aos membros do laboratório Schulenburg por seus conselhos e apoio.
Materiais
| Name | Company | Catalog Number | Comments |
| COMSOL | COMSOL | multiphysics simulation software |
Referências
- Schulenburg, H., Félix, M. -. A. The natural biotic environment of Caenorhabditis elegans. Genetics. 206 (1), 55-86 (2017).
- Petersen, C., Dirksen, P., Prahl, S., Strathmann, E. A., Schulenburg, H. The prevalence of Caenorhabditis elegans across 1.5 years in selected North German locations: the importance of substrate type, abiotic parameters, and Caenorhabditis competitors. BMC Ecology. 14 (1), 4 (2014).
- Petersen, C., et al. Travelling at a slug's pace: possible invertebrate vectors of Caenorhabditis nematodes. BMC Ecology. 15 (1), 19 (2015).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. -. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences. 113 (27), 3941-3949 (2016).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14 (1), 38 (2016).
- Johnke, J., Dirksen, P., Schulenburg, H. Community assembly of the native C. elegans microbiome is influenced by time, substrate and individual bacterial taxa. Environmental Microbiology. 22 (4), 1265-1279 (2020).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485 (2017).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3 Genes|Genomes|Genetics. 10 (9), 3025-3039 (2020).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2019).
- Kissoyan, K. A. B., et al. Exploring effects of C. elegans protective natural microbiota on host physiology. Frontiers in Cellular and Infection Microbiology. 12, 775728 (2022).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10 (1), 604 (2019).
- Kissoyan, K. A. B., et al. Natural C. elegans microbiota protects against infection via production of a cyclic lipopeptide of the viscosin group. Current Biology. 29 (6), 1030-1037 (2019).
- Watson, E., MacNeil, L. T., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Integration of metabolic and gene regulatory networks modulates the C. elegans dietary response. Cell. 153 (1), 253-266 (2013).
- Watson, E., et al. Interspecies systems biology uncovers metabolites affecting C. elegans gene expression and life history traits. Cell. 156 (4), 759-770 (2014).
- MacNeil, L. T., Watson, E., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Diet-induced developmental acceleration independent of TOR and insulin in C. elegans. Cell. 153 (1), 240-252 (2013).
- O'Donnell, M. P., Fox, B. W., Chao, P. -. H., Schroeder, F. C., Sengupta, P. A neurotransmitter produced by gut bacteria modulates host sensory behaviour. Nature. 583 (7816), 415-420 (2020).
- Snoek, B. L., et al. A multi-parent recombinant inbred line population of C. elegans allows identification of novel QTLs for complex life history traits. BMC Biology. 17 (1), 24 (2019).
- Avery, L., Shtonda, B. B. Food transport in the C. elegans pharynx. Journal of Experimental Biology. 206 (14), 2441-2457 (2003).
- Zhang, J., et al. A delicate balance between bacterial iron and reactive oxygen species supports optimal C. elegans development. Cell Host & Microbe. 26 (3), 400-411 (2019).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Félix, M. -. A., Duveau, F. Population dynamics and habitat sharing of natural populations of Caenorhabditis elegans and C. briggsae. BMC Biology. 10 (1), 59 (2012).
- Barrière, A., Félix, M. -. A. Temporal dynamics and linkage disequilibrium in natural Caenorhabditis elegans populations. Genetics. 176 (2), 999-1011 (2007).
- Dolgin, E. S., Félix, M. -. A., Cutter, A. D. Hakuna Nematoda: genetic and phenotypic diversity in African isolates of Caenorhabditis elegans and C. briggsae. Heredity. 100 (3), 304-315 (2008).
- Douglas, A. E. Simple animal models for microbiome research. Nature Reviews Microbiology. 17 (12), 764-775 (2019).
- Barrière, A., Félix, M. -. A. Isolation of C. elegans and related nematodes. WormBook. , 1-19 (2014).
- Weisburg, W. G., Barns, S. M., Pelletier, D. A., Lane, D. J. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 173 (2), 697-703 (1991).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77 (1), 71-94 (1974).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. Molecular Ecology. 31 (8), 2327-2347 (2022).
- Haber, M. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: Evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2004).
- Watson, E., et al. Metabolic network rewiring of propionate flux compensates vitamin B12 deficiency in C. elegans. eLife. 5, 17670 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados