Method Article
מיקרוסקופיה ברזולוציה גבוהה במיוחד של קומפלקס סינפטונמלי בתוך נבט Caenorhabditis elegans
In This Article
Summary
מיקרוסקופיה ברזולוציה גבוהה יכולה לספק תובנה מפורטת על ארגון הרכיבים בתוך קומפלקס סינפטונמלי במיוזיס. כאן אנו מדגימים פרוטוקול לפתרון חלבונים בודדים של קומפלקס סינפטונמלי Caenorhabditis elegans .
Abstract
במהלך מיוזה, כרומוזומים הומולוגיים חייבים לזהות זה את זה ולהיצמד זה לזה כדי לאפשר הפרדה נכונה שלהם. אחד מאירועי המפתח שמבטיחים את האינטראקציה של כרומוזומים הומולוגיים הוא הרכבת הקומפלקס הסינפטונמלי (SC) בפרופאזה מיוטית I. אף על פי שאין כמעט הומולוגיה של רצף בין רכיבי חלבונים בתוך ה-SC בין מינים שונים, המבנה הכללי של ה-SC נשמר מאוד במהלך האבולוציה. במיקרוגרפים של אלקטרונים, ה-SC מופיע כמבנה משולש, דמוי סולם, המורכב מיסודות או צירים רוחביים, חוטים רוחביים ויסוד מרכזי.
עם זאת, זיהוי מדויק של לוקליזציה של רכיבים בודדים בתוך המתחם על ידי מיקרוסקופיית אלקטרונים כדי לקבוע את המבנה המולקולרי של SC נותר מאתגר. לעומת זאת, מיקרוסקופיה פלואורסצנטית מאפשרת זיהוי של רכיבי חלבון בודדים בתוך המכלול. עם זאת, מכיוון שרוחב ה-SC הוא ~100 ננומטר בלבד, לא ניתן לפתור את תת-המבנה שלו על ידי מיקרוסקופיה פלואורסצנטית קונבנציונלית מוגבלת עקיפה. לפיכך, קביעת הארכיטקטורה המולקולרית של ה-SC דורשת טכניקות של מיקרוסקופיית אור ברזולוציה גבוהה במיוחד, כגון מיקרוסקופיית תאורה מובנית (SIM), מיקרוסקופיה של דלדול פליטה מגורה (STED) או מיקרוסקופיית לוקליזציה של מולקולה בודדת (SMLM).
כדי לשמור על המבנה והאינטראקציות של רכיבים בודדים בתוך ה- SC, חשוב להתבונן במכלול בסביבה הקרובה לסביבה הטבעית שלה בתאי הנבט. לכן, אנו מדגימים פרוטוקול אימונוהיסטוכימיה והדמיה המאפשר לחקור את תת-המבנה של ה-SC ברקמת נבט של Caenorhabditis elegans שלמה, עם SMLM ומיקרוסקופיית STED. קיבוע ישיר של הרקמה לכיסוי מפחית את תנועת הדגימות במהלך ההדמיה וממזער סטיות בדגימה כדי להשיג את הרזולוציה הגבוהה הדרושה כדי לדמיין את תת-המבנה של ה- SC בהקשר הביולוגי שלו.
Introduction
הפחתת מספר הכרומוזומים בחצי במהלך המיוזה היא המפתח ליצירת צאצאים בריאים באורגניזמים המתרבים מינית. כדי להשיג הפחתה זו במספר הכרומוזומים, כרומוזומים הומולוגיים חייבים להזדווג ולהפריד במהלך מיוזה I. כדי להבטיח הפרדה מדויקת של כרומוזומים הומולוגיים, תאי הנבט עוברים פרופאזה I מורחבת, שבמהלכה כרומוזומים הומולוגיים מזווגים, סינפסה ומתאחדים מחדש כדי ליצור קשרים פיזיקליים בין הומולוגים1. ה- SC התגלה כמבנה המרכזי שהוא המפתח לוויסות ההתקדמות הנכונה באמצעות פרופאזה מיוטית2.
ה-SC הוא קומפלקס שהמבנה הכללי שלו נשמר מבחינה אבולוציונית, למרות שיש מעט הומולוגיה בין מרכיבי החלבון שלו. ה-SC זוהה לראשונה במיקרוגרפים של אלקטרונים כמבנה משולש, דמוי סולם, המורכב משני יסודות או צירים רוחביים, אזור מרכזי שנוצר על ידי חוטים רוחביים, ויסוד מרכזי 3,4. קביעת הארגון של רכיבים בודדים בתוך המתחם היא המפתח לקידום הבנתנו את תפקיד ה- SC במהלך פרופאזה מיוטית.
אורגניזם המודל C. elegans מתאים באופן אידיאלי לחקור את המבנה והתפקוד של SC מכיוון שהחיידקים שלו מכילים מספר רב של גרעינים מיוטיים עם SCs5 מורכבים במלואם. מחקרים גנטיים וביוכימיים גילו כי צירי הכרומוזומים נוצרים על ידי שלושה קומפלקסים שונים של קוהסין6,7 וארבעה חלבוני תחום HORMA הנקראים HTP-1/2/3 ו-HIM-3 7,8,9,10,11 ב-C. elegans. באזור המרכזי של ה-SC זוהו עד כה שישה חלבונים המכילים תחומים סליליים מפותלים 12,13,14,15,16,17. כדי לגשר על המרחק בין שני הצירים, SYP-1, -5 ו-6- מתעמעמים באופן ראש בראש (איור 1), בעוד ששלושה חלבונים נוספים מייצבים את האינטראקציה שלהם ביסוד המרכזי16,17,18,19.
קבלת תובנה מפורטת על הארגון של חלבונים אלה חיונית להבנת הפונקציות הרבות של SC במהלך מיוזה. מכיוון שרוחב האזור המרכזי של ה-SC הוא רק ~100 ננומטר, לא ניתן לפתור את תת-המבנה שלו על ידי מיקרוסקופיה פלואורסצנטית מוגבלת עקיפה. עם זאת, הדמיה של רכיבים בתוך מבנה בגודל זה ניתנת להשגה בקלות על ידי מיקרוסקופיה ברזולוציה גבוהה. ואכן, מיקרוסקופיית תאורה מובנית (SIM), מיקרוסקופיית הרחבה 20, מיקרוסקופיית דלדול פליטה מגורה (STED) 21, ומיקרוסקופיית לוקליזציה של מולקולות בודדות (SMLM)22,23 התגלו ככלים חיוניים לחקר הארכיטקטורה המולקולרית של ה-SC על פני מינים 16,24,25,26,27,28,29, 30.
כדי להתגבר על מגבלת הרזולוציה, מיקרוסקופיית STED מסתמכת על כיסוי הנקודה המוגבלת בדיפרקציה של אור הפליטה עם קרן בצורת סופגנייה מלייזר STED, אשר תיאורטית מכווצת את פונקציית התפשטות הנקודות לממדים מולקולריים31,32. עם זאת, הרזולוציה הניתנת למעשה להשגה על ידי STED בתוך דגימות ביולוגיות נשארת בטווח של כמה עשרות ננומטרים ב- xy33.
רזולוציה גבוהה עוד יותר בדגימות ביולוגיות ניתן להשיג עם טכניקות SMLM. SMLM רותם את תכונות המצמוץ של פלואורופורים ספציפיים כדי לפתור אובייקטים ברמת תת-עקיפה על ידי הפרדת פלואורופורים חופפים מרחבית בזמן. לאחר מכן הדגימה מצולמת שוב ושוב כדי ללכוד תת-קבוצות שונות של פלואורופורים. המיקום של הפלואורופורים בתוך הדגימה נקבע לאחר מכן על ידי התאמת פונקציית התפשטות הנקודות (PSF) לאותות המתקבלים בכל התמונות, מה שיכול לפתור מבנים עד 15 ננומטר23,34.
יחד, התמונות המקומיות מקודדות את מיקומם של כל הפלואורופורים. הרזולוציה של SMLM נקבעת על ידי צפיפות הסימון והמאפיינים המצמוץ של הפלואורופור. על פי קריטריון ניקוויסט-שאנון, אי אפשר לפתור באופן אמין אובייקטים שהם פחות מפי שניים מהמרחק הממוצע בין תווית לתווית. לפיכך, יש צורך בצפיפות תיוג גבוהה להדמיה ברזולוציה גבוהה. עבור SC ב- C. elegans, ניתן להשיג צפיפות תיוג גבוהה על ידי שימוש בתגי אפיטופ המוצמדים לאתרים ספציפיים של חלבונים אנדוגניים באמצעות עריכת גנום. לאחר מכן ניתן להכתים את תגי האפיטופ בצפיפות גבוהה באמצעות נוגדנים חד שבטיים ספציפיים בעלי זיקות גבוהות19,30. יחד עם זאת, המחזור של פלואורופורים בודדים חייב להיות קצר מספיק כדי להבטיח שפלואורופורים חופפים מרחבית לא יילכדו בו זמנית35.
בשל שתי דרישות אלה, פתרון המבנה של קומפלקסים מקרומולקולריים גדולים כגון SC דורש הדמיה של מספר גדול מספיק של תמונות, ולכן יכול לקחת כמה שעות. המלכודת של זמני הדמיה ארוכים היא שדגימות נוטות להיסחף עקב תנועה של השלב או זרמים קטנים בתוך מאגר הדגימה; אפילו תנועות קטנות בסדר גודל של 10 ננומטר מזיקות ברזולוציית NM ויש לתקן אותן. עם זאת, שיטות תיקון הסחף הנפוצות אינן חזקות מספיק כדי לכסות במדויק תמונות של שני ערוצים שצולמו ברצף36. זה בעייתי מכיוון ששאלות ביולוגיות מבקשות לעתים קרובות זיהוי מדויק ולוקליזציה של מטרות מרובות בתוך אותה דגימה. כדי לעקוף בעיות אלה, פותחו שיטות כגון הדמיה יחסותית. הדמיה רציומטרית מאפשרת הדמיה סימולטנית של פלואורופורים מרובים עם ספקטרום עירור ופליטה חופפים, עם הקצאה עוקבת של כל אות שזוהה לפלואורופור המתאים לו בהתבסס על יחס העוצמות בערוצים נפרדים ספקטרלית37,38.
בנוסף, לימוד הארגון של קומפלקסים מקרומולקולריים כגון SC דורש מידע תלת-ממדי (3D). כדי להשיג רזולוציית-על בשלושה ממדים (3D-SMLM), עדשה גלילית משולבת בנתיב האופטי של האור הנפלט המעוותת את צורת ה-PSF של פלואורופור בהתאם למרחקו ממישור המוקד. לפיכך, ניתן לבצע אקסטרפולציה של המיקום המדויק של פלואורופור במישור z על ידי ניתוח צורת אות הפליטה שלו35,39. שילוב ההתקדמות הזו ב-SMLM מאפשר הדמיה של הארגון התלת-ממדי של קומפלקסים מקרומולקולריים, כולל ה-SC.
Protocol
1. הכנת פתרונות וכיסויים
הערה: עיין בטבלת החומרים לקבלת פרטים הקשורים לכל החומרים והריאגנטים ובטבלה 1 להרכב התמיסות המשמשות בפרוטוקול זה.
- כיסויים מצופים פולי-L-ליזין
- הכינו 0.01% (w/v) פולי-ל-ליזין (ראו טבלה 1).
- יש לשטוף כיסוי מדויק (קוטר 24 מ"מ; 0.17 ± 0.005 מ"מ, מס' 1.5) באתנול למשך 10-30 דקות. שטפו את הכיסוי עם ddH2O כדי להסיר אתנול, והשאירו את הכיסוי להתייבש בטמפרטורת החדר.
- פלזמה לנקות את כיסוי באמצעות מנקה פלזמה.
הערה: ניקוי פלזמה מגביר את ההידרופיליות של הכיסוי ומקל על השלבים הבאים. אם חומר ניקוי פלזמה אינו זמין, ניתן לדלג על שלב זה, אם כי הדבר עשוי לדרוש התאמת עוצמת הקול ו/או הריכוז של תמיסת הפולי-ל-ליזין. שינוי זה לא נבדק. - יש להניח טיפה אחת (120 μL) של 0.01% (w/v) poly-L-ליזין על הכיסוי. דגירה במשך 10 דקות בטמפרטורת החדר.
- לאחר הדגירה, יש לשטוף את המכסה ב-ddH2O ולייבש בטמפרטורת החדר. יש לאחסן בטמפרטורה של 4°C עד חודש אחד.
- F(ab')2 שברים מצומדים לצבעים אורגניים פלואורסצנטיים
- הוסף בסדר הבא לצינור PCR: 10 μL של 0.6-0.7 מ"ג/מ"ל F(ab')2 שבר ב- PBS, 1 μL של 0.1 M NaHCO 3 (pH8.3 ) ו- 1 μL של 1 mM succinimidyl (NHS) אסטר תגובתי פלואורופור ב- DMSO (יחס טוחן של F(ab')2:צבע הוא ~ 1:17). מערבבים היטב על ידי צנרת למעלה ולמטה.
- לדגור במשך שעה אחת בטמפרטורת החדר.
- הפרד את שבר F(ab')2 מהצבע הריאקטיבי החופשי הנותר באמצעות עמודת התפלה (7K MWCO) בהתאם למפרטי היצרן. השתמש ב- 1x PBS לשיווי משקל של העמודה ו- elution של מקטע F(ab')2 המסומן.
- אחסן את שבר F(ab')2 המסומן ב-4 °C למשך עד 3 חודשים.
הערה: זמני אחסון של יותר מ-3 חודשים לא נבדקו.
2. דיסקציה וקיבוע
הערה: הליכי הדיסקציה והקיבוע משתנים מפרוצדורות16,40 שהומלצו בעבר כדי לקבל דגימות אופטימליות למיקרוסקופיה ברזולוציה גבוהה.
- דיסקציה
- בחרו תולעי C. elegans תואמות גיל (שגודלו בטמפרטורה של 20 מעלות צלזיוס במחקר זה) לטיפה של 30 μL של EBTT (1x Egg buffer41 עם 0.2% חומר ניקוי לא יוני, טבלה 1) על מכסה (22 מ"מ x 22 מ"מ, מס' 1). הנח את משטח הכיסוי על מגלשת זכוכית למניפולציה קלה יותר. יש לשטוף עם 30 μL של EBTT על ידי פיפטינג למעלה ולמטה מספר פעמים. הסר 30 μL של התמיסה כדי להשאיר טיפה של 30 μL על הכיסוי.
הערה: יש להוסיף כמויות קטנות של חומר ניקוי לא-יוני לכל התמיסות שבהן התולעים עוברות פיפטה כדי למנוע מהתולעים להידבק לקצוות הפלסטיק. - השתמשו בלהב אזמל כדי לחתוך את הראשים ו/או את הזנבות של התולעים כדי לבלוט את הגונד (איור 2A).
- בחרו תולעי C. elegans תואמות גיל (שגודלו בטמפרטורה של 20 מעלות צלזיוס במחקר זה) לטיפה של 30 μL של EBTT (1x Egg buffer41 עם 0.2% חומר ניקוי לא יוני, טבלה 1) על מכסה (22 מ"מ x 22 מ"מ, מס' 1). הנח את משטח הכיסוי על מגלשת זכוכית למניפולציה קלה יותר. יש לשטוף עם 30 μL של EBTT על ידי פיפטינג למעלה ולמטה מספר פעמים. הסר 30 μL של התמיסה כדי להשאיר טיפה של 30 μL על הכיסוי.
- קיבוע
- הזיזו 30 μL של תמיסה מקובעת (טבלה 1) לתוך טיפת התולעים המנותחות ופיפט למעלה ולמטה לערבוב.
הערה: צנרת למעלה ולמטה כמה פעמים עשויה לעזור לשחרר יותר גונדות. - תקן במשך דקה אחת בדיוק לאחר הוספת הפתרון המקבע.
- עצור את הקיבוע על ידי העברת התולעים לתוך צינור PCR מלא TBST (טבלה 1). מעבירים את התולעים בנפח קטן ככל האפשר (~15 μL).
- סובבו את צינור ה-PCR על צנטריפוגה קטנה (2,000 × גרם, 10 שניות). יש להסיר את הסופר-נטנט ולשטוף 2x עם 200 μL של TBST כל אחד.
- יש לשטוף עם 200 μL של PBST (טבלה 1) במשך 5-10 דקות. חזור על שלבים 2.1.1 עד 2.2.5 עבור עד ארבע דגימות תוך שמירה על הדגימות שנותחו על קרח.
הערה: אם אתה מעבד יותר מארבע דגימות, המשך בשלבים 2.2.6 עד 2.2.7 לאחר כל ארבע דגימות כדי להבטיח שקיבוע הדגימה יישאר עקבי בכל הדגימות. - סובבו את הדגימות המנותחות על צנטריפוגה קטנה (2,000 × גרם, 10 שניות), הסירו את ה-PBST והוסיפו 50-100 מיקרון ליטר של מתנול קר (-20 מעלות צלזיוס).
אזהרה: מתנול הוא רעיל. יש ללבוש ציוד מגן ולהימנע משאיפה. - מערבבים על ידי צנרת למעלה ולמטה ומשאירים את הדגימות במתנול למשך 30-60 שניות. לשטוף את הדגימות 2x ב 200 μL של PBST.
הערה: אם אתה מעבד יותר מארבע דגימות, המשך בניתוח הדגימות הנותרות (שלבים 2.1.1 עד 2.2.7). - לשטוף את הדגימות בפעם השלישית עם 200 μL של PBST.
- הזיזו 30 μL של תמיסה מקובעת (טבלה 1) לתוך טיפת התולעים המנותחות ופיפט למעלה ולמטה לערבוב.
3. דגירה של נוגדנים
- חסימת
- חסום את הדגימות בתמיסת חסימה אחת (טבלה 1) למשך 45-60 דקות בטמפרטורת החדר.
הערה: זמן הדגירה יכול לנוע בין 30 דקות בטמפרטורת החדר למספר ימים ב -4 מעלות צלזיוס (הבדיקה נעשתה עד 3 ימים).
- חסום את הדגימות בתמיסת חסימה אחת (טבלה 1) למשך 45-60 דקות בטמפרטורת החדר.
- פתרון נוגדנים ראשוני
- דילול נוגדנים נגד HTP-3 (עוף42) ואנטי-HA (עכבר) (או הנוגדנים לפי בחירה) לפתרונות העבודה (1:250 עבור SMLM ו-1:1,000 עבור דגימות מיקרוסקופיית STED) בתמיסת חסימה אחת.
הערה: נוגדנים המשמשים לתיוג דגימות SMLM מרוכזים יותר מאשר עבור דגימות STED מכיוון שצפיפות תיוג גבוהה יותר מומלצת עבור מיקרוסקופ SMLM. - סובבו את הדגימות על צנטריפוגה קטנה (2,000 × גרם, 10 שניות), הסירו את המאגר החוסם והוסיפו 30-50 מיקרון של תמיסת הנוגדנים העיקרית. יש לדגור למשך הלילה בטמפרטורה של 4 מעלות צלזיוס (עדיפות) או למשך 1-2 שעות בטמפרטורת החדר.
- לאחר הדגירה, לשטוף 3 x 5-15 דקות עם PBST.
- דילול נוגדנים נגד HTP-3 (עוף42) ואנטי-HA (עכבר) (או הנוגדנים לפי בחירה) לפתרונות העבודה (1:250 עבור SMLM ו-1:1,000 עבור דגימות מיקרוסקופיית STED) בתמיסת חסימה אחת.
- תמיסת עבודה של שברי F(ab')2 המצומדים לצבע פלואורסצנטי
- דלל את שברי F(ab')2 המסומנים (שלב 1.2.4) לפתרונות העבודה (1:100 עבור SMLM ו- 1:1,000 עבור דגימות מיקרוסקופיית STED) בפתרון חסימה 1x.
הערה: עבור שתי טכניקות סופר-רזולוציה, נעשה שימוש בזוגות פלואורופור שדווחו בעבר, כלומר AlexaFluor647/CF680 עבור SMLM ו-AlexaFluor594/Abberior STAR635P עבור STED. AlexaFluor647 ו-STAR645P שימשו לתיוג שברי אנטי-עכבר (Fab')2 כדי להתמקד ב-C-terminus של SYP-5, ו-CF680/AlexaFluor594 עם תווית אנטי-עוף (Fab')2 כדי להתמקד ב-HTP-3. - סובבו את הדגימות על צנטריפוגה קטנה (2,000 × גרם, 10 שניות), הסירו את ה-PBST והוסיפו 30-50 μL של תמיסת נוגדנים משנית. דגירה במשך 30 דקות עד 2 שעות בטמפרטורת החדר (עדיף) או לילה ב 4 °C (60 °F). לשטוף 3 x 5-15 דקות עם PBST.
- דלל את שברי F(ab')2 המסומנים (שלב 1.2.4) לפתרונות העבודה (1:100 עבור SMLM ו- 1:1,000 עבור דגימות מיקרוסקופיית STED) בפתרון חסימה 1x.
4. הרכבת דוגמאות על מכסה
- פוסט-פיקסינגציה
הערה: עיבוד דוגמאות בנפרד באמצעות שלבים 4.1.1-4.2.1.- סובבו את הדגימות המוכתמות והסירו את הסופר-נטנט. הוסף 50 μL של PBST0.2 והעבר את התולעים המוכתמות על מכסה 22 מ"מ x 22 מ"מ מס '1.
הערה: יש להשתמש ב-PBST 0.2 טרי עם0.2 % חומר ניקוי לא-יוני (טבלה 1) לשלב זה כדי למנוע מהתולעים להיצמד לכיסוי. - Pipet 5.7-6.3 μL של תמיסה postfixative על כיסוי poly-L-ליזין.
הערה: יש להביא תחילה את כיסויי Poly-L-ליזין המאוחסנים בטמפרטורה של 4°C לטמפרטורת החדר. - סילקו את התולעים המנותחות באותו נפח (5.7-6.3 מיקרול) והעבירו אותן לטיפת הקיבוע על מכסה הפולי-L-ליזין (איור 2B).
הערה: בשלב זה ובשלב הבא, חשוב מאוד לשמור על הרקמה המנותקת במרכז הכיסוי המצופה פולי-L-ליזין. זה חשוב במיוחד אם מרכיבים את הדוגמאות במחזיק מותאם אישית כך שיתאים למיקרוסקופ SMLM שנבנה בהתאמה אישית המשמש כאן (ראו שלב 5.1, איור 2B). - כסו את הדגימה בכיסוי קטן (קוטר 12 מ"מ, איור 2B). הסר עודפי נוזלים באמצעות פיסת נייר סינון קטנה (איור 2B). תקן במשך 3-5 דקות בחדר חשוך.
- סובבו את הדגימות המוכתמות והסירו את הסופר-נטנט. הוסף 50 μL של PBST0.2 והעבר את התולעים המוכתמות על מכסה 22 מ"מ x 22 מ"מ מס '1.
- "פיצוח הקפאה"
- הקפיאו את הדגימות על-ידי הנחתן על גוש אלומיניום בקרח יבש (איור 2B).
הערה: בלוק האלומיניום חייב להיות מקורר היטב בקרח היבש לפני הנחת הדגימות עליו. המשך עם postfixation של הדוגמאות הנותרות (שלבים 4.1.1 עד 4.2.1). הדגימה צריכה להיות על קרח יבש לפחות 20 דקות או עד שעה לפני השלב הבא (4.2.2). - הסירו את הכיסוי הקטן יותר באמצעות סכין גילוח (איור 2B).
הערה: עבור STED, המשך לשלב 5.2.1. עבור SMLM, המשך לשלב 4.2.3. - טבלו את הכיסוי בצינור חרוטי של 50 מ"ל המכיל PBS קר כקרח (מועדף) או -20°C מתנול למשך כ-10 שניות.
הערה: הטמפרטורה היא גורם חשוב מאוד לשלב זה. לכן, יש להשתמש ב-PBS שמופשר טרי או נשמר באמבט קרח/אתנול. - מניחים את הכיסוי לתוך באר של צלחת בת שש בארות מלאה במאגר PBST. מוציאים את ה-PBST מהבארות ומוסיפים PBS טרי. השאירו את הדגימות ב- PBS למשך 5 דקות.
הערה: הזיזו את ה-PBS על דופן הבאר כדי למנוע פגיעה וניתוק של הדגימות. - יש לשטוף עם PBS טרי ולהשאיר את הדגימות בטמפרטורה של 4 מעלות צלזיוס עד להדמיה.
הערה: הדגימות יציבות למשך עד שבועיים, אך התוצאות הטובות ביותר מושגות אם הדגימות מצולמות תוך יומיים. - לפני ההדמיה, הערך את איכות הדגימה המורכבת תחת מיקרוסקופ סטריאו.
הערה: חיידקים המותקנים בהצלחה מחוברים ביציבות ללא תנועה ניכרת ביחס לכיסוי. חיידקים מחוברים בצורה גרועה יתעופפו בתמיסת החיץ.
- הקפיאו את הדגימות על-ידי הנחתן על גוש אלומיניום בקרח יבש (איור 2B).
5. הדמיה
- מיקרוסקופיית לוקליזציה של מולקולה בודדת
הערה: התמונות נרכשו במרכז ההדמיה של EMBL באמצעות מיקרוסקופ לוקליזציה של מולקולה בודדת שנבנה בהתאמה אישית סביב גוף מותאם אישית, כפי שדווח בעבר38,43עם התכונות הייחודיות שצוינו ב- טבלת חומרים; עיין https://www.embl.org/about/info/imaging-centre- רכישת כיול חרוזים תלת מימדי
- הכינו כיסוי מדויק (קוטר 24 מ"מ; 0.17 ± 0.005 מ"מ, מס' 1.5) עם חרוזי פלואורסצנט דבקים של 100 ננומטר כפי שתואר קודםלכן 38,44.
- הנח את דגימת הכיול משלב 5.1.1.1 על מחזיק דגימה.
- הוסיפו טיפת שמן טבילה על מטרת השמן הנקי 100x/1.5 והרכיבו את דגימת הכיול על המיקרוסקופ.
- בתוך MicroManager 2 45,46, ציין15-20 מיקומים במדגם הכיול.
- בתוך חלון תוסף EMU47, הגדר את רכישת תמונת z-stack עבור כל אחד מהמיקומים משלב 5.1.1.5.
הערה: כאן, עדשה גלילית מורכבת מספקת את האסטיגמציה הנדרשת להדמיה תלת-ממדית, ונרכשו 201 פרוסות z עבור כל מיקום המשתרע על הטווח שבין -1 מיקרומטר ל-1 מיקרומטר, בהפרש של 10 ננומטר. תאורת לייזר של 2 קילוואט/ס"מ2 640 ננומטר שימשה למשך 25 אלפיות השנייה עבור כל פרוסת z. - רכוש את תמונות z-stack של חרוזי פלואורסצנט בגודל 100 ננומטר דרך נתיב אופטי זהה שישמש לרכישת תמונות לדוגמה בשלב 5.1.11.
- באמצעות פלטפורמת ניתוח מיקרוסקופיה ברזולוציה גבוהה (SMAP48), צור מודל cspline של פונקציית התפשטות הנקודות הניסויית (PSF) שישמש להתאמת נתוני 3D-SMLM בשלב 5.1.13.
- הכן את מחזיק המדגם. עבור המחזיק המותאם אישית המשמש כאן ומשתמש בטבעת מגנטית ליצירת תא ההדמיה (איור 2B), עטוף את הטבעת המגנטית בפרפילם.
הערה: לחלופין, ניתן להשתמש בשקופית מיקרוסקופ עם חלל שקע קעור כדי להרכיב דגימות למיקרוסקופים עם מחזיקי שקופיות. - הכן 1 מ"ל של מאגר הדמיה44 (טבלה 1).
- קח כיסוי אחד משלב 4.2.6 והנח אותו במחזיק בהתאמה אישית. תקן את הכיסוי במחזיק באמצעות הטבעת המגנטית העטופה בפארפילם (שלב 5.1.2).
- פזרו בעדינות את מאגר ההדמיה (שלב 5.1.3) בתא שנוצר על-ידי הטבעת המגנטית שעל גבי הדגימה (איור 2B). אטמו את החדר עם חתיכת פרפילם.
- כדי להרכיב את הדגימה, הוסיפו טיפה אחת של שמן טבילה על מטרת השמן הנקי 100x/1.5. מבלי להכניס אוויר לשמן הטבילה, הניחו בעדינות את מחזיק הדגימה עם הדגימה המותקנת (שלב 5.1.5) על במת המיקרוסקופ.
הערה: לפני הנחת הדגימה על המיקרוסקופ, נקה את החלק התחתון של הכיסוי עם רקמה ו-70% אתנול. - באמצעות חלון תוסף EMU 47 בתוך MicroManager 245,46, הזז את שלב הפייזו עד שהאות מלייזר נעילת המיקוד מזוהה בפוטודיודה הרביע (QPD).
הערה: כדי לשמור על מיקוד קבוע לאורך זמן ההדמיה, נעילת המיקוד מושגת על-ידי השתקפות פנימית כוללת של לייזר מצומד לסיבים כמעט אינפרה-אדומים מהכיסוי וזיהוי רגיש לגובה לאחר מכן בפוטודיודה מרובעת (QPD). אות ה-QPD סיפק שליטה בלולאה סגורה על תושבת הפייזו של העדשה האובייקטיבית. - קבל תמונת מישור מוקד אחורי עם לייזר עירור של 640 ננומטר בהספק נמוך (כלומר, 1-5%) כדי לאשר את היעדר בועות האוויר בשמן הטבילה.
הערה: הסר את הדגימה מהשלב אם זוהתה בועת אוויר. נקו את החלק התחתון של הכיסוי ואת המטרה וחזרו על שלבים 5.1.6-5.1.8. אחרת, המשך לנעול את המוקד בתוך תוכנת EMU47. - לוקליזציה של רקמת הגונד באמצעות תאורת השדה הבהיר. באמצעות תאורה בעוצמה נמוכה של 640 ננומטר, התמקדו בחלק של הרקמה המכיל מתיחות SC רבות.
הערה: אין להתמקד במבנים הנמצאים במרחק של יותר מ-2 מיקרומטר מהכיסוי. אין להשתמש בעוצמת לייזר גבוהה יותר כדי לאתר את הדגימה, מכיוון שהדבר עלול להפוך חלק מהפלואורופורים למצב מהבהב בטרם עת. כאן, 1 קילוואט / ס"מ2 שימש במצב עולה עם פולס מוגדר 1,000. - המשך לחשוף את הדגימה עם תאורה של 640 ננומטר בהקרנה גבוהה (27 קילוואט/ס"מ2) במשך ~30 שניות עד שיושג קצב מצמוץ מתאים (סרטון משלים 1).
- רכוש 200,000 פריימים עם זמן חשיפה של 20 אלפיות השנייה באמצעות כלי הרכישה הרב-ממדי ב- MicroManager 245,46.
- בינתיים, הגדר את הפעלת ה- UV באמצעות אפשרות ההפעלה של תוסף EMU38,47 כדי לשמור על קצב ההבהוב הרצוי.
הערה: השתמש בלייזר UV בהקרנה של 3 קילוואט/ס"מ2 במצב עולה עם אורך פולס מרבי מוגדר ל-10,000. - בצע שחזור תמונה SMLM ועיבוד לאחריה.
הערה: כדי לבנות מחדש תמונות מנתוני SMLM גולמיים, עיין בשיטות שפורסמו. הנתונים המוצגים כאן עובדו באמצעות תוכנת SMAP48,49. שחזור תמונה פתור במיוחד, הקצאת ערוץ, תיקון סחף וסינון של לוקליזציות עם דיוק לוקליזציה לקוי ומסנן סבירות מרבי בוצעו בתוכנת SMAP48.
- רכישת כיול חרוזים תלת מימדי
- מיקרוסקופיית דלדול פליטה מגורה
הערה: התמונות נרכשו במערכת המיקרוסקופית המשולבת STED המצוידת בלייזר אור לבן, בלייזר STED פועם של 775 ננומטר, ובמודול FALCON Fluorescence Lifetime IM (טבלת חומרים) במרכז ההדמיה EMBL (https://www.embl.org/about/info/imaging-centre).- הנח טיפה של 20 μL של מדיום הרכבה (טבלת חומרים) על שקופית מיקרוסקופ. קח כיסוי אחד משלב 4.2.2 והנח את הדגימה בעדינות על המגלשה הפונה למדיום ההרכבה (איור 2B).
הערה: יש להימנע מהכנסת כיסי אוויר לאמצעי ההרכבה. - תנו למדיום ההרכבה לרפא בן לילה.
הערה: צלמו את הדגימות ביום שלמחרת או שמרו אותן בטמפרטורה של 4 מעלות צלזיוס עד להדמיה. - כדי להרכיב את הדגימה, הוסף טיפה אחת של שמן טבילה על מכסה הדגימה משלב 5.2.2. הניחו בעדינות את הדגימה על במת המיקרוסקופ באמצעות מטרת שמן בגודל 100x/1.40.
- התמקדו בדגימה ואתרו את רקמת הנבט באמצעות תאורת השדה הבהיר.
- באמצעות תוכנת המיקרוסקופ, ציין את אזור העניין שעבורו תירכש תמונת TauSTED.
- בחר את לייזרי העירור ואת העוצמה המתאימה שלהם המשמשת כדי לעורר את הפלואורופורים המשמשים בדגימה.
הערה: כאן, הלייזר של 580 ננומטר בעוצמה של 4% שימש לדימוי שברי נוגדנים משניים של AlexaFluor 594-מצומדים F(ab')2, ו-635 ננומטר בעוצמה של 3% כדי לצלם שברי F(ab') 2 מצומדים של STAR 635P. - באמצעות תוכנת המיקרוסקופ, בחר עוצמת לייזר מתאימה לדלדול STED והגדר את זיהוי התמונה.
הערה: כאן, עוצמת הלייזר המדלדלת STED של 775 ננומטר נקבעה ל-40%. הגלאי שימש במצב ספירה עם ערך רווח של 10 לזיהוי פוטונים, עם קצב סריקה של 100 הרץ, ובגודל פיקסל של 17 ננומטר. הצטברות של ארבעה קווים שימשה לרכישת TauSTED.
- הנח טיפה של 20 μL של מדיום הרכבה (טבלת חומרים) על שקופית מיקרוסקופ. קח כיסוי אחד משלב 4.2.2 והנח את הדגימה בעדינות על המגלשה הפונה למדיום ההרכבה (איור 2B).
תוצאות
כדי לדמות את ה-SC בתוך רקמת הנבט של C. elegans על-ידי SMLM, השתמשנו ב-3D-SMLM בעל יחס של 2 צבעים כדי למקם את HTP-3, רכיב של צירי הכרומוזומים, ואת C-terminus של הנימה הרוחבית SYP-5 המתויגת באופן אנדוגני עם תג המגלוטינין (HA). המיקום של שני החלבונים בתוך SC של C. elegans נקבע בעבר על ידי מחקרים אחרים16,30.
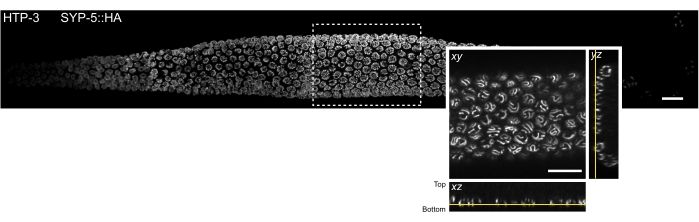
כדי למזער את פיזור האור ואת הסטיות האופטיות הטמונות בדגימות ביולוגיות עבות, דמיינו את חתך ה-z התחתון ביותר של גרעינים מיוטיים המכילים את ה-SCs (איור 3, קווים צהובים). עבור כל תמונה שנרכשה, מיקום הבמה של piezo של מישור ההדמיה סומן ביחס למיקום שלב piezo כאשר המטרה התמקדה בכיסוי. זה איפשר את חישוב המרחק piezo מן הכיסוי. דגימות שהורכבו בהצלחה מחוברות ביציבות קרוב לכיסוי ושומרות על צורת הגונד (כלומר, הרקמה אינה מרוסקת בין שני הכיסויים במהלך שלב הפוסט-פיקסציה). ניתן להעריך בקלות את איכות הרכבה של הדגימה תחת מיקרוסקופ סטריאו מכיוון שגונדות מחוברות היטב אינן מראות כל תנועה בתמיסה (שלב 4.2.6). עם זאת, בשל הסטוכסטיות של תהליך ההרכבה, רקמת הגונד לא בהכרח תהיה מונחת שטוחה לחלוטין על הכיסוי. לכן, המישור התחתון של הגרעינים המכילים SCs עשוי להימצא במרחקים משתנים ביחס לכיסוי בתוך אותה גונדה.
כדי להמחיש כיצד הרזולוציה משתנה בהתאם להצמדת הרקמה לכיסוי, רכשנו תמונות במרחקי piezo שונים לכיסוי. כדי להעריך את האיכות של תמונה בודדת, חושבו עקומות המתאם של טבעת פורייה (FRC)50,51 והרזולוציה נקבעה באמצעות תוסף FRCResolution בתוכנת SMAP 48. שני גרעינים מייצגים שחולצו משתי תמונות נפרדות של 3D-SMLM שצולמו במרחקים שונים לכיסוי מוצגים באיור 4. ב-SCs הממוקמים קרוב לכיסוי, צירי הכרומוזומים וסיומת C של SYP-5::HA נפתרים היטב בכל שלושת הממדים (איור 4A, 0.8 מיקרומטר מהכיסוי). כדי לפתור שני מבנים המופרדים על ידי מרחק נתון, רזולוציית FRC שהושגה חייבת בדרך כלל להיות קטנה ממחצית המרחק הזה ברזולוציה הצירית.
כדי להפריד את אותם מבנים לרוחב, יש להשיג אפילו ערכי רזולוציית FRC קטנים יותר. ואכן, בדגימות הממוקמות בסמיכות לכיסוי, רזולוציית ה- FRC היא 38 ננומטר עבור ערוץ AlexaFluor 647 ו- 34 ננומטר עבור ערוץ CF680, ולכן הרבה מתחת למרחק הצפוי של 84 ננומטר בין ה- C-termini של SYP-516. לפיכך, רזולוציה זו פותרת בקלות את הארגון של ה-SC לא רק בראייה פרונטלית אלא גם בראייה רוחבית (איור 4B i,ii). לעומת זאת, הרזולוציה מתדרדרת ב-SCs הממוקמים במרחק של 5 מיקרומטר מהכיסוי עקב פיזור אור וסטיות כדוריות (איור 4B). רזולוציות ה-FRC במרחק זה יורדות ל-47 ננומטר (AlexaFluor 647) ו-41 ננומטר (CF680), מה שלא יכול לפתור באופן מלא את ה-C-termini של SYP-5. מאחר שהסטיות האופטיות פוגעות ברזולוציה הצידית בצורה חמורה יותר מאשר הרזולוציה הצירית, רצועות HTP-3 ו-SYP-5 אינן נפתרות עוד בבירור בחתך הרוחב של התצוגה הצידית בדגימות הממוקמות במרחק של 5 מיקרומטר מהכיסוי (איור 4B ii). השוואת רזולוציית ה-FRC של תמונות שנרכשו במרחקי piezo שונים מהכיסוי גילתה שהרקמה המצולמת צריכה להיות במרחק של לא יותר מ-2 מיקרומטר מהכיסוי (איור 5). תוצאה זו מדגישה את החשיבות של ביצוע נכון של שלב postfixation, שבמהלכו יש להצליב את הרקמה בהצלחה לציפוי הפולי-L-ליזין של הכיסוי.
כדי להדגים את הרזולוציה הניתנת להשגה באמצעות טכניקה אחרת של רזולוציית-על, דמיינו גם SCs ברקמת נבט שלמה קבועה עם מיקרוסקופיית TauSTED. איור 6A מציג תמונות TauSTED עם הרזולוציה הגבוהה והנמוכה ביותר שהושגה במחקר זה, כפי שהוערך מפרופילי קווים של ה-SC במבט חזיתי (איור 6B). בשני הגרעינים, יכולנו לפתור את שני פסי הלוקליזציה של HTP-3 בצירי הכרומוזומים ושל C-termini של SYP-5 באזור המרכז, ולהוכיח כי הרזולוציה שניתן להשיג ב- TauSTED באמצעות פרוטוקול ממוטב זה היא מתחת ל-84 ננומטר. בתנאים אופטימליים (איור 6A, למעלה), יכולנו לפתור את ה-C-termini בתצוגות מוטות מעט של ה-SC שהופרדו ב-50 ננומטר בלבד (איור 6A, מלבן צהוב ו-6C).

איור 1: סכמטי של ארגון הקומפלקס הסינפטונמלי ב-Caenorhabditis elegans. הקריקטורה מציגה מבנה פשוט של ה-SC ב-C. elegans המגשר בין שני כרומוזומים הומולוגיים (אפור). המבנה מוצג בתצוגות חזיתיות, לרוחב וחתך. צירי הכרומוזומים מוצגים כפסים אדומים ואילו חוטים רוחביים מוצגים בציאן. חלבוני נימה רוחביים (SYP-1, 5, 6 ב- C. elegans) מכוונים באופן ראש בראש (גרפיקת מקל כדור ציאן) באזור המרכז כדי לגשר על המרחק בין שני הצירים. המרחקים הצפויים בין הצירים לבין C-termini של חוטים רוחביים מסומנים. קיצור: SC = קומפלקס סינפטונמלי. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
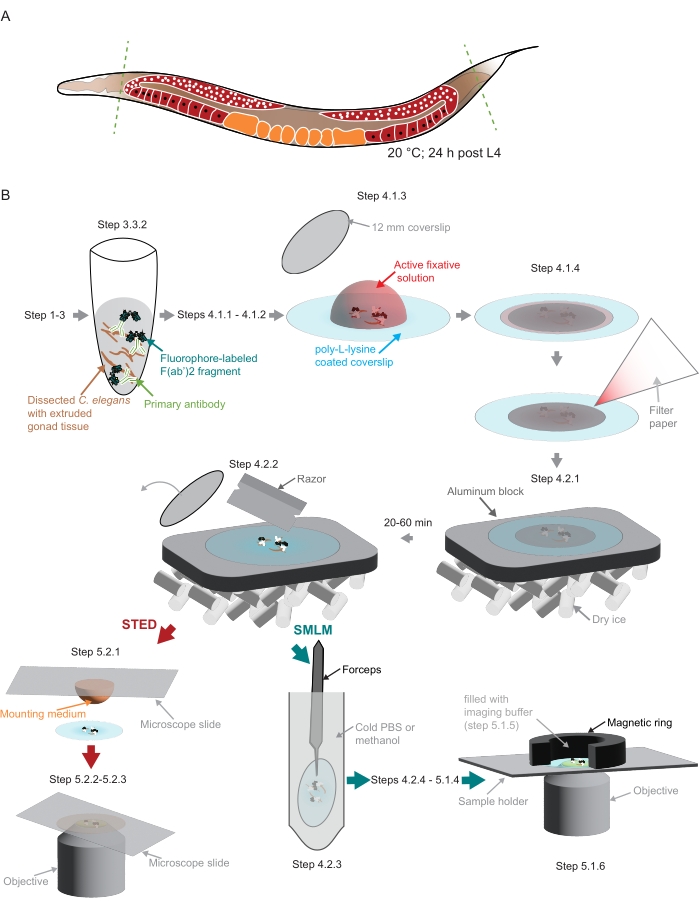
איור 2: המחשה של הכנת המדגם ששימשה במחקר . (A) בוגרים צעירים מסוג C. elegans מנותחים בראשם או בזנבו (קווים ירוקים ומקווקווים) ומעובדים כמתואר בפרוטוקול. (B) שלבים בודדים של השיטה מסומנים בגרפיקה המחוברת בחצים אפורים. קיצורים: STED = דלדול פליטה מגורה; SMLM = מיקרוסקופיית לוקליזציה של מולקולה בודדת; PBS = מלח חצוב פוספט. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
איור 3: מיקום מקטע הרקמה שניתן לצפות בו על-ידי מיקרוסקופיית לוקליזציה של מולקולה בודדת. MIP של תמונה קונפוקלית של דיסק מסתובב של גונד הר שלם C. elegans. הרקמה הוכתמה עבור HTP-3 ו- C-terminus של SYP-5 (SYP-5::HA), והאות המשולב מוצג באפור. תמונות קונפוקליות בודדות נתפרו באמצעות תוסף פיג'י52 לתפירת גריד/אוסף כדי ליצור תמונה של הגונד כולו. הכניסה מציגה תצוגת xy של מישור z התחתון ביותר המכיל את ה- SCs. הלוקליזציה של מישור זה מוצגת בתצוגות אורתוגונליות של קטע הרקמה המצוין על ידי מלבן בתמונת MIP של הגונד (קווים צהובים). סרגלי קנה מידה = 10 מיקרומטר. קיצורים: MIP = הטלת עוצמה מקסימלית; SCs = קומפלקסים סינפטונמליים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
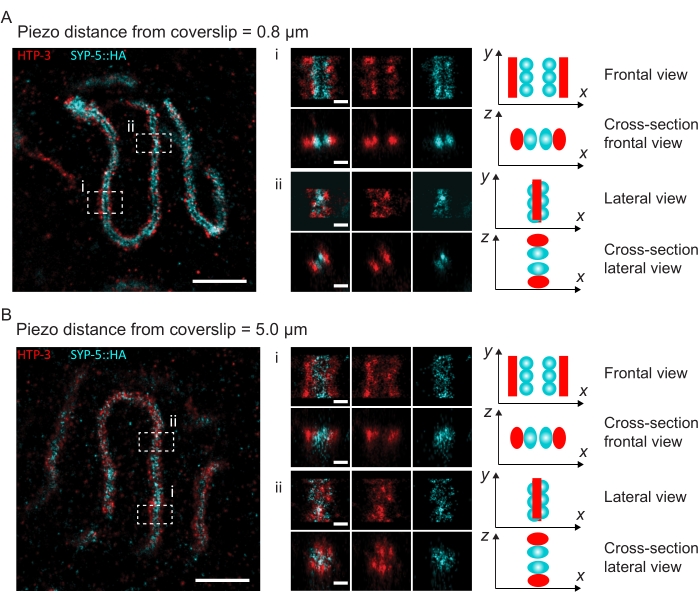
איור 4: מיקרוסקופיית לוקליזציה של מולקולה בודדת של HTP-3 וה-C-termini של ה-SYP-5. (A,B) משמאל: תמונות SMLM המציגות גרעיני פכיטן מוכתמים עבור HTP-3 (אדום) ואת C-terminus של SYP-5 (SYP-5::HA, ציאן) (סרגל קנה מידה = 1 מיקרומטר). מרכז: תמונות מוגדלות של אזורי עניין המצוינים ב- A וב- B עם תצוגות חתך תואמות המוצגות מתחת לכל תמונה (i, ii; סרגל קנה מידה = 100 ננומטר). המתיחות של ה-SC בתוך תמונות מוגדלות מסובבות כדי לכוון את צירי הכרומוזומים במקביל לציר ה-y. ימין: ייצוג גרפי של לוקליזציה של החלבונים בעלי העניין בתוך ה-SC המתאר את הכיוון של ה-SC באזורים המוגדלים המוצגים במרכז האיור. קיצורים: SMLM = מיקרוסקופיית לוקליזציה של מולקולה בודדת; SC = קומפלקס סינפטונמלי. נתונים גולמיים לשחזור תמונות SMLM זמינים דרך מסד הנתונים BioStudies60 (מזהה גישה: S-BIAD504). אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
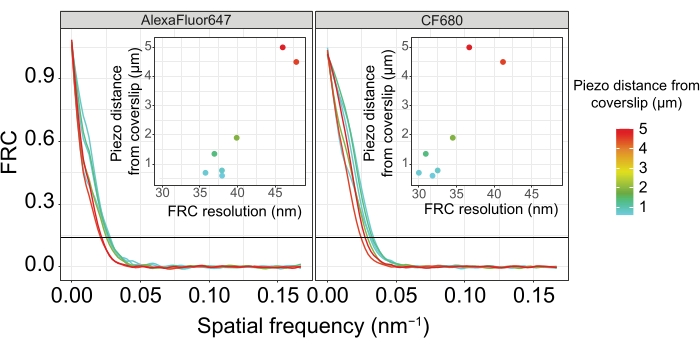
איור 5: רזולוציית המתאם של טבעת פורייה של תמונות מיקרוסקופיה של לוקליזציה של מולקולה בודדת תלויה במרחק של מישור z המצולם ממישור הכיסוי. קווים צבעוניים מציגים עקומות FRC של תמונות שנרכשו במרחקים שונים (כפי שמתואר בסרגל הצבע) מהכיסוי. סף 1/7 המשמש לקביעת רזולוציית FRC מסומן על ידי קו אופקי שחור. כניסות מראות את התלות של רזולוציית FRC במרחק הפיאזו מהכיסוי. העלילה בוצעה על ידי סקריפט R שנכתב בהתאמה אישית (גרסה 4.1.2, קובץ משלים 1) שבו העקומות המקוריות הוחלקו עם פונקציות מחבילת "ggplot2". קיצורים: FRC = מתאם טבעת פורייה; SMLM = מיקרוסקופיית לוקליזציה של מולקולה בודדת; SC = קומפלקס סינפטונמלי. נתונים עבור עקומות FRC ונתוני SMLM זמינים דרך מסד הנתונים BioStudies60 (מזהה גישה: S-BIAD504). אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.

איור 6: מיקרוסקופיית דלדול פליטה מגורה המשופרת על-ידי מידע פלואורסצנטי מבוסס אורך חיים (TauSTED) פותרת שני תחומי לוקליזציה הן עבור HTP-3 והן עבור C-terminus של SYP-5. (A) שתי תמונות מייצגות של TauSTED מראות גרעיני פכיטן מוכתמים עבור HTP-3 (אדום) ו-C-terminus של SYP-5 (SYP-5::HA, ציאן) עם הגדרה מבנית גבוהה יותר (עליונה) ותחתונה (למטה) (סרגל קנה מידה = 1 מיקרומטר). המלבנים מסמנים אזורים עם C-termini פתור של SYP-5 בחזית (לבן) ותצוגה מוטה מעט (צהוב) של SC. (B,C) התפלגות של HTP-3 (אדום) ו- C-terminus של SYP-5 (ציאן) אות שנפתר על ידי TauSTED. פרופילי קו של אזורי עניין המכילים את ה- SC בתצוגות חזיתיות (B) או מוטות מעט (C) מוצגים כקווים מלאים עם עוצמה מנורמלת לערך המרבי. פרופילי קו נוצרו באמצעות פיג'י ImageJ. קווים מקווקווים ב-B מציגים את הנתונים הממוצעים של כל חלבון. קו הציאן העבה ב- C מתאים לפרופיל הקו עם המרחק הקצר ביותר בין ה- C-termini של SYP-5 . כדי לקבוע את המרחקים בין הנוגדנים המכוונים לחלבונים ספציפיים, פרופילי הקו (n = 9 (B), n = 7 (C)) הותאמו לגאוס כפול באמצעות סקריפט R שנכתב בהתאמה אישית (גרסה 4.1.2, קובץ משלים 1). המרחק הממוצע ± סטיית התקן (B) והטווח עם הערך המינימלי המודגש בהדגשה (C) מסומנים מעל כל תרשים, בהתאמה. קיצורים: STED = מיקרוסקופיה של דלדול פליטה מגורה; SC = קומפלקס סינפטונמלי. תמונות מוצגות ונקודות נתונים של פרופילי קו משורטטים זמינות דרך מסד הנתונים BioStudies60 (מזהה גישה: S-BIAD504). אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
טבלה 1: הרכב המאגרים והפתרונות המשמשים בפרוטוקול זה. אנא לחץ כאן כדי להוריד טבלה זו.
סרטון משלים 1: רכישת מיקרוסקופיה של לוקליזציה של מולקולה בודדת. סרטון המציג פלואורופורים מהבהבים בקצב המתאים (מוצגות 50 פריימים, סרגל קנה מידה = 5 מיקרומטר, 20 אלפיות השנייה למסגרת). אנא לחץ כאן כדי להוריד סרטון זה.
קובץ משלים 1: סקריפט ניתוח נתונים. אנא לחץ כאן כדי להוריד קובץ זה.
Discussion
הארגון דמוי הסולם של ה-SC, החיוני לרקומבינציה נכונה ולהפרדה של כרומוזומים הומולוגיים, נצפה לראשונה לפני כמעט 70 שנה במיקרוסקופיית אלקטרונים 3,4. בעוד שהארגון הכולל של ה- SC נפתר בקלות במיקרוסקופיית אלקטרונים, לוקליזציה של רכיבים בודדים בתוך קומפלקס זה דורשת גישה ממוקדת יותר. עם רוחבו של ~ 100 ננומטר בלבד, לא ניתן לפתור את תת-המבנה של ה- SC על ידי מיקרוסקופיה פלואורסצנטית קונבנציונלית. עם זאת, מיקרוסקופיה ברזולוציה גבוהה הפכה למניע מרכזי לתגליות חדשות על המבנה והתפקוד של הקומפלקס הסינפטונמלי 16,19,24,25,26,27,28,29,30. כדי להקל על מחקר זה, הדגמנו הליך הרכבה המאפשר ללמוד את הארכיטקטורה של SC בתוך C. elegans gonad רקמה עם SMLM ו STED מיקרוסקופיה.
שלב קריטי למיטוב הרזולוציה בהדמיית SMLM הוא קישור ישיר של רקמת הנבט לכיסוי מצופה פולי-L-ליזין (שלב 4). ההצמדה הקוולנטית של הרקמה לכיסוי חיונית כדי להפחית את התנועות בתוך הדגימה שיגרמו לסחף גדול ויהפכו את ההדמיה לאורך פרקי זמן ארוכים עבור SMLM לבלתי אפשרית. בנוסף, אפילו חיבור לא אופטימלי שמשאיר את הגרעינים המכילים SCs במרחק מהכיסוי מוביל לירידה משמעותית ברזולוציה הניתנת להשגה כתוצאה מסטיות כדוריות (איור 4). לחלופין, לחיבור הקוולנטי המשמש כאן, רקמת נבט מוכתמת יכולה גם להיות משותקת בין שני כיסויים אטומים בטיפה קטנה של מאגרהדמיה 19,30. עם זאת, שיטת אימוביליזציה זו מפחיתה מאוד את נפח מאגר ההדמיה בדגימה מ-1 מ"ל המשמש בפרוטוקול הממוטב כאן למספר μL בלבד, מה שיגרום להחמצת מאגר ההדמיה ויפחית מאוד את הזמן שבו ניתן לצלם את הדגימה 38,53,54.
זמני רכישה ארוכים הן עבור מיקרוסקופיית SMLM והן עבור מיקרוסקופיית STED מגבילים את השימוש בשיטות אלה להדמיה של דגימות קבועות כימית. כאן, קיבוע paraformaldehyde מבטיח כי המבנה של SC נשמר במהלך הכנת המדגם והדמיה. עם זאת, למרות אמצעי הזהירות שננקטו כאן כדי לדמות את ה-SC בתוך רקמה שלמה, המבנה המתקבל של ה-SC לאחר הקיבוע אינו בהכרח זהה למבנה במצבו הטבעי בתוך אורגניזם חי. יתר על כן, מכיוון שתמונה אחת של ה-SC הקבוע מייצגת "תמונת מצב" יחידה של המבנה הביולוגי, גישה זו נותרת עיוורת לדינמיקה של המבנה המקומי in vivo.
עם זאת, מידע על הדינמיקה והשונות של מבנים מקרומולקולריים יכול גם להתקבל על ידי רכישת לא רק אחד אלא רבים "תמונות". בעוד שגישה זו יכולה לפתור שינויים במבנה של SC במהלך pachytene19, ישנם מספר גורמים המגבילים את מספר התמונות שניתן לרכוש מדגימה אחת שהוכנה באמצעות פרוטוקול זה. ראשית, כוחות הלייזר הגבוהים המשמשים במהלך רכישת התמונה מובילים להלבנה קבועה של הפלואורופורים ומונעים הדמיה של אזורי עניין סמוכים או מישורי z מרובים, ובכך מפחיתים משמעותית את מספר התמונות שניתן לרכוש מדגימה אחת. שנית, צפיפות הדגימה/רקמה על הכיסוי שהוכן בשיטה זו נמוכה, מה שמגביל משמעותית את מספר התמונות שניתן לרכוש מכיסוי יחיד. צפיפות הדגימה הנמוכה אוסרת גם על שימוש בצינורות אוטומטיים לרכישת תמונות שעזרו לשפוך אור על שאלות ביולוגיות אחרות 34,55,56,57,58,59. עם זאת, צפיפות המדגם יכולה להיות מוגברת מעט על ידי משתמש מנוסה.
הפרוטוקול המוצג כאן מותאם להשגת צפיפות תיוג גבוהה הנחוצה להשגת רזולוציה אופטימלית ב- SMLM35. בעוד שפרוטוקולים קודמים מצמידים את הרקמה באופן קוולנטי לכיסוי לפני חיסון16, פרוטוקול חדש זה מצליב את הרקמה לכיסוי רק לאחר שהדגימות הוכתמו בתמיסה. שינוי זה מאפשר לנוגדנים המשמשים להדבקה חיסונית לגשת באופן חופשי לרקמה מכל הצדדים, בעוד שהחיבור הקוולנטי של הרקמה לכיסוי עשוי להגביל את הגעת הנוגדנים לגרעינים הקרובים ביותר לכיסוי, ובכך להפחית את מידת התווית. יחד, השינויים המתוארים כאן משפרים את הרזולוציה מ-40-50 ננומטר (רזולוציית FRC)16 ל-30-40 ננומטר (פרוטוקול זה).
חשוב לציין כי בעוד שצפיפות תיוג גבוהה וריכוז גבוה של נוגדנים חיוניים ל-SMLM, מצאנו שתמונות מיקרוסקופיות STED טובות יותר מתקבלות באמצעות ריכוזי נוגדנים נמוכים יותר (שלב 3). ברזולוציה של עשרות ננומטרים, גודל המולקולות המשמשות לתיוג החלבון המעניין הופך לחשוב יותר ויותר. לכן השתמשנו בשברי F(ab')2 שגודלם מחצית מגודלם של נוגדנים באורך מלא. השיפור בניגודיות המקומית עקב מקור אות קטן יותר, ולכן הרזולוציה שהושגה על ידי שינוי זה בהשוואה לשימוש בנוגדנים משניים באורך מלא, אפשרו את הרזולוציה של שני C-termini של SYP-5 באזור המרכז על ידי TauSTED, אשר אינם נפתרים על ידי STED קונבנציונאלי באמצעות נוגדנים באורך מלא (16 ונתונים לא מוצגים). אנו צופים כי פרוטוקול ממוטב זה להדמיית SCs ב-C. elegans germlines שלמים יקל על חקירת הקשר בין מבנה לתפקוד של ה-SC במהלך מיוזה.
Disclosures
המחברים מצהירים על היעדר ניגוד עניינים.
Acknowledgements
ברצוננו להודות לג'ונאס ריס ולמעבדת ריס על שיתוף מאגרי הדמיה להדמיית SMLM. אנו מודים גם ליומי קים על זן C. elegans המשמש בפרוטוקול זה ולאבי פ. דרנבורג על הנוגדן נגד HTP-3. אנו מודים למרקו לאמפה וסטפן טרג'ונג ממתקן מיקרוסקופיית האור המתקדם ב- EMBL היידלברג על תמיכתם בשימוש במיקרוסקופ הקונפוקלי Olympus iXplore SPIN SR. עבודה זו נתמכה על ידי המעבדה האירופית לביולוגיה מולקולרית ו- Deutsche Forschungsgemeinschaft (DFG, קרן המחקר הגרמנית - 452616889, SK). אנו מכירים את הגישה והשירותים הניתנים על ידי מרכז ההדמיה במעבדה האירופית לביולוגיה מולקולרית (EMBL IC), הנתמכים בנדיבות על ידי קרן Boehringer Ingelheim.
Materials
| Name | Company | Catalog Number | Comments |
| 100x/1.5 oil objective | Olympus | UPLAPO100XOHR | UPLAPO100XOHR |
| 2-mercaptoethylamine (MEA) | Sigma-Aldrich | 30070-10G | Dissolved in MilliQ water to 5 M solution, pH 8.7 adjusted with HCl. Aliquoted to a single-use volume, frozen, and kept at -80 °C. |
| Additional 640 nm booster laser | Toptica | IBEAM-SMART-640-S-HP | |
| AlexaFluor 594, NHS ester | ThermoFischer Scientific | A37572 | Dissolved in DMSO to 1 mM solution, aliquoted to single use volume, frozen and kept at -80 °C |
| AlexaFluor 647, NHS ester | ThermoFischer Scientific | A37573 | Dissolved in DMSO to 1 mM solution, aliquoted to single use volume, frozen and kept at -80 °C |
| anti-HA | Thermo Fisher Scientific | 2-2.2.14 | Mouse monoclonal, 1:250 (SMLM), 1:1,000 (STED Microscopy) |
| anti-HTP-3 | a gift from Abby F. Dernburg | MacQueen et al., 2005 | Chicken polyclonal, 1:250 (SMLM), 1:1,000 (STED Microscopy) |
| Caenorhabditis elegans strain YKM349 | a gift from Yumi Kim | Hurlock et al., 2020 | syp-5(kim9[syp-5::HA]) I; meIs8[pie-1p::GFP::cosa-1, unc-119(+)] II |
| CF6680, NHS ester | Biotium | 92139 | Dissolved in DMSO to 1mM solution, aliquoted to single use volume, frozen and kept at -80 °C |
| Circular cover glass 12 mm No. 1 | Menzel-Gläser; VWR | 631-0713 | |
| Circular cover glass 24 mm No. 1.5 | Carl Roth | PK26.1 | |
| Cylindrical lenses | Thorlabs | LJ1516RM-A, LK1002RM-A | |
| Egg Buffer (10x) | Edgar 1995 | 250 mM HEPES, 1.18 M NaCl, 480 mM KCl, 20 mM EDTA, 5 mM EGTA, pH 7.4 | |
| Ethanol (absolute for analysis) | Merck | 64-17-5 | |
| F(ab’)2 fragment anti-chicken IgY | Jackson Immunoresearch | AB_2340347 | Donkey polyclonal, 1:100 (SMLM), 1:1,000 (STED Microscopy) |
| F(ab’)2 fragment anti-mouse IgG | Jackson Immunoresearch | AB_2340761 | Donkey polyclonal, 1:100 (SMLM), 1:1,000 (STED Microscopy) |
| Fisherbrand Microscope slides T/F Ground 0.8-1.0 mm thick | Fisher scientific | 7107 | |
| Gauge Worm Pick 30 diameter 0.254 mm - Iridium 10% | Kisker | 789265 | |
| Glucose oxidase/Catalase enzyme mix (GlOX/Cat ) | a gift from Jonas Ries | Hoess, Mund, Reitberger, & Ries, 2018 | 20x, 1916 U/mL glucose oxidase (Sigma G7141), 42350 U/mL catalase (Sigma C3155), 50 mM Tris-HCl pH 8.0, 51% glycerol, MilliQ water. Stored at -20 °C. |
| Imaging buffer base | a gift from Jonas Ries | Hoess, Mund, Reitberger, & Ries, 2018 | 50 mM Tris-HCl, pH 8.0, 10 mM NaCl, 10% D-Glucose. Aliquoted to a single-use volume (950 μL), frozen, and kept at -80 °C. |
| Invitrogen ProLong Glass Antifade Mountant | ThermoFischer Scientific | P36982 | |
| Leica Stellaris 8 STED FALCON | Leica | N/A | The microscope is equiped with the latest generation white light laser, a 775nm pulsed STED laser, the FALCON Fluorescence Lifetime IMaging module, HC PL APO CS2 100x/1.40 oil objective, and Leica HyD X detector. The system is capable of FLIM module enhanced Tau-STED which measures the specific fluorescence lifetime of a dye and is therefore capable of removing background signal based on differences in fluorescence lifetimes of the dyes, and dye conditions in the sample. Additionally, the resolution is increased by accounting for the variation of fluorescence lifetimes in different areas of the depletion donut. |
| Longwave channel emission filter | AHF Analysentechnik | F47-702 | 700/100 nm bandpass |
| Methanol (absolute for analysis) | Merck | 67-56-1 | |
| NaHCO3 | Sigma-Aldrich/Merck | S5761-500G | 100 mM NaHCO3, pH 8.3 |
| Near-infrared fiber-coupled laser | Toptica | IBEAM-SMART-PT-CD | Custom Design, 808 nm - 75mW |
| Objective lens piezo mount (PIFOC ) | Physik Instrumente | P-726.1.CD | 100 µm travel range |
| Orca Fusion BT sCMOS camera | Hamamatsu | C15440-20UP | |
| PCR tubes | Greiner Bio-One | 673283 | 0.2 mL |
| Phosphate Saline Buffer (PBS 10x) | N/A | 137 mM NaCl, 2.7 mM KCl, 10 mM Na2HPO4, 1.8 mM KH2PO4, pH 7.4 | |
| Pierce 16% formaldehyde (w/v), methanol free | ThermoFischer Scientific | 28906 | 16% formaldehyde is transferred from the original glass ampule into 1.5 mL tube and kept at room temperature. |
| PlasmaPrep2 plasma cleaner | GaLa Instrumente GmbH | N/A | |
| Poly-L-lysine hydrobromide | Sigma-Aldrich/Merck | P2636-25MG | 0.1% w/v solution was prepared in Milli-Q water, and stored in aliquots at -20 °C. |
| Primary dichroic (illumination reflecting) | AHF Analysentechnik | F73-866S | quad bandpass @ 405, 488, 561, 640 |
| Quadnotch filter | AHF Analysentechnik | F40-072 | 405/488/561/640 nm |
| Quadrant photodiode (QPD) | Laser Components | SD197-23-21-041, LC301DQD-PV | |
| Razor blades | Apollo Herkenrath Solinger | N/A | |
| Refractive beam shaper | AdlOptica | PiShaper 6_6_VIS | |
| Roche Blocking Reagent | Roche | 11096176001 | 10x solution was prepared according to recommendation. Frozen aliquots were stored at -20 °C. |
| Scalpel blade (Feather brand #11, No. 3) | Heinz Herenz Medizinalbedarf GmbH | 1110911 | |
| Scalpel removal box | Fisher scientific | 10002-50 | |
| Secondary dichroic (emission reflecting) | AHF Analysentechnik | F38-785S | 750 nm longpass |
| Shortpass filter | Semrock | BSP01-785R-25 | 750 nm |
| Shortwave channel emission filter | AHF Analysentechnik | F37-677 | 676/37 nm bandpass |
| Single molecule localization microscope | EMBL Imaging Centre | Diekmann et al., 2020 with modifications | The microscope provides widefield epi-illumination via a single-mode fiber-coupled laser engine, additional booster laser, and refractive beam shaper to provide a uniform illumination field (Stehr et al, 2019). Widefield images are captured on a sCMOS camera and appropriate relay optics for a system magnification of 61x and a pixel size of 106 nm. For ratiometric imaging of spectrally overlapping far-red dyes, an image splitter produces two spectrally distinct images on the camera (splitting dichroic: 665 nm long pass, shortwave channel emission filter: 676/37 nm bandpass, longwave emission filter: 700/100 nm bandpass. An additional 405/488/561/640 nm quadnotch filter and 750 nm shortpass filter are common to the two paths and provide additional laser blocking). A compound cylindrical lens provides the astigmatism required for 3D imaging. To maintain a fixed focus across acquisitions exceeding 2 hours in time (comprising 200 000 - 250 000 images), focus locking is achieved by total internal reflection of a near-infrared fiber-coupled laser from the coverslip and subsequent height sensitive detection on a quadrant photodiode (QPD). The QPD signal provided closed-loop control of the objective lens piezo mount. For access to this microscope, refer to https://www.embl.org/about/info/imaging-centre or contact ic-contact@embl.de |
| Single-mode fiber-coupled multi-laser engine | Toptica | iCHROME MLE-LFA-HP | Provides widefield epi-illumination of 100 mW at 405, 488, 561, 640 nm |
| Splitting dichroic | AHF Analysentechnik | F48-665SG | 665 nm long pass |
| Square cover glass 22 x 22 mm No.1 | Menzel-Gläser; VWR | 630-2882 | |
| STAR 635P, NHS ester | Abberior | ST635P-0002-1MG | Dissolved in DMSO to 1 mM solution, aliquoted to single use volume, frozen and kept at -80 °C |
| Stereo microscope Stemi 305 Stand K LAB | Zeiss | N/A | |
| Tetramisole hydrochloride | Sigma-Aldrich/Merck | T1512-2G | 1% (w/v) solution was prepared in Milli-Q water. Frozen aliquots were stored at -20 °C. Thawed aliquot was kept at 4 °C and used for several months. |
| TetraSpeck Microspheres | ThermoFischer Scientific | T7279 | 0.1 µm, fluorescent blue/green/orange/dark red |
| Tris Saline Buffer (TBS 10x) | N/A | 200 mM Tris-HCl, 1.5 M NaCl, pH 7.5 | |
| TWEEN 20 | Sigma-Aldrich/Merck | P9416-50ML | Kept at room temperature in original packaging. |
| WormStuff worm pick | Kisker | 789277 | |
| XY microscope stage | Smaract | N/A | Custom Design |
| Zeba Micro Spin Desalting Column | ThermoFischer Scientific | 89877 | 7K MWCO, 75 µL |
References
- Zickler, D., Kleckner, N. Meiotic chromosomes: integrating structure and function. Annual Review of Genetics. 33, 603(1999).
- Ur, S. N., Corbett, K. D. Architecture and dynamics of meiotic chromosomes. Annual Review of Genetics. 55, 497-526 (2021).
- Fawcett, D. W. The fine structure ot chromosomes in the meiotic prophase of vertebrate spermatocytes. The Journal of Biophysical and Biochemical Cytology. 2 (4), 403-406 (1956).
- Moses, M. J. Chromosomal structures in crayfish spermatocytes. The Journal of Biophysical and Biochemical Cytology. 2 (2), 215-218 (1956).
- Hillers, K. J., Jantsch, V., Martinez-Perez, E., Yanowitz, J. L. Meiosis. WormBook. , 433-434 (2017).
- Pasierbek, P., et al. A Caenorhabditis elegans cohesion protein with functions in meiotic chromosome pairing and disjunction. Genes & Development. 15 (11), 1349-1360 (2001).
- Severson, A. F., Ling, L., Van Zuylen, V., Meyer, B. J. The axial element protein HTP-3 promotes cohesin loading and meiotic axis assembly in C. elegans to implement the meiotic program of chromosome segregation. Genes & Development. 23 (15), 1763-1778 (2009).
- Zetka, M. C., Kawasaki, I., Strome, S., Müller, F. Synapsis and chiasma formation in Caenorhabditis elegans require HIM-3, a meiotic chromosome core component that functions in chromosome segregation. Genes & Development. 13 (17), 2258-2270 (1999).
- Martinez-Perez, E. HTP-1-dependent constraints coordinate homolog pairing and synapsis and promote chiasma formation during C. elegans meiosis. Genes & Development. 19 (22), 2727-2743 (2005).
- Couteau, F., Zetka, M. HTP-1 coordinates synaptonemal complex assembly with homolog alignment during meiosis in C. elegans. Genes & Development. 19 (22), 2744-2756 (2005).
- Goodyer, W., et al. HTP-3 Links DSB Formation with Homolog Pairing and Crossing Over during C. elegans Meiosis. Developmental Cell. 14 (2), 263-274 (2008).
- Colaiácovo, M. P., et al. Synaptonemal complex assembly in C. elegans is dispensable for loading strand-exchange proteins but critical for proper completion of recombination. Developmental Cell. 5 (3), 463-474 (2003).
- MacQueen, A. J., Colaiácovo, M. P., McDonald, K., Villeneuve, A. M. Synapsis-dependent and -independent mechanisms stabilize homolog pairing during meiotic prophase in C. elegans. Genes & Development. 16 (18), 2428-2442 (2002).
- Smolikov, S., et al. Synapsis-defective mutants reveal a correlation between chromosome conformation and the mode of double-strand break repair during Caenorhabditis elegans meiosis. Genetics. 176 (4), 2027-2033 (2007).
- Smolikov, S., Schild-Prüfert, K., Colaiácovo, M. P. A yeast two-hybrid screen for SYP-3 interactors identifies SYP-4, a component required for synaptonemal complex assembly and chiasma formation in Caenorhabditis elegans meiosis. PLoS Genetics. 5 (10), 1000669(2009).
- Hurlock, M. E., et al. Identification of novel synaptonemal complex components in C. Elegants. The Journal of Cell Biology. 219 (5), (2020).
- Zhang, Z., et al. Multivalent weak interactions between assembly units drive synaptonemal complex formation. The Journal of Cell Biology. 219 (5), (2020).
- Schild-Prüfert, K., et al. Organization of the synaptonemal complex during meiosis in Caenorhabditis elegans. Genetics. 189 (2), 411-421 (2011).
- Köhler, S., Wojcik, M., Xu, K., Dernburg, A. F. The interaction of crossover formation and the dynamic architecture of the synaptonemal complex during meiosis. bioRxiv. , (2020).
- Chen, F., Tillberg, P. W., Boyden, E. S. Expansion microscopy. Science. 347 (6621), 543-548 (2015).
- Klar, T. A., Jakobs, S., Dyba, M., Egner, A., Hell, S. W. Fluorescence microscopy with diffraction resolution barrier broken by stimulated emission. Proceedings of the National Academy of Sciences. 97 (15), 8206-8210 (2000).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-796 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Schücker, K., Holm, T., Franke, C., Sauer, M., Benavente, R. Elucidation of synaptonemal complex organization by super-resolution imaging with isotropic resolution. Proceedings of the National Academy of Sciences. 112 (7), 2029-2033 (2015).
- Cahoon, C. K., et al. Superresolution expansion microscopy reveals the three-dimensional organization of the Drosophila synaptonemal complex. Proceedings of the National Academy of Sciences. 114 (33), 6857-6866 (2017).
- Zwettler, F. U., et al. Tracking down the molecular architecture of the synaptonemal complex by expansion microscopy. Nature Communications. 11 (1), 1-11 (2020).
- Yoon, S., Choi, E. H., Kim, J. W., Kim, K. P. Structured illumination microscopy imaging reveals localization of replication protein A between chromosome lateral elements during mammalian meiosis. Experimental & Molecular Medicine. 50 (8), 1-12 (2018).
- Prakash, K., et al. Superresolution imaging reveals structurally distinct periodic patterns of chromatin along pachytene chromosomes. Proceedings of the National Academy of Sciences. 112 (47), 14635-14640 (2015).
- Xu, H., et al. Molecular organization of mammalian meiotic chromosome axis revealed by expansion STORM microscopy. Proceedings of the National Academy of Sciences. 116 (37), 18423-18428 (2019).
- Köhler, S., Wojcik, M., Xu, K., Dernburg, A. F. Superresolution microscopy reveals the three-dimensional organization of meiotic chromosome axes in intact Caenorhabditis elegans tissue. Proceedings of the National Academy of Sciences. 114 (24), 4734-4743 (2017).
- Hell, S. W. Far-field optical nanoscopy. Science. 316 (5828), 1153-1158 (2007).
- Hein, B., Willig, K. I., Hell, S. W. Stimulated emission depletion (STED) nanoscopy of a fluorescent protein-labeled organelle inside a living cell. Proceedings of the National Academy of Sciences. 105 (38), 14271-14276 (2008).
- Jahr, W., Velicky, P., Danzl, J. G. Strategies to maximize performance in STimulated Emission Depletion (STED) nanoscopy of biological specimens. Methods. 174, 27-41 (2019).
- Thevathasan, J. V., et al. Nuclear pores as versatile reference standards for quantitative superresolution microscopy. Nature Methods. 16 (10), 1045-1053 (2019).
- Xu, K., Shim, S. -H., Zhuang, X. Super-resolution imaging through stochastic switching and localization of single molecules: an overview. Far-Field Optical Nanoscopy. , 27-64 (2013).
- Wang, Y., et al. Localization events-based sample drift correction for localization microscopy with redundant cross-correlation algorithm. Optics Express. 22 (13), 15982(2014).
- Winterflood, C. M., Platonova, E., Albrecht, D., Ewers, H. Dual-color 3D superresolution microscopy by combined spectral-demixing and biplane imaging. Biophysical Journal. 109 (1), 3-6 (2015).
- Diekmann, R., et al. Optimizing imaging speed and excitation intensity for single molecule localization microscopy. Nature Methods. 17 (9), 909(2020).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-dimensional super-resolution imaging by stochastic optical reconstruction microscopy. Science. 319 (5864), 810-813 (2008).
- Phillips, C. M., McDonald, K. L., Dernburg, A. F. Cytological analysis of meiosis in Caenorhabditis elegans. Methods in Molecular Biology. 558, 171-195 (2009).
- Edgar, L. G. Blastomere culture and analysis. Methods in Cell Biology. 48, 303-321 (1995).
- MacQueen, A. J., et al. Chromosome sites play dual roles to establish homologous synapsis during meiosis in C. elegans. Cell. 123 (6), 1037-1050 (2005).
- Stehr, F., Stein, J., Schueder, F., Schwille, P., Jungmann, R. Flat-top TIRF illumination boosts DNA-PAINT imaging and quantification. Nature Communications. 10 (1), 1-8 (2019).
- Hoess, P., Mund, M., Reitberger, M., Ries, J. Dual-color and 3D super-resolution microscopy of multi-protein assemblies. Methods in Molecular Biology. 1764, 237-251 (2018).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer Control of microscopes using µManager. Current Protocols in Molecular Biology. 92 (1), 14-20 (2010).
- Edelstein, A. D., et al. Advanced methods of microscope control using µManager software. Journal of Biological Methods. 1 (2), 10(2014).
- Deschamps, J., Ries, J. EMU: reconfigurable graphical user interfaces for Micro-Manager. BMC Bioinformatics. 21 (1), 1-13 (2020).
- Ries, J. SMAP: a modular super-resolution microscopy analysis platform for SMLM data. Nature Methods. 17 (9), 870-872 (2020).
- Li, Y., et al. Global fitting for high-accuracy multi-channel single-molecule localization. Nature Communications. 13 (1), 1-11 (2022).
- Nieuwenhuizen, R. P. J., et al. Measuring image resolution in optical nanoscopy. Nature Methods. 10 (6), 557-562 (2013).
- Banterle, N., Bui, K. H., Lemke, E. A., Beck, M. Fourier ring correlation as a resolution criterion for super-resolution microscopy. Journal of Structural Biology. 183 (3), 363-367 (2013).
- Preibisch, S., Saalfeld, S., Tomancak, P. Globally optimal stitching of tiled 3D microscopic image acquisitions. Bioinformatics. 25 (11), 1463-1465 (2009).
- Shi, X., Lim, J., Ha, T. Acidification of the oxygen scavenging system in single-molecule fluorescence studies: in situ sensing with a ratiometric dual-emission probe. Analytical Chemistry. 82 (14), 6132-6138 (2010).
- Olivier, N., Keller, D., Rajan, V. S., Gönczy, P., Manley, S. Simple buffers for 3D STORM microscopy. Biomedical Optics Express. 4 (6), 885-899 (2013).
- Mund, M., et al. Superresolution microscopy reveals partial preassembly and subsequent bending of the clathrin coat during endocytosis. bioRxiv. , (2022).
- Mund, M., et al. Systematic nanoscale analysis of endocytosis links efficient vesicle formation to patterned actin nucleation. Cell. 174 (4), 884-896 (2018).
- Sabinina, V. J., et al. Three-dimensional superresolution fluorescence microscopy maps the variable molecular architecture of the nuclear pore complex. Molecular Biology of the Cell. 32 (17), 1523-1533 (2021).
- Cieslinski, K., et al. Nanoscale structural organization and stoichiometry of the budding yeast kinetochore. bioRxiv. , (2021).
- Sieben, C., Banterle, N., Douglass, K. M., Gönczy, P., Manley, S. Multicolor single-particle reconstruction of protein complexes. Nature Methods. 15 (10), 777-780 (2018).
- Sarkans, U., et al. The BioStudies database—one stop shop for all data supporting a life sciences study. Nucleic Acids Research. 46, (2018).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved