È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Purificazione di Hsp104, una proteina Disaggregase
In questo articolo
Riepilogo
Qui, descriviamo un protocollo per la purificazione del altamente attivi Hsp104, un esamerica AAA + proteine da lievito, che le coppie idrolisi dell'ATP per disaggregazione delle proteine. Questo schema sfrutta un costrutto His6-tag per la purificazione di affinità E. coli Seguita da cromatografia a scambio anionico, His6-tag rimozione di proteasi TEV, e le dimensioni di esclusione cromatografia.
Abstract
Hsp104 è un esamerica AAA + 1 da proteine del lievito, che le coppie idrolisi dell'ATP alle proteine disaggregazione 2-10 (Fig. 1). Questa attività conferisce due vantaggi chiave selettiva. In primo luogo, rinaturazione di aggregati disordinati di Hsp104 consente la sopravvivenza di lievito, dopo varie sollecitazioni misfolding di proteine, tra cui shock termico 3,5,11,12. In secondo luogo, rimodellamento di cross-beta fibrille amiloidi da Hsp104 lievito permette di sfruttare una miriade di prioni (amiloidi infettive) come un serbatoio di benefici e ereditabile variazione fenotipica 13-22. Sorprendentemente, Hsp104 rimodella direttamente preamyloid oligomeri e fibrille amiloidi, compresi compresi quelli di proteine prioniche il lievito Sup35 e Ure2 23-30. Questo amiloide-rimodellamento funzionalità è un aspetto specializzato di lievito Hsp104. La E. ortologo coli, ClpB, non riesce a oligomeri preamyloid rimodellare o fibrille amiloidi 26,31,32.
Ortologhi Hsp104 si trovano in tutti i regni della vita tranne, stranamente, gli animali. Infatti, se le cellule animali possiedono un sistema enzimatico che le proteine coppie disaggregazione di rinaturazione (piuttosto che degrado) rimane sconosciuto 33-35. Così, noi e altri hanno proposto che Hsp104 potrebbe essere sviluppato come agente terapeutico per varie malattie neurodegenerative legate alla misfolding di proteine specifiche in oligomeri preamyloid tossici e fibrille amiloidi 4,7,23,36-38. Non ci sono trattamenti che riguardano direttamente la specie aggregate associati a queste malattie. Eppure, Hsp104 dissolve oligomeri tossici e fibrille amiloidi composto da alfa-sinucleina, che sono collegati con la malattia di Parkinson 23 così come le forme di PrP amiloide 39. Importante, Hsp104 riduce l'aggregazione delle proteine e migliora neurodegenerazione in modelli di roditori della malattia di Parkinson 23 e la malattia di Huntington 38. Idealmente, per ottimizzare la terapia e minimizzare gli effetti collaterali, Hsp104 sarebbe progettato e potenziato per rimodellare selettivamente aggregati specifici centrale per la patologia in questione 4,7. Tuttavia, la limitata comprensione strutturale e meccanicistica di come Hsp104 disaggrega ad un repertorio eterogeneo di strutture aggregate e proteine non correlate frustra questi tentativi 30,40-42.
Per comprendere la struttura e il meccanismo di Hsp104, è essenziale per lo studio della proteina pura e ricostituire la sua attività disaggregase con componenti minimi. Hsp104 è una proteina 102kDa con un pI di ~ 5.3, che hexamerizes in presenza di ADP e ATP, oppure a concentrazioni di proteine in assenza di 43-46 nucleotidi. Qui, descriviamo un protocollo ottimizzato per la purificazione di molto attiva, stabile Hsp104 da E. coli. L'utilizzo di E. coli permette semplificata produzione su larga scala e il nostro metodo può essere eseguita in modo rapido e affidabile per Hsp104 numerose varianti. Il nostro protocollo aumenta Hsp104 purezza e semplifica la sua 6 tag rimozione rispetto ad un metodo di purificazione precedente da E. coli 47. Inoltre, il nostro protocollo è più facile e conveniente di due protocolli più recenti 26,48.
Protocollo
1. Espressione di Hsp104
- Il plasmide utilizzato per la purificazione in E. coli, pPROEX-HTB-Hsp104, contiene la Hsp104 open-lettura telaio sotto il controllo del promotore inducibile trc 26. Il plasmide produce Hsp104 con un N-terminale suoi 6-tag che possono essere rimossi per scissione della proteasi TEV. Trasforma pPROEX-HTB-Hsp104 in codone ottimizzato E. coli BL21-CodonPlus RIL-cellule (Stratagene, Agilent Technologies) utilizzando una procedura tipica trasformazione batterica (per esempio in base alle istruzioni del produttore). E 'importante utilizzare un codone ottimizzato E. coli ceppo Hsp104 perché ha un pregiudizio insolito codone.
- Inoculare una cultura 2XYT 100 ml (Ricetta USB.: 16g / l di peptone di caseina, 10 g / l estratto di lievito, 5 g / L di NaCl, pH 7,0) integrato con 100μg/mL ampicillina e cloramfenicolo 34μg/mL con trasformanti fresca e crescere overnight a 37 ° C con agitazione a 200gpm.
- Inoculare 10 mL della cultura durante la notte in 6 X 1L 2XYT con 100μg/mL 34μg/mL ampicillina e cloramfenicolo in fiaschi 2L. Agitare a 250rpm a 37 ° C fino OD 600 = ,4-0,6. A smettere di tremare e ridurre la temperatura a 15 ° C. Consentono alle cellule di equilibrare unagitated per 30min nell'incubatrice fino a raggiungere 15 ° C. Una volta a 15 ° C viene raggiunta indurre l'espressione delle proteine con l'aggiunta di IPTG ad una concentrazione finale di 1 mm. Riprendi agitazione a 250rpm notte (12-16 ore).
2. Raccolta e lisi delle cellule
- Harvest cellule mediante centrifugazione (in un rotore preraffreddato) a 4.000 rpm per 20min a 4 ° C (usiamo un Sorvall RC 3BP centrifuga +). I passi successivi devono essere eseguiti immediatamente, perché Hsp104 attività è diminuito quando E. coli sono congelati. Inoltre, tutti i passi successivi devono essere eseguiti su ghiaccio o a 4 ° C.
- Pellet cellulari risospendere in prechilled (su ghiaccio) 10 ml tampone di lisi (40mm HEPES-KOH pH 7,4, 500mm KCl, 20 mM MgCl 2, 2.5% (w / v) glicerolo, 20 mM imidazolo, 5 micron pepstatina A, completo cocktail inibitore della proteasi (1 EDTA senza tablet/50mL), e 2mM β-mercaptoetanolo).
- Lisare le cellule con una stampa francese (Emulsiflex) omogeneizzatore che utilizza uno scambiatore di calore immerso in acqua ghiacciata. Prima dell'uso, assicurarsi che tutti i grumi di cellule sono state solubilizzate. Dopo equilibrante omogeneizzatore con tampone di lisi, due passa attraverso il cilindro con una pressione di 15.000-18.000 psi è sufficiente per lisi completa. Se una stampa francese non è disponibile, l'incubazione con lisozima seguita da sonicazione può essere utilizzato (vedi discussione). Salva un piccolo campione di cellule lisate per l'analisi SDS-PAGE (1ml) (lisato in fig. 2).
- Rimuovere i detriti cellulari mediante centrifugazione a 16.000 rpm per 20min a 4 ° C (usiamo un RC5C Sorvall centifuge +). Conservare il supernatante per la fase successiva e salvare un piccolo campione (1ml) di supernatante per SDS-PAGE analisi (Load Ni in fig. 2).
3. Hsp104 Purificazione
- Mix surnatante dal punto 2.4 con 12mL (2 ml Ni-Sepharose perle per 1L di cellule) del 50% impasto di buffer di lisi equilibrata Ni-Sepharose perline (GE).
- Ruotare il campione lentamente per 3 ore a 4 ° C in modo tale che Ni-Sepharose rimane fluttuante e schiumare è ridotto al minimo. Tempi di incubazione più brevi sono possibili, ma diminuisce la resa. A 4 ° C in presenza di inibitori della proteasi, il degrado si verifica poco, e un 3 ore al Ni-Sepharose tempo di incubazione non diminuisce l'attività del prodotto finale. Raccogliere Ni-Sepharose tramite centrifugazione per 2 minuti a 4 ° C a 2.000 rpm (centrifuga Eppendorf 5810R). Salva un piccolo campione di surnatante per SDS-PAGE analisi (1ml) e scartare il resto (flusso Ni Thru (FT) in fig. 2).
- Lavare i recuperate Ni-Sepharose con 25 volumi di colonna di tampone di lavaggio (40mm HEPES-KOH pH 7,4, 150mm KCl, 20 mM MgCl 2, 2.5% (w / v) glicerolo, 20 mM imidazolo, 2mM β-mercaptoetanolo), 5 volumi di colonna tampone di lavaggio con KCl 1M per rimuovere i contaminanti legato elettrostaticamente a Hsp104, e 25 volumi di colonna di tampone di lavaggio. Raccogliere Ni-Sepharose dopo ogni lavaggio mediante centrifugazione per 2 minuti a 4 ° C a 2.000 rpm (centrifuga Eppendorf 5810R). Dopo ogni ciclo di lavaggio e centrifugazione, rimuovere tampone per aspirazione.
- Per eluire Hsp104, mescolare Ni-Sepharose con 1 ml di tampone di eluizione (40mm HEPES-KOH pH 7,4, 150mm KCl, 20 mM MgCl 2, 2.5% (w / v) glicerolo, 350mm imidazolo, 2mM β-mercaptoetanolo) per 1 ml e Ni-Sepharose rotazione a 4 ° C per 20 minuti. L'alta concentrazione imidazolo interrompe il suo tallone-Ni 6 interazione. Rimuovere Ni-Sepharose con colonne vuote 1,2 ml di spin (Bio-Rad) tramite centrifugazione per 2 minuti a 4 ° C a 2.000 rpm (centrifuga Eppendorf 5810R). Salva 1ml di eluato per SDS-PAGE analisi (eluato Ni in fig. 2). Per ogni litro di cellule, ~ 15 mg di Hsp104 si ottiene in questa fase.
- Buffer eluato scambio in Buffer Q (20 mM Tris-HCl pH 8, EDTA 0,5 mm, 5mm MgCl 2, 50 mM NaCl) usando MWCO 30.000 Amicon Ultra (Millipore) 15 ml di unità concentratore centrifugo. In primo luogo, concentrato di proteine di ~ 1,5 ml quindi aggiungere 14.5mL di tampone di Q per diluire proteina di 10 volte. Ripetere 3 volte per lo scambio di buffer. L'aumento del pH e sale a bassa assicura che Hsp104 ha una carica negativa alto.
- Purificare Hsp104 tramite cromatografia a scambio anionico utilizzando un buffer equilibrato Q colonna Risorsa Q 6 ml (GE). Prima dell'iniezione, la proteina viene filtrata attraverso un basso legame proteico Millex GP PES membrana 0.22μM siringa filtro (Millipore). Ci avvaliamo di una portata di 1mL/min. Lavare via debolmente proteine di legame con 1 volume colonna del 20% Buffer Q + (20 mM Tris-HCl pH 8, EDTA 0,5 mm, 5mm MgCl 2, 1M NaCl). Eluire Hsp104 con sfumatura lineare (20% -50% Buffer Q +) più di 5 volumi di colonna (Fig. 3). Raccogliere frazioni da 1 ml. Hsp104 e la maggior parte delle varianti tipicamente eluire a ~ 400mm NaCl (31mS/cm). Salva campioni delle frazioni di picco (1ml) per SDS-PAGE analisi (Fig. 3, nel riquadro).
- Per rimuovere la sua 6-tag, proteine di scambio con il concentratore centrifugo Amicon come descritto sopra (vedi punto 3.5) nel buffer della scissione (20 mM HEPES-KOH pH 7,4, KCl 140mm, e 10mM MgCl 2). Usa proTEV proteasi (Promega) o AcTEV proteasi (Invitrogen) secondo le istruzioni del produttore. Per Hsp104, abbiamo trovato che il più alto rapporto di proteasi TEV: Hsp104 sono necessari per la scissione totale. Usiamo un rapporto di 1 unità di proteasi per 12μg Hsp104. Scissione deve essere eseguita a 30 ° C per 2-4 ore, seguita da una incubazione 16 ore a 4 ° C. Concentrazione finale Hsp104 dovrebbe essere tra 20-75μM monomero. Riducono le rimanenti Hsp104 His6 marcatura ed proteasi TEV con Ni-Sepharose (GE) con l'aggiunta di Ni-Sepharose in eccesso alla quantità di Hsp104 nella reazione della scissione (assumere perle può legare 15 mg di proteina per ml di resina imballato). Rimuovere perline con colonne di rotazione vuoto (Bio-Rad) per centrifugazione per 2 minuti a 2.000 rpm a 4 ° C. Raccogliere i campioni prima e dopo la scissione (1ml) per SDS-PAGE analisi per garantire la scissione, e la rimozione delle proteine uncleaved (Fig. 4).
- Scambio in dimensione del buffer di esclusione (20 mM HEPES-KOH pH 7,4, 140mm KCl, 10mM MgCl 2, 0.1mm EDTA, 1 mM DTT) utilizzando MWCO 30.000 Amicon Ultra (Millipore), come descritto al punto 3.5.
- Ulteriori purificare Hsp104 via dimensione-cromatografia di esclusione. Usa esclusione Superose dimensione del buffer equilibrata 6 o 200 Superdex colonna (sia da GE) (Fig. 5). Prima dell'iniezione, la proteina viene filtrata attraverso un basso legame proteico Millex GP PES membrana 0.22μM siringa filtro (Millipore). Per meno di 10 mg di proteine, le colonne di dimensioni 10/300 (24ml) può essere utilizzato e le frazioni di 0,5 ml sono stati raccolti. Per i campioni maggiori di 10 mg, la colonna dimensione 12/60 (120ml) dovrebbe essere usato e 1 ml frazioni vengono raccolte. Fare riferimento alle istruzioni del produttore per flussi e volumi di campione da caricare. Dopo la purificazione, frazioni Hsp104 sono riuniti come mostrato in fig. 5 e concentrata in Unità Concentratore Amicon centrifuga (Millipore). Hsp104 viene memorizzato come descritto di seguito. A causa della perdita di materiale a separare Hsp104 prodotti di degradazione e contaminanti la resa finale del purificata full-length Hsp104 è ~ 1-3mg per litro di partenza cultura.
4. Hsp104 Disaggregase attività
- Dopo la purificazione, è buona per valutare Hsp104 attività in un test di disgregazione. Tipicamente, ci avvaliamo di un saggio di luciferasi riattivazione 2 (Fig. 6). In questo saggio, Hsp104 in collaborazione con il sistema di chaperone Hsp70 (Hsp70 e Hsp40) disaggrega urea-denaturato aggregati luciferasi lucciola 2. Solubile luciferasi catalizza l'ossidazione della luciferina ad oxyluciferin, una reazione che libera la luce. Dalla luminescenza monitoraggio, la riattivazione relativa, e quindi disaggregazione, di luciferasi può essere determinato. Hsp104 deve essere in grado di collaborare in sinergia con il co-chaperone Hsp70 sistema. La quantità di luminescenza recuperato dipende dalla Hsp70 esatta: coppia Hsp40 utilizzati. Noi abitualmente utilizzano Hsp72 e Hsp40 (disegni Assay). Come minimo, attivo Hsp104 dovrebbe produrre un aumento di 5 volte nella riattivazione della luciferasi rispetto alla riattivazione di Hsp72: Hsp40 solo (Fig. 6). Hsp104 preparati che non riescono a raggiungere questo livello di attività vengono scartati.
5. Hsp104 archiviazione
- Per la conservazione a breve termine, Hsp104 può essere conservato a 4 ° C in formato esclusione buffer. Tuttavia, l'attività si ridurrà dopo 2-3 giorni a 4 ° C e bruscamente dopo 1 settimana a 4 ° C. Per i saggi di disaggregazione si consiglia di Hsp104 deve essere utilizzato il più presto possibile dopo la purificazione. Idealmente, Hsp104 deve essere utilizzato immediatamente per le analisi disaggregazione amiloide.
- Se le proteine devono essere conservati a lungo termine, Hsp104 viene scambiato nella soluzione per la conservazione (20 mM HEPES-KOH pH 7,4, 140mm KCl, 10mM MgCl 2, 0.1mm EDTA, 1mM DTT, 10% glicerolo (w / v)). Aliquote 100μl sono a scatto congelato in azoto liquido e conservati a -80 ° C. Cicli di gelo-disgelo ridurre drasticamente Hsp104 attivitàe deve essere evitato. Si consiglia di scongelare lentamente Hsp104 sul ghiaccio. -Amiloide disaggregase attività si ridurrà drasticamente dopo 1 mese a -80 ° C.
6. Risultati Rappresentante e figure:
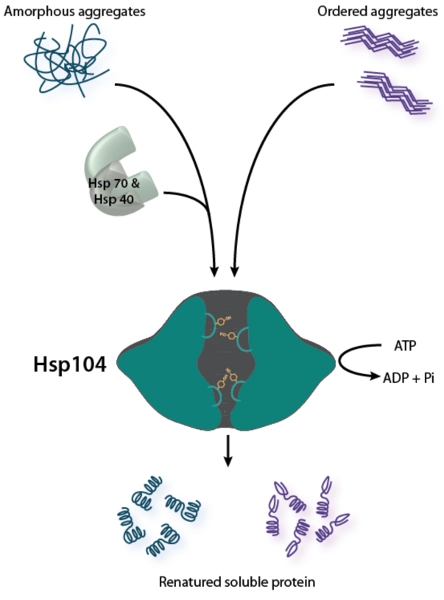
Figura 1. Hsp104 è un disaggregase bifunzionali. Disaggregazione degli aggregati disordinati (mostrato a sinistra) richiede la cooperazione del sistema chaperone Hsp70 (Hsp70 e Hsp40) 2. Rimodella Hsp104 ordinato aggregati amiloidi (illustrato a destra) senza l'ausilio di Hsp70 e Hsp40 in vitro, ma Hsp70 e Hsp40 può migliorare Hsp104 attività contro amiloide 26,28. Per entrambe le tipologie di strutture aggregate, Hsp104 coppie idrolisi dell'ATP per traslocazione del substrato attraverso il suo canale centrale per promuovere disaggregazione. Tirosina-cuscinetto loop pori coinvolgere e substrato navetta attraverso il canale centrale 49-52.

Figura 2. SDS-PAGE analisi di Ni-sefarosio passo affinità purificazione. Lisato, carico Ni, Ni FT e campioni di eluato Ni sono stati frazionati mediante SDS-PAGE utilizzando un 4-20% Tris-HCl 1,0 millimetri Criterion gel (Bio-Rad) e colorate Coomassie . Si noti che non tutti i Hsp104 è in grado di legarsi al Ni-sefarosio. Ampia Gamma di marcatori di peso molecolare (Bio-Rad) sono mostrati (corsia sinistra).
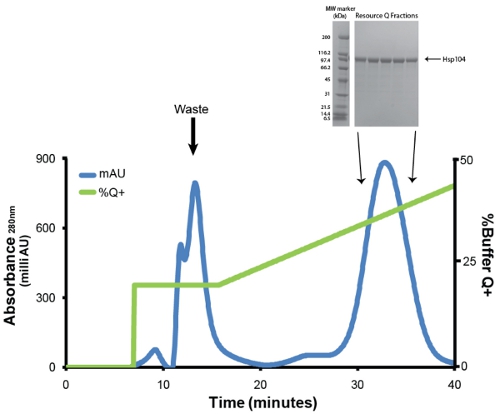
Figura 3. Risorsa purificazione Q di Ni-sefarosio eluato. Traccia blu rappresenta l'assorbanza a 280 nm e traccia verde rappresenta% Buffer Q + (massimo al 50%). Il primo picco, che eluisce al 20% + Q contiene impurità, prodotti di degradazione e piegato impropriamente Hsp104. Il secondo picco e le principali contiene accuratamente piegati e attiva Hsp104. Portata era 1ml/min. Gradiente che va dal 20-50% Q + è di 30 minuti o 5 volumi di colonna. Riquadro: frazioni di picco vengono risolte mediante SDS-PAGE analisi utilizzando un 4-20% Tris-HCl 1,0 millimetri Criterion gel (Bio-Rad) e colorate Coomassie. Ampia Gamma di marcatori di peso molecolare (Bio-Rad) sono indicati (a sinistra).

Figura 4. SDS-PAGE analisi del passo clivaggio della proteasi proTEV. Suoi 6-Hsp104 dalla purificazione delle risorse Q è stato trattato con proteasi proTEV per 4 ore a 30 ° C e poi 16 ore a 4 ° C. I campioni sono stati frazionati mediante la SDS-PAGE utilizzando un 4-20% Tris-HCl 1,0 millimetri Criterion gel (Bio-Rad) e colorate Coomassie. Si noti che proTEV spaccati Hsp104 migra più rapidamente. TEV proteasi e uncleaved Hsp104 sono state esaurite con batterie Ni-Sepharose come descritto al punto 3.7. Ampia Gamma di marcatori di peso molecolare (Bio-Rad) sono indicati (a sinistra).
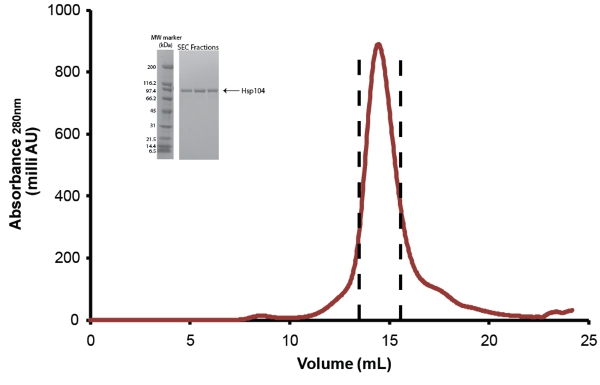
Figura 5. Taglia-esclusione di purificazione Hsp104. Spaccati Hsp104 è stato ulteriormente purificato attraverso una colonna 6 gel filtrazione Superose (10/300, GE). = Portata 0.4ml/min. Il picco tra le linee tratteggiate rappresentano frazioni pool. Riquadro: frazioni di picco vengono risolte mediante SDS-PAGE analisi utilizzando un 4-20% Tris-HCl 1,0 millimetri Criterion gel (Bio-Rad) e colorate Coomassie. Ampia Gamma di marcatori di peso molecolare (Bio-Rad) sono indicati (a sinistra).
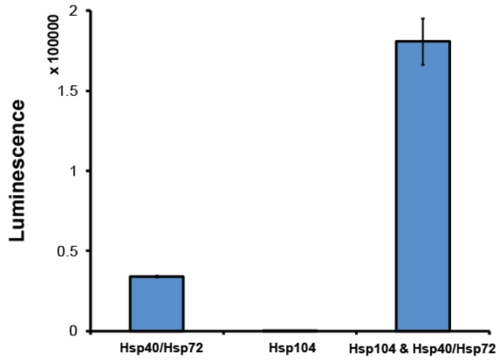
Figura 6. Saggio riattivazione luciferasi. Denaturato aggregati lucciola luciferasi (50nm) sono state incubate sia con Hsp72 e Hsp40 (1μM) (entrambi dal design Assay), Hsp104 (6μM monomero) o Hsp104, Hsp72 e Hsp40 per 90min a 25 ° C. Luciferasi denaturato è solo completamente riattivato in presenza di entrambi i Hsp104 e Hsp40/Hsp72. Luminescenza recuperato viene misurato su una piastra lettore Infinite M1000 (Tecan). Valori rappresentano le medie ± SEM (n = 3).
Discussione
Timeline: per la massima attività Hsp104 raccomandiamo che il sistema di purificazione intero essere completato il più rapidamente possibile. Tuttavia, il numero di passaggi di purificazione rende un programma impegnativo che non può sempre essere pratico. Se le fasi di purificazione vengono eseguite più rapidamente possibile, il tempo a partire dalla fine di espressione durante la notte fino alle 2-4 ore di incubazione a 30 ° C con proteasi TEV è di circa 9-11 ore. Un posto potenziale per mettere in pausa ...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo lavoro è stato sostenuto da una borsa di studio del NIH (5T32GM008275-22) e un americano comunione Heart Association predoctoral (a EAS), una Chimica-Biologia Fellowship interfaccia dal NIH (2T32GM071339-06A1) (MED); e sovvenzioni da parte NIH (1DP2OD002177-01 e NS067354-0110), Il Ellison Medical Foundation e la Fondazione Bill e Melinda Gates Foundation (per JS).
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | |
|---|---|---|---|
| BL21-CodonPlus-RIL cellule competenti | Stratagene, Agilent Technologies | 230255 | |
| 2XYT brodo | USB | 75864 | |
| Completi, mini, EDTA senza inibitori della proteasi compresse | Roche | 1836170 | |
| Un pepstatina | Sigma | P4265 | |
| Ni-Sepharose 6 flusso veloce | GE Healthcare | 17-5318-02 | |
| Amicon Ultra-15 unità centrifughe filtro (MWCO 30.000) | Millipore | UFC903008 | |
| Risorsa Q - colonna 6ml | GE Healthcare | 17-1179-01 | |
| proTEV proteasi | Promega | V6052 | |
| AcTEV proteasi | Invitrogen | 12575015 | |
| Superose sei 10/300 GL | GE Healthcare | 17-5172-01 | |
| Hsp40 | Assay Designs | SPP-400 | |
| Hsp72 | Assay Designs | ADI-NSP-555 |
Riferimenti
- Erzberger, J. P., Berger, J. M. Evolutionary relationships and structural mechanisms of AAA+ proteins. Annu Rev Biophys Biomol Struct. 35, 93-114 (2006).
- Glover, J. R., Lindquist, S. Hsp104, Hsp70, and Hsp40: A Novel Chaperone System that Rescues Previously Aggregated Proteins. Cell. 94, 73-82 (1998).
- Parsell, D. A., Kowal, A. S., Singer, M. A., Lindquist, S. Protein disaggregation mediated by heat-shock protein Hsp104. Nature. 372, 475-478 (1994).
- Vashist, S., Cushman, M., Shorter, J. Applying Hsp104 to protein-misfolding disorders. Biochem Cell Biol. 88, 1-13 (2010).
- Parsell, D. A., Sanchez, Y., Stitzel, J. D., Lindquist, S. Hsp104 is a highly conserved protein with two essential nucleotide-binding sites. Nature. 353, 270-273 (1991).
- Doyle, S. M., Wickner, S. Hsp104 and ClpB: protein disaggregating machines. Trends Biochem Sci. 34, 40-48 (2009).
- Shorter, J. Hsp104: a weapon to combat diverse neurodegenerative disorders. Neurosignals. 16, 63-74 (2008).
- Glover, J. R., Lum, R. Remodeling of protein aggregates by Hsp104. Protein Pept Lett. 16, 587-597 (2009).
- Mogk, A., Haslberger, T., Tessarz, P., Bukau, B. Common and specific mechanisms of AAA+ proteins involved in protein quality control. Biochem Soc Trans. 36, 120-125 (2008).
- Grimminger-Marquardt, V., Lashuel, H. A. Structure and function of the molecular chaperone Hsp104 from yeast. Biopolymers. 93, 252-276 (2010).
- Sanchez, Y., Lindquist, S. L. HSP104 required for induced thermotolerance. Science. 248, 1112-1115 (1990).
- Sanchez, Y., Taulien, J., Borkovich, K. A., Lindquist, S. Hsp104 is required for tolerance to many forms of stress. Embo J. 11, 2357-2364 (1992).
- Alberti, S., Halfmann, R., King, O., Kapila, A., Lindquist, S. A systematic survey identifies prions and illuminates sequence features of prionogenic proteins. Cell. 137, 146-158 (2009).
- Halfmann, R., Alberti, S., Lindquist, S. Prions, protein homeostasis, and phenotypic diversity. Trends Cell Biol. 20, 125-1233 (2010).
- Shorter, J., Lindquist, S. Prions as adaptive conduits of memory and inheritance. Nat Rev Genet. 6, 435-450 (2005).
- True, H. L., Berlin, I., Lindquist, S. L. Epigenetic regulation of translation reveals hidden genetic variation to produce complex traits. Nature. 431, 184-187 (2004).
- True, H. L., Lindquist, S. L. A yeast prion provides a mechanism for genetic variation and phenotypic diversity. Nature. 407, 477-483 (2000).
- Tyedmers, J., Madariaga, M. L., Lindquist, S. Prion switching in response to environmental stress. PLoS Biol. 6, e294-e294 (2008).
- Chernoff, Y. O., Lindquist, S. L., Ono, B., Inge-Vechtomov, S. G., Liebman, S. W. Role of the chaperone protein Hsp104 in propagation of the yeast prion-like factor [psi+]. Science. 268, 880-884 (1995).
- Halfmann, R., Lindquist, S. Epigenetics in the extreme: prions and the inheritance of environmentally acquired traits. Science. 330, 629-632 (2010).
- Satpute-Krishnan, P., Langseth, S. X., Serio, T. R. Hsp104-dependent remodeling of prion complexes mediates protein-only inheritance. PLoS Biol. 5, e24-e24 (2007).
- Sweeny, E. A., Shorter, J. Prion proteostasis: Hsp104 meets its supporting cast. Prion. 2, 135-140 (2008).
- Lo Bianco, C. Hsp104 antagonizes alpha-synuclein aggregation and reduces dopaminergic degeneration in a rat model of Parkinson disease. J Clin Invest. 118, 3087-3097 (2008).
- Narayanan, S., Walter, S., Reif, B. Yeast prion-protein, sup35, fibril formation proceeds by addition and substraction of oligomers. Chembiochem. 7, 757-765 (2006).
- Savistchenko, J., Krzewska, J., Fay, N., Melki, R. Molecular chaperones and the assembly of the prion Ure2p in vitro. J Biol Chem. 283, 15732-15739 (2008).
- Shorter, J., Lindquist, S. Hsp104 catalyzes formation and elimination of self-replicating Sup35 prion conformers. Science. 304, 1793-1797 (2004).
- Shorter, J., Lindquist, S. Destruction or potentiation of different prions catalyzed by similar Hsp104 remodeling activities. Mol Cell. 23, 425-438 (2006).
- Shorter, J., Lindquist, S. Hsp104, Hsp70 and Hsp40 interplay regulates formation, growth and elimination of Sup35 prions. Embo J. 27, 2712-2724 (2008).
- Doyle, S. M. Asymmetric deceleration of ClpB or Hsp104 ATPase activity unleashes protein-remodeling activity. Nat Struct Mol Biol. 14, 114-122 (2007).
- Wendler, P. Atypical AAA+ subunit packing creates an expanded cavity for disaggregation by the protein-remodeling factor Hsp104. Cell. 131, 1366-1377 (2007).
- Hinault, M. P. Stable alpha-synuclein oligomers strongly inhibit chaperone activity of the Hsp70 system by weak interactions with J-domain co-chaperones. J Biol Chem. 285, 38173-38182 (2010).
- Tipton, K. A., Verges, K. J., Weissman, J. S. In vivo monitoring of the prion replication cycle reveals a critical role for Sis1 in delivering substrates to Hsp104. Mol Cell. 32, 584-591 (2008).
- Bieschke, J., Cohen, E., Murray, A., Dillin, A., Kelly, J. W. A kinetic assessment of the C. elegans amyloid disaggregation activity enables uncoupling of disassembly and proteolysis. Protein Sci1. 8, 2231-2241 (2009).
- Cohen, E., Bieschke, J., Perciavalle, R. M., Kelly, J. W., Dillin, A. Opposing activities protect against age-onset proteotoxicity. Science. 313, 1604-1610 (2006).
- Cohen, E. Reduced IGF-1 signaling delays age-associated proteotoxicity in mice. Cell. 139, 1157-1169 (2009).
- Cushman, M., Johnson, B. S., King, O. D., Gitler, A. D., Shorter, J. Prion-like disorders: blurring the divide between transmissibility and infectivity. J Cell Sci. 23, 1191-11201 (2010).
- Carmichael, J. Bacterial and yeast chaperones reduce both aggregate formation and cell death in mammalian cell models of Huntington's disease. Proc Natl Acad Sci. 97, 9701-9705 (2000).
- Vacher, C., Garcia-Oroz, L., Rubinsztein, D. C. Overexpression of yeast hsp104 reduces polyglutamine aggregation and prolongs survival of a transgenic mouse model of Huntington's disease. Hum Mol Genet. 14, 3425-3433 (2005).
- Liu, Y. H. Heat Shock Protein 104 Inhibited the Fibrillization of Prion Peptide 106-126 and Disassembled Prion peptide 106-126 Fibrils in vitro. Int J Biochem Cell Biol. , (2011).
- Wendler, P., Saibil, H. R. Cryo electron microscopy structures of Hsp100 proteins: crowbars in or out. Biochem Cell Biol. 88, 89-96 (2010).
- Wendler, P. Motor mechanism for protein threading through Hsp104. Mol Cell. 34, 81-92 (2009).
- Lee, S., Sielaff, B., Lee, J., Tsai, F. T. CryoEM structure of Hsp104 and its mechanistic implication for protein disaggregation. Proc Natl Acad Sci. 107, 8135-8140 (2010).
- Parsell, D. A., Kowal, A. S., Lindquist, S. Saccharomyces cerevisiae Hsp104 protein. Purification and characterization of ATP-induced structural changes. J Biol Chem. 269, 4480-4487 (1994).
- Schirmer, E. C., Queitsch, C., Kowal, A. S., Parsell, D. A., Lindquist, S. The ATPase activity of Hsp104, effects of environmental conditions and mutations. J Biol Chem. 273, 15546-15552 (1998).
- Schirmer, E. C., Ware, D. M., Queitsch, C., Kowal, A. S., Lindquist, S. L. Subunit interactions influence the biochemical and biological properties of Hsp104. Proc Natl Acad Sci. 98, 914-919 (2001).
- Hattendorf, D. A., Lindquist, S. L. Cooperative kinetics of both Hsp104 ATPase domains and interdomain communication revealed by AAA sensor-1 mutants. EMBO J. 21, 12-21 (2002).
- Schirmer, E. C., Lindquist, S. Purification and properties of Hsp104 from yeast. Methods Enzymol. 290, 430-444 (1998).
- Hattendorf, D. A., Lindquist, S. L. Analysis of the AAA sensor-2 motif in the C-terminal ATPase domain of Hsp104 with a site-specific fluorescent probe of nucleotide binding. Proc Natl Acad Sci. 99, 2732-2737 (2002).
- Lum, R., Niggemann, M., Glover, J. R. Peptide and protein binding in the axial channel of Hsp104. Insights into the mechanism of protein unfolding. J Biol Chem. 283, 30139-30150 (2008).
- Lum, R., Tkach, J. M., Vierling, E., Glover, J. R. Evidence for an unfolding/threading mechanism for protein disaggregation by Saccharomyces cerevisiae Hsp104. J Biol Chem. 279, 29139-29146 (2004).
- Tessarz, P., Mogk, A., Bukau, B. Substrate threading through the central pore of the Hsp104 chaperone as a common mechanism for protein disaggregation and prion propagation. Mol Microbiol. 68, 87-97 (2008).
- Weibezahn, J. Thermotolerance requires refolding of aggregated proteins by substrate translocation through the central pore of ClpB. Cell. 119, 653-665 (2004).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon