È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Immunolocalizzazione della membrana esterna proteine mediante citometria di flusso di mitocondri isolati
In questo articolo
Riepilogo
Qui descritto è un metodo per rilevare e quantificare proteine di membrana mitocondriale esterna da immunomarcatura di mitocondri isolati da tessuto roditore e l'analisi mediante citometria a flusso. Questo metodo può essere esteso a valutare aspetti funzionali di sottopopolazioni mitocondriali.
Abstract
Metodi per rilevare e monitorare mitocondriali componenti proteiche della membrana esterna nei tessuti animali sono di vitale importanza per lo studio della fisiologia mitocondriale e fisiopatologia. Questo protocollo descrive una tecnica in cui i mitocondri isolati da tessuto roditori sono immunolabeled e analizzati mediante citometria a flusso. I mitocondri sono isolate dal midollo spinale di roditori e sottoposti ad una fase di arricchimento rapido in modo da rimuovere mielina, un importante contaminante di frazioni mitocondriali preparate da tessuto nervoso. Mitocondri isolati vengono poi etichettati con un anticorpo di scelta e di un anticorpo secondario coniugato fluorescente. L'analisi per flusso citometria verifica la relativa purezza delle preparazioni mitocondriali da colorazione con un colorante specifico mitocondriale, seguita da rilevamento e la quantificazione di proteine immunolabeled. Questa tecnica è rapido, quantificabile e ad alta velocità, consentendo l'analisi di centinaia di migliaia di mitocondri per campione. È applicabile per valutare romanzo proteins sulla superficie mitocondriale in condizioni fisiologiche normali, così come le proteine che possono diventare mislocalized a questo organello durante patologia. È importante sottolineare che questo metodo può essere accoppiato a coloranti indicatori fluorescenti a riferire su determinate attività di sottopopolazioni mitocondriali ed è fattibile per i mitocondri del sistema nervoso centrale (cervello e midollo spinale), così come il fegato.
Introduzione
I mitocondri sono organelli altamente dinamiche che subiscono vari cicli di fissione e fusione, sono trasportati ai siti di domanda di alta energia e di rispondere rapidamente agli stimoli fisiologici 1. Poiché è sempre più riconosciuto che i mitocondri all'interno di diversi tessuti, anche diversi compartimenti cellulari, hanno profili funzionali distinti, nuovi metodi sono necessari per identificare questi sottoinsiemi mitocondriali distinti.
Microscopia fornisce un mezzo attraverso il quale i singoli mitocondri possono essere visualizzati e la presenza di una proteina in o nei mitocondri può essere determinato mediante immunofluorescenza 2. Tuttavia, l'analisi quantitativa con questo metodo è laborioso ed è più adatto per gli esperimenti che utilizzano linee cellulari immortalizzate o primari. Lo studio dei mitocondri individuale derivate da tessuti è molto più difficile e la maggior parte dei metodi non consentono una facile identificazione di sottoinsiemi mitocondriali in concomitanza con la valuzione della funzione mitocondriale 3.
Per far fronte a questo ostacolo, un nuovo metodo per immunolabel mitocondri isolati da tessuti di roditori e successivamente analizzati mediante citometria a flusso è stato sviluppato. Questo permette la rapida individuazione e la quantificazione delle proteine localizzate alla membrana esterna mitocondriale, che rispetto all'analisi al microscopio, è molto meno laborioso e permette l'analisi di migliaia di mitocondri in un singolo campione. Questo test può essere applicato per controllare il destino e la quantità relativa di proteine di membrana esterna mitocondriale che si pensa siano costitutivamente presente alla mitocondri, l'assunzione di proteine alla superficie mitocondriale, o la rilevazione di proteine mislocalized ai mitocondri in condizioni patologiche. Inoltre, l'incorporazione di coloranti convenzionali indicatori fluorescenti consente la valutazione simultanea di alcuni aspetti della funzione mitocondriale in mitocondriale distintosottopopolazioni.
Protocollo
Animali utilizzati in questo studio sono stati trattati in stretta conformità ad un protocollo (N08001CVsr) approvato dal du Centre Hospitalier de l'Université de Montréal (CRCHUM) Comitato istituzionale Centre de Recherche per la protezione degli animali che segue gli standard nazionali, come delineato dalla Canadian Consiglio su Animal Care (CCAC).
Preparare tutti i reagenti necessari per eseguire questo protocollo (Tabella 1). Tutti gli altri dettagli riguardanti attrezzature, forniture e fornitori possono essere trovati nella lista dei materiali.
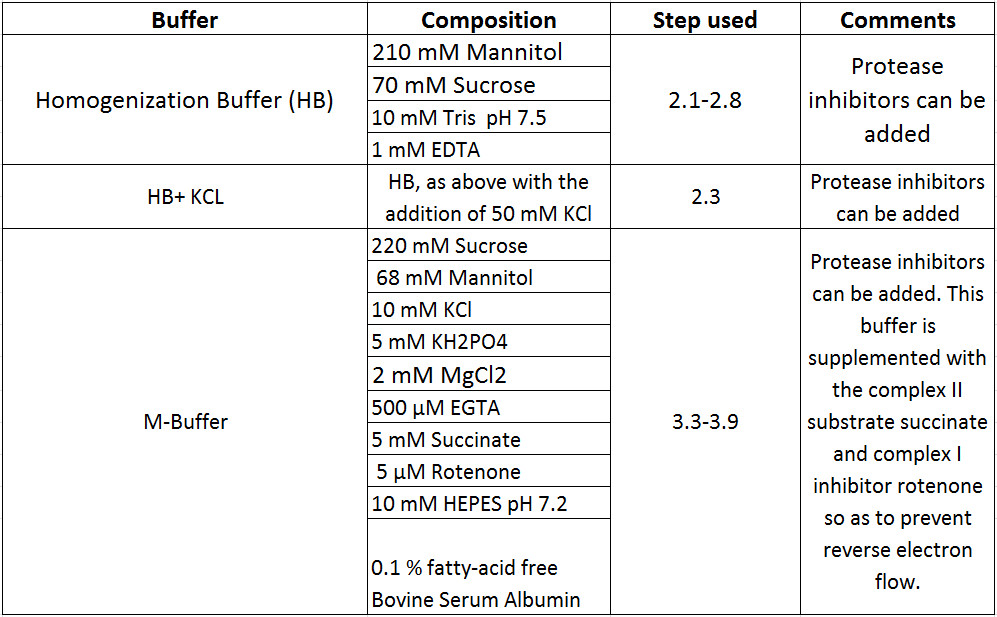
Tabella 1 Buffer Composizioni.
1 Raccolta di Rat Spinal Cord
- Profondamente anestetizzare il ratto (Sprague Dawley) con il 4% isoflorane. Verificare l'anestesia da una mancanza di riflesso su di schiacciamento di the zampa anteriore. Euthanize il ratto per decapitazione mediante ghigliottina. Questo metodo di eutanasia è preferito rispetto ad altri, che potrebbero distorcere il midollo spinale.
- Tagliare la pelle della schiena per esporre la colonna vertebrale. Tagliare la colonna vertebrale con le forbici osso appena sopra l'osso pelvico. Visualizzare l'apertura della colonna vertebrale.
- Inserire una siringa da 10 ml, con una pipetta punta di 200 microlitri (collegato tramite la fusione poco più di fiamma), riempita con tampone fosfato (PBS), nella colonna vertebrale.
- Lavare il midollo spinale, applicando una quantità media di pressione allo stantuffo.
- Se è presente del sangue sul midollo spinale, risciacquare con PBS prima di procedere alla fase successiva.
NOTA: Questo metodo è stato validato anche per il cervello e il fegato. Se tra cui questi tessuti, raccogliere la metà del cervello dal ratto eutanasia e un pezzo di fegato pari in peso al midollo spinale. Tutti gli altri passaggi rimangono identici.
2 Isolamento del midollo spinale Mitochondria (Adattato da Vande Velde et al. 4)
- Raccogliere tutto il midollo spinale intatta e posto in 5 ml omogeneizzatore vetro con 5 volumi (~ 3,25 ml) Omogeneizzazione Buffer (HB). Per il recupero ottimale dei mitocondri isolati, eseguire tutti i passaggi su ghiaccio o in camera fredda. Omogeneizzare i tessuti a mano fino a quando rimangono senza grandi pezzi di tessuto, circa otto colpi. Mettere omogenato in due (2 ml) o tre (1,7 ml) microprovette. Centrifuga 1.300 xg per 10 min a 4 ° C in una microcentrifuga da banco.
- Recuperare il surnatante e posto in un tubo ultracentrifuga 5 ml. Aggiungere 750 ml (~ 0,5 volumi) HB alla provetta-pellet contenente e risospendere delicatamente il pellet. Ripetere la centrifugazione e risospensione passi altre due volte. Pool tutti i surnatanti (S1a e S1b) nella stessa 5 ml tubo ultracentrifuge sopra. Questo passaggio serve per eliminare piccoli detriti.
- Centrifugare riunito S1 utilizzando un'ultracentrifuga dotato di marciume oscillante secchioo e centrifugare a 17.000 xg per 15 min a 4 ° C.
- Tenere surnatante (S2) per l'ulteriore elaborazione se la frazione citosolica è di interesse (ad esempio per le analisi Western blot). Risospendere il pellet (P2), la frazione mitocondriale grezzo, in 4 ml HB + KCl 50 mM. Centrifugare 17.000 xg per 15 min a 4 ° C in un rotore oscillante. Eliminare il supernatante e risospendere delicatamente il pellet (P3) in 800 ml HB.
NOTA: I mitocondri vengono lavati con HB + 50 mM KCl per rimuovere eventuali contaminanti mitocondrialmente associati non specifici. - In un nuovo 5 ml tubo Ultracentrifuge, aggiungere esattamente 800 ml di sedimento risospeso (P3).
- Per aggiungere questo tubo, 200 ml di Iodixanolo (media gradiente di densità), creando così una concentrazione finale del 12% Iodixanolo. Miscelare il contenuto della provetta delicatamente, ma accuratamente mediante pipettaggio con un P1000. Aggiunta di Iodixanolo prima, e poi pellet risospeso (P3) può essere preferibile per facilitare un'accurata miscelazione. Centrifugare in un ultracentrifuge dotato di un rotore benna oscillante a 17.000 xg per 15 min a 4 ° C.
- NOTA: Fegato non contiene mielina e, pertanto, questo passaggio non è necessario se solo il fegato è in fase di elaborazione. Tuttavia, se il fegato sta per essere eseguita in concomitanza con mitocondri del SNC, si raccomanda di trattare tutti i tessuti allo stesso modo.
- Aspirare lo strato di mielina nella parte superiore del tubo e con attenzione rimuovere e scartare il surnatante. Pellet potrebbe essere allentato. Risospendere il pellet in 4 ml di HB. Centrifugare nuovamente a 17.000 xg per 10 min a 4 ° C. Eliminare il supernatante e risospendere il pellet in 4 ml di HB. Ripetere la centrifugazione e rimuovere il surnatante.
- Risospendere il pellet finale (P7) in 100-200 ml HB e trasferire in una provetta da 1,7 ml microcentrifuga. Questo campione contiene mitocondri isolati.
- Procedere alla proteina quantificazione. Diluire i campioni e la curva standard nel 2% sodio dodecil solfato (SDS) per assicurare un'adeguata solubilizzazione dei mitocondri During proteina quantificazione.
3. immunomarcatura di mitocondri isolati per Citometria a flusso
- Per ogni mix colorazione da testare, pipetta 25 mg di mitocondri isolati in una provetta da 1,7 ml microcentrifuga. Includere un campione senza macchia in ogni esperimento; e per ogni anticorpo, essere sicuri di includere un campione per il controllo isotipico appropriato.
- Centrifugare a 17.000 xg per 2 min a 4 ° C in una microcentrifuga da banco.
- Rimuovere il surnatante e risospendere mitocondri isolati in 50 microlitri mitocondri Buffer (Buffer M) supplementato con 10% di BSA libera acidi grassi per 15 min a 4 ° C (blocco passo).
NOTA: Durante l'etichettatura, effettuare incubazioni in frigorifero a 4 ° C. - Aggiungere l'anticorpo primario (coniglio anti-MFN2, 20 mg per ml) per tubo e incubare per 30 minuti a 4 ° C.
NOTA: Determinare la concentrazione ottimale di ciascun anticorpo empiricamente mediante titolazione. A causa della variabilità della concentrazione e / o purezza, diversi lotti dello stesso anticorpo dello stesso produttore possono portare a risultati diversi; pertanto è necessaria titolazione per ciascun nuovo lotto di anticorpi. - Lavare anticorpo primario non legato: Centrifugare a 17.000 xg per 2 min a 4 ° C. Rimuovere il surnatante e risospendere delicatamente il pellet in 200 ml M Buffer. Centrifugare a 17.000 xg per 2 min a 4 ° C. Rimuovere il surnatante e risospendere il pellet in 50 microlitri M tampone.
- Aggiungere l'anticorpo secondario (Donkey anti-coniglio IgG ficoeritrina (PE), 0,5 mg per ml) di tubo e incubare i campioni per 30 minuti a 4 ° C, al riparo dalla luce.
- Lavare anticorpo secondario non legato: Centrifugare a 17.000 xg per 2 min a 4 ° C. Rimuovere il surnatante e risospendere il pellet in 200 ml M Buffer. Centrifugare a 17.000 xg per 2 min a 4 ° C. Rimuovere il surnatante e risospendere il pellet in 500 microlitri M tampone.
- Per assicurare gli eventi sono in realtà mitocondri, macchia isolato mitocondri concolorante mitocondri specifico fluorescente per 15 minuti a temperatura ambiente, al riparo dalla luce. Se si desidera la colorazione di altri parametri funzionali (potenziale transmembrana mitocondriale o di produzione superossido), passare al punto 4 In caso contrario, passare al punto 5 per l'acquisizione.
NOTA: È importante verificare che lo spettro di emissione della anticorpo secondario è compatibile con quella dei coloranti funzionali. Ad esempio, se la verifica purezza mitocondriale con un colorante commerciale con proprietà spettrali simili a potenziale transmembrana con FITC e tetramethylrhodamine estere metilico (TMRM), un anticorpo secondario praticabile sarebbe allophycocyanin (APC: Ex 650 nm / em 660 nm). Aggiungere controlli compensazioni, cioè, un campione immunolabeled o tinto con un unico fluoroforo, quando applicabile. - Trasferire in un tubo adatto per il flusso di carico citometro. (Per facilitare la piccola dimensione del campione, un tubo di microtitolazione è collocato all'interno 3 ml citofluorimetro tubo.) Conservare i campioni in ghiaccio e procedere immediatamente allacitofluorimetro per l'acquisizione.
4 Saggio mitocondriale potenziale transmembrana mitocondriale superossido e Produzione per Citometria a flusso
- Verificare che i mitocondri isolati hanno un potenziale transmembrana intatto dalla colorazione con 100 nM TMRM (Ex 548 nm / 574 nm Em) 5, al punto 3.8, per 15 minuti a temperatura ambiente, al riparo dalla luce. Per il confronto del potenziale transmembrana tra i campioni e popolazioni, l'uso di concentrazioni inferiori / non-tempra di TMRM (da 1 a 30 Nm), può essere più appropriato 6.
- Come controllo per TMRM colorazione, macchia mitocondri isolati con 100 nM TMRM in presenza di 100 mM carbonilico cianuro m-cloro fenil idrazina (CCCP), un disaccoppiatore mitocondriale che depolarizzare mitocondri. La concentrazione di CCCP necessaria per depolarizzare i mitocondri può essere inferiore se si utilizzano più basse concentrazioni di colorante.
- Verificare che i mitocondri isolati producono superossido mitocondriale da staining con un appropriato indicatore superossido mitocondriale 7, anche al punto 3.8, per 15 minuti a temperatura ambiente, al riparo dalla luce.
- Come controllo per la produzione di superossido mitocondriale, macchia mitocondri isolati con colorante in presenza di 10 mM Antimicina A, un inibitore del complesso III della catena respiratoria che aumenterà la produzione di superossido mitocondriale.
5. acquisizione e analisi dei immunolabeled mitocondri isolati da Citometria a flusso
- Strumento costituito: tensioni Passare dalla lineare alla modalità log per facilitare l'analisi dei mitocondri isolati e tensioni previste (FSC: 450; SSC: 250). Assicurarsi che gli eventi sono raccolti in FSC modo A (area) così come FSC-W (larghezza) e FSC-H (altezza), per poter escludere doppietti (cioè due eventi, passano attraverso il rivelatore allo stesso tempo ) nella raccolta di post-analisi dei dati. Impostare il numero di eventi da raccogliere a 100.000. Acquisire i controlli di compensazione, Se applicabile.
- Acquisizione dati: Prima di acquisizione dei dati, evitare vortex campioni. Invece Mescolare agitando leggermente il tubo. Inizialmente la raccolta di eventi a bassa pressione, durante gating. Gate popolazione totale. Regolare le tensioni di istogrammi di conseguenza, di solito il picco del campione non colorato corrisponderà al secondo decennio (10 2). Una volta che cancelli sono stabiliti, ei campioni vengono elaborati, la pressione può essere commutata in alto.
- Analisi: Visualizza doppietti tracciando FSC-W contro FSC. Identificare canottiere e doppiette. Gate canottiere. Selezionare la popolazione mitocondriale da gating su eventi che sono macchiati positivamente con un colorante mitocondriale selettivo.
- Determinare l'etichettatura sfondo da controllo isotipico. Utilizzando il campione di controllo isotipico, determinare la percentuale della popolazione di etichettatura mitocondriale positivo per gli anticorpi MFN2.
Risultati
I mitocondri derivate da midollo spinale di ratto possono essere immunolabeled con un anticorpo mirato al Mitofusin2 (MFN2), una proteina coinvolta nella fusione della membrana esterna dei mitocondri 8. Dopo aver isolato e marcatura con un anticorpo specifico MFN2 e un anticorpo secondario coniugato fluorescente, mitocondri vengono elaborati mediante citometria di flusso (Figura 1). Dopo l'acquisizione dei dati, i campioni vengono analizzati mediante citometria a flusso software di analis...
Discussione
E 'sempre più evidente che i mitocondri sono i principali attori sia normale fisiologia e patologia. Mentre immunoblotting può determinare quali proteine si trovano all'interno dei mitocondri né sulla superficie mitocondriale in una certa condizione, questo metodo relazioni sulla media dell'intera popolazione. Questo metodo non può fornire informazioni sulle abbondanze relative delle sottopopolazioni mitocondriali o sottoinsiemi. Mentre è stato precedentemente ipotizzato che tutti i mitocondri son...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo Laurie Destroismaisons e Sarah Peyrard per un supporto tecnico e Dr.Alexandre Prat per l'accesso al citofluorimetro. Vorremmo inoltre ringraziare il dottor Timothy Miller per il suo contributo per quanto riguarda la rimozione della mielina dai preparativi. Questo lavoro è stato supportato dal Canadian Institutes of Health Research (CIHR) Neuromuscular Research Partnership, Canadian Foundation for Innovation, ALS Society of Canada, la Fondazione Frick per la SLA Research, CHUM Fondazione e Fonds de la Recherche en Santé du Québec (CVV). Sia CVV e NA sono studiosi di ricerca del Fonds de la Recherche en Santé du Québec e CIHR nuovi ricercatori. SP è sostenuto dalla Tim Noël Studentship dalla ALS Society of Canada.
Materiali
| Name | Company | Catalog Number | Comments |
| Rats | Charles River | Strain code 400 | Adult (9 weeks to 18 weeks) male or female rats can be used for the isolation protocol. Weight of rats is dependent on gender and age (males between 300 to 500 g and females between 200 to 350 g) are typically used. |
| Dounce homogenizer | Kontes Glass Co. | 885450-0022 | Duall 22 |
| Microcentrifuge | Thermo Scientific | Sorvall Legend Micro 17 R | |
| Ultra-Clear Ultracentrifuge tubes | Beckman Coulter | 344057 | Transparent, thin walled |
| Sorvall Ultracentrifuge | Thermo Scientific | Sorvall WX UltraSeries | |
| AH-650 rotor and buckets | Thermo Scientific | ||
| Opti-prep | Axis-Shield | 1114542 | Iodixanol, density gradient medium |
| Fatty acid free Bovine Serum Albumin | Sigma | A8806 | Must be fatty acid free for mitochondria |
| Sodium succinate dibasic hexahydrate | Sigma | S9637 | |
| Rabbit anti-Mitofusin2 antibody | Sigma | M6319 | |
| Rabbit IgG | Jackson Immuno Research | 011-000-003 | |
| Anti-Rabbit IgG PE | eBioscience | 12-4739-81 | |
| Micro titer tube | Bio-Rad | 223-9391 | For sample acquisition by flow cytometry |
| MitoTrackerGreen (MTG) | Invitrogen | M7514 | 100 nM: Ex 490 nm/Em 516 nm |
| TMRM | Invitrogen | T668 | 100 nM: Ex 548 nm/Em 574 nm |
| CCCP | Sigma | C2759 | |
| MitoSOX Red | Invitrogen | M36008 | 5 µM: Ex 540 nm/Em 600 nm |
| Antimycin A | Sigma | A8874 | |
| LSR II flow cytometer | BD | ||
| BD FACSDiva Software | BD | ||
| FlowJo | TreeStar Inc. | Software used for analysis | |
| BCA protein assay kit | Pierce/Thermo Scientific | 23225 | Bradford assay is not recomended as it is not compatible with high concentrations of SDS |
Riferimenti
- Itoh, K., Nakamura, K., Iijima, M., Sesaki, H. Mitochondrial dynamics in neurodegeneration. Trends Cell Biol. 23, 64-71 (2013).
- Narendra, D., Tanaka, A., Suen, D. F., Youle, R. J. Parkin is recruited selectively to impaired mitochondria and promotes their autophagy. J Cell Biol. 183, 795-803 (2008).
- Zorov, D. B., Kobrinsky, E., Juhaszova, M., Sollott, S. J. Examining intracellular organelle function using fluorescent probes: from animalcules to quantum dots. Circ Res. 95, 239-252 (2004).
- Vande Velde, C., Miller, T., Cashman, N. R., Cleveland, D. W. Selective association of misfolded ALS-linked mutant SOD1 with the cytoplasmic face of mitochondria. Proc Natl Acad Sci U S A. 105, 4022-4027 (2008).
- Loew, L. M., Tuft, R. A., Carrington, W., Fay, F. S. Imaging in five dimensions: time-dependent membrane potentials in individual mitochondria. Biophys J. 65, 2396-2407 (1993).
- Perry, S. W., Norman, J. P., Barbieri, J., Brown, E. B., Gelbard, H. A. Mitochondrial membrane potential probes and the proton gradient: a practical usage guide. Biotechniques. 50, 98-115 (2011).
- Xu, X., Arriaga, E. A. Qualitative determination of superoxide release at both sides of the mitochondrial inner membrane by capillary electrophoretic analysis of the oxidation products of triphenylphosphonium hydroethidine. Free Radic Biol Med. 46, 905-913 (2009).
- Otera, H., Ishihara, N., Mihara, K. New insights into the function and regulation of mitochondrial fission. Biochim Biophys Acta. 1833, 1256-1268 (2013).
- de Brito, O. M., Scorrano, L. Mitofusin 2 tethers endoplasmic reticulum to mitochondria. Nature. 456, 605-610 (2008).
- Wikstrom, J. D., Twig, G., Shirihai, O. S. What can mitochondrial heterogeneity tell us about mitochondrial dynamics and autophagy. Int J Biochem Cell Biol. 41, 1914-1927 (2009).
- Pickles, S., Destroismaisons, L., Peyrard, S. L., Cadot, S., Rouleau, G. A., Brown, R. H., Julien, J. P., Arbour, N., Vande Velde, C. Mitochondrial damage revealed by immunoselection for ALS-linked misfolded SOD1. Hum Mol Genet. 22, 3947-3959 (2013).
- Frank, S., Gaume, B., Bergmann-Leitner, E. S., Leitner, W. W., Robert, E. G., Catez, F., Smith, C. L., Youle, R. J. The role of dynamin-related protein 1, a mediator of mitochondrial fission, in apoptosis. Dev Cell. 1, 515-525 (2001).
- West, A. P., Brodsky, I. E., Rahner, C., Woo, D. K., Erdjument-Bromage, H., Tempst, P., Walsh, M. C., Choi, Y., Shadel, G. S., Ghosh, S. TLR signalling augments macrophage bactericidal activity through mitochondrial ROS. Nature. 472, 476-480 (2011).
- Abad, M. F., Di Benedetto, G., Magalhães, P. J., Filippin, L., Pozzan, T. Mitochondrial pH monitored by a new engineered green fluorescent protein mutant. J Biol Chem. 279, 11521-11529 (2004).
- Murphy, A. N., Bredesen, D., Cortopassi, G., Wang, E., Fiskum, G. Bcl-2 potentiates the maximal calcium uptake capacity of neural cell mitochondria. Proc Natl Acad Sci U S A. 93, 9893-9898 (1996).
- Tantama, M., Martinez-François, J. R., Mongeon, R., Yellen, G. Imaging energy status in live cells with a fluorescent biosensor of the intracellular ATP-to-ADP ratio. Nat Commun. 4, 2550 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon