È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Proprietà di trasporto di ibuprofene Encapsulated in ciclodestrina Nanosponge idrogel: un protone HR-MAS NMR Spettroscopia di studio
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
I regimi di movimento di ibuprofene incapsulata in nanospugne beta-ciclodestrina rete polimero sono studiati con spin-echo tecnica (PGSE) NMR campo pulsato-gradiente. Sintesi, purificazione, droga caricamento, esecuzione della sequenza di impulsi NMR e analisi dei dati per calcolare lo spostamento quadratico medio del farmaco in diversi tempi di osservazione sono descritte in dettaglio.
Abstract
La reticolazione chimica di β-ciclodestrina (β-CD) con etilendiamminotetraacetico dianidride (EDTA) ha portato a polimeri ramificati denominati nanospugne ciclodestrina (CDNSEDTA). Due diverse preparazioni sono descritti con 1: 4 e 1: 8 CD-EDTA rapporti molari. I polimeri reticolati corrispondenti sono stati contattati con soluzione acquosa 0,27 M di sale sodico ibuprofene (IP) che conduce al omogenee, incolori, droga caricata idrogel.
I sistemi sono stati caratterizzati da alta risoluzione angolo di magia filatura (MAS HR-) spettroscopia NMR. Spettroscopia NMR campo pulsato gradient spin echo (PGSE) è stato utilizzato per determinare lo spostamento quadratico medio (MSD) del IP nel gel polimerico a diversi tempi di osservazione t d. I dati sono stati ulteriormente trattati al fine di studiare la dipendenza dal tempo della MSD: MSD = f (t d). La metodologia proposta è utile per caratterizzare i diversi regimi di diffusione che,in linea di principio, il soluto può verificarsi all'interno del idrogel, vale a dire la diffusione normale o anomala. I protocolli completi inclusa la preparazione del polimero e purificazione, l'ottenimento di idrogel droga-caricata, la preparazione del campione NMR, la misura di MSD mediante spettroscopia HR-MAS NMR e trattamento finale per ottenere la dipendenza dal tempo della MSD vengono qui riportati e discussi . Gli esperimenti presentati rappresentano un caso paradigmatico ed i dati sono discussi in termini di approccio innovativo per la caratterizzazione delle proprietà di trasporto di un ospite incapsulato all'interno di un host polimerico di potenziale applicazione per la consegna della droga.
Introduzione
Vi è un crescente interesse nella progettazione e nella formulazione di sistemi polimerici in grado di inglobamento, tramite interazioni non covalenti, piccole molecole con potenziale attività biochimica. Tali materiali dovrebbero trovare applicazioni nel trasporto del principio attivo bersaglio selettivo e rilasciare su azione di stimoli esterni, quali variazioni di pH, temperatura, ecc In questo contesto, idrogel si sono rivelati materiali versatili e potenti per nanomedicina in vista del rilascio controllato di farmaci 1. La formazione di idrogel polimerici può essere realizzato collegando le catene macromolecolari da i) fisiche, interazioni non covalenti quali legami idrogeno, ii) covalente reticolazione delle catene che portano ad una rete tridimensionale in grado di rigonfiare in presenza di una soluzione acquosa o iii) una combinazione dei due metodi citati 2-4.
Una classe particolarmente versatile di tre dimensional, polimeri rigonfiabili per l'incapsulamento di specie organiche ed inorganiche possono essere ottenuti partendo da naturale β-ciclodestrina (β-CD) tramite condensazione con opportuni, derivati attivati di un acido tetracarbossilico 5 - 8 generatore nanospugne ciclodestrina (CDN). La sintesi, caratterizzazione e applicazione di CDNS è un tema di ricerca consolidata del nostro gruppo. I risultati degli ultimi anni indicano che CDNS mostrano proprietà intriganti di gonfiore, assorbimento / inclusione di sostanze chimiche, e il rilascio di piccole molecole di droga, con applicazioni in rilascio controllato di principi attivi farmaceutici 9 - 11 e chimica ambientale 12 - 14.
Date queste premesse, due grandi questioni da affrontare riguardano il carico efficiente del composto attivo nel gel polimerico e una migliore comprensione della mobilità soluti nelle matrici gel 15 . La letteratura fornisce entrambi gli studi e le teorie legate ai meccanismi di diffusione di piccole molecole nelle reti macromolecolari 16,17 sperimentali. Eco spin-(PGSE) spettroscopia Pulsed campo gradiente NMR è un metodo strutturale consolidata ampiamente utilizzato per studiare la diffusione traslazionale di piccole molecole in solventi 18 o l'auto-diffusione dei liquidi puri. I recenti sviluppi di alta risoluzione angolo di magia filatura (HR-MAS), la tecnologia NMR hanno permesso di raccogliere dati ad alta risoluzione NMR di molecole di telefonia mobile in sospensioni eterogenei 19, gel e polimeri rigonfiabili 20,21. In effetti, l'apparato sperimentale che combina HR-MAS spettroscopia NMR e la sequenza di impulsi PGSE fornisce un'opportunità unica per osservare le molecole di soluto in ambiente molecolare del padrone di casa. I dati importanti sulle proprietà di trasporto della molecola farmaco intrappolato all'interno di una matrice di gel possono così essere ottenuti. dati sperimentali di alta qualità possono quindi essere ottened consentendo un design più razionale dei sistemi host-guest nanostrutturati.
Nel presente lavoro descriviamo i protocolli dettagliati per le seguenti fasi: i) sintesi e la purificazione di due differenti formulazione di CDNS reticolato con polimeri EDTA (Figura 1), denominata CDNSEDTA, e caratterizzata da diversi CD / cross molare linker rapporto: 1: 4 (CDNSEDTA 1: 4) e 1: 8 (CDNSEDTA 1: 8); ii) la preparazione di idrogeli droga-caricata per entrambi CDNSEDTA 1: 4 e CDNSEDTA 1: 8. In questa fase abbiamo utilizzato, come molecola modello di farmaco, il popolare non-steroidei anti-infiammatori sale di sodio ibuprofene (IP); iii) l'indagine approfondita delle proprietà di trasporto di IP all'interno del CDNSEDTA idrogeli tramite spettroscopia NMR PGSE-HRMAS. Il metodo qui proposto si basa sulla misura dello spostamento quadratico medio (MSD) del farmaco incapsulato all'interno idrogel seguita dall'analisi della dipendenza dal tempo della MSD.
noi wish sottolineare che la metodologia sopra descritta - che si concentra sulla dipendenza dal tempo della MDS del farmaco nella matrice - fornisce un più ampio spettro di informazioni rispetto al metodo consolidato basato sulla determinazione di un solo coefficiente di diffusione del farmaco. Abbiamo recentemente dimostrato 21 che questo approccio ha permesso per la discriminazione dei normali e anomale regimi di diffusione sperimentati da IP confinati in idrogel CDN.
Riteniamo quindi che la descrizione step-by-step di sintesi dei polimeri / purificazione, formazione degli idrogel droga-caricato, HR-MAS NMR caratterizzazione e l'elaborazione dei dati dei dati MDS, è un toolkit potente per gli scienziati interessati a caratterizzare i sistemi nanostrutturati per la confino e il rilascio di piccole molecole.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Sintesi di CDNSEDTA Polimeri
- Dry β-ciclodestrina (β-CD) in forno a 80 ° C per 4 ore prima dell'uso. Secco 500 ml di dimetilsolfossido (DMSO) e 100 ml di trietilammina (Et 3 N) oltre setacci molecolari (4 A) per 24 ore prima di utilizzarli nel protocollo.
- Introdurre 25 ml di DMSO in un pallone da ml un girocollo-bottom 50. Sotto agitazione magnetica, aggiungere 5.675 g di β-CD (5 mmoli). Per ridurre la formazione di grumi, aggiungere la polvere β-CD in piccole porzioni a DMSO.
- Dopo circa 30 minuti, aggiungere 6 ml di Et 3 N per la soluzione omogenea con un 10 ml pipetta graduata. Mantenere la miscela sotto agitazione per 15 minuti a temperatura ambiente. Immergere il pallone in un bagno d'acqua a temperatura ambiente.
NOTA: La reazione tra β-CD e EDTA è esotermica. Pertanto, immergendo il matraccio nel bagno dell'acqua favorisce lo scambio termico evitando il surriscaldamento della miscela di reazione. - Aggiungere 5.124 g (20 mmol, preparazione di CDNSEDTA 1: 4) o 10.248 g (40 mmol, preparazione di CDNSEDTA 1: 8) di EDTA-dianidride sotto forte agitazione.
- Dopo 3 ore, rimuovere il materiale solido (CDNSEDTA 1: 4 o CDNSEDTA 1: 8) dalla muffola con una spatola e schiacciare grossolanamente con un mortaio e pestello.
- Lavare il materiale solido su carta da filtro con acetone a temperatura ambiente (100 ml x 5 volte), con HCl 0,1 M (200 ml x 5 volte) e acqua deionizzata (200 ml x 3 volte).
- Infine, asciutta tutto il materiale solido in aria a temperatura ambiente per 48 ore, schiacciare finemente in un mortaio e pestello e poi tenerlo sotto vuoto (<15 mbar) per 2 ore a 45 ° C.
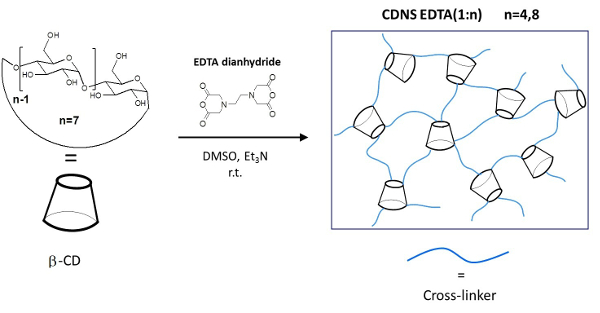
Figura 1:. Rappresentazione schematica dei polimeri CDNSEDTA via sintetica schematica. Sinistra: struttura molecolare del monomero β-ciclodestrina (β-CD) e agente reticolante EDTA-dianidride. Sulla freccia condizioni di reazione complessive. Destra:. Schizzo del polimero reticolato Si prega di cliccare qui per vedere una versione più grande di questa figura.
2. HR-MAS NMR Misure
- HR-MAS NMR Preparazione del campione
- Preparare una soluzione 0,27 M di sale sodico ibuprofene (IP) in acqua deuterato (99,8%).
- Aggiungere 20 mg di CDNSEDTA 1: 4 e 2 mg di carbonato di sodio anidro (Na 2 CO 3) a 150 microlitri della soluzione preparata al punto 2.1.1) in una fiala di vetro da 2 ml. Mescolare il contenuto della fiala con una piccola spatola per omogeneizzare. Attendere 2 ore prima di utilizzare il gel formato con questa procedura. Ripetere questo punto per il CDNSEDTA 1: polimero 8.
- Inserire il gel in un rotore NMR 5 millimetri adatto per esperimenti HR-MAS NMR utilizzando una piccola spatola. La quantità totale di gel da utilizzare dipende dal volume interno del rotore (12 microlitri raccomandato).
- HR-MAS 1 Esperimenti H NMR
- Impostare i seguenti parametri strumentali: velocità del rotore di filatura di 4 KHz presso l'unità di controllo pneumatico MAS, temperatura del campione a 305 K in unità di temperatura variabile.
- Acquisire il 1 H HR-MAS NMR spettri di ibuprofene in CDNSEDTA (1: 4) e CDNSEDTA (1: 8) sistemi polimerici mediante un convenzionale sequenza di un impulso sulla risonanza protonica.
- Creare un nuovo set di dati. Fare clic sulla scheda "AcquPars". Selezionare il PULPROG: ZG.
- Selezionare il numero di scansioni (NS = 4) e il ritardo tra di loro (D1 = 5 sec) .Set la larghezza spettrale (SW = 8 ppm), il dominio del tempo (TD = 16K) e il guadagno del ricevitore (RG = 32 ).
- Tipo "zg" sulla console e ci sarà un decadimento libero dell'induzione (FID) sullo schermo. Per elaborare i dati fare clic sulla scheda "ProcPars". Impostare la dimensione spettrale (SI = 32K), una funzione di finestra di moltiplicazione esponenziale (WDW = EM) e la linea di ampliamento (LB = 1).Tipo "ft" per eseguire la trasformazione di Fourier. Fase dello spettro utilizzando la scheda di fase sullo schermo. Ottenere un alta risoluzione spettro ben risolto.

Figura 2: le coppie di impulsi bipolari longitudinale Eddy Current Delay (BPPLED) Sequenza di impulsi Rappresentazione schematica della sequenza di impulsi utilizzato per eseguire gli esperimenti PFGSE.. Il ciclo di fase per gli impulsi di 90 ° è: P1: (0) 16, P2: (0022) 4, P3: (0) 4 (2) 4 (1) 4 (3) 4, P4: (0202 2020 1313 3131 ), P5: (0) 4 (2) 4 (1) 4 (3) 4. I 180 ° impulsi sono + x. (modificato da ref.18) Clicca qui per vedere una versione più grande di questa figura.
- HR-MAS 1 H NMR Esperimenti PGSE
NOTA: Gli esperimenti PGSE vengono eseguiti utilizzando la sequenza di impulsi BPPLED 18 riportato in Figura 2. Questa è una pseudo esperimento bidimensionale con una rampa di pendenza crescente linearmente dal 2% al 100% nella dimensione indiretta. L'intensità del segnale viene attenuato in funzione del tempo di diffusione Δ e δ pulsazioni gradiente. L'ottimizzazione di questi parametri è richiesto prima di eseguire correttamente un esperimento PGSE. L'ottimizzazione avviene eseguendo alcune misure 1D in cui Δ si mantiene costante, mentre δ è vario.- parametri di ottimizzazione
- Creare un nuovo set di dati - numero di esperimento 1. Fare clic sulla scheda "AcquPars". Selezionare il PULPROG: ledbpgp2s1d la sequenza di impulsi 1D per l'ottimizzazione diffusione.
- Selezionare il numero di scansioni (NS = 16) e il ritardo di tempo tra di loro (D1 = 10 sec). Impostare la larghezza spettrale (SW = 8 ppm), tegli dominio del tempo (TD = 16K) e il guadagno del ricevitore (RG = 32).
- Impostare Δ (D20 nella sequenza) uguale ad un valore costante e δ (P30) ad un valore di prova. Inizia valore Δ = 50 msec, δ = 3 msec (valore massimo consentito per gli strumenti ad alta risoluzione).
- Leggere il valore di frequenza spettrale (SFO1) dall'esperimento 1 H e utilizzare ora tale valore. Impostare la forza di gradiente GPZ6 al 2%. Ripetere il punto 2.2.2.3. Utilizzare questo spettro come riferimento per l'ottimizzazione.
- Nello stesso insieme di dati creare il numero di esperimento 2. Osservare tutti i parametri sperimentali. Aumentare la forza di gradiente GPZ6 al 95%. Ripetere il punto 2.2.2.3. Confrontare questo spettro con lo spettro di riferimento utilizzando l'icona doppia indicazione e osservare il cambiamento nell'intensità del segnale.
NOTA: Uno spettro ben attenuato dovrebbe avere intensità segnale residuo circa il 5% rispetto al spettro di riferimento. Se l'intensità del segnale è perso, ridurre il valore di δ e rescrostata la sezione 2.3.1 procedura dal punto 2.3.1.3 fino a quando viene trovato il giusto valore per δ. - Ripetere la procedura di ottimizzazione dei parametri nella sezione 2.3.1 per tutti i cinque Δ valori.
NOTA: scegliere cinque valore Δ = 50, 80, 110, 140 e 170 msec e ottimizzato il δ corrispondente al 3, 2.7, 2.4, 2.1, 1.8 msec (per IP in CDNSEDTA 1: 8) e δ a 2.7, 2.4 , 2, 1.7, 1.4 (per IP nella CDNSEDTA 1: 4).
- Acquisizione del 2D Diffusione set di dati
- Negli stessi set di dati creare il numero di esperimento 3, verranno caricati tutti i parametri sperimentali 1D. Tipo "eda". Selezionare il PULPROG: ledbpgp2s la sequenza di impulsi 2D e cambiare il parmode a 2D.
- Set FnMODE = QF. Impostare il dominio TD volta in dimensione F2 uguale a 32, il numero di passi di gradiente. Tutti gli altri parametri sono impostati correttamente. Tipo "DOSY" e la rampa gradiente verrà generata e memorizzata in un file. la starte e valori finali della rampa (2 - 95) sono indicati come parametri di input. L'acquisizione è ormai avviato.
- parametri di ottimizzazione
- Elaborazione dati
- Tipo "XF2" per eseguire la trasformazione di Fourier nella dimensione F2. Tipo "ABS2" per eseguire la correzione della linea di base nella dimensione F2. Tipo "setdiffparm" per richiamare i parametri sperimentali (Í caratteristica δ, e l'elenco di pendenza) per la prossima fase di lavorazione.
- Fai clic su "modulo di rilassamento T1 / T2" nella scheda di analisi e definire le vette essere montati utilizzando il primo spettro dell'esperimento 2D. Definire le gamme di punta ed eseguire il montaggio. Le intensità di segnale in ogni fase del gradiente applicato si ottengono.
NOTA: Le intensità di segnale I (q, t d), per ogni valore Δ, dipende dalle variabili sperimentali: applicato impulso depositato gradiente (g), variabile temporale (δ), il rapporto magnetogyric (γ) q = (γgδ) secondo la foequazione llowing:
con l'molecolare MSD = z 2. - Esportare le intensità di segnale in un foglio di calcolo e di eseguire una misura lineare dei dati per ottenere il valore di z 2 per ogni osservati tempo di diffusione t d.
NOTA: Il valore di MSD è legata al t d tempo di osservazione secondo:
- Eseguire la trama log-log di z 2 contro t d per ciascun valore sperimentale t d. Il valore dell'esponente α è la pendenza della regressione lineare. Una discussione più esaustiva degli aspetti fisici delle equazioni sopra riportate possono essere trovate in rif. 21 e ivi riferimenti.
NOTA: A seconda del valore delle α esponente, il regime di diffusione è definita come: i) isotropo diffusione incontrollata di α = 1, ii) subdiffusiv anomalae regime per 0 <α <1, iii) regime superdiffusive anomala per α> 1.
Access restricted. Please log in or start a trial to view this content.
Risultati
Abbiamo dapprima applicato questa metodologia alla molecola droga IP disciolto in soluzione acquosa per verificare la validità di questo approccio. Una descrizione completa dei risultati rappresentativi si possono trovare in rif. 21. Piuttosto, ci concentreremo qui sugli aspetti metodologici e l'approccio dadi e bulloni per la raccolta e l'analisi dei dati. Figura 3 mostra, in scala semilogaritmica, il segnale sperimentale normalizzato decade I (q, t d)
Access restricted. Please log in or start a trial to view this content.
Discussione
Presentiamo una metodologia sperimentale per determinare il regime diffusione di una piccola molecola farmaco incapsulato all'interno di due formulazioni rappresentative degli idrogel CDNSEDTA. HR-MAS PGSE NMR permette la determinazione dello spostamento quadratico medio delle piccole molecole in un dato tempo di diffusione (dell'ordine di pochi millisecondi fino al secondo), poi distanze monitoraggio nelle scale micrometriche. Nell'intervallo osservato (50 - 170 msec) si osserva un solo tipo di movimento pe...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
The authors gratefully acknowledge PRIN 2010-2011 NANOMED prot. 2010 FPTBSH and PRIN 2010-2011 PROxy prot. 2010PFLRJR_005 for funding.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| HR-MAS probe | BRUKER | N/A | Probe for NMR measurements on semi-solid samples |
| NMR Spectrometer | BRUKER | DRX 500 | FT NMR spectrometer for liquid ans semi-solis state |
| β-cyclodextrin (β-CD) | Alfa-Aesar | J63161 | Reagent |
| Ethylenediaminetetracetic (EDTA) dianhydride | Sigma-Aldrich | 332046 | Reagent |
| Dimethylsulfoxide (DMSO) | Alfa-Aesar | D0798 | Solvent |
| Triethylamine | Sigma-Aldrich | 471283 | Base (reagent) |
| Ibuprofen (IP) sadium salt | Sigma-Aldrich | I1892 | Antinflammatory drug |
| Excel 2010 | Microsoft | N/A | speadsheet for data analysis |
| Origin 8 SR0 | OriginLab Co. | speadsheet for data analysis |
Riferimenti
- Sharpe, L. A., Daily, A. M., Horava, S. D., Peppas, N. A. Therapeutic applications of hydrogels in oral drug delivery. Expert Opin. Drug Deliv. 11, 901-915 (2014).
- Hennik, W. E., van Nostrum, C. F. Novel crosslinking methods to design hydrogels. Adv. Drug Deliv. Rev. 54, 13-36 (2002).
- Yu, L., Ding, J. D. Injectable hydrogels as unique biomedical materials. Chem. Soc. Rev. 37, 1473-1481 (2008).
- Ma, M., Kuang, Y., Gao, Y., Zhang, Y., Gao, P., Xu, B. Aromatic-Aromatic Interactions Induce the Self-Assembly of Pentapeptidic Derivatives in Water To Form Nanofibers and Supramolecular Hydrogels. J. Am. Chem. Soc. 132, 2719-2728 (2010).
- Trotta, F., Tumiatti, W. Patent WO. , 03/085002 (2003).
- Trotta, F., Tumiatti, W., Cavalli, R., Zerbinati, O., Roggero, C. M., Vallero, R. Ultrasound-assisted synthesis of cyclodextrinbased nanosponges. Patent WO. , 06/002814 (2006).
- Trotta, F., Cavalli, R. Characterization and applications of new hyper-cross-linked cyclodextrins. Compos. Interface. 16, 39-48 (2009).
- Cavalli, R., Trotta, F., Tumiatti, W. Cyclodextrin-based nanosponges for drug delivery. J. Incl. Phenom. Macrocycl. Chem. 56, 209-213 (2006).
- Trotta, F., et al. Cyclodextrin-based nanosponges as a vehicle for antitumoral drugs. Patent WO. , 09/003656 (2009).
- Vyas, A., Shailendra, S., Swarnlata, S. Cyclodextrin based novel drug delivery systems. J. Incl. Phenom. Macrocycl. Chem. 62, 23-42 (2008).
- Swaminathan, S., Vavia, P. R., Trotta, F., Torne, S. Formulation of beta-cyclodextrin based nanosponges of itraconazole. J. Incl. Phenom. Macrocycl. Chem. 57, 89-94 (2007).
- Mamba, B. B., Krause, R. W., Malefetse, T. J., Gericke, G., Sithole, S. P. Cyclodextrin nanosponges in the removal of organic matter to produce water for power generation. Water SA. 34, 657-660 (2008).
- Mamba, B. B., Krause, R. W., Malefetse, T. J., Nxumalo, E. N. Monofunctionalized cyclodextrin polymers for the removal of organic pollutants from water. Environ.Chem. Lett. 5, 79-84 (2007).
- Mhlanga, S. D., Mamba, B. B., Krause, R. W., Malefetse, T. J. Removal of organic contaminants from water using nanosponge cyclodextrin polyurethanes. J. Chem. Technol. Biot. 82, 382-388 (2007).
- Lehmann, S., Seiffert, S., Richtering, W. Spatially Resolved Tracer Diffusion in Complex Responsive Hydrogels. J. Am. Chem. Soc. 134, 15963-15969 (2012).
- Ferrer, G. G., Pradas, M. M., Ribelles, J. L. G., Colomer, F. R., Castilla-Cortazar, I., Vidaurre, A. Influence of the nature of the porous confining network on the sorption, diffusion and mechanical properties of hydrogel IPNs. Eur. Polym. J. 46, 774-782 (2010).
- Santoro, M., Marchetti, P., Rossi, F., Perale, G., Castiglione, F., Mele, A., Masi, M. Smart approach to evaluate drug diffusivity in injectable agar-carbomer hydrogels for drug delivery. J. Phys. Chem B. 115, 2503-2510 (2011).
- Johnson, C. S. Diffusion ordered nuclear magnetic resonance spectroscopy: principles and applications. Prog. Nucl. Magn. Reson. Spectroscopy. 34, 203-256 (1999).
- Viel, S., Ziarelli, F., Caldarelli, S. Enhanced diffusion-edited NMR spectroscopy of mixtures using chromatographic stationary phases. Proc. Natl. Acad. Sci. U. S. A. 100, 9696-9698 (2003).
- Alam, T. M., Hibbs, R. M. Characterization of heterogeneous solvent diffusion environments in anion exchange membranes. Macromolecules. 47, 1073-1084 (2014).
- Ferro, M., Castiglione, F., Punta, C., Melone, L., Panzeri, W., Rossi, B., Trotta, F., Mele, A. Anomalous diffusion of Ibuprofen in cyclodextrin nanosponges hydrogels: an HR-MAS NMR study. Beilstein J. Org. Chem. 10, 2715-2723 (2014).
- Wolf, G., Kleinpeter, E. Pulsed Field Gradient NMR Study of Anomalous Diffusion in a Lecithin-Based Microemulsion. Langmuir. 21, 6742-6752 (2005).
- Rossi, F., Castiglione, F., Ferro, M., Marchini, P., Mauri, E., Moioli, M., Mele, A., Masi, M. Drug-Polymer interactions in hydrogel-based drug-delivery systems: an experimental and theoretical study. Chem. Phys. Chem. , (2015).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon