Method Article
"Fegato-on-a-Chip" colture di epatociti primari e cellule di Kupffer per infezione da Virus dell'epatite B
In questo articolo
Riepilogo
L'obiettivo del presente protocollo è quello di fornire una guida dettagliata per eseguire esperimenti di 3D "fegato-on-a-chip" infezione con virus dell'epatite B.
Abstract
Nonostante l'eccezionale infettività del virus dell'epatite B (HBV) in vivo, dove solo tre genomi virali possono comportare una cronicità di scimpanzé infettati sperimentalmente, più modelli in vitro richiedono diverse centinaia di migliaia di genomi virali per la cellula in al fine di avviare un infezione transitoria. Inoltre, 2D statici colture di epatociti umani primari (PHH) consentono solo studi di breve durata a causa della loro rapida dedifferenziazione. Qui, descriviamo culture di fegato-on-a-chip 3D di PHH, monocolture o in cocultures con altre cellule di fegato-residente parenchimale. Questi offrono un miglioramento significativo nello studio a lungo termine infezioni da HBV con risposte di host fisiologico delle cellule. Oltre a facilitare gli studi di efficacia del farmaco, analisi tossicologica e indagini sulla patogenesi, questi sistemi di cultura microfluidici consentono la valutazione delle terapie curative per l'infezione da HBV volti ad eliminare covalentemente chiuso, circolare (ccc) del DNA. Presentato il metodo descrive il set-up di monocolture PHH e PHH/Kupffer cellule co-culture, loro infezione con HBV purificato e l'analisi della risposta ospite. Questo metodo è particolarmente indicato per la valutazione degli effetti a lungo termine dell'infezione da HBV, combinazioni di trattamento e patogenesi.
Introduzione
Lo studio di HBV è stato complicato dalla scarsa suscettibilità dei sistemi di coltura, che richiedono diverse centinaia di migliaia di copie del genoma HBV per cella per avviare l' infezione1. Inoltre, epatociti umani primari sono generalmente estremamente fragili e rapidamente dedifferentiate durante le colture convenzionali2. Questo è dovuto principalmente al fatto che le superfici in plastica piane e dura non imitare gli ambienti naturali extracellulari trovati all'interno del fegato e la generale mancanza di ossigenazione delle culture in assenza di circolazione di microfluidica. Culture tradizionali dell'epatocita statico sulle piastre rivestite con collagene rapidamente dedifferentiate e perdono la loro suscettibilità all' infezione di HBV3. Qui, descriviamo il set-up e l'infezione di PHH coltivate in 3D culture di fegato-on-a-chip, che sono enormemente vantaggiose sopra le colture statiche 2D convenzionali PHH sulle piastre rivestite con collagene grazie alla loro estesa competenza funzionale e metabolica, facilitando colture a lungo termine di almeno 40 giorni4. In questo sistema, PHH sono seminati su impalcature rivestite con collagene, che sono continuamente irrorate con terreno di coltura per la fornitura di ossigeno e sostanze nutritive alle cellule. Anche se sistemi di cultura alternativa per PHH basati su complessi cocultures di fibroblasti murini o sviluppo 3D in sferoidi sono stati convalidati e sono suscettibili di infezione da HBV con molteplicità di infezione di 500 equivalenti di genoma (GE) di HBV per la cellula, 3D fegato-on-a-chip culture rimangono il sistema unico modello in vitro suscettibile di 0.05 GE di HBV per cella4. Questo è inoltre sostenuto dalla necessità dell'utilizzo di alte concentrazioni di solfossido dimetilico (DMSO) e polietilenglicole (PEG) per stabilire l'infezione da HBV in queste culture, che non è indispensabile per l'infezione dei sistemi di coltura di fegato-on-a-chip 3D 4. tra i principali tratti distintivi dell'HBV infezione è la piscina di cccDNA, che agisce come il modello trascrizionale per tutti de novo–prodotto virioni5,6. Anche se cccDNA può essere rilevata in convenzionale dell'epatocita culture7,8, rimane poco chiaro se la regolazione del cccDNA ed eventuali approcci terapeutici volti a sua eliminazione sono ricapitolati parzialmente o completamente dedifferentiated epatociti. Abbiamo dimostrato che cccDNA funzionalmente è formata nelle culture di fegato-on-a-chip 3D, risponde a stimoli fisiologici e possa essere mirato interferendo con l'accessibilità del macchinario trascrizionale al genoma cccDNA4.
Risposta dell'ospite all'infezione di HBV in 3D mimica di fegato-on-a-chip quelli osservati in pazienti con infezione da HBV, che consentano l'identificazione di biomarcatori per infezione, così come successo terapeutico. Tra le caratteristiche uniche delle culture di fegato-on-a-chip è la capacità di valutare le risposte ospite a lungo termine tra PHH e altre cellule parenchimale all'interno del fegato, incluse Kupffer cellule4e cellule stellate9fegato sinusoidale cellule endoteliali10,11. Questo offre l'opportunità unica per valutare interazioni cellula/cellula in un microambiente 3D complessa.
Inoltre, il periodo di coltura estesa di questa piattaforma facilita la valutazione dei trattamenti farmacologici sequenziale e il loro impatto sulla persistenza di HBV, che non sono possibili utilizzando sistemi di coltura convenzionale dell'epatocita.
Questo protocollo descrive come 3D fegato-on-a-chip culture vengono generate, monocolture di PHH o per cocultures di PHH con cellule di Kupffer. Inoltre, descriviamo la produzione di HBV purificata per gli studi di basso-molteplicità di infezione, come pure l'analisi successiva di host e risposte virali.
Protocollo
1. montaggio e l'equilibratura delle piastre
- Assicurarsi che sia il compressore e la pompa del vuoto connesso con la piattaforma di LiverChip siano accesi. Eseguire il montaggio e l'equilibratura delle piastre in una classe II gabinetto.
- Asetticamente montare le piastre di microfluidica posizionando una membrana sterile tra la piastra di base e l'aggiunta di ben contenente piastra superiore (Figura 1a).
- Assicurarsi che la membrana sterile senza intoppi riposa su due perni della piastra di base, da compromessi posizionamento irregolare membrana la circolazione di microfluidica.
- Aggiungere un coperchio piastra sterile e serrare le viti alla base della piastra utilizzando una coppia di precisione automatizzati per 33 lb usando una spirale sequenza di serraggio. Garantire che tutte le viti siano serrate a 35 lb utilizzando una coppia di serraggio manuale. Durante questo passaggio, assicurarsi che le viti siano serrate simmetricamente (Figura 1a).
- Preriscaldare dell'epatocita semina Media contenente Williams E media, degli epatociti primari scongelamento e placcatura integratori, 5% siero bovino fetale (FBS) e 1 µM desametasone a 37 ° C prima di innesco.
- Adescare la piastra completamente montata inserendola all'interno del bacino di lavaggio e l'aggiunta di 400 µ l dell'epatocita semina medio al lato serbatoio di ciascun pozzetto. Garantire che la piastra scatta il bacino di lavaggio completamente.
- Avviare il flusso verso l'alto per 3,5 min a 1 µ l/s. La funzione di successo della circolazione microfluidica può essere accertata attraverso gli indicatori rossi sul lato della piastra (Figura 1a).
- Una volta che il mezzo viene pompato al lato di crescita delle cellule della piastra, assicurando il corretto montaggio del canale di flusso, aggiungere un ulteriore 1,2 mL di semina medio dell'epatocita.
- Accuratamente il piano di trasferimento nell'alloggiamento all'interno di un incubatore a 37 ° C e 5% CO2 e avviare il flusso verso l'alto ad una portata di 1 µ l/s per 16 h (Figura 1C).
- Il piano di trasferimento al molo di lavaggio ed eliminare bolle nel pozzo pipettando delicatamente su e giù.
- Aggiungere uno sterile, tondo carta da filtro, seguita da un'impalcatura di attaccamento cellulare e un anello di ritegno, a ciascun pozzetto usando pinze sterili. Premere giù ogni bene con uno stantuffo sterile per bloccare l'anelli di fermo e ponteggi in posizione (Figura 1b).
- Aspirare tutto il mezzo e aggiungere 400 µ l di terreno semina dell'epatocita preriscaldati delicatamente sopra il patibolo e avviare il flusso verso il basso per 3,5 min a 1 µ l/s.
- Aspirato tutto il mezzo pompato fuori dal lato del serbatoio della piastra. Questo passaggio è necessario sostituire il mezzo contenuto nel canale di flusso.
- Aggiungere 1,4 mL di semina a ciascun pozzetto medio prima di restituire la piastra al dock per completare il volume totale per pozzetto dell'epatocita. Il volume per pozzetto è ora 1,6 mL (1,4 mL nel pozzo e 0,2 mL nel canale di flusso).
2. lo scongelamento e semina degli epatociti per monocolture
- Preriscaldare lo scongelamento medio e medio semina dell'epatocita a 37 ° C prima del disgelo un flaconcino di PHH secondo le istruzioni del fornitore dell'epatocita. Utilizzare una centrifuga a temperatura ambiente durante questa fase per evitare sbalzi di temperatura. Eseguire lo scongelamento e semina degli epatociti in una classe di governo II.
- Risospendere le cellule in 1 mL di terreno di semina dell'epatocita e contare le celle utilizzando trypan blu. Assicurarsi che la vitalità delle cellule è superiore al 90%.
- Mantenere le cellule su ghiaccio fino a quando non vengono aggiunti ai pozzetti.
- Trasferire la piastra equilibrata e completamente assemblata nel Dock di lavaggio e aspirare tutte le medie dei pozzi.
- Aggiungere 600.000 epatociti in ciascun pozzetto in un volume di 500 µ l di medie semina dell'epatocita.
- Avviare il flusso verso il basso con una portata di 1 µ l/s e portare il volume totale nel pozzo a 1,6 mL aggiungendo 900 µ l di semina medio dell'epatocita.
- Trasferire la piastra per l'alloggiamento all'interno di un incubatore a 37 ° C e 5% CO2.
- Avviare il flusso verso il basso con una portata di 1 µ l/s per 8h, seguita da un'inversione del flusso verso l'alto ad una portata di 1 µ l/s per 8 h.
- Il piano di trasferimento al molo di lavaggio ed aspirare tutto il mezzo dai pozzi.
- Aggiungere 400 µ l di mezzo di mantenimento dell'epatocita (Williams E supplementato con supplementi di manutenzione dell'epatocita e 100 nM desametasone) per ogni bene e avvio il flusso verso il basso con una portata di 1 µ l/s per 3,5 min.
- Aspirare tutto il mezzo dal serbatoio e aggiungere 1,4 mL di mezzo di mantenimento dell'epatocita (Figura 1D).
- Sostituire il mezzo con mezzo di mantenimento dell'epatocita ogni 48 h. Per garantire che tutte le medie nel pozzo viene sostituito, è necessario eseguire una fase di lavaggio prima dell'aggiunta di mezzo di mantenimento dell'epatocita fresco.
- Per la fase di lavaggio, il piano di trasferimento al molo di lavaggio, aspirare tutto il mezzo dai pozzetti e aggiungere 400 µ l di terreno di manutenzione.
- Avviare il flusso verso il basso a 1 µ l/s per un minimo di 3,5 aspirato tutto il mezzo che compaiono sul lato serbatoio dei pozzi.
- Aggiungere 1,4 mL di mezzo di manutenzione degli epatociti e, all'interno di un incubatore a 37 ° C e 5% CO2, trasferire la piastra nell'alloggiamento e avviare il flusso verso l'alto ad una portata di 1 µ l/s per 48 h (Figura 1f).
Nota: Per monoculture dell'epatocita usati come comandi per cocultures, al fine di garantire condizioni controllate, viene utilizzato un secondo tipo di mezzo di manutenzione, che è specifico per l'uso in cocultures con cellule di Kupffer umane primarie. 48 h dopo la sostituzione dell'epatocita semina medium con mezzo di mantenimento dell'epatocita (3 ° giorno post-semina), il mezzo di manutenzione regolare dell'epatocita verrà sostituito con mezzo di manutenzione coculture II, soprattutto nelle monocolture di PHH quando si confrontano per cocultures sia PHH Kupffer cellule, come i componenti medi differiscono leggermente.
3. lo scongelamento e la semina delle cellule di Kupffer ed epatociti per co-colture
- Al fine di garantire un confronto accurato dei risultati, sempre confrontare cocultures cellula PHH/Kupffer a monocolture PHH
- Per cocultures delle cellule PHH e Kupffer, scongelare un flaconcino di cellule di Kupffer nel avanzati di Dulbecco modificato medio dell'Aquila (AdDMEM) senza desametasone ma completati con lo scongelamento degli epatociti primari e placcatura integratori (coculture semina medio) secondo alle istruzioni del fornitore. Eseguire lo scongelamento e la semina delle cellule di Kupffer ed epatociti in una classe di governo II.
- Risospendere le cellule in 1 mL di coculture semina medio e contare le celle utilizzando trypan blu. Assicurarsi che la vitalità delle cellule è superiore al 90%.
- Mantenere le cellule sul ghiaccio prima di aggiungerli ai pozzetti per evitare l'adesione delle cellule.
- Seguire le istruzioni riportate nei punti 2.1 – 2.3 per lo scongelamento di epatociti umani primari.
- Trasferire la piastra equilibrata e completamente assemblata nel Dock di lavaggio e aspirare tutte le medie dei pozzi.
- Aggiungere 60.000 cellule di Kupffer e/o 600.000 epatociti in ciascun pozzetto in un volume totale di 250 µ l di coculture semina medio.
- Avviare il flusso verso il basso con una portata di 1 µ l/s e 900 µ l di coculture semina medio in ciascun pozzetto.
- Trasferire la piastra nell'alloggiamento all'interno di un incubatore a 37 ° C e 5% CO2.
- Avviare il flusso verso il basso con una portata di 1 µ l/s per 8 h, seguito da inversione di flusso nella direzione verso l'alto ad una portata di 1 µ l/s per 8 h.
- Il piano di trasferimento al molo di lavaggio ed aspirare tutto il mezzo dai pozzi.
- Aggiungere 400 µ l di mezzo di manutenzione coculture I (AdDMEM senza desametasone ma completato con supplementi di manutenzione dell'epatocita) per ogni bene e avvio il flusso verso il basso con una portata di 1 µ l/s per 3,5 min.
- Aspirare tutto il mezzo dal lato serbatoio e aggiungere 1,4 mL di mezzo di manutenzione coculture ho in ciascun pozzetto.
- Trasferire la piastra nell'alloggiamento all'interno di un incubatore a 37 ° C e 5% CO2 e avviare il flusso verso l'alto ad una portata di 1 µ l/s per 48 h.
- Il piano di trasferimento al molo di lavaggio ed aspirare tutto il mezzo dai pozzi. Aggiungere 400 µ l di terreno di coculture manutenzione II (Williams E medio senza desametasone ma completati con 100 nM idrocortisone e dell'epatocita manutenzione i supplementi) e avviare il flusso verso il basso a 1 µ l/s per 3,5 min.
- Aspirare tutto il mezzo che compaiono sul lato serbatoio dei pozzi.
- Aggiungere 1,4 mL di mezzo di manutenzione coculture II e trasferire la piastra nell'alloggiamento all'interno di un incubatore a 37 ° C e 5% CO2.
- Avviare il flusso verso l'alto ad una portata di 1 µ l/s per 48 h (Figura 1e).
- Sostituire il mezzo ogni 48 h con mezzo di manutenzione coculture II. Per garantire che tutte le medie nel pozzo sono sostituito, è necessario eseguire una fase di lavaggio prima dell'aggiunta di terreno nuovo.
- Per lavare, il piano di trasferimento al molo di lavaggio, aspirare tutto il mezzo dai pozzetti e aggiungere 400 µ l di terreno di coculture manutenzione II.
- Avviare il flusso verso il basso a 1 µ l/s per un minimo di 3,5 aspirato tutto il mezzo che compaiono sul lato serbatoio dei pozzi.
- Aggiungere 1,4 mL di mezzo di manutenzione coculture II e trasferire la piastra nell'alloggiamento all'interno di un incubatore a 37 ° C e 5% CO2e avviare il flusso verso l'alto ad una portata di 1 µ l/s per 48 h (Figura 1f).
4. produzione di un Virus di epatite B per gli studi di infezione
- Eseguire questa sezione del protocollo in un laboratorio III livello di contenimento. Fare la semina, le modifiche medie, medium collezione e concentrazione di virus in una classe di governo II.
- La cultura di cellule produttrici di HBV (ad es., HepDE19, HepAD38) in boccette del 5-strato di T1000 rivestite con collagene in 120 mL di DMEM/F12 completo (10% FBS, penicillina/steptomycin, aminoacidi non essenziali, 500 μg/mL G418 e tetraciclina 1 μg/mL) fino a raggiungere confluency di 90%.
- Modificare il mezzo per mezzo di induzione (DMEM completo senza tetraciclina) per indurre la produzione di HBV.
- Il volume medio completo ogni 48 h per 12 giorni post-diritto di recesso della tetraciclina di raccogliere e conservare a 4 ° C.
- Filtrare il mezzo raccolto attraverso un filtro a 0,45 µm bottiglia superiore.
- Aggiungere sterile PEG 8000 in tampone fosfato salino (PBS) al mezzo raccolto a una concentrazione finale di 4% w/w, miscelare capovolgendo x-10 8x e incubare a 4 ° C per 16 h. centrifuga a 10.000 x g per 1 h a 4 ° C per raccogliere il virus PEG-precipitata e risospendere il pellet in PBS contenente 10% FBS.
- Combinare il virus PEG-precipitata da tutti i punti di tempo raccolto e lo strato sopra un cuscino di saccarosio del 20%. Centrifugare a 140.000 x g per 16 h a 4 ° C, utilizzando un rotore SW28.
- Aspirare il supernatante e risospendere il pellet in PBS completati con 10% FBS e aliquotare e conservare a-80 ° C.
- Determinare il numero di copie di HBV DNA presente nel surnatante di HBV DNA qPCR (passaggio 6).
5. infezione delle culture 3D con HBV
- Eseguire le infezioni in una classe II gabinetto all'interno di un laboratorio III livello di contenimento.
- 3 giorni dopo la semina le monocolture o cocultures, scongelare il numero richiesto di HBV-contenente le aliquote a temperatura ambiente e diluire la dose necessaria virus in 1,8 mL di mezzo di mantenimento dell'epatocita o mezzo di manutenzione coculture II per pozzetto, rispettivamente.
- Questo virus diluito 1,8 mL è sufficiente per la fase di lavaggio (400 µ l) e la sostituzione del mezzo nel pozzo (1,4 mL). Tuttavia, la necessaria molteplicità di infezione deve essere adattate all'account per il volume finale della cultura di 1,6 mL.
- Il piano di trasferimento al molo di lavaggio ed aspirare tutto il mezzo dai pozzi. Aggiungere 400 µ l di terreno contenente HBV e avviare il flusso verso il basso a 1 µ l/s per un minimo di 3,5 aspirare tutti i media che compaiono sul lato serbatoio dei pozzi.
- Aggiungere 1,4 mL di HBV-contenente medio/coculture manutenzione Media II per bene e trasferire la piastra nell'alloggiamento all'interno di un incubatore a 37 ° C e 5% CO2. Avviare il flusso verso il basso con una portata di 1 µ l/s per 8h, seguita da un'inversione di direzione ascendente con una portata di 1 µ l/s.
- 24 h dopo l'aggiunta di HBV, il piano di trasferimento al molo di lavaggio ed aspirare tutto il mezzo dai pozzi.
- Lavare ogni bene nella piastra 3 x con il mezzo corrispondente, secondo il tipo di cultura come indicato passaggi 2.12 – 2,14, per eliminare virus avanzi dal pozzo. In contrasto con passaggi 2.12 – 2,14, aggiungere 1,6 mL di terreno in ogni pozzetto all'account per il volume supplementare da campionare per escludere il riporto di inoculo (Figura 1 g).
- In seguito questi passaggi di lavaggio, è necessario raccogliere 200 µ l di terreno da ogni pozzetto per confermare la rimozione completa dell'inoculo HBV mediante la quantificazione del DNA di HBV extracellulare.
- Trasferire la piastra per l'alloggiamento all'interno di un incubatore a 37 ° C e 5% CO2e avviare il flusso verso l'alto ad una portata di 1 µ l/s per 48 h.
- 48 ore più tardi, raccogliere il volume ben completo per l'analisi a valle, seguita da tre lavaggi in mezzo di mantenimento dell'epatocita come descritto in procedura: 2.12 – 2,14. Sostituire il mezzo e lavare ogni bene 3 x ogni 48 h fino alla terminazione sperimentale.
6. quantificazione del DNA di HBV extracellulare
- Isolare il DNA totale dai surnatanti della cultura secondo le istruzioni del produttore con l'aggiunta di 1 µ g di vettore RNA in un laboratorio III livello di contenimento per garantire l'inattivazione di virus in campioni prima di spostarli in un'altra area.
- Preparare un mix master contenente miscela master PCR quantitativa, 600 nM forward primer, primer inverso di 600 nM e 300 nM della sonda.
- 7 µ l di master mix in ciascun pozzetto di una piastra 384 pozzetti.
- Aggiungere 5 µ l di campioni di DNA in duplicato, un controllo di modello di no e duplicati di diluito serialmente HBV contenenti genoma plasmide standard basato su (per esempio, pCMV-HBV) che vanno da 109 copie per reazione di 102 copie per reazione in ciascun pozzetto della piastra di qPCR.
- Posizionare un coperchio adesivo sopra la piastra e garantire che ogni bene è sigillati correttamente.
- Centrifugare la piastra per 1 min a 300 x g.
- Avviare la qPCR eseguito secondo le istruzioni del produttore. Le condizioni di ciclo di PCR in tempo reale sono 95 ° C per 10 minuti, seguiti da 40 cicli di 95 ° C per 15 s e 60 ° C per 1 min.
- Quantificare il numero di copie di HBV DNA all'interno di campioni incogniti secondo la curva standard.
7. quantificazione di HBV intracellulare Pregenomic (pg) RNA
- Isolare il RNA totale dalle impalcature secondo le istruzioni del produttore. Al fine di garantire la lisi cellulare completo, vortexare ciascuna impalcatura 3x per 30 s seguita da centrifugazione a 300 x g per 1 min tra ogni Vortex. Eseguire la lisi delle cellule in laboratorio III per garantire l'inattivazione di virus in campioni prima di spostarli in un'altra area del livello di contenimento.
- Trascrivere il cDNA dal RNA isolato secondo le istruzioni del produttore.
- Le condizioni di ciclo per la retrotrascrizione sono 25 ° C per 10 minuti, 37 ° C per 120 min e 85 ° C per 5 min.
- Mantenere i campioni di cDNA a 4 ° C per breve periodo o a-20 ° C per la conservazione a lungo termine.
- Preparare miscele master per pgRNA e mix master PCR quantitativa RPS11 contenente primer forward e reverse per pgRNA e RPS11 (usato come gene housekeeping) ad una concentrazione finale di 0,2 µM.
- Aggiungere 7,5 µ l di master mix e 2,5 µ l di cDNA per pozzetto su una piastra 384 pozzetti. Misura RPS11 e pgRNA di ogni campione in duplice copia e includere un controllo di modello di no bene per entrambi i geni.
- Posizionare un coperchio adesivo sopra la piastra e garantire che ogni bene è sigillato completamente.
- Centrifugare la piastra per 1 min a 300 x g.
- Inserire la piastra il ciclatore qPCR e avviare la qPCR eseguire utilizzando il protocollo standard di PCR quantitativo secondo le istruzioni del produttore. Le condizioni di ciclo di PCR in tempo reale sono 50 ° C per 2 min, 95 ° C per 2 min, seguiti da 40 cicli di 95 ° C per 15 s e 60 ° C per 1 min.
- Calcolare l'espressione di pgRNA rispetto al RPS11.
8. immunofluorescenza dell'antigene virale
- Rimuovere l'anello di ritegno dal pozzo e il patibolo con il forcipe in una classe di governo II nel laboratorio III livello di contenimento.
- Difficoltà i ponteggi contenenti cellule con paraformaldeide al 4% in 1 mL di PBS per 30 min a temperatura ambiente in laboratorio III terzo livello di contenimento. Le seguenti operazioni possono essere eseguite in un'area diversa.
- Lavare i ponteggi 3 x con 1 mL di PBS.
- Permeabilize le celle utilizzando 0.1% Triton X 100 in 1 mL di PBS per 1 h a temperatura ambiente.
- Lavare i ponteggi 3 x con 1 mL di PBS.
- Legame non specifico di blocco mediante incubazione delle impalcature con BSA 1% in 1 mL di PBS per 16 h a 4 ° C.
- Lavare i ponteggi 3 x con 1 mL di PBS.
- Eseguire l'anticorpo primario che macchia usando antigene core del virus di coniglio anti-epatite B ad una diluizione di 1: 200 in 1% BSA in 1 mL di PBS per 16 h a 4 ° C.
- Lavare i ponteggi 1 x con 0.1% Tween in 1 mL di PBS (PBS-Tween) e 3 volte con 1 mL di PBS.
- Eseguire l'anticorpo secondario che macchia usando capra anti-coniglio IgG (H + L) Croce-adsorbito Alexa Fluor 594-coniugato anticorpo secondario ad una diluizione di 1:2,000 in 1% BSA in 1 mL di PBS per 16 h a 4 ° C.
- Lavare i ponteggi 1 x con 1 mL di 0,1% PBS-Tween e 3 volte con 1 mL di PBS.
- Colorante di contrasto i ponteggi utilizzando DAPI in 1 mL di PBS ad una concentrazione di 2 µ g/mL per 10 min a temperatura ambiente.
- Lavare i ponteggi 1 x con 1 mL di 0,1% PBS-Tween e 3 volte con 1 mL di PBS.
- Trasferire le impalcature di un vetrino da microscopio e montarlo per l'imaging.
- Immagine i ponteggi utilizzando un microscopio a fluorescenza.
9. umano dell'albumina ELISA
- Eseguire questa sezione del protocollo in una classe II gabinetto allocata in laboratorio III terzo livello di contenimento, se lavora con materiali infettivi.
- Per valutare la fattibilità e la funzionalità metabolica di PHH, valutare la produzione di albumina umana da ELISA.
- Cappotto 96 pozzetti con 50 µ l di anticorpo di capra anti-umano diluito 1: 800 nel tampone di rivestimento (carbonato/bicarbonato di 100 mM, pH 9,6). Coprire le piastre e incubare per 2 ore a 37 ° C o una notte a 4 ° C.
- L'anticorpo di rivestimento dalla piastra di aspirare e lavare 4 volte con 200 µ l di 0.05% PBS-Tween.
- Aggiungere 200 µ l di tampone bloccante (1% BSA in PBS), coprire le piastre e li Incubare per 1h a 37 ° C o conservare a 4 ° C per 3 mesi. Per l'archiviazione a lungo termine, aggiungere sodio azide 0.05% per il tampone di bloccaggio.
- Aspirare il tampone bloccante e lavare 1x con 200 µ l di 0.05% PBS-Tween.
- Aggiungere 50 µ l di campioni precedentemente diluiti per pozzetto (1: 100). Il diluente del campione contiene 1% BSA in 0.05% PBS-Tween. Incubare per 1h a 37 ° C o una notte a 4 ° C.
Nota: Incubare i campioni allo stesso tempo come i campioni. Una gamma di concentrazione di 500 – 0,488 ng/mL (diluizioni seriali 1:2) è raccomandato. Eseguire tutte le diluizioni seriali di albumina umana nel diluente. - Aspirare i campioni dalla piastra e lavare 4 volte con 200 µ l di 0.05% PBS-Tween.
- Aggiungere 50 µ l di anticorpo anti-umano dell'albumina HRP-coniugato di capra precedentemente diluito 1: 10.000 nel diluente. Incubare per 2 ore a 37 ° C o una notte a 4 ° C.
- Aspirare l'anticorpo dalla piastra e lavarlo 6x con 200 µ l di 0.05% PBS-Tween.
- Aggiungere 100 µ l di Reagente TMB e, non appena i più alti standard sono completamente sviluppati, aggiungere 100 µ l 1 M H2così4 per interrompere la reazione colorimetrica.
- Leggere l'assorbanza a 450 nm in un lettore di piastra a 96 pozzetti per l'analisi.
10. interleuchina (IL) 6 e fattori di necrosi del tumore (TNF) α produzione in 3D co-colture
- Eseguire questa sezione del protocollo in una classe II gabinetto allocata in laboratorio III terzo livello di contenimento, se lavora con materiali infettivi.
- Quantificare la produzione IL6 e TNF per valutare la funzionalità e la vitalità delle cellule di Kupffer primarie. Per indurre la produzione di queste citochine dalle cellule di Kupffer, trattare cocultures con 1 lipopolysaccharide µ g/mL (LPS) 9 d post-semina in mezzo di manutenzione di coculture II per 48 h.
- Al giorno 11 post-semina, raccolto il mezzo da ogni pozzetto e conservare a-80 ° C.
- Misurare la concentrazione di IL6 e TNFα nel mezzo di coltura mediante un'analisi appropriata e secondo le istruzioni del produttore.
Risultati
Descriviamo una piattaforma semplice e versatile per la coltura a lungo termine delle cellule di Kupffer umane primarie e/o epatociti e loro infezione con HBV. Cellule umane primarie sono seminate su impalcature di polistirolo rivestite con collagene all'interno di un gruppo di piastra microfluidica, che continuamente perfuses le cellule con crescita medio (Figura 1a).
PHH, che sono solitamente solo stabile per un periodo di tempo limitato in sistemi di coltura convenzionale, può essere mantenuta in modo funzionale per lunghi periodi di tempo. Albumina umana, che è secreta dagli epatociti funzionali ed è considerato il migliore indicatore per la valutazione del metabolismo epatico, è stabile e altamente espressi da culture 3D fino al giorno 40 post-semina (Figura 2). Per cocultures, vitalità e funzionalità delle cellule di Kupffer possono essere valutate dalla secrezione di citochine specifiche (ad es., IL6 e TNF). Per misurare la produzione di citochine, l'uso di mezzi di rilevamento basato su acquisizione LPS-stimolazione di cocultures è raccomandato (Figura 3).
Le cellule formano microtissues epatica, di solito entro 3 giorni dalla semina del PHH, dimostrando Duttuli Biliari funzionale e polarizzazione cellulare completo (Figura 2). Oltre mantenendo il loro fisiologico metabolismo cellulare, queste culture diventano eccezionalmente suscettibili di infezione da HBV. HBV DNA e altri marcatori virali, a differenza di altri sistemi di coltura, diventano facilmente rilevabile da post-infezione giorno 2 (Figura 4). Oltre a secreti marcatori di infezione virale, impalcature degli epatociti contenenti possono essere estratto dalle colture e utilizzate per la rilevazione di immunofluorescenza di antigeni virali (ad es., HBsAg, HBcAg) (Figura 4). Dove culture dell'epatocita convenzionali richiedono l'inoculazione con almeno 500 HBV GE per cella e l'aggiunta di 2% DMSO e 4% PEG, minor come 0,05 HBV GE sono in grado di avviare l'infezione in 3-d culture senza il requisito di DMSO o PEG (Figura 4).
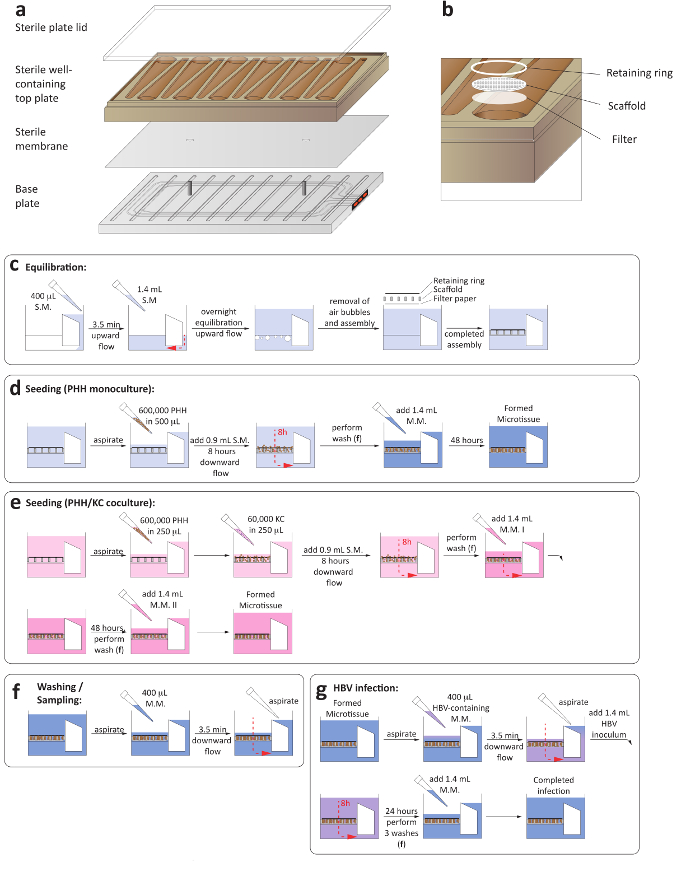
Figura 1: set-up delle culture di fegato-on-a-chip 3-d. (un) questo è un layout schematico per l'assemblaggio della piastra di coltura al fine di garantire l'istituzione di microfluidica circolazione. (b) questo pannello mostra una vista ravvicinata dei pozzi di cultura, tra cui la carta da filtro, impalcatura e anello di ritegno. (c) questo pannello mostra il processo di equilibrazione piastra prima della semina le culture. I prossimi due pannelli mostrano il processo di semina per (d) dell'epatocita monocolture e cellulare dell'epatocita/Kupffer (e) cocultures. (f) questo pannello mostra i passaggi di lavaggio coinvolti nei cambiamenti medi. (g) questo pannello mostra l'assetto di infezione di HBV, compresa la rimozione dell'inoculo. S.M. = semina medio, M.M. = mezzo di manutenzione. Clicca qui per visualizzare una versione più grande di questa figura.
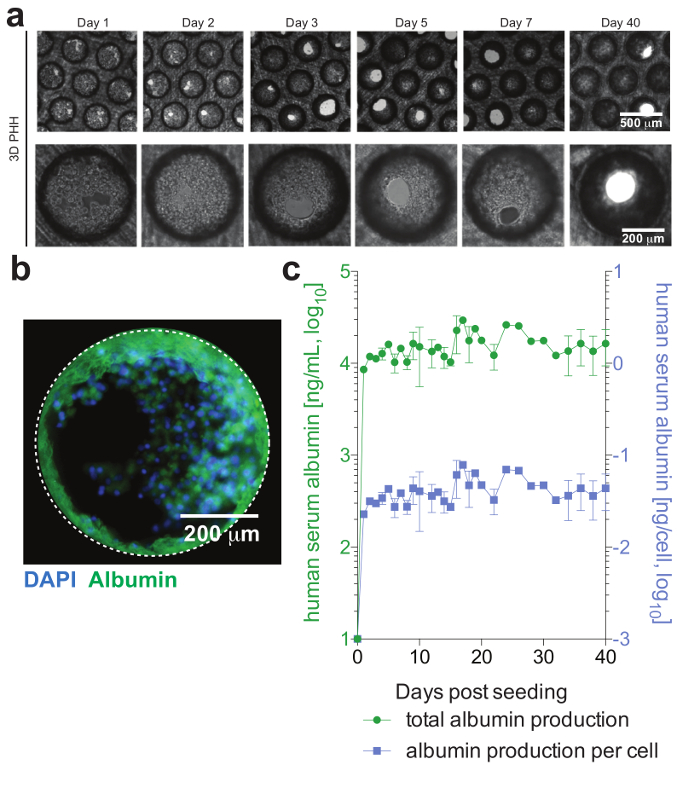
Figura 2: microtessuti epatico formazione e dell'epatocita attuabilità. (un) questo pannello mostra le immagini di campo chiaro longitudinale delle monocolture 3D dell'epatocita dimostrando microtessuti formazione segue semina. (b) questo pannello mostra le immagini di immunofluorescenza delle culture per nuclei (blu) e albumina umana (verde). (c) questo pannello mostra longitudinale totale albumina, come pure per la cellula adeguando la produzione di albumina, durante 40 giorni delle monocolture dell'epatocita, come determinato da ELISA. I dati riportati sono la media ± SD Questa figura è adattata da Ortega-Prieto et al.4. Clicca qui per visualizzare una versione più grande di questa figura.
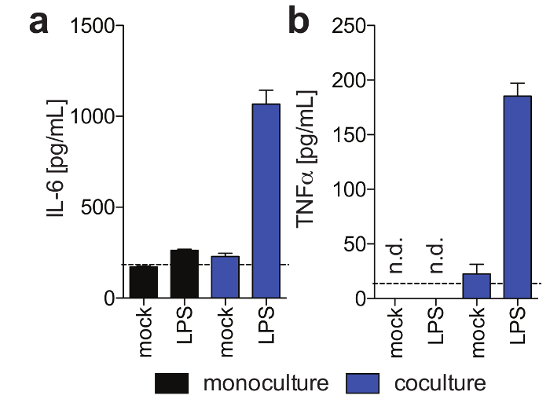
Figura 3: funzionalità di cellule di Kupffer in 3-d cocultures. Questi pannelli mostrano la secrezione di (un) IL6 e (b) TNFα in monocolture dell'epatocita e cellulare dell'epatocita/Kupffer cocultures 11 giorni post-semina in risposta ai LPS esogenicamente aggiunto al giorno 9 post semina, come determinato tramite umano magnetico Dosaggio di Luminex. Questa figura è adattata da Ortega-Prieto et al.4. Clicca qui per visualizzare una versione più grande di questa figura.
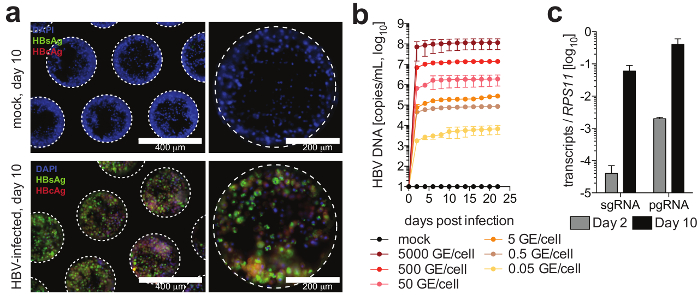
Figura 4: infezione da HBV nel fegato-on-a-chip culture. (un) questo pannello mostra la rilevazione di microscopia di immunofluorescenza di HBcAg (rosso), HBsAg (verde) e nuclei (blu) 10 giorni che seguono l'infezione delle colture con HBV. (b) questo pannello mostra la sensibilità delle colture all'infezione di HBV utilizzando diverse molteplicità di infezione, come determinato da una quantificazione del DNA di HBV nei sovranatante. (c) questo pannello mostra la quantificazione dell'accumulo longitudinale di HBV pgRNA riguardante il gene housekeeping RPS11. I dati riportati sono la media ± SD Questa figura è adattata da Ortega-Prieto et al.4. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Le sfide nel mantenimento di colture a lungo termine di PHH hanno guidato lo sviluppo di diversi modelli di cultura con una maggiore funzionalità e longevità, ciascuno che esibiscono differenziali vantaggi e svantaggi. Ora è ampiamente riconosciuto che culture statiche 2D di PHH sono che imita alcuni aspetti della biologia dell'epatocita per quantità molto limitata di tempo. Così, bioerodibili cocultures12,13, sferoide culture14,15, e16,di culture di fegato-on-a-chip 3D17 stanno rapidamente sostituendo questi sistemi più di base. Soprattutto quando lo studio delle malattie infettive, che hanno si con il loro ospite di utilizzare specifici microambienti, il requisito per fornire ambienti fisiologici è sostenuto dalla natura spesso impegnativa di coltura umana-Tropico malattie infettive, tra cui il virus dell'epatite C, HBV e la malaria.
La fase più critica nell'esecuzione 3D fegato-on-a-chip culture è la qualità dei tipi cellulari primarie inizialmente provenienza. Queste cellule devono essere testate per la loro capacità di aderenza e solo placcabili PHH lotti dovrebbero essere utilizzati al fine di garantire la formazione di tessuto di successo e la generazione di cultura. Anche se PHH appena isolato può essere utilizzato, loro crioconservazione è solitamente complicato e richiede speciali congelatori tasso-controllato.
In contrasto con le colture 2D statiche convenzionali, il background genetico di host è trascurabile per quanto riguarda la suscettibilità all'infezione da HBV, e tutti i donatori dell'epatocita testata-finora sono in grado di stabilire l'infezione di HBV4.
Anche se derivato paziente HBV stabilisce infezioni delle culture 3D, è imperativo utilizzare PEG-precipitata e saccarosio cuscino-purificato HBV ogni volta utilizzando viscoelastica HBV produttore cella linee per la generazione di inoculi virali. Surnatanti delle cellule direttamente applicati alle culture di fegato-on-a-chip 3D, sia attraverso la presenza di fattori inibitori o a causa di un'incompatibilità di fattori di crescita presenti con epatociti, non facilmente provocare infezione. Inoltre, quando si seleziona paziente-derivato inoculi virali, solo siero dovrebbe essere usato, poiché al plasma inevitabilmente coagula e ostruisce la circolazione di microfluidica della piattaforma cultura.
Indipendentemente dal fatto l'inoculo virale utilizzato, garantendo la vitalità cellulare e la differenziazione, nonché a garantire la rimozione completa dell'inoculo iniziale da HBV, è chiave a successo studi di infezione a lungo termine. Il modo più conveniente per fare questo è campionamento culture dopo la rimozione dell'inoculo virale, come pure la misurazione dei livelli di albumina sierica umana per tutto il periodo di cultura. Della nota, allo stesso modo per tutte le altre piattaforme descritte, l'infezione da HBV, una volta stabilito, non prontamente diffuso a cellule non infette. Il meccanismo di fondo per questo rimane evasivo, poiché l'infezione da HBV in vivo prontamente infetta la maggior parte degli epatociti all'interno del fegato.
Per quanto riguarda cocultures delle cellule PHH e Kupffer, si consiglia di eseguire prove del lotto delle cellule stellate per valutare la secrezione di IL6 e TNFα in risposta alla stimolazione dei LPS, poiché non tutti i donatori di cellule di Kupffer disponibili in commercio hanno una reattività uguale.
Cosa importante, per tutti i trattamenti farmacologici o infezione iniziale delle culture con HBV, il volume totale del pozzo (1,4 mL), anche a partire dal canale microfluidico (0,2 mL), deve tener conto per il calcolo delle concentrazioni di farmaco o dell'inoculo. Al fine di garantire un dosaggio preciso, viene eseguita una fase di lavaggio con mezzo contenente HBV o farmaci al fine di adescare il canale microfluidico.
La piattaforma utilizzata utilizza 600.000 PHH per pozzetto, che assicura gli epatociti multistrati all'interno di impalcature. Anche se il numero di cellulare può essere variato, la concentrazione di cellule selezionate garantisce risultati ottimali. Il formato di piastra detiene un totale di 12 ponteggi, che può essere aggiornata a 36 impalcature. Tuttavia, a causa dei requisiti di microfluidica, scalabilità di numeri ben più alti non è possibile fino ad oggi.
Utilizzando questi approcci, culture possono essere mantenute con prestazioni ottimali delle cellule per almeno 40 giorni, che, finora, offre opportunità senza precedenti per valutare nuovi farmaci candidati, come pure di studiare l'interazione complessa tra differenti delle cellule epatici popolazioni durante l'infezione da HBV.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Quest'opera è stata finanziata da una sovvenzione di Starter dal Consiglio europeo della ricerca (637304), un Wellcome Trust Investigator Award (104771/Z/14/Z) e dalle innovazioni Bio CN.
Materiali
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| William's E Medium, no phenol red | GIBCO | A12176-01 | |
| Hepatocyte Thaw Medium | GIBCO | CM7500 | |
| Primary Hepatocyte Thawing and Plating Supplements | GIBCO | CM3000 | |
| Primary Hepatocyte Maintenance Supplements | GIBCO | CM4000 | |
| DMEM/F-12 | GIBCO | 11320-033 | |
| Advanced DMEM | GIBCO | 12491023 | |
| DPBS, no calcium, no magnesium | GIBCO | 14190-144 | |
| MEM Non-Essential Amino Acids (NEAA) 100x | GIBCO | 11140050 | |
| Penicillin-Streptomycin (10,000 U/mL) | GIBCO | 15140-122 | |
| Fetal Bovine Serum, USA origin, Heat Inactivated, sterile-filtered, suitable for cell culture | SIGMA | 12106C | |
| Hydrocortisone | SIGMA | H0888 | |
| Trypan blue | Merck | T8154 | |
| Collagen from calf skin | Merck | C9791 | |
| G418 | SIGMA | G418-RO | |
| Tetracycline | SIGMA | T3258 | |
| Polyethylene glycol 8000 | SIGMA | P2139 | |
| Sucrose | SIGMA | SO389 | |
| Sodium carbonate anhydrous | SIGMA | 451614-25G | |
| Sodium bicarbonate | SIGMA | S5761 | |
| Sodium azide | SIGMA | S2002-5G | |
| Sulfuric acid, 99.999% | SIGMA | 339741 | |
| 4% Paraformaldehyde | SIGMA | 252549 | |
| Triton-X 100 | SIGMA | X100 | |
| Tween 20 | SIGMA | P1379 | |
| DAPI | SIGMA | D9564 | |
| Albumin (human) | SIGMA | A9731 | |
| Fisher BioReagent Bovine Serum Albumin, Fraction V, Heat Shock Treated | Fisher Scientific | BP9701-100 | |
| ProLong Gold Antifade Mountant | Invitrogen | P36930 | |
| TaqMan Universal Master Mix II, no UNG | Applied Biosystems | 4440040 | |
| SYBR Select Master Mix | Applied Biosystems | 4472903 | |
| Lipopolysaccharide from Escherichia coli K12 | InvivoGen | tlrl-eklps | |
| Name | Company | Catalog Number | Comments |
| Kits/Consumables | |||
| Sterile membrane | CN Bio innovations | LC-SC | |
| LiverChip Perfusion cell culture plate | CN Bio innovations | LC12 | |
| LiverChip culture plate lid | CN Bio innovations | LC-SC | |
| Sterile round filter paper | CN Bio innovations | LC-SC | |
| Cell attachment scaffold | CN Bio innovations | LC-SC | |
| Retaining ring | CN Bio innovations | LC-SC | |
| Sterile plunger | CN Bio innovations | LC-ST | |
| Dneasy blood & tissue kit | Qiagen | 69506 | |
| Rneasy mini kit | Qiagen | 74106 | |
| Human Magnetic Luminex assay | R&D Systems | ||
| 1-Step Ultra TMB-ELISA Substrate Solution | ThermoFisher Scientific | 34028 | |
| High Capacity cDNA Reverse Transcription Kit | ThermoFisher Scientific | 4368814 | |
| QIAamp Viral RNA Mini Accessory Set | Qiagen | 1048147 | Containing RNA carrier |
| Millicell HY 5-layer cell culture flask, T-1000, sterile | Millipore (Merck) | PFHYS1008 | |
| MicroAmp Optical 384-Well Reaction Plate with Barcode | Life technologies | 4309849 | |
| MicroAmp Optical Adhesive Film | Life technologies | 4311971 | |
| Clear Flat-Bottom Immuno Nonsterile 96-Well Plates | ThermoFisher Scientific | 442404 | |
| Sealing Tape for 96-Well Plates | ThermoFisher Scientific | 15036 | |
| Nalgene Rapid-Flow Sterile Disposable Bottle Top Filters with PES Membrane | ThermoFisher Scientific | 295-3345 | |
| Fisherbrand Microscopic Slides with Ground Edges, Twin Frost | Fisher Scientific | FB58628 | |
| Tube, Thinwall, Ultra-Clear, 38.5 mL, 25 x 89 mm | Beckman Coulter | 344058 | |
| Name | Company | Catalog Number | Comments |
| Primary cells / Cell lines | |||
| Human Plateable Hepatocytes, Transporter Qualified | Thermo Fisher Scientific | HMCPTS | |
| Cryopreserved Human Kupffer Cells | Thermo Fisher Scientific | HUKCCS | |
| HepDE19 cell line | Haitao Guo (Indiana University, IN, USA) | ||
| Name | Company | Catalog Number | Comments |
| Primers/Probes/Standards | |||
| HBV DNA forward primer | Invitrogen | 5'-GTGTCTGCGGCGTTTTATCA-3' | |
| HBV DNA reverse primer | Invitrogen | 5'-GACAAACGGGCAACATACCTT-3' | |
| HBV DNA probe | Invitrogen | 5'FAM-CCTCTKCATCCTGCTGCTATGCCTCATC-3'TAMRA | |
| pgRNA forward primer | Invitrogen | 5'-GAGTGTGGATTCGCACTCC-3' | |
| pgRNA reverse primer | Invitrogen | 5'-GAGGCGAGGGAGTTCTTCT-3' | |
| RPS11 forward primer | Invitrogen | 5'-GCCGAGACTATCTGCACTAC-3' | |
| RPS11 reverse primer | Invitrogen | 5'-ATGTCCAGCCTCAGAACTTC-3' | |
| pCMV-HBV | Professor Christoph Seeger (Fox Chase Cancer Centre, PA, USA) | ||
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| Anti-Hepatitis B virus core antigen IgG fraction (polyclonal) | DAKO | discontinued | Lot 10102505 |
| Human Albumin Antibody, A80-129A | Bethyl Laboratories. inc | A80-129A | |
| Human Albumin cross-adsorbed Antibody, A80-229P | Bethyl Laboratories. inc | A80-229P | |
| Goat anti-Rabbit IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 594 | ThermoFisher Scientific | # A-11072 | Lot 1431810 |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| LiverChip Vacuum pump | CN Bio innovations | LC-PN | |
| LiverChip Pneumatic Hookup | CN Bio innovations | LC-PN | |
| LiverChip platform | CN Bio innovations | LC-PN | |
| LiverChip plate washing dock | CN Bio innovations | ||
| Autoclavable metal forceps | VWR | 232-0106 | |
| Vortex Genie 2 | Scientific industries | SKU: SI-0236 | |
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A95765 | |
| Heraeus Multifuge X3R Centrifuge | Thermo Scientific | 75004500 | |
| SAM-12 Medical Suction High Vacuum High Flow | MGE worldwide | SAM12/01010101 | |
| NUAIRE 5800 SERIES incubator | NUAIRE | NU-5841 | |
| Automated precision torgue | CN Bio innovations | ||
| Manual torque | CN Bio innovations | ||
| LiverChip compressor | CN Bio innovations | ||
| Luminex LX-200 Instrument with xPONENT 3.1 | Luminex | ||
| Millipore Hand-Held Magnetic Separator Block | ThermoFisher Scientific | Millipore™ 40-285 | |
| FluoStar Optima Plate Reader | BMG Labtech | ||
| KOLVER Precision electric screwdrivers | VTECH ltd | FAB10RE/FR | |
| KOLVER Power supply | VTECH ltd | EDU1FR | |
| BAMBI VTS75D | Air Equipment | Discontinued | |
| Integra Vacuboy | INTEGRA | ||
| ViiA 7 Real-Time PCR System with 384-Well Block | ThermoFisher Scientific | 4453536 |
Riferimenti
- Verrier, E. R., Colpitts, C. C., Schuster, C., Zeisel, M. B., Baumert, T. F. Cell Culture Models for the Investigation of Hepatitis B and D Virus Infection. Viruses. 8 (9), (2016).
- Elaut, G., et al. Molecular mechanisms underlying the dedifferentiation process of isolated hepatocytes and their cultures. Current Drug Metabolism. 7 (6), 629-660 (2006).
- Konig, A., et al. Kinetics of the bile acid transporter and hepatitis B virus receptor Na+/taurocholate cotransporting polypeptide (NTCP) in hepatocytes. Journal of Hepatology. 61 (4), 867-875 (2014).
- Ortega-Prieto, A. M., et al. 3D microfluidic liver cultures as a physiological preclinical tool for hepatitis B virus infection. Nature Communications. 9 (1), 682 (2018).
- Allweiss, L., Dandri, M. The Role of cccDNA in HBV Maintenance. Viruses. 9 (6), (2017).
- Guo, J. T., Guo, H. Metabolism and function of hepatitis B virus cccDNA: Implications for the development of cccDNA-targeting antiviral therapeutics. Antiviral Research. 122, 91-100 (2015).
- Lucifora, J., et al. Direct antiviral properties of TLR ligands against HBV replication in immune-competent hepatocytes. Scientific Reports. 8 (1), 5390 (2018).
- Hosel, M., et al. Hepatitis B Virus Activates Signal Transducer and Activator of Transcription 3 Supporting Hepatocyte Survival and Virus Replication. Cellular and Molecular Gastroenterology and Hepatology. 4 (3), 339-363 (2017).
- Mazza, G., et al. Rapid production of human liver scaffolds for functional tissue engineering by high shear stress oscillation-decellularization. Scientific Reports. 7 (1), 5534 (2017).
- Hang, T. C., Lauffenburger, D. A., Griffith, L. G., Stolz, D. B. Lipids promote survival, proliferation, and maintenance of differentiation of rat liver sinusoidal endothelial cells in vitro. American Journal of Physiology-Gastrointestinal and Liver Physiology. 302 (3), G375-G388 (2012).
- Hwa, A. J., et al. Rat liver sinusoidal endothelial cells survive without exogenous VEGF in 3D perfused co-cultures with hepatocytes. The FASEB Journal. 21 (10), 2564-2579 (2007).
- Khetani, S. R., Bhatia, S. N. Microscale culture of human liver cells for drug development. Nature Biotechnology. 26 (1), 120-126 (2008).
- March, S., et al. Micropatterned coculture of primary human hepatocytes and supportive cells for the study of hepatotropic pathogens. Nature Protocols. 10 (12), 2027-2053 (2015).
- Bell, C. C., et al. Characterization of primary human hepatocyte spheroids as a model system for drug-induced liver injury, liver function and disease. Scientific Reports. 6, 25187 (2016).
- Tong, W. H., et al. Constrained spheroids for prolonged hepatocyte culture. Biomaterials. 80, 106-120 (2016).
- Griffith, L. G., Wells, A., Stolz, D. B. Engineering liver. Hepatology. 60 (4), 1426-1434 (2014).
- Domansky, K., et al. Perfused multiwell plate for 3D liver tissue engineering. Lab Chip. 10 (1), 51-58 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon