Method Article
Generazione di organoidi intestinali derivati da hiPSC per applicazioni di modellizzazione dello sviluppo e delle malattie
In questo articolo
Riepilogo
Questo protocollo consente la differenziazione delle cellule pluripotenti umane in organoidi intestinali. Il protocollo imita il normale sviluppo umano differenziando le cellule in una popolazione di endoderma definitivo, endoderma dell'intestino posteriore e quindi epitelio intestinale. Ciò rende il protocollo adatto sia per lo studio dello sviluppo intestinale che per le applicazioni di modellizzazione delle malattie.
Abstract
Gli organoidi intestinali derivati da hiPSC sono strutture epiteliali che si auto-assemblano da cellule differenziate in strutture 3D complesse, rappresentative dell'epitelio intestinale umano, in cui presentano strutture simili a cripte/villi. Qui, descriviamo la generazione di organoidi intestinali derivati da hiPSC mediante la differenziazione graduale di hiPSC nell'endoderma definitivo, che viene poi posteriorizzato per formare l'epitelio dell'intestino posteriore prima di essere trasferito in condizioni di coltura 3D. L'ambiente di coltura 3D è costituito da matrice extracellulare (ECM) (ad esempio, Matrigel o altra ECM compatibile) integrata con SB202190, A83-01, Gastrin, Noggin, EGF, R-spondina-1 e CHIR99021. Gli organoidi subiscono un passaggio ogni 7 giorni, durante il quale vengono interrotti meccanicamente prima di essere trasferiti alla matrice extracellulare fresca e lasciati espandere. La QPCR e l'immunocitochimica confermano che gli organoidi intestinali derivati da hiPSC contengono tipi di cellule epiteliali intestinali mature, tra cui cellule caliciformi, cellule di Paneth ed enterociti. Inoltre, gli organoidi mostrano evidenza di polarizzazione per espressione di villina localizzata sulla superficie apicale delle cellule epiteliali.
Gli organoidi risultanti possono essere utilizzati per modellare lo sviluppo intestinale umano e numerose malattie intestinali umane, tra cui le malattie infiammatorie intestinali. Per modellare l'infiammazione intestinale, gli organoidi possono essere esposti a mediatori infiammatori come TNF-α, TGF-β e LPS batterico. Gli organoidi esposti a citochine proinfiammatorie mostrano un fenotipo infiammatorio e fibrotico in risposta. L'accoppiamento di cellule sane rispetto a hiPSC derivate da pazienti con IBD può essere utile per comprendere i meccanismi che guidano l'IBD. Ciò potrebbe rivelare nuovi bersagli terapeutici e nuovi biomarcatori per aiutare nella diagnosi precoce della malattia.
Introduzione
Le proprietà delle cellule staminali pluripotenti (PSC), come l'auto-rinnovamento e la capacità di differenziarsi con qualsiasi tipo di cellula del corpo umano, le rendono strumenti preziosi nello studio dello sviluppo, della patologia delle malattie e dei test farmacologici1. Le cellule staminali pluripotenti indotte umane (hiPSC) sono particolarmente utili per gli studi di modellizzazione della malattia in quanto possono essere derivate dai pazienti, catturando direttamente il genoma responsabile del fenotipo 2,3 della malattia. Tali hiPSC possono essere differenziate in base al tipo di cellula interessata dal difetto genetico, consentendo un attento esame del meccanismo molecolare della malattia4.
I protocolli di differenziamento per le PSC umane mirano a dirigere il differenziamento delle cellule attraverso le principali fasi di sviluppo attraverso l'attivazione o l'inibizione di specifiche vie di segnalazione che governano l'impegno e la specificazione del lignaggio. Il mantenimento di hiPSC in uno stato pluripotente richiede livelli moderati di segnalazione dell'Activina A (Act-A), mentre un alto dosaggio di Act-A per 3 giorni impegna l'hiPSC a un destino endodermico (DE)definitivo 5,6. Le vie Act-A e Wnt dirigono l'identità antero-posteriore del DE. La segnalazione da parte di Act-A induce marcatori dell'intestino anteriore (FG) come HHEX, HNF4α e GATA4, bloccando l'espressione dei geni dell'intestino posteriore (HG) come CDX2. La segnalazione Wnt induce la posteriorizzazione della DE, che poi adotta un profilo di espressione genica HG 7,8. Una volta stabilita l'identità delle cellule HG, la differenziazione può essere spostata da 2D a 3D e indirizzata verso la formazione di organoidi intestinali.
Gli organoidi intestinali sono tipicamente coltivati in un sistema di coltura basato su matrice extracellulare 3D (ad esempio, Matrigel o altri ECM compatibili)9, composto da laminina, collagene IV ed entactina e arricchito con fattori di crescita come EGF, FGF, PDGF e IGF-1 per contribuire alla sopravvivenza e alla proliferazione del supporto. Gli organoidi vengono coltivati in un terreno definito contenente Gastrina, Noggin e CHIR per stimolare e sostenere la crescita e la proliferazione delle cellule staminali intestinali durante la coltura a lungo termine.
Dopo che le cellule epiteliali intestinali sono state incorporate nella matrice extracellulare, le cripte intestinali iniziano a formarsi ed espandersi formando infine sferoidi. Questi maturano in strutture organoidi che imitano il funzionamento fisiologico dell'epitelio intestinale. Gli organoidi possono in genere essere coltivati per più di 1 anno senza una perdita significativa dei profili funzionali e di espressione genica. Il passaggio è richiesto su base settimanale utilizzando la digestione enzimatica degli organoidi in frammenti più piccoli che poi si auto-riassemblano in organoidi completi.
Le linee di organoidi consolidate possono essere utilizzate come modello affidabile di numerosi disturbi legati all'intestino, tra cui il morbo di Crohn, la colite ulcerosa e i tumori del colon-retto 10,11,12,13. Questo è un modello preferito alle cellule animali in quanto esprimono geni umani legati a questi disturbi e rispondono a stimoli esterni più simili a quelli che si verificano nei tessuti umani in vivo.
Protocollo
NOTA: Tutte le colture tissutali descritte di seguito devono essere eseguite in una cappa a flusso laminare di Classe II.
1. Differenziazione delle cellule staminali pluripotenti indotte umane (hiPSCs) nell'endoderma dell'intestino posteriore
- Per rivestire un pallone di coltura cellulare con matrice extracellulare, calcolare prima la quantità di matrice necessaria. Di seguito è riportato un esempio di piastra a 12 pozzetti rivestita con Matrigel. Calcolare la quantità necessaria di matrice extracellulare per rivestire una piastra a 12 pozzetti. Usa la seguente formula:
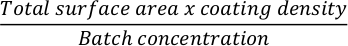
NOTA: Rivestire le piastre di coltura cellulare con matrice extracellulare a scelta. Se si utilizza Matrigel, utilizzare una concentrazione di 0,035 mg/cm2 24 h prima della semina per la differenziazione. Controllare la concentrazione in batch della matrice extracellulare sulla scheda del prodotto e preparare aliquote più piccole prima degli esperimenti secondo le istruzioni del produttore. - Diluire la quantità necessaria di matrice extracellulare in terreni DMEM freddi utilizzando una pipetta p1000.
- Mescolare bene con uno stripette da 5 ml.
- Aggiungere 500 μL di matrice diluita a ciascuna piastra a pozzetti. Agitare delicatamente la piastra per distribuire uniformemente la matrice extracellulare diluita e incubare per almeno 12 ore a 37 °C.
- Prima di seminare le cellule sulla piastra, lavare ogni pozzetto con 500 μL di PBS per rimuovere la matrice extracellulare in eccesso. Ciò impedirà il distacco cellulare durante la differenziazione.
- Lasciare l'ultimo lavaggio nei pozzetti fino al momento della semina per evitare l'essiccazione della matrice extracellulare.
- Per seminare la differenziazione delle hiPSC verso l'endoderma definitivo (DE) o l'intestino posteriore (HG), prelevare un matraccio di hiPSCs coltivate in un terreno di mantenimento a scelta (ad esempio: essential 8 media) e aspirare il terreno. (In questo caso, seminiamo cellule a 25.000 cellule/cm2).
NOTA: La densità di semina è fondamentale per il successo della differenziazione e deve essere ottimizzata per ogni singola linea cellulare hiPSC. - Lavare le cellule nel matraccio con 5 mL di PBS. Aspirare il PBS.
- Aggiungere 2,5 mL di soluzione di dissociazione cellulare (ad es. TrypLE) e lasciare a RT per 4 minuti. Aspirare la soluzione di dissociazione cellulare e picchiettare delicatamente il pallone per staccare le cellule.
- Lavare il matraccio con 5 mL di terreno DMEM riscaldato a 37 °C e raccogliere tutte le cellule in una provetta da 15 mL.
- Prelevare 10 μL di soluzione cellulare in sospensione e misurare la densità cellulare con un emocitometro.
- Prelevare una quantità sufficiente di sospensione cellulare per raccogliere 1,05 x 106 cellule e metterla in una provetta da 15 ml.
- Centrifugare a 160 x g per 3 min. Durante la centrifugazione delle cellule, aspirare PBS dalla piastra a 12 pozzetti rivestita di matrice extracellulare.
- Dopo la centrifugazione, aspirare il surnatante e risospendere il pellet cellulare in 12 mL di terreno di mantenimento con inibitore ROCK (10 μM).
- Utilizzando una pipetta p1000, aggiungere 1 mL di sospensione cellulare a ciascun pozzetto della piastra da 12 pozzetti. Risospendere bene le cellule per garantire una distribuzione uniforme tra i pozzetti.
- Agitare delicatamente la piastra per distribuire le cellule all'interno dei pozzetti della piastra, ma evitare di far roteare il terreno in quanto concentrerà le cellule al centro dei pozzetti.
- Porre in un'incubatrice a 37 °C, 5% di CO2 .
- Sostituire il terreno con un terreno di mantenimento (ad es. terreno essenziale 8) solo (senza inibitore ROCK) 24 ore dopo la semina.
- Per iniziare la differenziazione in DE, preparare i terreni basali dell'endoderma secondo la Tabella 1.
NOTA: All'inizio della differenziazione le cellule dovrebbero avere una confluenza compresa tra il 60 e l'80%. - Preparare 12 mL di terreno DE aggiungendo Activina A (100 ng/mL) e Wnt3 (50 ng/mL) al terreno basale dell'endoderma.
- Riscaldare il supporto a 37 °C.
- Aspirare i terreni da ciascun pozzetto della piastra o del pallone di coltura tissutale.
- Iniziare la differenziazione DE aggiungendo 1 mL di terreno DE a ciascun pozzetto della piastra a 12 pozzetti.
- A 24 ore dall'inizio della differenziazione DE (D1 DE), preparare nuovi terreni DE ed eseguire il cambio dei terreni.
- Ripetere il passaggio 1.24 a 48 h (D2 DE). Se in questa fase è presente molta morte cellulare, lavare tutti i pozzetti con 500 μL di PBS prima di cambiare il terreno.
NOTA: Per confermare il successo della differenziazione dell'endoderma, eseguire la citometria a flusso per valutare l'espressione di SOX17. Normalmente ci aspettiamo che il >80% delle cellule sia positivo a SOX17 entro D3 DE. Se l'espressione di SOX17 non è ottimale, la densità di semina cellulare deve essere ottimizzata, così come le concentrazioni di Act-A. - Iniziare la differenziazione HG a questo punto, cioè 72 ore dopo l'inizio della differenziazione DE. Preparare 12 mL di terreno HG aggiungendo CHIR99021 (3 μM) e RA (1 μM) ai terreni basali dell'endoderma (Tabella 1).
NOTA: A D3 DE la cella dovrebbe aver formato un monostrato uniforme. La differenziazione ottimale della DE è fondamentale per le successive fasi di differenziazione. - Aspirare i terreni DE.
- Iniziare la differenziazione HG aggiungendo 1 mL di terreno HG a ciascun pozzetto della piastra a 12 pozzetti.
- Continua la differenziazione per 4 giorni con cambi di media giornalieri.
NOTA: Per determinare il successo della posteriorizzazione della DE, eseguire la citometria a flusso per valutare l'espressione di CDX2. Con D4 del differenziamento HG normalmente ci aspettiamo che il >80% delle cellule sia CDX2 positivo. Se la posteriorizzazione non è ottimale, la concentrazione di CHIR99021 deve essere ottimizzata. - Per raccogliere un campione per l'estrazione dell'RNA, aspirare i terreni di differenziazione e lavare il pozzetto con 500 ul di PBS.
- Aspirare PBS e aggiungere un volume adeguato di tampone di lisi cellulare da un kit di estrazione dell'RNA.
- Utilizzando una pipetta p1000, raschiare il fondo del pozzetto per garantire la lisi di tutte le cellule.
- Aspirare il lisato cellulare e metterlo in una provetta pulita.
- Procedere all'estrazione dell'RNA o congelare il lisato a -20 °C fino al momento dell'estrazione dell'RNA.
- Per l'immunocolorazione, aspirare i terreni di differenziazione e lavare i pozzetti con 500 μL di PBS.
- Aspirare il PBS e aggiungere 500 μL di PFA al 4%.
NOTA: Il PFA è tossico. Utilizzare DPI appropriati e seguire le procedure di laboratorio locali per lo smaltimento del PFA. - Incubare a 4 °C per 20 min.
- Rimuovere il PFA e lavare i pozzetti con 500 μL di PBS tre volte.
- Lasciare l'ultimo lavaggio PBS sulle cellule fino al momento di eseguire l'immunocolorazione.
2. Passaggio degli organoidi intestinali
- Preparare i terreni basali intestinali secondo la Tabella 2.
- Per trasferire da una coltura cellulare 2D a una 3D, utilizzare una piastra a 6 pozzetti per generare cellule DE da hiPSCS. Staccare il monostrato di cellule dalla piastra a 6 celle utilizzando una striscia da 5 ml.
- Raccogliere le cellule in una provetta da centrifuga da 15 ml, prima di centrifugarle a 400 x g per 1 minuto per produrre un pellet cellulare.
- Risospendere il pellet cellulare in terreni di crescita intestinali contenenti fattori di crescita: SB202190 (10 μM), A83-01 (500 nM), Gastrina (10 nM), Noggin (100 ng/μL), EGF (500 ng/μL), R-Spondina1 (100 ng/mL), CHIR99021 (6 μM), inibitore ROCK (10 μM).
- Aggiungere un volume appropriato di matrice extracellulare in base al numero di pozzetti in cui viene placcato. Questo può essere calcolato utilizzando le equazioni seguenti.
[1] Volume totale della matrice/terreno extracellulare (μL) = 30 μL * numero di pozzetti della piastra a 48 pozzetti
[2] Volume richiesto di matrice extracellulare (μL) = Risposta da [1] * 2/3
[3] Volume richiesto di fluido (μL) = Risposta da [1] * 1/3 - Aggiungere 30 μL di sospensione cellulare al centro di ciascun pozzetto di una piastra a 48 pozzetti.
- Incubare la piastra in un incubatore a 37 °C per almeno 5 minuti per consentire alla matrice extracellulare di solidificarsi.
- Una volta che le cupole della matrice extracellulare si sono solidificate, aggiungere 300 μL di terreno di crescita intestinale contenente tutti i fattori di crescita in ciascun pozzetto.
- Riportare la piastra in un incubatore a 37 °C con il 5% di CO2 .
NOTA: Se dopo 48 ore non è possibile osservare organoidi e/o c'è una morte cellulare significativa, potrebbe essere necessario ottimizzare le concentrazioni di ROCKi e NOGGIN. - Per far passare gli organoidi, osservare le cellule al microscopio ottico per valutare la densità e le dimensioni degli organoidi e determinare se hanno bisogno di essere passati.
- Sostituire i terreni di coltura cellulare con PBS ghiacciato.
NOTA: Le colture di organoidi dovranno essere divise ogni 5-7 giorni circa. Il passaggio delle colture di organoidi intestinali è necessario quando c'è un accumulo di detriti cellulari evidente all'interno del lume dell'organoide e un ingiallimento del mezzo di crescita intestinale circostante. - Staccare meccanicamente gli organoidi e la sfera della matrice extracellulare dalla piastra utilizzando un movimento di raschiamento con una striscia da 5 ml.
- Raccogliere gli organoidi da ciascun pozzetto in una provetta da centrifuga da 15 ml.
- Centrifugare la sospensione di organoidi a 400 x g per 1 minuto per pellettare gli organoidi. Aspirare il surnatante fino a raggiungere la sommità del pellet cellulare, facendo attenzione quando si raggiunge lo strato visibile della matrice extracellulare.
- Risospendere il pellet in 15 mL di PBS ghiacciato.
NOTA: Questo passaggio serve a lavare la matrice extracellulare rimanente che non è stata rimossa nella centrifuga iniziale. - Centrifugare a 400 x g per 1 min. Aspirare il terreno fino al pellet, contenente gli organoidi.
- Risospendere in 1 ml di PBS ghiacciato. Utilizzando una pipetta p200, rompere manualmente gli organoidi intatti pipettando su e giù più volte.
NOTA: Osservare le dimensioni degli organoidi utilizzando il microscopio ottico per determinare se devono essere ulteriormente dissociati. - Aggiungere 9 mL di terreno senza fattori di crescita.
- Centrifugare a 400 x g per 1 min. Aspirare il surnatante fino a quando gli organoidi non si incastrano.
- Calcola la quantità richiesta di matrice e terreno extracellulare utilizzando le equazioni seguenti:
[1] Volume totale della matrice/terreno extracellulare (μL) = 30 μL * numero di pozzetti della piastra a 48 pozzetti
[2] Volume richiesto di matrice extracellulare (μL) = Risposta da [1] * 2/3
[3] Volume richiesto di fluido (μL) = Risposta da [1] * 1/3 - Risospendere il pellet di organoide nel volume calcolato di terreno intestinale con fattori di crescita (incluso l'inibitore di Noggin e ROCK).
- Aggiungere il volume richiesto di matrice extracellulare in questa sospensione cellulare e risospendere per garantire una distribuzione uniforme degli organoidi.
- Pipettare 30 μL di questa sospensione al centro di ciascun pozzetto di una piastra da 48 pozzetti (preferibilmente preriscaldata in un incubatore a 37 °C).
- Rimettere la piastra in un incubatore a 37 °C per 5 minuti fino a quando la matrice extracellulare non si è solidificata.
- Preparare terreni intestinali con fattori di crescita (+ inibitore ROCK) (circa 17 mL per piastra da 48 pozzetti).
- Aggiungere 300 μL a ciascun pozzetto.
- Incubare a 37 °C al 5% di CO2 .
- Dopo il passaggio, aspirare il terreno per gli organoidi intestinali e sostituirlo con terreno intestinale fresco con fattori di crescita (senza inibitore di Noggin e ROCK) ogni 2-4 giorni.
NOTA: Se dopo 48 ore non è possibile osservare organoidi e/o c'è una morte cellulare significativa, potrebbe essere necessario ottimizzare le concentrazioni di ROCKi e NOGGIN.
NOTA: Gli organoidi intestinali rispondono a una serie di mediatori dell'infiammazione in vivo. Il TNFα è una proteina di segnalazione cellulare coinvolta in diversi processi infiammatori. - Per innescare una risposta infiammatoria negli organoidi intestinali, preparare il TNFα a una concentrazione di 40 ng/mL solo nei terreni basali.
- Aspirare i terreni da una piastra a 48 pozzetti.
- Aggiungere 300 μL di terreno basale preparato contenente 40 ng/mL di TNFα.
- Incubare la piastra a 37 °C per 48 ore per replicare un ambiente pro-infiammatorio.
NOTA: Gli organoidi intestinali rispondono a una serie di mediatori dell'infiammazione in vivo. Il TNFα è una proteina di segnalazione cellulare coinvolta in diversi processi infiammatori. - Smaltire le cellule rimanenti mediante aspirazione in una trappola sottovuoto contenente il 5% di Trigene.
Risultati
Uno schema del protocollo di differenziazione è mostrato nella Figura 1A. Il giorno 1 del protocollo, le hiPSC dovrebbero essere compatte e formare piccole colonie con una confluenza totale di circa il 50-60%. 24 ore dopo l'induzione del differenziamento verso le cellule DE iniziano a migrare lontano dalle colonie di cellule staminali per formare un monostrato di cellule. Questo continua nei 3 giorni successivi e dovrebbe formare un monostrato completo su D3 di differenziazione DE (Figura 1). L'espressione genica deve essere monitorata nel corso del differenziamento con marcatori di pluripotenza (OCT4, NANOG, SOX2) altamente espressi su D0 e rapidamente sottoregolati durante il differenziamento di DE. Durante il differenziamento DE l'espressione di T dovrebbe raggiungere il picco su D1, seguita da EOMES e MIXL su D2. Su D2 i geni DE (SOX17, FOXA2, GATA4, CXCR4) dovrebbero iniziare ad essere espressi e raggiungere il picco in D3 (Figura 2 e Figura 3). Le cellule dovrebbero essere un monostrato di DE D3 e possono quindi essere posteriorizzate, nell'endoderma dell'intestino posteriore. Durante l'evento di posteriorizzazione le strutture 3D inizieranno a formarsi già a partire da D2. Tuttavia, a volte inizieranno ad apparire solo in D4 o non appariranno affatto; questo non è sempre indicativo del fatto che le cellule procedano o meno e formino organoidi intestinali (Figura 1). Durante la specifica HG, l'espressione di CDX2 e HNF4a dovrebbe essere indotta e aumentare nel tempo (Figura 3).
Dopo il trasferimento di fogli di cellule 2D nella matrice extracellulare, si osserveranno grumi di cellule 2D per le prime 24 ore. Dopo 48 ore, i fogli di cellule dovrebbero iniziare ad auto-organizzarsi in strutture sferoidi 3D più compatte che sono inizialmente piccole (Figura 4A), quindi aumentano gradualmente di dimensioni e complessità nel corso di 7-10 giorni di coltura (Figura 4B e Figura 4C). Gli organoidi non devono essere fatti passare fino a quando non hanno raggiunto una morfologia organoide/sferoide chiara con epitelio evidente con il lume rivolto verso il centro dell'organoide/sferoide (Figura 4D). In questa fase, l'immunocitochimica può essere utilizzata per confermare l'espressione di marcatori intestinali come la villina e CDX2 (Figura 5). Non tutti i grumi di cellule 2D si svilupperanno in organoidi e ci saranno alcune cellule morte contaminanti all'interno della matrice extracellulare. Questi fogli morti di cellule dovrebbero essere ignorati fino a quando le cellule sopravvissute non hanno formato grandi organoidi e sono pronte per il passaggio.
Per modellare l'infiammazione, il TNFα può essere aggiunto al terreno di coltura tissutale per 24-48 ore. Dopo l'incubazione con molecole proinfiammatorie, gli organoidi vengono raccolti utilizzando la stessa tecnica utilizzata per il loro isolamento e passaggio e quindi lisati utilizzando un'applicazione compatibile con il tampone cellulare come la QPCR o il western blotting. Se sono necessarie esposizioni più brevi, gli organoidi devono prima essere rimossi dalla matrice extracellulare ed esposti al TNFα in sospensione utilizzando una provetta da 1,5 mL. Il trattamento degli organoidi intestinali con TNFα per 48 ore induce tipicamente l'espressione di marcatori pro-infiammatori (TNFα, IL1B, IL8, IL23) mentre influenza negativamente l'espressione dei marcatori epiteliali intestinali (LGR5, VIL) (Figura 6).
| Mezzi basali dell'endoderma | 50 ml | |
| RPMI 1640 | 48,5 mL | |
| Supplemento B27 | 1 mL | |
| 1% NEAA | 0,5 mL | |
Tabella 1: Composizione dei mezzi basali endodermici per la differenziazione dell'endoderma.
| Mezzi basali intestinali | 50 ml | |
| DMEM/F12 avanzato | 46,5 mL | |
| Tampone HEPES | 0,5 mL | |
| GlutaMAX | 0,5 mL | |
| Nicotinammide | 0,5 mL | |
| Supplemento N2 | 0,5 mL | |
| Supplemento B27 | 1,0 mL | |
| Penna/Streptococco | 0,5 mL | |
Tabella 2: Composizione dei terreni basali intestinali per la coltura di organoidi intestinali
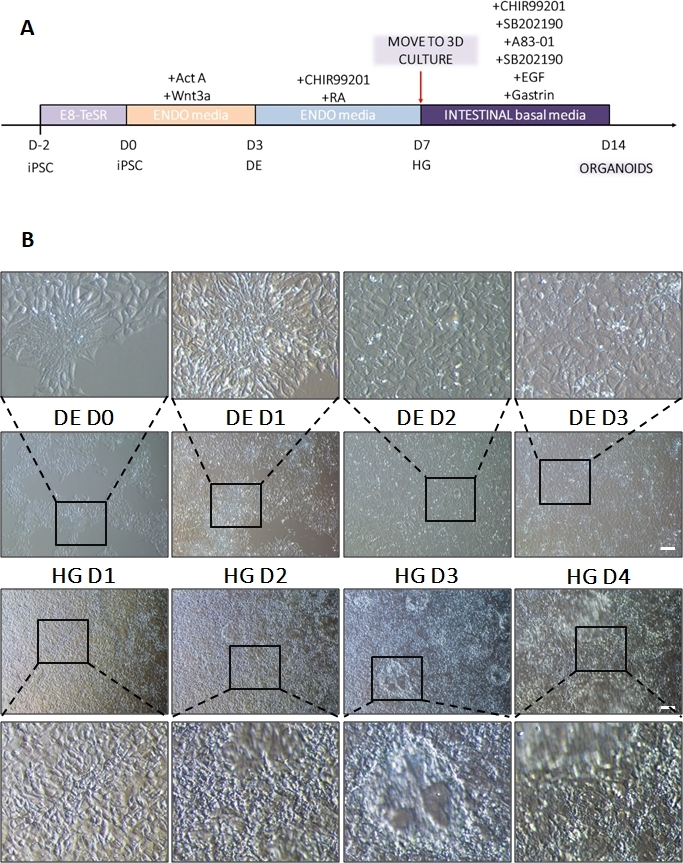
Figura 1: Cambiamenti morfologici durante la differenziazione di hiPSC attraverso l'endoderma definitivo alla linea dell'intestino posteriore.
(A) Panoramica schematica del protocollo di differenziamento intestinale. Questa linea cellulare di hiPSC forma colonie sciolte di piccole cellule con un elevato rapporto nucleo/citoplasma. Man mano che il differenziamento procede, le cellule subiscono cambiamenti coerenti con la transizione dal fenotipo epiteliale a quello mesenchimale e per DE D3 formano un monostrato uniforme. Una volta che i segnali appropriati vengono forniti, le cellule DE si allungano e formano un monostrato più denso con sferoidi 3D che appaiono non appena HD D3, ma questo dipende dalla linea cellulare utilizzata e non è un requisito per la transizione alla coltura 3D (B). Barra graduata: 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
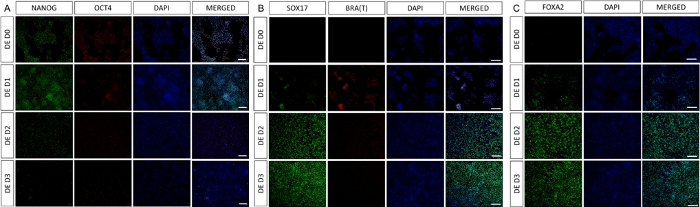
Figura 2: La differenziazione delle hiPSC nell'endoderma HG induce l'espressione dei geni endodermici.
L'immunocolorazione di hiPSC che si differenzia nell'endoderma definitivo mostra cambiamenti nell'espressione di TF a livello proteico. I marcatori di pluripotenza (NANOG e OCT4) sono sottoregolati da DE D3 (A). L'espressione del marcatore mesendodermico BRA(T) è presente in D1 del protocollo (B) e i TF specifici per DE SOX17 e FOXA2 compaiono in D2 (B e C). Barra graduata: 200 mm. Fare clic qui per visualizzare una versione più grande di questa figura.
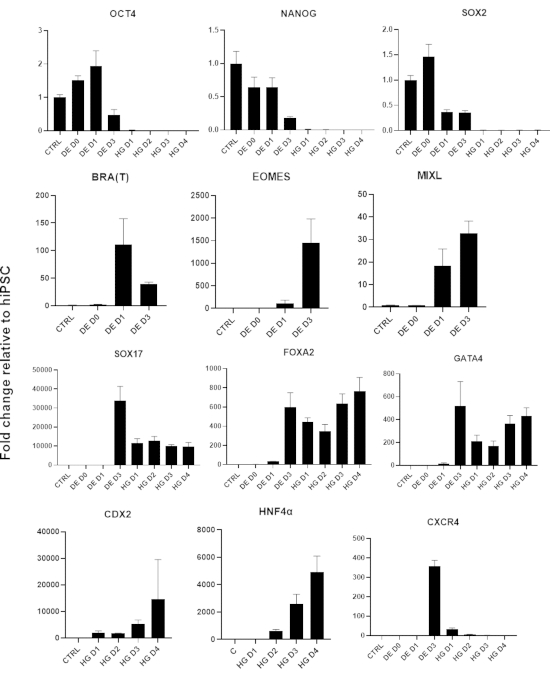
Figura 3: Cambiamenti dell'espressione genica mediante qPCR durante la differenziazione di hiPSC nell'endoderma dell'intestino posteriore (HG).
I geni associati alla pluripotenza sono sottoregolati (OCT4, NANOG, SOX2) seguiti dall'espressione transitoria dei geni del mesendoderma (T, EOMES, MIXL1) e infine dall'espressione dei geni DE (SOX17, FOXA2, CXCR4) e dell'intestino posteriore (CDX2, GATA4, HNF4a). Dati presentati come media ±SD. Fare clic qui per visualizzare una versione più grande di questa figura.
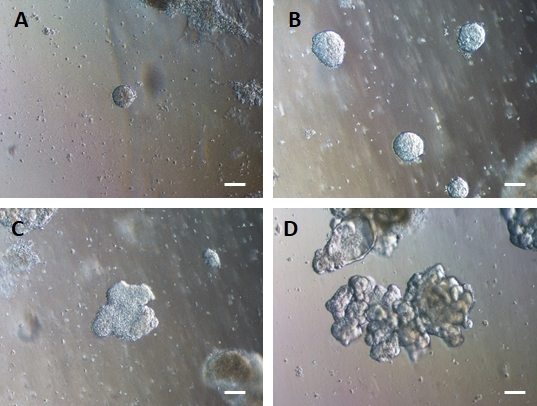
Figura 4: L'endoderma HG si auto-assembla per formare organoidi intestinali 3D in una coltura di matrice extracellulare 3D.
L'endoderma HG viene trasferito in un'opportuna coltura di matrice extracellulare 3D e forma inizialmente piccoli grumi solidi di cellule (A). I grumi di endoderma HG si espandono in 7-10 giorni di coltura (B) e poi diventano asimmetrici e iniziano a formare un epitelio più complesso (C) dando infine origine a organoidi con una chiara morfologia epiteliale e una superficie luminale rivolta verso il centro dell'organoide (D). Barre graduate = 50 μm Fare clic qui per visualizzare una versione più grande di questa figura.
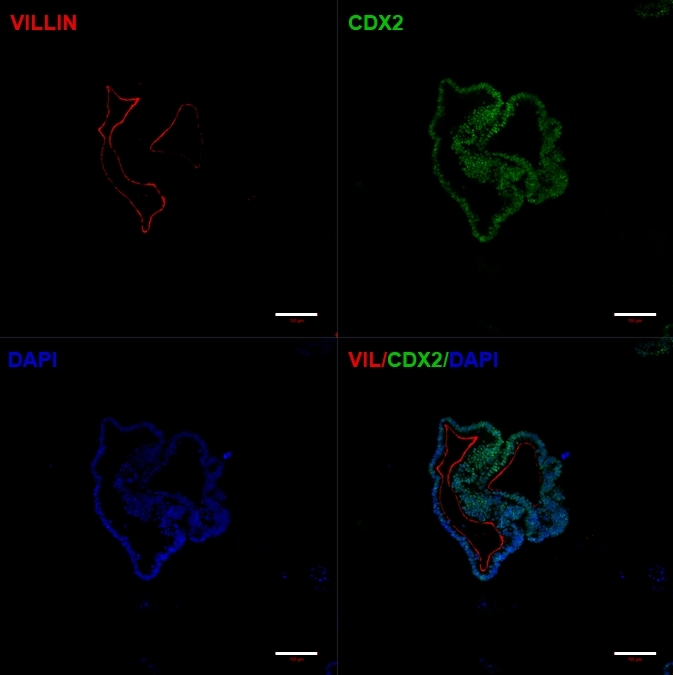
Figura 5: Gli organoidi intestinali derivati da hiPSC esprimono marcatori intestinali.
Immunocitochimica che mostra l'espressione di CDX2 e Villina. Barre di scala = 100 μm Fare clic qui per visualizzare una versione più grande di questa figura.
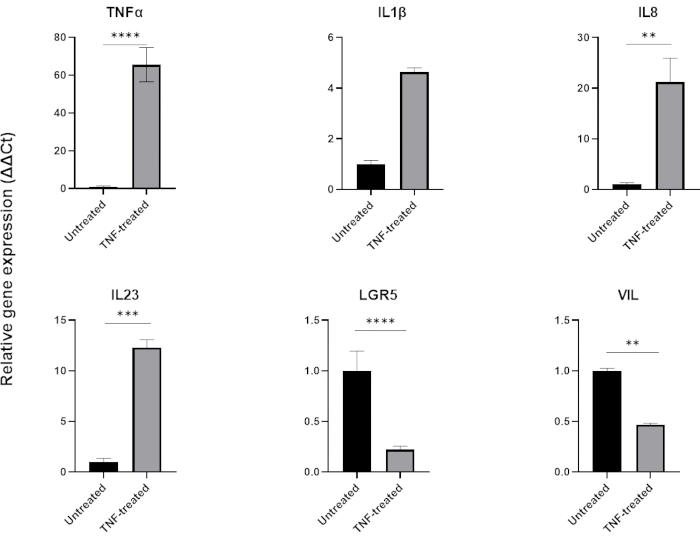
Figura 6: Effetto del TNFα sul profilo infiammatorio e sull'espressione delle cellule intestinali di organoidi intestinali sani.
Profilo infiammatorio di organoidi sani del colon dopo 48 ore di trattamento con TNFα (40 ng/mL). L'espressione di marcatori pro-infiammatori (TNFα, IL-8 e IL-23) aumenta in seguito all'esposizione a TNFα, mentre allo stesso tempo l'espressione di marcatori epiteliali intestinali (LGR5, VIL) è sottoregolata. Le analisi statistiche sono state eseguite mediante test t di studente su due lati. I dati sono espressi come media ± SD di ciascun gruppo. *P < 0,01; **P < 0,001; P < .0001. (n=3). Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Qui descriviamo un protocollo per la differenziazione di cellule pluripotenti umane in organoidi intestinali umani. Dimostriamo il loro utilizzo per studiare l'infiammazione; tuttavia, questo può essere applicato a vari contesti, e accoppiato con approcci di editing genetico CRISPR/Cas914, su qualsiasi background genetico. Una volta differenziati, seguendo la naturale sequenza di differenziazione evolutiva dell'endoderma definitivo, dell'endoderma dell'intestino posteriore e quindi dell'epitelio intestinale, gli organoidi risultanti possono essere continuamente coltivati e fatti passare per più di 12 mesi.
Un aspetto critico di questo protocollo è la densità iniziale di placcatura delle cellule staminali indifferenziate prima della differenziazione dell'endoderma. Se questo non è sufficientemente ottimizzato, è probabile che le cellule muoiano durante la fase iniziale di differenziazione DE (se le cellule sono troppo scarse) o riducano l'efficienza della differenziazione DE (se le cellule sono troppo dense). La corretta densità iniziale deve essere ottimizzata per la linea cellulare utilizzata e la densità corretta deve generare un monostrato entro la fine di DE D3. La citometria a flusso dovrebbe essere utilizzata per determinare l'efficienza della specifica DE e in genere vediamo il >80% delle cellule positive per SOX17 e/o CXCR4. Quando il numero di cellule SOX17 positive è inferiore al 60%, l'efficienza del patterning HG è influenzata, il che si traduce nella formazione di un minor numero di organoidi quando viene trasferito alla matrice extracellulare. Ciò alla fine causerà il fallimento delle colture di organoidi risultanti. Per determinare se il patterning da DE a HG ha avuto successo, valutiamo il numero di cellule CDX2 positive mediante citometria a flusso e in genere ci aspettiamo di vedere il >80% di cellule positive. Anche in questo caso, se il numero di cellule CDX2 positive scende al di sotto del 50%, ciò avrà un effetto negativo sul numero di organoidi intestinali che vengono generati quando vengono trasferiti alla coltura di matrice extracellulare 3D.
Dopo il trasferimento di monostrati 2D in coltura 3D, 24-48 ore dopo il trasferimento dovrebbero apparire piccole sfere compatte. Possono comparire grandi fogli di cellule morte a seconda dell'efficienza di differenziazione per la linea cellulare utilizzata. Invece di passare immediatamente le colture per rimuovere questi detriti, permettiamo agli organoidi di formarsi completamente e sviluppare la loro struttura più complessa e piegata. Aspettare 7-10 giorni prima di tentare il primo passaggio assicura che siano presenti cellule in divisione sufficienti per generare molti nuovi organoidi intestinali. Eventuali detriti ancora presenti nella coltura possono essere facilmente rimossi durante il processo di passaggio facendo ruotare lentamente le miscele di organoidi/detriti dissociati in una provetta con una velocità sufficiente per pellettare gli organoidi ma lasciare fogli di cellule che galleggiano nel terreno. I terreni e i detriti cellulari possono quindi essere aspirati in modo che rimanga solo il pellet degli organoidi.
Il limite di questo approccio è che i tipi di cellule derivate da hiPSC spesso non sono completamente maturi in termini di espressione genica e profili funzionali. Per determinare se il tessuto intestinale derivato da hiPSC è adatto per applicazioni specifiche, gli organoidi devono essere caratterizzati per diversi tipi di cellule, tra cui enterociti (VIL), cellule enteroendocrine (neurog3), cellule caliciformi (MUC2), cellule di amplificazione transitoria (CD133), cellule di paneth (FZD5) e cellule staminali LGR5+ (LGR5) per determinare la composizione cellulare dell'organoide.
Nel complesso, il principale vantaggio di questo protocollo rispetto a molti altri protocolli di differenziazione degli organoidi è che questa piattaforma di coltura è molto conveniente grazie alla sostituzione di diverse proteine ricombinanti e preparazioni di terreni condizionati con piccole molecole 15,16. Il differenziamento in HG è molto semplice e veloce e può essere applicato sia a cellule staminali embrionali umane che a cellule staminali pluripotenti indotte con risultati identici. Se seguito rigorosamente e ottimizzato per le linee cellulari utilizzate, fornisce una piattaforma modello relativamente semplice, priva di cellule mesenchimali contaminanti, che può quindi essere applicata per studiare l'epitelio intestinale in una varietà di contesti, tra cui l'infiammazione e le interazioni ospite-patogeno7. La modellazione della fibrosi intestinale potrebbe essere studiata fornendo stimoli pro-fibrotici e quindi valutando l'espressione di proteine della matrice extracellulare come collagene, laminina e fibronectina mediante QPCR, western blot ed ELISA. L'utilizzo di tecniche di editing genetico CRISPR/Cas9 su linee di cellule staminali indifferenziate prima del differenziamento consente la creazione di organoidi di knockout genico o di sovraespressione proteica che potrebbero essere utilizzati per creare organoidi specifici per la malattia e modelli di malattia più complessi 14,17,18.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
NH è finanziato dall'MRC (MR/S009930/1) e dal Wellcome Trust (204267/Z/16/Z), PD è finanziato dall'MRC PhD DTP, KLF è finanziato dal BBSRC iCASE.
Materiali
| Name | Company | Catalog Number | Comments |
| A83-01 | Tocris | 2939 | |
| Activin A | R&D | 338-AC | |
| Advanced DMEM/F12 (1X) | Life Technologies | 12654-010 | |
| B27 supplement | Gibco | 17504044 | |
| CHIR99021 | Sigma | SML1046-5MG | |
| Epidermal Growth Factor | R&D Systems | 236-EG-01M | |
| Gastrin | Sigma Aldrich | G9145 | |
| GlutaMAX (100X) | Life Technologies | 15630-056 | |
| Growth Factor reduced Matrigel | BD | ||
| HEPES Buffer solution (1M) | Life Technologies | 15630-080 | |
| N2 Supplement (100X) | Gibco | 17502-048 | |
| N-acetyl-cysteine | Sigma Aldrich | A7250 | |
| Nicotinamide | Sigma Aldrich | N0636 | |
| Noggin | R&D Systems | 6057-NG | |
| Non-essential amino acids | Gibco | 11140-050 | |
| Paraformaldehyde | VWR | 9713.5 | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| Phosphate Buffered Saline | Gibco | 14190-094 | |
| Retinoic Acid | Sigma | 302-79-4 | |
| ROCK inhibitor | Tocris | 1254/1 | |
| ROCK inhibitor Y-27632 | Tocris | 1254 | |
| RPMI | Sigma | R8758-500ml | |
| R-Spondin-1 | Peprotech | 120-38 | |
| SB202190 | Tocris | 1264 | |
| TrypLe Express | Gibco | 12604-021 | |
| Wnt 3a | R&D | 5036-WN |
Riferimenti
- Thomson, J. A., et al. Embryonic stem cell lines derived from human blastocysts. Science. 282, 1145-1147 (1998).
- Takahashi, K., Yamanaka, S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell. 126, 663-676 (2006).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131, 861-872 (2007).
- Rashid, S. T., et al. Modeling inherited metabolic disorders of the liver using human induced pluripotent stem cells. Journal of Clinical Investigation. 120, 3127-3136 (2010).
- Vallier, L., Alexander, M., Pedersen, R. A. Activin/Nodal and FGF pathways cooperate to maintain pluripotency of human embryonic stem cells. Journal of Cell Science. 118, 4495-4509 (2005).
- Jaremko, K. L., Marikawa, Y. Regulation of developmental competence and commitment towards the definitive endoderm lineage in human embryonic stem cells. Stem Cell Research. 10, 489-502 (2013).
- Forbester, J. L., et al. Interaction of Salmonella enterica Serovar Typhimurium with Intestinal Organoids Derived from Human Induced Pluripotent Stem Cells. Infection and Immunity. 83, 2926-2934 (2015).
- Hannan, N. R., et al. Generation of multipotent foregut stem cells from human pluripotent stem cells. Stem Cell Reports. 1, 293-306 (2013).
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459, 262-265 (2009).
- de Souza, H. S., Fiocchi, C. Immunopathogenesis of IBD: current state of the art. Nature Reviews Gastroenterology & Hepatology. 13, 13-27 (2016).
- Tripathi, K., Feuerstein, J. D. New developments in ulcerative colitis: latest evidence on management, treatment, and maintenance. Drugs in Context. 8, 212572 (2019).
- Ganesh, K., et al. A rectal cancer organoid platform to study individual responses to chemoradiation. Nature Medicine. 25, 1607-1614 (2019).
- Fair, K. L., Colquhoun, J., Hannan, N. R. F. Intestinal organoids for modelling intestinal development and disease. Philosophical Transactions of the Royal Society B: Biological Sciences. 373, (2018).
- Cuevas-Ocaña, S., Yang, J. Y., Aushev, M., Schlossmacher, G., Bear, C. E., Hannan, N. R. F., Perkins, N. D., Rossant, J., Wong, A. P., Gray, M. A. A Cell- Based Optimised Approach for Rapid and Efficient Gene Editing of Human Pluripotent Stem Cells. Int. J. Mol. Sci. 24, 10266 (2023).
- Spence, J. R., et al. Directed differentiation of human pluripotent stem cells into intestinal tissue in vitro. Nature. 470, 105-109 (2011).
- Sato, T., et al. Long-term Expansion of Epithelial Organoids From Human Colon, Adenoma, Adenocarcinoma, and Barrett's Epithelium. Gastroenterology. 141, 1762-1772 (2011).
- Giacalone, J. C., et al. CRISPR-Cas9-Based Genome Editing of Human Induced Pluripotent Stem Cells. Current Protocols in Stem Cell Biology. 44, 1-22 (2018).
- Bruntraeger, M., Byrne, M., Long, K., Bassett, A. R., Luo, Y. . CRISPR Gene Editing: Methods and Protocols. , 153-183 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon