È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Coltura di periciti capillari cerebrali per misurazioni citosoliche del calcio e studi di imaging del calcio
In questo articolo
Riepilogo
I periciti capillari cerebrali sono attori essenziali nella regolazione delle proprietà della barriera ematica-encefalica e del flusso sanguigno. Questo protocollo descrive come i periciti capillari cerebrali possono essere isolati, coltivati, caratterizzati rispetto al tipo di cellula e applicati per le indagini sulla segnalazione intracellulare del calcio con sonde fluorescenti.
Abstract
I periciti sono associati alle cellule endoteliali e ai piedi finali astrocitici in una struttura nota come unità neurovascolare (NVU). La funzione del pericita capillare cerebrale non è completamente nota. I periciti sono stati suggeriti per essere coinvolti nello sviluppo capillare, nella regolazione della tenuta barriera endoteliale e nell'attività della trancytosis, nella regolazione del tono capillare e nel svolgere ruoli cruciali in alcune patologie cerebrali.
I periciti sono difficili da indagare nel cervello intatto a causa delle difficoltà nel visualizzare i processi nel parenchima cerebrale, così come la vicinanza alle altre cellule dell'NVU. Il presente protocollo descrive un metodo per l'isolamento e la coltura dei periciti capillari del cervello bovino primario e il loro successivo utilizzo negli studi di imaging del calcio, in cui possono essere studiati gli effetti degli agonisti coinvolti nella segnalazione cerebrale e nelle patologie. I frammenti capillari corticali sono autorizzati ad attaccarsi al fondo dei contenitori di coltura e, dopo 6 giorni, cellule endoteliali e periciti sono cresciuti dai frammenti capillari. Le cellule endoteliali vengono rimosse mediante tripsinazione delicata e i periciti vengono coltivati per altri 5 giorni prima della passaging.
I periciti isolati vengono seminati in piastre di coltura da 96 porcili e caricati con il colorante indicatore del calcio (Fura-2 acetoxymethyl (AM)) per consentire misurazioni dei livelli di calcio intracellulare in una configurazione del lettore di lastre. In alternativa, i periciti vengono seminati su coverlips e montati in camere cellulari. Dopo il caricamento con l'indicatore del calcio (Cal-520 AM), l'imaging vivo di calcio può essere eseguito utilizzando la microscopia confocale a una lunghezza d'onda di eccitazione di 488 nm e una lunghezza d'onda di emissione di 510-520 nm.
Il metodo qui descritto è stato utilizzato per ottenere le prime misurazioni intracellulari del calcio dai periciti capillari cerebrali primari, dimostrando che i periciti sono stimolati tramite ATP e sono in grado di contrarsi in vitro.
Introduzione
I periciti capillari cerebrali, insieme alle cellule endoteliali e agli astrociti, costituiscono l'NVU1,2,3. Le cellule endoteliali, che formano la base strutturale dei capillari, formano lunghi tubi cilindrici con un diametro di 5-8 μm. Le cellule endoteliali sono sporadicamente coperte di periciti e circondate da sporgenze da astrociti; l'endfeet astrocita.
La barriera ematico-encefalica (BBB), situata ai capillari cerebrali, è il sito principale per lo scambio di nutrienti, gas e prodotti di scarto tra il cervello e il sangue. La BBB protegge anche il cervello dalle neurotossine endogene ed esogene e funge da barriera per la consegna di un gran numero di composti farmacologici. La funzione barriera è un'area di interesse, oltre che un ostacolo, per le aziende farmaceutiche che sviluppano farmaci del sistema nervoso centrale (SNC). Ciò ha suscitato un grande interesse nello studio delle cellule dell'NVU nella coltura4. Gli astrociti cerebrali e le cellule endoteliali sono stati coltivati e caratterizzati in una serie di studi, mentre gli studi e i protocolli per la coltura dei periciti sono scarsi.
Protocolli pubblicati in precedenza hanno descritto in una certa misura la generazione di colture di periciti capillari cerebrali, utilizzando una serie di approcci diversi come l'immunopanning5,i mezzi ad alto e basso glucosio6,lo smistamento cellulare attivato fluorescente7,la centrifugazione del gradiente didensità 8,ecc. Sebbene questi metodi sembrino sufficienti per ottenere colture di periciti, alcuni richiedono molto tempo, costano caro e i periciti ottenuti potrebbero non essere ideali a causa del numero di passaggi di coltura che possono dis differenziare i periciti9. Inoltre, il potenziale dei periciti coltivati negli studi di segnalazione in vitro è stato finora abbastanza inesplorato.
Il presente lavoro si concentra sulla generazione di colture di periciti da capillari cerebrali bovini isolati e sulla successiva configurazione per misurazioni e studi di imaging dei cambiamenti nel calcio intracellulare, un importante secondo messaggero intracellulare. Descriviamo brevemente l'isolamento dei capillari dalla materia grigia corticale (per i dettagli vedi Helms et al.10) e l'isolamento e la coltura dei periciti in pura monocoltura senza contaminazione con cellule endoteliali o gliali. Forniamo quindi un protocollo per la semina di periciti in piastre da 96 porcili e protocolli di carico per la sonda di calcio Fura-2 AM. Infine, mostriamo come i periciti possono essere utilizzati nell'imaging confocale in tempo reale nelle camere di coltura del microscopio e descriviamo i protocolli per questo.
Protocollo
1. Preparazione di tamponi e soluzioni per la coltivazione cellulare
- Preparare la soluzione di stock di collagene sciogliendo 5 mg di collagene IV dalla placenta umana in 50 mL di PBS durante la notte a 4 °C. Aliquota la soluzione stock in porzioni da 5 mL e conservare a -20 °C.
- Preparare la soluzione di stock di fibronectina sciogliendo 5 mg di fibronectina in 5 mL di acqua sterile durante la notte. Conservare le scorte di fibronectina in aliquote di 500 μL a -20 °C. Durante lo scongelamento, aggiungere PBS a un volume finale di 50 mL per preparare la soluzione di lavoro e conservarla a 4 °C.
- Preparare il mezzo completo di Dulbecco Modified Eagle Medium (DMEM) aggiungendo 50 mL di siero bovino fetale (FBS), 5 mL di amminoacidi nonessenziali MEM e 5 mL di penici la streptomicina (0,1 g/L di streptomicina solfato e 100.000 U/L penicillina G di sodio) a 500 mL di DMEM.
- Preparare 5 mg/mL di soluzione di stock di eparina sciogliendo il sale di sodio eparina in PBS e passarlo attraverso un filtro da 0,2 μm per la sterilizzazione. Conservare la soluzione stock a 4 °C.
- Preparare il mezzo di crescita (GM) immediatamente prima dell'uso; mescolare 10 ml di DMEM-comp e 250 μL di soluzione di soluzione di eparina per pallone T75.
2. Isolamento dei capillari dal cervello fresco dei bovini
NOTA: I capillari cerebrali bovini sono isolati e coltivati come descritto in precedenza (Helms et al.10).
- Raccogli cervelli dai vitelli, di età non superiore ai 12 mesi, da un mattatoio e porta direttamente in laboratorio sul ghiaccio.
- Rimuovere le meningi e raccogliere tutta la materia grigia dal cervello usando un bisturi. Identificare le meningi come il film che copre il cervello e la materia grigia dal suo colore grigio.
- Utilizzare una smerigliatrice di tessuto Dounce da 40 mL per omogeneizzare la materia grigia nel Mezzo aquila modificato (DMEM) di Dulbecco. Riempire la parte sottile della smerigliatrice di tessuto 1/5 con sospensione di materia grigia e aggiungere DMEM fino a riempire la parte sottile.
- Separare i capillari dalle cellule libere e dai pezzi di tessuto più piccoli filtrando l'omogeneato attraverso un filtro a rete di nylon da 160 μm. Svuotare i filtri con DMEM-comp. Recuperare i capillari e raggruppare le sospensioni in tubi di centrifugazione da 50 ml.
- Rimescolare i capillari in DMEM-comp e aggiungere una miscela enzimatica di DNasi I (170 U/mL), collagenasi di tipo III (200 U/mL) e tripside (90 U/mL). Lasciare la sospensione per 1 h in un bagno d'acqua a 37 °C per la digestione dei capillari.
- Eseguire la sospensione attraverso un filtro a rete da 200 μm e rimorsi in FBS con solfossido di dimetile al 10% (DMSO). Congelare i capillari durante la notte a -80 °C e spostarli in azoto liquido il giorno dopo per lo stoccaggio a lungo termine.
NOTA: Il protocollo può essere messo in pausa qui.
3. Semina e coltivazione di capillari bovini
- Giorno 0: Mescolare 0,7 mL di collagene IV stock con 6,3 mL di PBS. Aggiungere la soluzione a un pallone T75 e lasciare il pallone per 2 ore a temperatura ambiente (RT) o lasciarlo durante la notte a 4 °C.
- Rimuovere la soluzione di collagene dal pallone e lavare tre volte con PBS.
- Aggiungere 7 ml di soluzione di lavoro con fibronectina e lasciare il pallone per 30 minuti a RT. Quindi, rimuovere la soluzione di fibronectina e seminare i capillari immediatamente dopo.
- Durante i 30 minuti di attesa, scongelare una fiala di capillari in un bagno d'acqua a 37 °C.
- Quando i capillari vengono scongelati, trasferire immediatamente in un tubo di centrifugazione con 30 ml di DMEM-comp e centrifugare per 5 min a 500 x g e RT. Rimuovere DMEM-comp dal tubo e sospendere di nuovo il pellet capillare in 10 ml di DMEM-comp fresco.
- Trasferire la sospensione da 10 mL nel pallone T75 rivestito e lasciare che i capillari aderiscano al fondo del pallone per 4-6 ore in un incubatore a 37 °C al 10% di CO2.
NOTA: Il tasso di crescita cellulare è più alto al 10% di CO2 piuttosto che al convenzionale 5% di CO2. - Dopo 4-6 ore di incubazione ispezionare il pallone al microscopio leggero. Le frazioni di capillari dovrebbero ora essere attaccate al fondo del pallone(Figura 1, giorno 0).
- Preparare GM e aspirare il mezzo DMEN-comp molto attento dai capillari e sostituirlo con 10 mL di GM appena fatto.
- Giorno 2: Rimuovere GM dai capillari e sostituirlo con 10 mL di GM appena fatto.
4. Isolamento dei periciti primari dai capillari cerebrali bovini
-
Giorno 4: Ispezionare i capillari al microscopio leggero.
NOTA: Il pallone dovrebbe ora essere circa il 60-70% confluente per fornire una quantità appropriata di periciti(Figura 1, giorno 4). In caso contrario, non è così; sostituire il GM con 10 ml di mezzo fresco e lasciare il pallone nell'incubatrice per un altro giorno. - Aspirare il mezzo e lavare delicatamente le cellule in PBS.
- Aggiungere 2 ml di tripsiderina-EDTA scongelata per le cellule endoteliali e lasciare il pallone nell'incubatrice per 1-3 minuti. Esenzi frequentemente il pallone e osserva con il microscopio durante questo periodo di tempo.
NOTA: Le cellule endoteliali devono arrotondare e staccarsi dal pallone; i periciti dovrebbero essere visibili come cellule con una morfologia "fantasma" ed essere ancora attaccati alla superficie del pallone. Si tratta di un passo difficile e importante. È essenziale rimuovere la maggior parte delle cellule endoteliali per evitare la contaminazione della monocoltura dei periciti, ma la tripsinazione prolungata può anche staccare i periciti. Il tempo di tripsinzizzazione può variare leggermente di volta in volta, ed è quindi della massima importanza osservare frequentemente il pallone con il microscopio durante il trattamento. - Toccare delicatamente il pallone, quando le cellule endoteliali hanno iniziato a arrotondare, per staccare le cellule endoteliali allentate.
- Per interrompere la tripinazione, aggiungere 10 ml di DMEM-comp al pallone. Lavare il pallone con cura alcune volte con il mezzo per rimuovere le cellule endoteliali. Aspirare la sospensione cellulare endoteliale dal pallone. Le cellule endoteliali possono ora essere utilizzate per altri scopi.
- Aggiungere 10 mL di DMEM-comp al pallone. Guarda al microscopio luminoso per assicurarti che i periciti siano ancora presenti e attaccati al fondo. Rimettere il pallone nell'incubatrice per consentire alla cultura arricchita di periciti di crescere.
NOTA: È importante osservare la cultura nei giorni successivi. Se c'è ancora una discreta quantità di cellule endoteliali che crescono un altro trattamento con tripina può essere eseguita. - Consentire alla monocoltura pericita di crescere con il cambiamento di DMEM-comp. mezzo ogni secondo giorno. Controllare la crescita delle cellule al microscopio luminoso(Figura 1, giorno 5-8).
5. Generazione e conservazione di una monocoltura di periciti bovini primari
-
Giorno 8-9: Ispezionare i capillari al microscopio luminoso
NOTA: I periciti avrebbero dovuto raggiungere la confluenza del 70-80% e crescere nelle isole del pallone(figura 1,giorno 9). Se la confluenza dei periciti è inferiore al 70%, consentire alle cellule di crescere per un altro giorno. I periciti non formeranno un monostrato completo come lo sarebbero le cellule endoteliali. - Aspirare DMEM-comp e lavare i periciti con 7 mL di PBS.
- Aggiungere 2 ml di tripside-EDTA al pallone e lasciarlo nell'incubatrice per 2-3 minuti. Posizionare frequentemente il pallone al microscopio luminoso per osservare quando i periciti si arrotondano e si staccano dal pallone. Quando i periciti hanno iniziato a arrotondare, il pallone può essere delicatamente toccato per staccare le cellule.
- Toccare delicatamente il pallone, quando i periciti hanno iniziato a arrotondare, per staccare le cellule.
- Aggiungere 10 ml di DMEM-comp al pallone per interrompere il processo di tripsinizzazione. Sciacquare il pallone un paio di volte con il mezzo per aiutare a staccare gli ultimi periciti.
- Trasferire la sospensione cellulare da 12 ml su un tubo di centrifugazione da 50 ml e riempire fino a 30 mL con DMEM-comp.
- Centrifugare la sospensione cellulare per 5 min a 500 x g e RT. Aspirare il DMEM-comp. con attenzione senza toccare il pellet cellulare. Resuspend il pellet cellulare in 3 mL di FBS con 10% DMSO.
- Trasferire la sospensione cellulare in crioviali; aggiungere 1 mL a ciascuno, quindi ci sarà un totale di 3 flal per T75-flask di periciti. Congelare i periciti a -80 °C durante la notte e spostarli in azoto liquido il giorno dopo per lo stoccaggio a lungo termine.
NOTA: Le cellule possono essere conteggiate prima del congelamento per una stima successiva della percentuale di sopravvivenza. Il protocollo può essere messo in pausa qui.
6. Creazione di una monocoltura pericita per esperimenti
- Rivestire un pallone T75 con collagene IV e fibronectina utilizzando la stessa procedura di cui al punto 3.1-3.4.
- Mentre il pallone viene rivestito con fibronectina, scongelare una fiala di periciti in un bagno d'acqua a 37 °C.
- Trasferire i periciti ora scongelati dal crioviale a un tubo di centrifugazione con 30 ml di DMEM-comp. Centrifugare la sospensione cellulare per 5 min a 500 x g,RT.
- Aspirare con cura il mezzo, lasciando il pellet cellulare nella parte inferiore del tubo. Sospendere di nuovo il pellet in DMEM-comp da 10 mL.
- Raccogliere e trasferire la sospensione cellulare nel pallone rivestito. Lasciare il pallone con periciti per crescere in un incubatore di 37 °C al 10% di CO2.
- Ogni secondo giorno, rinfrescare il mezzo con 10 mL di DMEM-comp fresco.
NOTA: Dopo 5 giorni di crescita, i periciti avrebbero dovuto raggiungere circa l'80% di confluenza. Se la confluenza è minore, lasciare che le cellule crescano per un altro giorno o due. Le cellule dovrebbero ora essere pronte per la semina per ulteriori esperimenti.
7. Semina di periciti in una piastra rivestita da 96 potte
- Diluire il collagene IV come descritto nella fase 3.1. Aggiungere 100 μL a ciascun pozzo in una piastra da 96 po' e incubare per 2 ore a RT o durante la notte a 4 °C.
- Aspirare la soluzione e lavare i pozzi tre volte con PBS.
- Aggiungere 100 μL di fibronettatina diluita ad ogni pozzo e incubare a RT per 30 min. Rimuovere la soluzione di fibronectina e utilizzare immediatamente la piastra.
NOTA: A seconda di quanto bene sta crescendo il lotto di periciti, dovrebbero esserci abbastanza cellule per seminare due piastre. - Eserti i periciti dall'incubatore e aspira il mezzo. Lavare le celle con PBS.
- Aggiungere 2 mL di tripside-EDTA ai periciti e seguire la stessa procedura del passaggio 5.3-5.6.
- Aspirare il mezzo, senza danneggiare il pellet cellulare e sospendere di nuovo il pellet in 1 mL di DMEM-comp fresco.
- Esezione di 12 μL di sospensione cellulare e aggiungere a una camera di conteggio. Al microscopio a luce, contare almeno 3 griglie 3x3 e utilizzare il numero medio di celle per griglia.
- Utilizzare l'equazione seguente per calcolare il volume della sospensione cellulare che deve essere aggiunto a ciascun pozzo per seminare 10.000 cellule per pozzo, nella piastra a 96 pozzi.
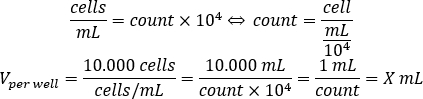
- Aggiungere DMEM-comp e il volume calcolato di sospensione cellulare in ogni pozzo per raggiungere un volume finale di 200 μL.
- Posizionare la piastra da 96 porsi in un incubatore a 37 °C al 10% di CO2. Lasciare crescere le cellule per 4 giorni con un cambio di mezzo dopo 2 giorni.
8. Preparazione di tamponi e soluzioni per l'imaging Ca2+
- L'autoclave copre camere di celle a puntini di sospensione e copripavimenti.
- Tampone di dosaggio: Aggiungere 1,19 g di HEPES a 500 mL di tampone HBSS per una concentrazione finale di 10 mM HEPES. Regolare il pH a 7,4.
- Preparare 20% (con v) Pluronic F127 + 1% (v/v) soluzione di olio di ricino polietossilato sciogliendo 0,5 g di soluzione Pluronic F127 in 2,5 mL di DMSO anidro in una fiala di vetro. Riscaldare a 40 °C per circa 30 min o fino a quando non si è sciolto e vortice. Aggiungere 25 μL di olio di ritore polietossilato e conservare a RT. Non congelare.
- Preparare 2 mM Fura-2 AM stock sciogliendo 1 mg in 500 μL di DMSO anidro. Conservare in aliquote di 20 μL a -20 °C protette dalla luce.
- Preparare 5 μM Fura-2 AM soluzione di carico mescolando 20 μL di 20% Pluronic F-127 + 1% soluzione di olio di ritore polietossilato con la soluzione di 20 μL di 2 mM Fura-2 AM aliquota. Aggiungere 500 μL di tampone di dosaggio e vortice. Aggiungere il buffer di dosaggio a un volume finale di 8 mL. La soluzione deve essere preparata immediatamente prima dell'uso e protetta dalla luce.
- Preparare 4 mM Cal-520 AM sciogliendo 1 mg in 226,7 μL di DMSO anidro. Conservare in aliquote di 20 μL a -20 °C protette dalla luce.
- Preparare 20 μM Cal-520 AM soluzione di carico mescolando 20 μL di 20% Pluronic F-127 + 1% soluzione di olio di ritore polietossilato con l'aliquota 20 μL 4 mM Cal-520. Aggiungere 500 μL di tampone di dosaggio e vortice. Aggiungere il buffer di dosaggio a un volume finale di 4 mL. La soluzione deve essere preparata immediatamente prima dell'uso e protetta dalla luce.
9. Caricamento di periciti con colorante per indicatori di calcio Fura-2 AM in una configurazione lettore di lastre
NOTA: Tutte le soluzioni devono essere a RT prima dell'inizio dell'esperimento.
- Estraere la piastra da 96 pozzi con le cellule dell'incubatore e aspirare il mezzo dai pozzi. Lavare le cellule due volte con tampone di dosaggio.
- Aggiungere 100 μL di soluzione di carico ad ogni pozzo e avvolgere la piastra con tinfoil, per evitare lo sbiancamento fotografico. Incubare per 45 minuti con 30 giri/min tremando a RT.
NOTA: Non caricare Fura-2 AM a 37 °C, in quanto potrebbe caricare scomparti interni. Ricordarsi di lasciare pozzi con celle nel buffer di dosaggio invece di caricare il buffer; questi sono gli "spazi vuoti" utilizzati per misurare la fluorescenza di fondo. - Aspirare il tampone di carico e lavare le cellule con tampone di dosaggio due volte. Aggiungere 100 μL di tampone di dosaggio fresco e lasciare che le cellule incubano per 30 minuti a RT; ciò consente una scollatura continua dell'AM-estere e quindi intrappolando Fura-2 AM all'interno delle cellule.
- Prima dell'imaging Ca2+,lavare e sostituire il tampone con 100 μL di tampone di dosaggio fresco.
10. Lettura a fluorescenza a piastra di periciti in una configurazione lettore di lastre
- Impostare la temperatura del lettore di lastre a 37 °C e trasferire la piastra da 96 pozzi con le celle nella posizione della piastra del campione. Posizionare la piastra del reagente con l'agonista nella posizione della piastra del reagente.
- Iniziare misurando il carico delle celle per garantire lo stesso carico di Fura-2 AM in tutti i pozzi.
- Eseguire le misurazioni con lunghezza d'onda di fluorescenza di eccitazione a 340 nm/380 nm e la lunghezza d'onda di emissione a 510 nm. Aggiungere 50 μL di agonista alla velocità di 150 μL/s dalla piastra del reagente a ciascun pozzo con celle nella posizione della piastra del campione.
- Salvare i dati ed esportali come file xlsx per ulteriori analisi. La figura 2 mostra la risposta citosolica Ca2+misurata come rapporto tra le due lunghezze d'onda di eccitazione nel tempo, dove la fluorescenza di fondo viene sottratta.
NOTA: Il lettore di lastre deve essere un lettore a doppia micropiastra con spazio per un "vassoio cellulare" e un "vassoio campione" e un sistema di pipettor integrato.
11. Semina di periciti in una camera cellulare rivestita per l'imaging dal vivo
NOTA: Le coperture possono anche essere posizionate sul fondo dei pozzi di coltura, rivestite e sementi con periciti come descritto sopra, e quindi montate nella camera prima degli esperimenti.
- Montare un coverslip nella camera cellulare e renderlo stretto per evitare perdite.
- Diluire il collagene IV come descritto nella fase 3.1. Aggiungere 500 μL a ogni camera cellulare e incubare per 2 ore a RT o durante la notte a 4 °C.
- Aspirare la soluzione di collagene e lavare le camere tre volte con 500 μL di PBS.
- Aggiungere 500 μL di fibronectina diluita ad ogni pozzo e incubare a RT per 30 minuti. Rimuovere la soluzione di fibronectina e utilizzare la camera cellulare subito dopo.
- Nel frattempo, esenzi il pallone con periciti confluenti e lava con 7 ml di PBS.
- Aggiungere 2 mL di tripside-EDTA ai periciti e seguire la stessa procedura del passaggio 5.3-5.6.
- Procedere seguendo gli stessi passaggi del passaggio 8.6-8.7.
- Utilizzare l'equazione seguente per calcolare il volume della sospensione cellulare, che deve essere aggiunta a ogni camera per seminare 90.000 celle per camera.
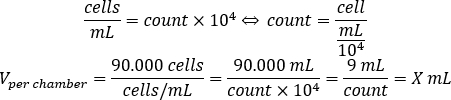
- Aggiungere DMEM-comp e il volume calcolato di sospensione cellulare in ogni camera per raggiungere un volume finale di 500 μL.
- Posizionare le camere cellulari nell'incubatore a 37 °C, 10% CO2. Lasciare crescere le cellule per 6 giorni (o fino a quando non sono confluenti).
NOTA: I periciti crescono più lentamente sui vetrini rispetto alla plastica; sono necessari più giorni di crescita.
12. Caricamento di periciti con colorante per indicatori di calcio Cal-520 AM per immagini dal vivo
NOTA: Tutte le soluzioni devono essere a RT prima dell'inizio dell'esperimento.
- Preparare il tampone di carico Cal-520 AM da 20 μM: Mescolare 20 μL di 20% Pluronic F-127 + 1% di soluzione di olio di rione polietossilato con l'aliquota 20 μL 4 mM Cal-520. Aggiungere 500 di tampone di dosaggio e vortice μL. Aggiungere il buffer di dosaggio a un volume finale di 4 mL. La soluzione deve essere preparata immediatamente prima dell'uso e protetta dalla luce.
NOTA: Proteggere le soluzioni contenenti Cal-520 AM dall'esposizione alla luce. - Esezione le camere cellulari dall'incubatore e aspirare il mezzo. Lavare le cellule due volte con tampone di dosaggio.
- Aggiungere 500 μL di tampone di carico a ogni camera e incubare a RT per 45 minuti.
- Aspirare il tampone di carico e lavare le cellule due volte con tampone di dosaggio.
- Aggiungere 500 μL di tampone di dosaggio fresco a ogni camera e incubare per 30 minuti a RT per consentire la scissione dell'AM-estere.
- Sostituire il buffer con 500 μL di tampone di dosaggio fresco prima di eseguire l'imaging dal vivo al microscopio confocale.
13. Imaging dal vivo di livelli ca2+intracellulari
NOTA: Una varietà di tipi di microscopio può essere utilizzata per l'imaging. È possibile utilizzare microscopi a scansione laser confocale verticale o invertita con sorgente di eccitazione appropriata (488 nm) e filtri di emissione (510-520 nm). Gli obiettivi dovrebbero essere adatti alla fluorescenza ed essere di alta qualità e ad alta apertura numerica (NA).
- Montare la camera cellulare sul palco del microscopio confocale il più delicato possibile, al fine di evitare disturbi delle cellule.
- Selezionare una lunghezza d'onda di eccitazione di 488 nm, emissione a 515 nm, acquisizione sequenziale di immagini con intervalli di 5 secondi, una dimensione dell'immagine XY di 512 x 512 pixel e misura per 2 minuti per misurare i segnali di calcio di base.
- Aggiungere 3 μL di ATP da 100 mM alla camera cellulare con una pipetta e continuare l'acquisizione sequenziale dell'immagine. Eseguire l'aggiunta lentamente e delicatamente per non disturbare la preparazione e spostare le cellule fuori fuoco.
- Osservare il grado di cambiamenti e aumentare l'intervallo di tempo nel tempo secondo necessità per circa 18 minuti fino a quando non si noterà alcun ulteriore cambiamento morfologico (Figura 3).
- Risparmia immagini time-lapse ed esportale come file TIFF e/o AVI per ulteriori analisi.
NOTA: Una fiala di periciti dovrebbe dare abbastanza cellule per la semina in piastre da 1-2 96 po 'e diverse coverlips, il che significa che puoi preparare le cellule per entrambi i tipi di misurazioni del calcio.
Risultati
I capillari cerebrali bovini sono stati isolati dal tessuto cerebrale fresco e la figura 1 presenta la semina capillare e la crescita cellulare nel corso dei giorni e la successiva purificazione dei periciti. I capillari sono completamente attaccati al pallone al giorno 1 e il giorno 2 la germogliazione endoteliale è diventata visibile(Figura 1, giorno 2). Dopo 4 giorni, la crescita cellulare è altamente distintiva
Discussione
In questo studio, abbiamo presentato un metodo per isolare i periciti primari dal cervello dei bovini. Il protocollo descritto consente impostazioni cultura di questo tipo di cella altrimenti piuttosto inaccessibile. La successiva coltura cellulare ottenuta era una popolazione quasi omogenea di periciti, con poca o nessuna contaminazione con cellule endoteliali e cellule gliali basate sulla morfologia cellulare e sull'espressione proteica12. Inoltre, abbiamo dimostrato un metodo semplice e diretto...
Divulgazioni
Gli autori non dichiarano interessi finanziari concorrenti.
Riconoscimenti
Gli autori desiderano riconoscere i finanziamenti dell'iniziativa di ricerca della Lundbeck Foundation sulle barriere cerebrali e la somministrazione di farmaci (RIBBDD) e della Simon Hougners Family Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| ATP | Tocris | 3245 | |
| Cal-520 AM | AAT Bioquest | 21130 | |
| Cell incubator | Thermo Fisher | ||
| Centrifuge | Thermo Fisher | Heraeus Multifuge 3SR+ | Standard large volume centrifuge for spinning down cells |
| Collagen IV | Sigma Aldrich | C5533 | |
| Confocal laser scanning microscope | Carl Zeiss | Zeiss LSM 510 | Inverted microscope |
| Counting chamber | FastRead | 102 | |
| Coverslip cell chamber | Airekacells | SC15022 | |
| Cremophor EL | Sigma Aldrich | C5135 | Formerly known as Kolliphor EL |
| DMSO | Sigma Aldrich | 471267 | |
| Dulbecco's Modified Eagles Medium | Sigma Aldrich | D0819 | |
| Fetal bovine serum (FBS) | PAA/GE Healthcare | A15-101 | |
| Fibronectin | Sigma Aldrich | F1141 | |
| Fura-2 AM | Thermo Fisher | F1201 | |
| Glass coverslips 22x22 mm | VWR International | 631-0123 | |
| HBSS | Gibco | 14065-049 | |
| Heparin | Sigma Aldrich | H3149 | |
| HEPES | AppliChem Panreac | A1069 | |
| Light microscope | Olympus | Olympus CK2 | Upright light microscope with phase contrast |
| MEM nonessential amino acids | Sigma Aldrich | M7145 | |
| Microplate Reader | BMG LabTech | NOVOstar | |
| PBS | Sigma Aldrich | D8537 | Phosphate-buffered saline |
| penicillin G sodium/streptomycin sulfate | Sigma Aldrich | P0781 | |
| Pluronic F127 | Sigma Aldrich | P2443 | |
| Trypsin-EDTA | Sigma Aldrich | T4299 | |
| T-75 flask | Sigma Aldrich | CLS3972 | |
| 96-well plate | Corning incorporated | 3603 |
Riferimenti
- Armulik, A., Genove, G., Betsholtz, C. Pericytes: Developmental, Physiological, and Pathological Perspectives, Problems, and Promises. Developmental Cell. 21 (2), 193-215 (2011).
- Abbott, N. J., Ronnback, L., Hansson, E. Astrocyte-endothelial interactions at the blood-brain barrier. Nature Reviews Neuroscience. 7 (1), 41-53 (2006).
- Mathiisen, T. M., Lehre, K. P., Danbolt, N. C., Ottersen, O. P. The Perivascular Astroglial Sheath Provides a Complete Covering of the Brain Microvessels: An Electron Microscopic 3D Reconstruction. Glia. 58 (9), 1094-1103 (2010).
- Helms, H. C., et al. In vitro models of the blood-brain barrier: An overview of commonly used brain endothelial cell culture models and guidelines for their use. Journal of Cerebral Blood Flow and Metabolism. 36 (5), 862-890 (2016).
- Zhou, L., Sohet, F., Daneman, R. Purification of Pericytes from Rodent Optic Nerve by Immunopanning. Cold Spring Harbor Protocols. , 608-617 (2014).
- Liu, G. H., et al. Isolation, Purification, and Cultivation of Primary Retinal Microvascular Pericytes: A Novel Model Using Rats. Microcirculation. 21 (6), 478-489 (2014).
- Nayak, R. C., Herman, I. M. Bovine retinal microvascular pericytes: isolation, propagation, and identification. Methods In Molecular Medicine. 46, 247-263 (2001).
- Nees, S., et al. Isolation, bulk cultivation, and characterization of coronary microvascular pericytes: the second most frequent myocardial cell type in vitro. American Journal of Physiology-Heart and Circulatory Physiology. 302 (1), H69-H84 (2012).
- Armulik, A., Abramsson, A., Betsholtz, C. Endothelial/pericyte interactions. Circulation Research. 97 (6), 512-523 (2005).
- Helms, H. C., Brodin, B., Milner, R. Methods in Molecular Biology. Cerebral Angiogenesis: Methods and Protocols. 1135, 365-382 (2014).
- Abbracchio, M. P., Burnstock, G., Verkhratsky, A., Zimmermann, H. Purinergic signalling in the nervous system: an overview. Trends in Neurosciences. 32 (1), 19-29 (2009).
- Cai, C., et al. Stimulation-induced rises in cerebral blood flow and local capillary vasoconstriction depend on conducted vascular responses in brain capillaries. PNAS. , (2018).
- Tigges, U., Welser-Alves, J. V., Boroujerdi, A., Milner, R. A novel and simple method for culturing pericytes from mouse brain. Microvascular Research. 84 (1), 74-80 (2012).
- Maier, C. L., Shepherd, B. R., Yi, T., Pober, J. S. Explant Outgrowth, Propagation and Characterization of Human Pericytes. Microcirculation. 17 (5), 367-380 (2010).
- Orlidge, A., Damore, P. A. Cell specific effects of glycosaminoglycans on the attachment and proliferation of vascular wall components. Microvascular Research. 31 (1), 41-53 (1986).
- Rhinehart, K., Zhang, Z., Pallone, T. L. Ca2+ signaling and membrane potential in descending vasa recta pericytes and endothelia. American Journal of Physiology-Renal Physiology. 283, F852-F860 (2002).
- Bintig, W., et al. Purine receptors and Ca2+ signalling in the human blood-brain barrier endothelial cell line hCMEC/D3. Purinergic Signalling. 8 (1), 71-80 (2012).
- Neuhaus, A. A., Couch, Y., Sutherland, B. A., Buchan, A. M. Novel method to study pericyte contractility and responses to ischaemia in vitro using electrical impedance. Journal of Cerebral Blood Flow and Metabolism. 37 (6), 2013-2024 (2017).
- Kamouchi, M., et al. Calcium influx pathways in rat CNS pericytes. Molecular Brain Research. 126 (2), 114-120 (2004).
- Das, A., et al. ATP Causes Retinal Pericytes to Contract In vitro. Experimental Eye Research. 46, 349-362 (1988).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon