È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analisi dei sistemi della risposta neuroinfiammatoria ed emodinamica alla lesione cerebrale traumatica
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo presenta metodi per caratterizzare la risposta neuroinfiammatoria ed emodinamica a lesioni cerebrali traumatiche lievi e per integrare questi dati come parte di un'analisi di sistemi multivariati utilizzando la regressione parziale dei minimi quadrati.
Abstract
Le lesioni cerebrali traumatiche lievi (mTBI) sono un problema significativo di salute pubblica. L'esposizione ripetuta a mTBI può portare a deficit funzionali cumulativi e di lunga durata. Numerosi studi del nostro gruppo e di altri hanno dimostrato che mTBI stimola l'espressione delle citochine e attiva la microglia, diminuisce il flusso sanguigno cerebrale e il metabolismo e compromette la reattività cerebrovascolare. Inoltre, diversi lavori hanno riportato un'associazione tra squilibri in questi marcatori neuroinfiammatori ed emodinamici e deficit cognitivi. Qui descriviamo in dettaglio i metodi per caratterizzare la risposta del tessuto neuroinfiammatorio ed emodinamico a mTBI nei topi. In particolare, descriviamo come eseguire un modello di caduta di peso di mTBI, come misurare longitudinalmente il flusso sanguigno cerebrale utilizzando una tecnica ottica non invasiva chiamata spettroscopia di correlazione diffusa e come eseguire un test immunologico multiplexed Luminex su campioni di tessuto cerebrale per quantificare citochine e fosfoproteine immunomodulatorie (ad esempio, all'interno delle vie MAPK e NFκB) che rispondono e regolano l'attività della microglia e di altre cellule immunitarie neurali. Infine, descriviamo in dettaglio come integrare questi dati utilizzando un approccio di analisi dei sistemi multivariati per comprendere le relazioni tra tutte queste variabili. Comprendere le relazioni tra queste variabili fisiologiche e molecolari ci consentirà in ultima analisi di identificare i meccanismi responsabili dell'mTBI.
Introduzione
Panoramica
Lievi lesioni cerebrali traumatiche (mTBI) hanno un impatto ~ 1,6-3,8 milioni di atleti all'anno1. Queste lesioni, comprese le lesioni sub-concussive e concussive, possono lasciare i pazienti con sintomi fisici, emotivi, psicologici e cognitivi transitori2. Inoltre, l'mTBI ripetitivo (rmTBI) sostenuto all'interno di una "finestra di vulnerabilità" può portare a gravità cumulativa e durata delle conseguenze cognitive che durano più a lungo degli effetti di un singolo mTBI da solo3 e, in definitiva, anche alla perdita permanente della funzione 4,5,6. Sebbene molti pazienti guariscano in un lasso di tempo relativamente breve (<1 settimana), il 10-40% dei pazienti soffre di effetti più duraturi di mTBI per > 1 mese, con alcuni che durano fino a 1 anno 3,7,8,9. Nonostante l'elevata prevalenza e le conseguenze durature di queste lesioni, i meccanismi di lesione sono poco conosciuti e non esistono strategie di trattamento efficaci.
Data l'elevata variabilità degli esiti dopo mTBI/rmTBI, una sfida nell'identificare i trigger molecolari in fase iniziale dal tessuto ottenuto negli studi terminali mTBI/rmTBI è la mancanza di dati longitudinali che dimostrino i "legami molecolari acuti" definitivi di questi trigger molecolari a esiti a lungo termine. Per superare questa sfida, il nostro gruppo ha scoperto che il flusso sanguigno cerebrale acutamente ridotto misurato acutamente utilizzando uno strumento ottico chiamato spettroscopia di correlazione diffusa (DCS), è fortemente correlato con l'esito cognitivo a lungo termine in un modello murino di rmTBI10. Utilizzando questo biomarcatore emodinamico, abbiamo dimostrato che i topi con un flusso sanguigno cerebrale acutamente basso (e, per estensione, un esito a lungo termine peggiore previsto) hanno concomitanti aumenti acuti della segnalazione fosfo neuronale all'interno delle vie MAPK e NFκB, aumenti dell'espressione neuronale delle citochine pro-infiammatorie e aumenti dell'espressione del fagocita/marcatore microgliale Iba111 . Questi dati suggeriscono un possibile ruolo per la segnalazione fosfo-neuronale, l'espressione di citochine e l'attivazione microgliale sia nella regolazione acuta del flusso sanguigno cerebrale dopo la lesione, sia nell'innescare una cascata di segnalazione che porta a disfunzioni neuronali e peggior esito cognitivo. Qui, descriviamo in dettaglio il nostro approccio per sondare simultaneamente sia l'ambiente emodinamico che neuroinfiammatorio dopo rmTBI e come integrare questi set di dati complessi. In particolare, delineiamo le procedure per quattro passaggi chiave di questo approccio globale: (1) un modello di caduta di peso di lieve lesione cerebrale traumatica, (2) valutazione del flusso sanguigno cerebrale con spettroscopia di correlazione diffusa, (3) quantificazione dell'ambiente neuroinfiammatorio e (4) integrazione dei dati (Figura 1). Di seguito, forniamo una breve introduzione a ciascuno di questi passaggi chiave per aiutare a guidare i lettori attraverso la logica alla base dei nostri metodi. Il resto del manoscritto fornisce un protocollo dettagliato per ciascuno di questi passaggi chiave.
Modello di calo di peso di lieve lesione cerebrale traumatica
Sebbene esistano molti eccellenti modelli preclinici di TBI lieve ripetitivo 12,13,14,15,16,17,18, impieghiamo un modello di trauma cranico chiuso a caduta di peso ben consolidato e clinicamente rilevante. Le caratteristiche principali di questo modello includono (1) impatto smussato del cranio / cuoio capelluto intatto seguito da rotazione illimitata della testa attorno al collo, (2) nessuna lesione cerebrale strutturale evidente, edema, danno da barriera emato-encefalica, morte cellulare acuta o perdita cronica del tessuto cerebrale e (3) deficit cognitivi persistenti (fino a 1 anno) che emergono solo dopo più colpi19 (Figura 2).
Valutazione del flusso sanguigno cerebrale con spettroscopia di correlazione diffusa
La spettroscopia di correlazione diffusa (DCS) è una tecnica ottica non invasiva che misura il flussosanguigno 5,20,21. In DCS, una sorgente di luce nel vicino infrarosso viene posizionata sulla superficie del tessuto. Un rilevatore viene posizionato a una distanza fissa dalla sorgente sulla superficie del tessuto per rilevare la luce che si è moltiplicata dispersa attraverso il tessuto (Figura 3). La dispersione dei globuli rossi in movimento fa sì che l'intensità della luce rilevata fluttui nel tempo. Un semplice modello analitico noto come teoria della diffusione della correlazione viene utilizzato per correlare queste fluttuazioni di intensità a un indice di flusso sanguigno (CBFi, Figura 4). Sebbene le unità di CBFi (cm2/s) non siano le tradizionali unità di flusso (mL/min/100 g), uno studio precedente sui topi ha dimostrato che il CBFi è fortemente correlato al flusso sanguigno cerebrale misurato mediante spin arterioso marcato MRI21.
Per riferimento, lo strumento DCS utilizzato qui è stato costruito internamente ed è composto da un laser a lunghezza di coerenza lunga 852 nm, una serie di 4 fotodiodi a valanga di conteggio dei fotoni e una scheda autocorrelatore hardware (singola tau, 8 canali, 100 ns tempo minimo di campionamento)21,22. I dati vengono acquisiti con software fatti in casa scritti in LabView. L'interfaccia animale per il dispositivo è costituita da una fibra sorgente multimodale da 400 μm (gamma di lunghezze d'onda 400-2200 nm, nucleo di silice pura, rivestimento rigido TECS) e una fibra del rilevatore monomodale da 780 nm (gamma di lunghezze d'onda 780-970 nm, nucleo di silice pura, rivestimento rigido TECS, 730 ± taglio della seconda modalità a 30 nm) distanziati di 6 mm e incorporati in un sensore nero stampato in 3D (4 mm x 8 mm, Figura 3).
Quantificazione dell'ambiente neuroinfiammatorio
Sebbene la neuroinfiammazione sia regolata da diversi processi cellulari, due meccanismi chiave rilevanti sono la segnalazione extracellulare da parte di citochine / chemochine e la segnalazione intracellulare da parte di fosfoproteine. Per studiare l'ambiente neuroinfiammatorio del cervello post-lesione, i cervelli vengono estratti da topi, microsezionati e citochine / chemochine e fosfoproteine vengono quantificate usando Luminex (Figura 5, Figura 6, Figura 7). I saggi immunologici multiplexed Luminex consentono la quantificazione simultanea di una collezione diversificata di queste proteine accoppiando saggi immunoassorbenti enzimatici (ELISA) a perline magnetiche marcate fluorescentmente. Per ogni proteina di interesse vengono utilizzati tag fluorescenti distinti e le perline di ciascun tag sono funzionalizzate con un anticorpo di cattura contro quella particolare proteina. Centinaia di perline per catturare ogni proteina vengono mescolate insieme, poste in una piastra da 96 pozzetti e incubate con il campione. Dopo l'incubazione del campione, viene utilizzato un magnete per intrappolare le perline nel pozzo mentre il campione viene lavato via. Successivamente, l'anticorpo di rilevamento biotinilato si lega all'analita di interesse per formare un sandwich anticorpo-antigene simile a un ELISA tradizionale, ma con l'ELISA per ogni proteina che si verifica su un diverso tallone marcato fluorescentemente. L'aggiunta di streptavidina coniugata con ficoeritrina (SAPE) completa ogni reazione. Lo strumento Luminex legge quindi le perline e separa il segnale in base a ciascun tag/proteina fluorescente.
Integrazione dei dati
A causa del gran numero di analiti (ad esempio, citochine) misurati nel test Luminex, l'analisi dei dati può essere difficile da interpretare se ogni proteina quantificata viene analizzata individualmente. Per semplificare l'analisi e catturare le tendenze osservate tra gli analiti, utilizziamo un metodo di analisi multivariata chiamato regressione parziale dei minimi quadrati (PLSR, Figura 8)23. PlSR funziona identificando un asse di pesi corrispondente a ciascuna proteina misurata (cioè citochine o fosfo-proteine, denominate "variabili predittive") che insieme spiegano in modo ottimale la co-varianza delle proteine misurate con una variabile di risposta (ad esempio, flusso sanguigno cerebrale). I pesi sono indicati come "carichi" e sono assemblati in un vettore noto come variabile latente (LV). Proiettando (indicato come "punteggio") i dati proteici misurati su ciascuno dei due LV, i dati possono essere ritracciati in termini di questi LV. Dopo aver calcolato il PLSR, utilizziamo una rotazione varimax per identificare un nuovo LV che massimizza la covarianza tra le proiezioni del campione sul LV e la variabile predittiva24. Questo approccio ci consente di definire LV1 come l'asse per il quale viene spiegata al meglio la varianza della variabile di risposta. LV2 massimizza la co-varianza tra la variabile di risposta e i dati residui di LV1, che possono essere associati alla variabilità biologica o tecnica tra i campioni. Infine, conduciamo una Convalida incrociata Leave One Out (LOOCV) per garantire che il modello PLSR non dipenda fortemente da un campione23.
In questo protocollo, dettagliamo i metodi per caratterizzare la risposta del tessuto neuroinfiammatorio ed emodinamico a mTBI. Il flusso di lavoro generale è descritto nella Figura 1. In questo protocollo, i topi sono soggetti a uno o più mTBI utilizzando un modello di trauma cranico chiuso a goccia di peso. Il flusso sanguigno cerebrale viene misurato longitudinalmente prima e in più punti temporali dopo l'infortunio. Nel momento di interesse per l'interrogazione dei cambiamenti neuroinfiammatori, l'animale viene eutanasia e il cervello viene estratto. Le regioni cerebrali di interesse sono isolate tramite microdissezione e quindi lisate per estrarre proteine. I lisati vengono quindi utilizzati sia per i saggi immunologici multiplexati Luminex di espressione di citochine e fosfoproteine, sia per Western blot. Infine, questo set di dati olistico è integrato utilizzando un'analisi di regressione parziale dei minimi quadrati.
Protocollo
Tutte le procedure per gli animali sono approvate dal Comitato istituzionale per la cura e l'uso degli animali della Emory University (IACUC) e hanno seguito le linee guida NIH per la cura e l'uso degli animali da laboratorio.
1. Modello di calo di peso di lieve lesione cerebrale traumatica
- Preparare la configurazione di caduta di peso. Montare una morsa su una superficie piana con un tubo guida di 1 m (diametro interno 2,54 cm) allineato verticalmente (controllare utilizzando un livello). Utilizzare un bullone da 54 g (diametro base del corpo di 0,95 cm, diametro della testa di 2 cm, lunghezza 10,2 cm) per l'impatto.
- Anestetizzare brevemente il mouse. Indurre il topo con il 4,5% di isoflurano in ossigeno al 100% per 45 secondi. Confermare una profondità sufficiente dell'anestesia dalla mancanza di una risposta al pizzico della punta.
- Indurre lesioni.
- Rimuovere rapidamente il mouse dall'anestesia e posizionare il mouse prono al centro di una membrana sottile (11,2 cm x 21,3 cm di tessuto).
- Usa entrambe le mani per tenere il tessuto teso con il mouse prono al centro. Fissare la coda del mouse sotto un pollice. Posizionare la testa del mouse sotto il tubo guida (Figura 2).
- Lascia cadere il bullone dalla parte superiore del tubo guida sull'aspetto dorsale della testa del topo, mirando all'impatto tra la parte posteriore degli occhi e la parte anteriore delle orecchie. All'impatto, il topo penetrerà nel tessuto, consentendo una rapida accelerazione della testa attorno al collo (Figura 2).
- Guarigione
- Dopo l'impatto, posizionare il mouse supino su un cuscinetto riscaldante a 37 °C nell'aria ambiente. Monitorare il recupero per 1 ora dopo l'infortunio. Entro 1 ora, i topi dovrebbero essere in grado di deambulare normalmente, trovare cibo e acqua e non mostrare deficit motori lordi.
NOTA: L'analgesia non viene utilizzata per approvazione del Comitato istituzionale per la cura e l'uso degli animali, il che è giustificato dall'influenza confondente dell'analgesia sui parametri di interesse (ad esempio, flusso sanguigno cerebrale, marcatori di infiammazione). La perdita di coscienza, definita come il tempo che intercorre tra la rimozione dall'anestesia e il tempo per ritrovare il riflesso raddrizzante, è prevista e dura tipicamente dai 20 ai 3 minuti (Tabella supplementare 1). Si possono osservare brevi episodi (<30 s) di apnea e/o attività simile alle convulsioni, in particolare dopo lesioni ripetitive alla testa distanziate una volta al giorno.
- Dopo l'impatto, posizionare il mouse supino su un cuscinetto riscaldante a 37 °C nell'aria ambiente. Monitorare il recupero per 1 ora dopo l'infortunio. Entro 1 ora, i topi dovrebbero essere in grado di deambulare normalmente, trovare cibo e acqua e non mostrare deficit motori lordi.
- Ripetere l'operazione se necessario. Questa lesione può essere ripetuta una volta al giorno, settimanalmente o mensilmente. Il numero e la spaziatura delle lesioni dipendono dalla gravità della lesione desiderata. In genere, impieghiamo cinque colpi distanziati una volta al giorno per indurre forti deficit nell'apprendimento spaziale e nella memoria.
NOTA: Studi precedenti hanno dimostrato che cinque colpi distanziati una volta al giorno sono sufficienti per indurre deficit nell'apprendimento spaziale e nella memoria che durano oltre 1 anno dopo l'infortunio senza edema, emorragia o lesioni strutturali evidenti al cervello19. I topi vengono pesati quotidianamente e attentamente monitorati per segni di disidratazione, deficit motori e perdita di appetito. Se disidratati, ai topi viene somministrato chow umido e un'iniezione sottocutanea di 1 ml di soluzione salina una volta al giorno. Al fine di prevenire sofferenze inutili e garantire un endpoint umano, i topi vengono eutanasizzati se: la disidratazione persiste o peggiora >24 ore dopo il trattamento salino post-sottocutaneo, il peso corporeo diminuisce di oltre il 20% rispetto al basale pre-lesione, compaiono deficit motori come il cerchio o il trascinamento della zampa e persistono >1 ora dopo l'infortunio.
2. Valutazione del flusso sanguigno cerebrale con spettroscopia di correlazione diffusa
- Acquisizione dati DCS
- Rimuovere i capelli sul cuoio capelluto. Poiché la DCS funziona meglio in assenza di peli, è necessario rimuovere la pelliccia sulla testa prima dell'inizio degli esperimenti. In genere, la depilazione viene eseguita 1-3 giorni prima dell'inizio dello studio.
- Indurre topi con il 4,5% di isoflurano in ossigeno al 100% per 45 secondi e mantenere con l'1-2% di isoflurano in ossigeno al 100%.
- Rasare la testa tra gli occhi e le orecchie. Quindi, utilizzare la crema depilatoria per rimuovere la pelliccia sulla testa come nella Figura 3.
- Lascia che l'animale si riprenda dall'anestesia su un cuscinetto riscaldante e poi torni in gabbia.
- Misurare il flusso sanguigno cerebrale con DCS. Per ridurre al minimo gli artefatti di movimento durante la misurazione, studiare i topi in breve anestesia isoflurano.
NOTA: monitorare visivamente la respirazione e la risposta al pizzico delle dita durante le misurazioni e regolare la concentrazione di isoflurano secondo necessità per garantire una profondità costante dell'anestesia. Variazioni significative nella profondità dell'anestesia potrebbero alterare il flusso sanguigno dati i noti effetti vasomodulatori dell'isoflurano25.- Indurre con il 4,5% di isoflurano in ossigeno al 100% per 45 secondi, quindi mantenere con l'1,0-1,75% di isoflurano nel 100% di ossigeno. Confermare una profondità sufficiente dell'anestesia dall'assenza di una risposta al pizzico della punta e dalla normale respirazione (tra ~ 60-80 respiri al minuto).
- Dopo un periodo di stabilizzazione di 2 minuti, appoggiare delicatamente il sensore DCS sull'emisfero destro in modo che il bordo superiore del sensore ottico si allinei con la parte posteriore dell'occhio e il lato del sensore si allinei lungo la linea mediana (Figura 3). Prendi una mano sopra il sensore per proteggerlo dalla luce della stanza. Acquisire 5 secondi di dati (acquisizione a 1 Hz).
- Riposiziona il sensore sull'emisfero sinistro e acquisisci 5 secondi di dati.
- Ripeti 3 volte / emisfero per tenere conto delle eterogeneità locali sotto la superficie del tessuto.
- Guarigione
- Rimuovere il mouse dall'anestesia e posizionarlo su un pad riscaldante.
- Dopo che il topo riacquista il suo riflesso di raddrizzamento, restituiscilo alla gabbia.
- Rimuovere i capelli sul cuoio capelluto. Poiché la DCS funziona meglio in assenza di peli, è necessario rimuovere la pelliccia sulla testa prima dell'inizio degli esperimenti. In genere, la depilazione viene eseguita 1-3 giorni prima dell'inizio dello studio.
- Analisi dei dati DCS
- Eseguire il controllo di qualità iniziale. Ogni fotogramma di dati DCS è costituito da una funzione
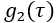 di autocorrelazione di intensità normalizzata misurata (Figura 4A) e da una frequenza di conteggio dei fotoni (kHz).
di autocorrelazione di intensità normalizzata misurata (Figura 4A) e da una frequenza di conteggio dei fotoni (kHz).- Per rimuovere i fotogrammi di dati con artefatti di movimento significativi, eliminate i fotogrammi di dati per i quali il
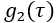 valore medio della coda della curva (ad esempio,
valore medio della coda della curva (ad esempio, 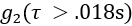 ) è > 1.005.
) è > 1.005. - Per rimuovere i fotogrammi di dati con scarso rapporto segnale-rumore, eliminare i fotogrammi di dati se la frequenza di conteggio dei fotoni rilevata è < 20 kHz.
- Per rimuovere i fotogrammi di dati con artefatti di movimento significativi, eliminate i fotogrammi di dati per i quali il
- Estrarre l'indice del flusso sanguigno cerebrale. Utilizzando fminsearch in Matlab, adatta ogni ith frame
 di dati misurati per CBFi(i). Restrict fits to
di dati misurati per CBFi(i). Restrict fits to 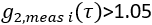 , e trova il valore di CBFi che riduce al minimo la seguente funzione di costo:
, e trova il valore di CBFi che riduce al minimo la seguente funzione di costo: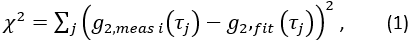
Dove la somma è sopra tutti i tempi di ritardo misurati,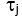 , ed
, ed 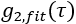 è la soluzione omogenea semi-infinita dell'equazione di diffusione di correlazione (Figura 4B):
è la soluzione omogenea semi-infinita dell'equazione di diffusione di correlazione (Figura 4B):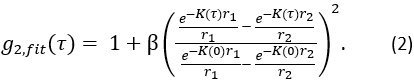
Qui β è un fattore di coerenza determinato dall'impostazione sperimentale,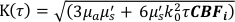 ,
, 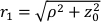 ,
, 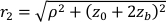 ,
, 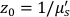 ,
, 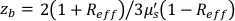 , Reff =0,493 per un indice tissutale presunto di rifrazione di 1,4, ρ è 6 mm, e μa e μ sono l'assorbimento e il coefficiente di scattering ridotto del tessuto (assunto come 0,25 e 9,4/cm, rispettivamente10,26,27).
, Reff =0,493 per un indice tissutale presunto di rifrazione di 1,4, ρ è 6 mm, e μa e μ sono l'assorbimento e il coefficiente di scattering ridotto del tessuto (assunto come 0,25 e 9,4/cm, rispettivamente10,26,27).
NOTA: poiché β possono variare di circa il 10% nel tempo, adattare contemporaneamente ogni frame di dati per β e CBFi . - Eseguire il controllo di qualità secondario. All'interno di ogni ripetizione (che consiste di 5 frame di dati), scartare i valori anomali. I valori anomali sono definiti come quei valori CBFi che non rientrano in 1,5 deviazioni standard del CBFmedio i per quella ripetizione. Se più di 1 punto dati viene identificato come anomalo, scartare l'intera ripetizione.
- Stimare l'indice medio del flusso sanguigno cerebrale: stimare un CBFi medio per emisfero prendendo la media tra tutti i frame di dati per tutte le ripetizioni (Figura 4C). Se non si osservano differenze emisferiche significative, mediare tra gli emisferi per ottenere una stima del CBFglobale medio i.
- Eseguire il controllo di qualità iniziale. Ogni fotogramma di dati DCS è costituito da una funzione
3. Quantificazione multiplexata di citochine e fosfoproteine mediante saggi luminex
- Estrazione tissutale
NOTA: La quantificazione delle citochine cerebrali e delle proteine fosfo-segnalanti utilizzando Luminex richiede l'estrazione dei tessuti.- Anestetizzare il mouse utilizzando il 4,5% di isoflurano in ossigeno al 100% per 1-2 minuti. Controllare il piano profondo dell'anestesia tramite la mancanza di una risposta al pizzico della punta. Eutanasia tramite decapitazione.
- Raccogli il tessuto.
- Rimuovi il cervello. In genere, fissare l'emisfero sinistro per l'istologia e microdissettare diverse regioni dell'emisfero destro all'interno della corteccia e dell'ippocampo (Figura 5).
- Posizionare i campioni sezionati in tubi di microcentrifuga, congelare il flash in azoto liquido. Per l'analisi delle proteine sensibili al congelamento, è ottimale suddividere le sezioni di tessuto prima del congelamento flash per evitare il successivo congelamento-scongelamento.
NOTA: il protocollo può essere messo in pausa e i campioni di tessuto possono essere conservati a -80 °C fino a quando non sono pronti per lisare i campioni. In alternativa, i campioni possono essere lisati e quindi conservati a -80 °C.
- Campioni di lisatura.
- Preparare il tampone di lisi aggiungendo inibitore della proteasi e 2 mM di fenilmetilsulfonil fluoruro al tampone di lisi.
- Aggiungere 150 μL del tampone di lisi mista per circa 3 μg di tessuto animale. Per riferimento, i campioni di tessuto della corteccia visiva del topo sono di circa 3 μg.
- Per omogeneizzare il tessuto, triturare meccanicamente il tessuto tubando su e giù ~ 15-20 volte usando una pipetta da 1000 μL. Per una triturazione ottimale del campione, è possibile utilizzare un pestello omogeneizzatore.
- Posizionare le provette campione su un rotatore per 30 minuti a 4°C.
- Centrifugare i campioni a 4°C per 10 minuti a circa 15.000 x g e raccogliere il surnatante. I campioni possono essere trattati immediatamente o conservati a -80°C per ulteriori analisi.
NOTA: I lisati campione preparati utilizzando questo protocollo sono compatibili con Western blot, da cui il marcatore fagocitario/microgliale Iba1 e/o il marcatore di attivazione astrocitaria GFAP possono essere analizzati per integrare l'analisi delle citochine e delle fosfo-proteine per gli studi di neuroinfiammazione11.
- Protocollo di immunodosaggio multiplex per citochine e fosfoproteine
NOTA: Sebbene nel complesso simili, ci sono alcune piccole differenze nei protocolli per i kit di citochine e fosfoproteine. Le differenze sono annotate in ogni passaggio. I passaggi per preparare i campioni per il test Luminex sono descritti di seguito.- Preparazione dei reagenti (giorno 1, stesso per le citochine e le fosfoproteine)
- Lasciare riscaldare i reagenti a temperatura ambiente (~30 min).
- Sonicate multiplex bottiglia di perline magnetiche per 30 secondi seguita da 1 minuto di vortice. Assicurarsi che le perle magnetiche multiplex siano schermate dalla luce con un foglio di alluminio o utilizzare bottiglie protettive leggere fornite.
- Preparare il tampone di lavaggio mescolando 0,1% Tween20 in 1xPBS o in alternativa utilizzare il tampone di lavaggio fornito nel kit.
- Preparazione di campioni di tessuto lisato (giorno 1, lo stesso per citochine e fosfoproteine)
- Se precedentemente congelati, rimuovere i campioni di tessuti lisati dal congelatore e lasciare scongelare sul ghiaccio (~ 20 min). Centrifugare i campioni per 10 minuti a 9.167 x g per rimuovere il precipitato.
- Preparare 25 μL di campione alla concentrazione ottimale di proteine determinata dall'analisi lineare dell'intervallo (vedere paragrafo 3.3). Per normalizzare il volume totale per tutti i campioni, diluire i campioni nel tampone di dosaggio fornito nel kit.
- Preparazione di 96 piastre di pozzetto (giorno 1, lo stesso per citochine e fosfoproteine)
- Utilizzare la piastra del pozzo 96 inclusa nel kit o una con un fondo sottile (ad esempio, Brand Tech).
- Aggiungere 200 μL di tampone di lavaggio (o 1x PBS, 0,1% Tween) in ogni pozzetto e mescolare sullo shaker per 10 minuti a 750 giri / min.
- Tampone di lavaggio decantante e toccare la piastra su un tovagliolo di carta per rimuovere i residui.
- Procedura di immunodosaggio per le citochine (giorno 1)
- Aggiungere quanto segue a ciascun pozzetto in ordine.
- Aggiungere 25 μL di tampone di dosaggio a tutti i pozzetti.
- Aggiungere 25 μL di buffer di analisi aggiuntivo SOLO ai pozzetti di fondo. Per ogni esecuzione sperimentale, avere almeno due pozzetti di sfondo. I pozzetti di fondo non hanno alcun campione caricato e definiscono l'intensità fluorescente letta dallo strumento senza campione.
- Aggiungere 25 μL di ciascun campione diluito ai pozzetti del campione corrispondenti.
- Aggiungere 25 μL di 1x perle magnetiche multiplex a tutti i pozzetti (Figura 6). Assicurati di vorticose perline per 1 minuto prima di aggiungere ai pozzetti.
- Aggiungere 25 μL di tampone di dosaggio a tutti i pozzetti.
- Sigillare la piastra con sigillante a piastre e coprire la piastra con un foglio di alluminio. Incubare durante la notte (12-16 h) a 2-8 °C.
- Aggiungere quanto segue a ciascun pozzetto in ordine.
- Procedura di immunodosaggio per le citochine (giorno 2)
- Posizionare la piastra del pozzo 96 sul separatore magnetico, assicurandosi che i pozzetti siano allineati con i magneti. Lasciare riposare per 2 minuti. Decantare il contenuto del pozzo mentre la piastra è ancora attaccata al separatore magnetico.
- Lavare la piastra 2 volte utilizzando i seguenti passaggi.
- Aggiungere 200 μL di tampone di lavaggio a ciascun pozzetto e posizionare sullo shaker per 2 minuti a temperatura ambiente.
- Posizionare la piastra del pozzo sul separatore magnetico per 2 minuti a temperatura ambiente.
- Decantare il contenuto del pozzo mentre la piastra del pozzo è ancora attaccata al separatore magnetico.
- Aggiungere 25 μL di anticorpo di rilevamento per pozzetto (Figura 6). Coprire con un foglio. Incubare per 1 ora su uno shaker a piastre (750 giri / min) a temperatura ambiente.
- Lasciare l'anticorpo di rilevamento e aggiungere 25 μL di streptavidina-ficoeritrina (SAPE) a ciascun pozzetto (Figura 6). Coprire con un foglio. Incubare per 30 minuti su scuotitore di piastre (750 giri / min) a temperatura ambiente.
- Posizionare la piastra del pozzo su un separatore magnetico e lasciare riposare per 2 minuti. Decantare il contenuto del pozzo e staccarsi dal separatore magnetico.
- Lavare bene la piastra due volte (vedere il punto 3.2.5.2).
- Aggiungere 75 μL di Luminex Drive Fluid (se si utilizza lo strumento MAGPIX) a ciascun pozzetto o buffer di analisi (se si utilizza uno strumento 3D 200 o FlexMap). Ri-sospendere le perline sullo shaker a piastre per 5 minuti a temperatura ambiente.
- Leggere su Luminex Instrument (MAGPIX, 200 o FlexMap 3D), facendo riferimento al manuale dell'utente per il corretto funzionamento (Figura 6).
- Procedura di immunodosaggio per fosfoproteine (giorno 1)
- Aggiungere quanto segue a ciascun pozzetto in ordine.
- Aggiungere 25 μL di tampone di dosaggio a tutti i pozzetti.
- Aggiungere 25 μL di buffer di analisi aggiuntivo SOLO ai pozzetti di fondo. Per ogni esecuzione sperimentale, si consiglia di avere almeno due pozzetti di sfondo. I pozzetti di fondo non hanno alcun campione caricato e definiscono l'intensità fluorescente letta dallo strumento senza campione.
- Aggiungere 25 μL di ciascun campione diluito a ciascun pozzetto del campione.
- Aggiungere 25 μL di 1x perle magnetiche multiplex a tutti i pozzetti (Figura 6).
NOTA: il kit di analisi Luminex fornisce perline magnetiche multiplex in soluzione madre 20x. Assicurarsi di vortex 20x stock multiplex soluzione di perline magnetiche per 2 minuti, quindi diluirla nel tampone di analisi a 1x soluzione. Vortex 1x sospensione magnetica multipla per 1 minuto prima di aggiungere ai pozzetti.
- Sigillare la piastra con sigillante a piastre e coprire la piastra con un foglio di alluminio. Incubare durante la notte (12-16 ore) a 2-8 °C.
- Aggiungere 25 μL di tampone di dosaggio a tutti i pozzetti.
- Aggiungere quanto segue a ciascun pozzetto in ordine.
- Procedura di immunodosaggio per fosfoproteine (giorno 2)
- Posizionare la piastra del pozzo su un separatore magnetico, assicurandosi che la piastra del pozzo sia completamente allineata con il separatore magnetico. Lasciare riposare per 2 minuti. Decantare il contenuto del pozzo mentre la piastra del pozzo è ancora attaccata al separatore magnetico.
- Lavare la piastra 2 volte (vedere il punto b nella procedura di immunodosaggio delle citochine giorno 2).
- Diluire l'anticorpo di rilevamento 20x stock in 1x soluzione in tampone di dosaggio. Aggiungere 25 μL di 1x anticorpo di rilevamento per pozzetto (Figura 6). Coprire con un foglio. Incubare per 1 ora su scuotitore a piastra (750 rpm) a temperatura ambiente.
- Posizionare la piastra da 96 pozzetti sul separatore magnetico e lasciare riposare per 2 minuti. Decantare il contenuto del pozzo, staccarsi dal separatore magnetico.
- Diluire 25x stock SAPE in tampone di analisi a 1x buffer. Aggiungere 25 μL di 1x SAPE (Figura 6). Coprire con un foglio e incubare per 15 minuti su scuotitore a piastra (750 giri / min) a temperatura ambiente.
- Lasciare il SAPE nei pozzetti e aggiungere 25 μL di tampone di amplificazione a ciascun pozzetto. Coprire con un foglio.
- Incubare per 15 minuti su scuotitore a piastre (750 giri / min) a temperatura ambiente.
- Posizionare la piastra del pozzo sul separatore magnetico per 2 minuti. Decantare il contenuto del pozzo e staccarsi dal separatore magnetico.
- Aggiungere 75 μL di Luminex Drive Fluid (se si utilizza lo strumento MAGPIX) o il buffer di analisi (se si utilizza lo strumento 3D 200 o FlexMap). Sospendere nuovamente le perline su uno shaker a piastre per 5 minuti a temperatura ambiente.
- Leggere sullo strumento Luminex (MAGPIX, 200 o FlexMap 3D), facendo riferimento al manuale dell'utente per il corretto funzionamento (Figura 6).
- Preparazione dei reagenti (giorno 1, stesso per le citochine e le fosfoproteine)
- Linearità della curva di diluizione del campione
- Preparazione dei campioni: Campioni di prova diluiti in serie con diverse concentrazioni di proteine totali. Per i tessuti cerebrali sfusi, caricare diluizioni seriali da 0-25 μg per le citochine e 0-12 μg per le fosfoproteine. La concentrazione totale di proteine può essere misurata utilizzando il test dell'acido bicinchoninico (BCA).
- Immunodosaggio multiplex: Eseguire il test Luminex (vedere paragrafo 3.2) su campioni selezionati.
- Analisi dei dati
- Tracciare l'intensità fluorescente per ogni proteina rispetto alla quantità di proteine caricate (Figura 7).
- Per ogni analita, identificare l'intervallo di proteine totali caricate per le quali la relazione tra la proteina totale e la lettura dell'intensità fluorescente è lineare (Figura 7).
- Per determinare la quantità di proteina totale che deve essere caricata per l'intero ciclo di analisi, identificare la porzione lineare della curva per ciascun analita e quindi selezionare una concentrazione proteica che rientri nell'intervallo lineare per la maggior parte degli analiti.
NOTA: Sebbene la maggior parte delle proteine condivida un intervallo lineare simile, gli intervalli lineari potrebbero non sovrapporsi per tutte le proteine. In questo caso, potrebbe essere necessario eseguire ogni campione più volte con diverse quantità di proteine totali caricate. In alternativa, i campioni non lineari possono essere esclusi dall'analisi. Inoltre, alcune proteine potrebbero non avere un intervallo lineare di sorta.
4. Regressione parziale dei minimi quadrati
NOTA: vengono forniti codice R di esempio e un foglio di calcolo dei dati di esempio per eseguire l'analisi parziale dei minimi quadrati.
- Preparazione dei dati: formattare i dati come mostrato nel foglio di calcolo dei dati di esempio fornito, "MyData". Includere i nomi delle variabili nella riga 1, i nomi di esempio nella colonna A, la variabile di risposta nella colonna B e tutte le variabili predittive nelle colonne C+. Riempire le ultime due righe con i dati di sfondo e impostare entrambi i nomi di esempio su "Sfondo".
- Regressione parziale dei minimi quadrati in RStudio
- Installa R da www.r-project.org (gratuito, open source).
- Installa RStudio Desktop da www.rstudio.com (licenza gratuita open source).
- Scaricare il codice R di esempio fornito con questa pubblicazione, "PLSR_Sample_Code.R" e salvarlo nella stessa cartella che contiene il foglio di calcolo dei dati. Aprire il file di codice in RStudio.
- Nella sezione Input utente , modificare "dataFileName" con il nome del foglio di calcolo dati.
- Eseguire i passaggi seguenti evidenziando la sezione di codice da eseguire e facendo clic su Esegui nell'angolo superiore destro dello script.
- Caricare i pacchetti R necessari, le funzioni, l'indirizzo della directory di lavoro e i valori di input dell'utente in RStudio (sottosezione "Preliminari").
- Caricare i dati in RStudio e preparare i dati grezzi per l'elaborazione sottraendo il segnale medio di fondo da tutte le misurazioni e z-scoring di ciascun analita (sottosezione "Lettura dei dati e sottrazione dello sfondo")(Figura 8A).
- Eseguire la regressione parziale dei minimi quadrati in RStudio utilizzando il pacchetto plsRglm v1.2.528 disponibile sulla Comprehensive R Archive Network (CRAN). Eseguire una rotazione varimax (pacchetto statistiche v3.6.2)23 nel piano LV1-LV2 per identificare un nuovo asse orizzontale che meglio separa i campioni in base alla variabile di risposta (sottosezione "PLS")(Figura 8B).
- Eseguire una convalida incrociata Leave One Out (LOOCV) in cui un campione viene lasciato iterativamente fuori dai dati e il modello PLSR viene ricalcolato. Calcolare la deviazione standard per i carichi di analiti su tutte le esecuzioni LOOCV (sottosezione "LOOCV").
- Crea grafici rappresentativi: Esegui il codice di esempio fornito come descritto sopra per creare grafici rappresentativi che esportano automaticamente come file PDF nella directory di lavoro (la cartella contenente i dati e i file di codice).
- Creare una mappa termica dei dati elaborati come mostrato nella Figura 8A (sottosezione "PLS"). Colora ogni voce lungo uno spettro definito da z-score. Ordina gli analiti in base all'ordine calcolato nella variabile latente di interesse.
- Creare un grafico dei punteggi con i punteggi LV1 tracciati lungo l'asse orizzontale e i punteggi LV2 tracciati lungo l'asse verticale, come illustrato nella Figura 8B (sottosezione "PLS"). Colora ogni punto dati in base alla sua misurazione della variabile di risposta per visualizzare la relazione tra ogni variabile latente e la variabile di risposta.
- Creare un grafico a barre che mostri i caricamenti per ciascuna delle variabili predittive per visualizzare in che modo ogni analita contribuisce alle variabili latenti, come illustrato nella Figura 8C (sottosezione "LOOCV").
- Creare un grafico che regredisca i punteggi LV1 rispetto alla variabile di risposta per visualizzare quanto bene il modello PLSR separa i campioni, come mostrato nella Figura 8D (sottosezione "PLS").
Risultati
I dati raccolti in precedenza sono stati presi da un lavoro precedente in cui un gruppo di otto topi C57BL / 6 sono stati sottoposti a tre lesioni alla testa chiuse (Figura 2) distanziate una volta al giorno11. In questo lavoro, il flusso sanguigno cerebrale è stato misurato con spettroscopia di correlazione diffusa 4 ore dopo l'ultima lesione (Figura 3, Figura 4). Dopo la valutazione del CBF post-lesione, g...
Discussione
Qui descriviamo in dettaglio i metodi per la valutazione della risposta emodinamica e neuroinfiammatoria a lesioni cerebrali traumatiche lievi ripetitive. Inoltre, abbiamo mostrato come integrare questi dati come parte di un'analisi di sistemi multivariati utilizzando la regressione parziale dei minimi quadrati. Nel testo seguente discuteremo alcuni dei passaggi chiave e delle limitazioni associate al protocollo, nonché i vantaggi / svantaggi dei metodi rispetto ai metodi esistenti.
M...
Divulgazioni
Nessuno.
Riconoscimenti
Questo progetto è stato sostenuto dal National Institutes of Health R21 NS104801 (EMB) e R01 NS115994 (LBW / EB) e Children's Healthcare of Atlanta Junior Faculty Focused Award (EMB). Questo lavoro è stato anche sostenuto dal Dipartimento della Difesa degli Stati Uniti attraverso i programmi di ricerca medica diretti dal Congresso sotto il premio n. W81XWH-18-1-0669 (LBW/EMB). Opinioni, interpretazioni, conclusioni e raccomandazioni sono quelle dell'autore e non sono necessariamente approvate dal Dipartimento della Difesa. Questo materiale si basa sul lavoro sostenuto dal National Science Foundation Graduate Research Fellowship Program nell'ambito della sovvenzione n. 1937971. Eventuali opinioni, risultati e conclusioni o raccomandazioni espresse in questo materiale sono quelle degli autori e non riflettono necessariamente le opinioni della National Science Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| Adjustable pipettes | any adjustable pipette | ||
| Aluminum foil | VWR | 89107-726 | |
| Bio-Plex cell lysis kit | C Bio-Rad | 171304012 | |
| BRAND BRANDplates pureGrade Microplates, Nonsterile | BrandTech | 781602 96 | |
| Complete mini protease inhibitor tablet | Sigma-Aldrich | 11836153001 | |
| Depilatory cream | Amazon | Nair | |
| DiH2O | VWR | VWRL0200-1000 | |
| Handheld magnetic separator block for 96 well flat bottom plates | Millipore Sigma Catalogue | 40-285 | |
| Hardware Autocorrelator Board | www.correlator.com | Flex05-8ch | |
| Isoflurane 250 mL | MED-VET INTERNATIONAL | RXISO-250 | |
| Kimwipe (11.2 x 21.3 cm) | VWR | 21905-026 | |
| Laboratory vortex mixer | VWR | 10153-838 | |
| LabView | National Instruments | LabVIEW | |
| Luminex 200, HTS, FLEXMAP 3D, or MAGPIX with xPONENT software | Luminex Corporation | ||
| Luminex Drive Fluid | Luminex | MPXDF-4PK | |
| Luminex sheath fluid | EMD Millipore | SHEATHFLUID | |
| MILLIPLEX MAP Mouse Cytokine/Chemokine Magnetic Bead Panel - Premixed 32 Plex - Immunology Multiplex Assay | Millipore Sigma | MCYTMAG-70K-PX32 | |
| MILLIPLEX MAPK/SAPK Signaling 10-Plex Kit-Cell Signaling Multiplex Assay | Millipore Sigma | 48-660MAG | |
| Mini LabRoller rotator | VWR | 10136-084 | |
| Phenylmethylsulfonyl fluoride | Sigma-Aldrich | P7626-1G | |
| Phosphate-buffered Saline (PBS) | VWR | 97064-158 | |
| Plate Sealer | VWR | 82050-992 | |
| Polypropylene microfuge tubes | VWR | 20901-547 | |
| Mini LabRoller | Millipore Sigma | Z674591 | |
| Reagent Reservoirs | VWR | 89094-668 | |
| R Programming Language | |||
| RStudio | www.rstudio.com | ||
| Sonicator | |||
| Titer plate shaker | VWR | 12620-926 | |
| Tween20 | Sigma-Aldrich | P9416-50ML | |
| 1 m acrylic guide tube | McMaster-Carr | 49035K85 | |
| 4 photon counting avalanche photodiode | Perkin-Elmer | SPCM-AQ4C-IO | |
| 400 um multimode source fiber | Thorlabs Inc. | FT-400-EMT | |
| 54 g bolt | Ace Hardware | 0.95 cm basic body diameter, 2 cm head diameter, 10.2 cm length | |
| 780 nm single mode detector fiber | Thorlabs Inc. | 780HP | |
| 852 nm long-coherence length laser | TOPTICA Photonics | iBeam smart |
Riferimenti
- Langlois, J. A., Rutland-Brown, W., Wald, M. M. The epidemiology and impact of traumatic brain injury: a brief overview. Journal of Head Trauma Rehabilitation. 21 (5), 375-378 (2006).
- Iraji, A., et al. Resting State Functional Connectivity in Mild Traumatic Brain Injury at the Acute Stage: Independent Component and Seed-Based Analyses. Journal of Neurotrauma. 32 (14), 1031-1045 (2015).
- Guskiewicz, K. M., et al. Cumulative effects associated with recurrent concussion in collegiate football players: the NCAA Concussion Study. Journal of the American Medical Association. 290 (19), 2549-2555 (2003).
- Longhi, L., et al. Temporal window of vulnerability to repetitive experimental concussive brain injury. Neurosurgery. 56 (2), 364-374 (2005).
- Committee on Sports-Related Concussions in Youth, Board on Children, Youth, and Families, Institute of Medicine, National Research Council. . Sports-Related Concussions in Youth: Improving the Science, Changing the Culture. , (2014).
- Barkhoudarian, G., Hovda, D. A., Giza, C. C. The Molecular Pathophysiology of Concussive Brain Injury - an Update. Physical Medicine and Rehabilitation Clinics of North America. 27 (2), 373-393 (2016).
- McCrory, P., et al. Consensus statement on concussion in sport--the 4th International Conference on Concussion in Sport held in Zurich, November 2012. Clinical Journal of Sport Medicine. 23 (2), 89-117 (2012).
- Belanger, H. G., Vanderploeg, R. D., Curtiss, G., Warden, D. L. Recent neuroimaging techniques in mild traumatic brain injury. Journal of Neuropsychiatry and Clinical Neurosciences. 19 (1), 5-20 (2007).
- Sours, C., Zhuo, J., Roys, S., Shanmuganathan, K., Gullapalli, R. P. Disruptions in Resting State Functional Connectivity and Cerebral Blood Flow in Mild Traumatic Brain Injury Patients. PLoS ONE. 10 (8), 0134019 (2015).
- Buckley, E. M., et al. Decreased Microvascular Cerebral Blood Flow Assessed by Diffuse Correlation Spectroscopy after Repetitive Concussions in Mice. Journal of Cerebral Blood Flow & Metabolism. 35 (12), 1995-2000 (2015).
- Sankar, S. B., et al. Low cerebral blood flow is a non-invasive biomarker of neuroinflammation after repetitive mild traumatic brain injury. Neurobiology of Disease. 124, 544-554 (2019).
- Vagnozzi, R., et al. Temporal window of metabolic brain vulnerability to concussions: mitochondrial-related impairment--part I. Neurosurgery. 61, 379-388 (2007).
- Longhi, L., et al. Temporal window of vulnerability to repetitive experimental concussive brain injury. Neurosurgery. 56, 364-374 (2005).
- Fujita, M., Wei, E. P., Povlishock, J. T. Intensity- and interval-specific repetitive traumatic brain injury can evoke both axonal and microvascular damage. Journal of Neurotrauma. 29, 2172-2180 (2012).
- Angoa-Perez, M., et al. Animal models of sports-related head injury: bridging the gap between preclinical research and clinical reality. Journal of Neurochemistry. 129, 916-931 (2014).
- Prins, M. L., Hales, A., Reger, M., Giza, C. C., Hovda, D. A. Repeat traumatic brain injury in the juvenile rat is associated with increased axonal injury and cognitive impairments. Developmental Neuroscience. 32, 510-518 (2010).
- Viano, D. C., Hamberger, A., Bolouri, H., Saljo, A. Concussion in professional football: animal model of brain injury--part 15. Neurosurgery. 64, 1162-1173 (2009).
- Kane, M. J., et al. A mouse model of human repetitive mild traumatic brain injury. Journal of Neuroscience Methods. 203, 41-49 (2012).
- Meehan, W. P., Zhang, J., Mannix, R., Whalen, M. J. Increasing Recovery Time Between Injuries Improves Cognitive Outcome After Repetitive Mild Concussive Brain Injuries in Mice. Neurosurgery. 71 (4), 885-892 (2012).
- Durduran, T., Yodh, A. G. Diffuse correlation spectroscopy for non-invasive, micro-vascular cerebral blood flow measurement. NeuroImage. 85, 51-63 (2014).
- Sathialingam, E., et al. Small separation diffuse correlation spectroscopy for measurement of cerebral blood flow in rodents. Biomedical Optics Express. 9 (11), 5719 (2018).
- Lee, S. Y., et al. Noninvasive optical assessment of resting-state cerebral blood flow in children with sickle cell disease. Neurophotonics. 6 (03), 1 (2019).
- Wang, H., Liu, Q., Tu, Y. Interpretation of partial least-squares regression models with VARIMAX rotation. Partial Least Squares. 48 (1), 207-219 (2005).
- Eriksson, L., Byrne, T., Johansson, E., Trygg, J., Vikström, C. Multi- and Megavariate Data Analysis Basic Principles and Applications. Umetrics Academy. , (2013).
- Conzen, P. F., et al. Systemic and regional hemodynamics of isoflurane and sevoflurane in rats. Anesthesia and Analgesia. 74 (1), 79-88 (1992).
- Durduran, T., Choe, R., Baker, W. B., Yodh, A. G. Diffuse optics for tissue monitoring and tomography. Reports on Progress in Physics. 73 (7), 076701 (2010).
- Lee, S. Y., et al. Small separation frequency-domain near-infrared spectroscopy for the recovery of tissue optical properties at millimeter depths. Biomedical Optics Express. 10 (10), 5362-5377 (2019).
- . plsRglm: Partial Least Squares Regression for Generalized Linear Models Available from: https://CRAN.R-project.org/package=pplsRglm (2019)
- White, B. R., Bauer, A. Q., Snyder, A. Z., Schlaggar, B. L., Lee, J. M., Culver, J. P. Imaging of functional connectivity in the mouse brain. PLoS One. 6, 16322 (2011).
- Buckley, E. M., Parthasarathy, A. B., Grant, P. E., Yodh, A. G., Franceschini, M. A. Diffuse correlation spectroscopy for measurement of cerebral blood flow: future prospects. Neurophotonics. 1 (1), 011009 (2014).
- Rowan, O., et al. Cerebrovascular reactivity measured in awake mice using diffuse correlation spectroscopy. Neurophotonics. 8 (1), (2021).
- Tate, J., Ward, G. Interferences in immunoassay. The Clinical Biochemist. Reviews. 25 (2), 105-120 (2004).
- Staples, E., Ingram, R. J. M., Atherton, J. C., Robinson, K. Optimising the quantification of cytokines present at low concentrations in small human mucosal tissue samples using Luminex assays. Journal of Immunological Methods. 394 (1-2), 1-9 (2013).
- Gierut, J. J., et al. Network-level effects of kinase inhibitors modulate TNF-α-induced apoptosis in the intestinal epithelium. Science Signaling. 8 (407), 129 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon