Method Article
Utilizzo del cervello dell'embrione di pulcino come modello per analisi in vivo ed ex vivo del comportamento delle cellule di glioblastoma umano
In questo articolo
Riepilogo
Gli embrioni di pulcino sono utilizzati per studiare i tumori cerebrali del glioblastoma umano (GBM) in ovo e in co-colture di fette cerebrali ex vivo. Il comportamento delle cellule GBM può essere registrato mediante microscopia time-lapse in co-colture ex vivo, ed entrambi i preparati possono essere analizzati all'endpoint sperimentale mediante analisi confocale 3D dettagliata.
Abstract
L'embrione di pulcino è stato un sistema modello ideale per lo studio dello sviluppo dei vertebrati, in particolare per le manipolazioni sperimentali. L'uso dell'embrione di pulcino è stato esteso per studiare la formazione di tumori cerebrali di glioblastoma umano (GBM) in vivo e l'invasività delle cellule tumorali nel tessuto cerebrale circostante. I tumori GBM possono essere formati mediante iniezione di una sospensione di cellule marcate fluorescentmente nel ventricolo del mesencefalo E5 (tectum ottico) in ovo.
A seconda delle cellule GBM, i tumori compatti si formano casualmente nel ventricolo e all'interno della parete cerebrale e gruppi di cellule invadono il tessuto della parete cerebrale. Sezioni di tessuto spesso (350 μm) di tecta fissa E15 con tumori possono essere immunocolorate per rivelare che le cellule invasori spesso migrano lungo i vasi sanguigni quando analizzate dalla ricostruzione 3D di immagini confocali z-stack. Le fette vive di mesencefalo e proencefalo E15 (250-350 μm) possono essere coltivate su inserti di membrana, dove le cellule GBM marcate con fluorescenza possono essere introdotte in posizioni non casuali per fornire co-colture ex vivo per analizzare l'invasione cellulare, che può anche verificarsi lungo i vasi sanguigni, per un periodo di circa 1 settimana. Queste co-colture ex vivo possono essere monitorate mediante microscopia time-lapse a fluorescenza a campo largo o confocale per osservare il comportamento delle cellule vive.
Le fette co-coltivate possono quindi essere fissate, immunocolorate e analizzate al microscopio confocale per determinare se l'invasione si è verificata o meno lungo i vasi sanguigni o gli assoni. Inoltre, il sistema di co-coltura può essere utilizzato per studiare potenziali interazioni cellula-cellula posizionando aggregati di diversi tipi e colori cellulari in diverse posizioni precise e osservando i movimenti cellulari. I trattamenti farmacologici possono essere eseguiti su colture ex vivo , mentre questi trattamenti non sono compatibili con il sistema in ovo . Questi due approcci complementari consentono analisi dettagliate e precise del comportamento delle cellule GBM umane e della formazione del tumore in un ambiente cerebrale vertebrato altamente manipolabile.
Introduzione
Gli studi in vitro sui comportamenti delle cellule tumorali sono spesso utilizzati per analizzare i potenziali meccanismi che operano durante il comportamento più complesso che si osserva durante la formazione del tumore e l'invasione cellulare nei modelli di xenotrapianto in vivo. Ad esempio, con il glioblastoma (GBM), studi in vitro hanno scoperto meccanismi di come L1CAM opera potenzialmente durante la formazione del tumore e l'invasione cerebrale in un nuovo modello di tumore cerebrale xenotrapianto di embrione di pulcino 1,2,3,4,5. Sebbene gli esperimenti in vitro e in vivo si completino a vicenda in modi utili, lasciano una lacuna sostanziale nel modo in cui i risultati possono essere correlati. Ad esempio, le analisi meccanicistiche della motilità delle cellule GBM su un piatto sono una situazione altamente artificiale e i modelli di xenotrapianto in vivo possono rivelare solo analisi statiche del punto temporale o dell'endpoint della formazione del tumore e del comportamento cellulare. Gli studi in vivo che utilizzano embrioni di roditori o pulcini non si prestano facilmente al monitoraggio del comportamento cellulare mentre le cellule invadono il tessuto cerebrale in questi modelli di xenotrapianto. Tuttavia, il modello di xenotrapianto di embrioni di pulcino ha dimostrato che la proteina di adesione L1CAM svolge un ruolo stimolante nella capacità invasiva delle cellule umane T98G GBM 2,5.
Una soluzione adeguata a questo problema può essere raggiunta collegando metodi sia in vivo che in vitro utilizzando un modello organotipico di coltura di fette di cervello, indicato come modello ex vivo . In questo modello ex vivo, il tessuto cerebrale vivo può essere mantenuto a uno spessore di diverse centinaia di micron per un massimo di alcune settimane, rendendo possibile l'impianto di cellule tumorali, osservare il loro comportamento nel tessuto reale nel tempo e quindi eseguire un'analisi dei marcatori più dettagliata all'endpoint dell'esperimento.
Un popolare metodo di coltura organotipica è stato quello di coltivare una fetta di cervello spessa diverse centinaia di micron sopra una membrana porosa traslucida o trasparente, lasciando il tessuto esposto all'aria, ma consentendo ai mezzi nutritivi di sostenere il tessuto da sotto la membrana (fare riferimento a Stoppini et al.6). Diverse varianti di questo metodo sono state utilizzate per diversi studi, incluso l'utilizzo di diversi mezzi o diversi inserti di membrana. Diversi inserti di membrana includono un inserto a membrana poroso (0,4 μm) di diametro 30 mm in un piatto di coltura da 35 mm 6 e inserti di coltura cellulare (0,4 μm) per piastre a6 pozzetti7. Diversi mezzi includono 50% MEM / HEPES + 25% siero di cavallo inattivato dal calore + 25% di soluzione salina bilanciata di Hanks (HBSS)8, 50% di media sierici ridotti + 25% di siero di cavallo + 25% di HBSS9, così come altri. Se viene utilizzata una membrana traslucida o trasparente insieme a cellule GBM marcate con fluorescenza, tali colture possono essere visualizzate dal basso utilizzando un microscopio a fluorescenza a campo largo invertito o confocale 10,11,12,13,14,15.
Mentre molti modelli di xenotrapianto di tumore cerebrale ortotopico in vivo e di coltura di fette di cervello organotipiche ex vivo sono stati stabiliti utilizzando roditori, come citato sopra, l'embrione di pulcino (Gallus gallus) è stato sottoutilizzato per questi scopi. Tuttavia, è stato dimostrato che l'embrione di pulcino può essere utilizzato come modello di xenotrapianto ortotopico in vivo per lo studio dell'invasione del glioma sia umano che di ratto 1,2,5. Le cellule xenotrapiantate nel cervello di embrioni di pulcino hanno mostrato modelli di invasione simili a quelli osservati nei modelli di roditori, supportando ulteriormente l'uso di embrioni di pulcino come modello in vivo per l'analisi delle cellule tumorali GBM. Gli embrioni di pulcino sono anche poco costosi, possono essere mantenuti più facilmente dei roditori (cioè nei loro gusci d'uovo in un incubatore di laboratorio) e sono molto più facili da lavorare, rendendoli un'opzione attraente per studi GBM in vivo a breve termine. Un recente articolo ha descritto l'uso di colture di fette di cervello di embrione di pulcino per la formazione e la crescita degli assoni durante il normale sviluppo del cervello in cui le fette erano vitali per almeno 7 giorni16. Tuttavia, manca l'uso di tali colture di fette di cervello di embrione di pulcino per l'analisi ex vivo del comportamento delle cellule GBM in un ambiente tissutale. In questo articolo, sono descritti sia il trapianto di cellule GBM umane e cellule staminali GBM (GSC) nel cervello precoce dell'embrione di pulcino in vivo, sia l'introduzione di cellule GBM su colture di cellule di cervello di embrioni di pulcino vivo ex vivo. Vengono inoltre forniti alcuni esempi rappresentativi dei tumori risultanti e dei modelli di invasione cellulare ottenuti da questi preparati.
Protocollo
Non era necessaria alcuna autorizzazione o approvazione presso l'Università del Delaware per svolgere questo lavoro.
1. Iniezione di cellule GBM nel tectum ottico del pulcino
- Preparazione di GSC e cellule GBM per l'iniezione
- Cultura SGC nei media dell'SGC (tabella 1). Linee cellulari GBM stabilite dalla coltura nei terreni GBM (Tabella 1).
- Risciacquare le cellule su piastre con soluzione salina tamponata con fosfato sterile (PBS) e introdurre 1 mL di soluzione di tripsina in un piatto da 10 cm. Lasciare in un incubatore di coltura cellulare per 2-3 minuti fino a quando le cellule iniziano a staccarsi.
- Inattivare la tripsina aggiungendo 10 ml di terreno di coltura appropriato contenente siero nel piatto da 10 cm e staccare le cellule pipettando su e giù. Inserire la sospensione cellulare in una provetta conica da centrifuga da 15 ml.
- Pellettare le celle mediante centrifugazione a 800 × g per 5 minuti a 4 °C.
- Aspirare il fluido dal pellet cellulare utilizzando una pipetta di vetro fine monouso collegata a un pallone a braccio laterale e a una pompa per vuoto. Risospendere le cellule in terreni di crescita appropriati ad una concentrazione di 10.000 cellule per microlitro e metterle in un tubo di microfuge o in un piccolo tubo a vite sul ghiaccio.
- Mescolare le cellule con una piccola quantità di colorante FCF Fast Green sterile all'1% (utilizzare un rapporto di 5 μL di colorante / 100 μL di sospensione cellulare).
- Iniezione di cellule GBM nel tectum ottico E5 in ovo. Cfr. figura supplementare 1.
- Incubare le uova di gallina fecondate in un'incubatrice umidificata, con l'estremità appuntita rivolta verso il basso, a 37,5 °C (il 1° giorno di incubazione è embrionale giorno 0 [E0]). Il 6 ° giorno di incubazione (E5), sterilizzare il guscio d'uovo spruzzando con etanolo al 70%.
- Usando un candelabro per uova, traccia lungo il perimetro dello spazio aereo sopra l'embrione con una matita e copri l'area delineata con nastro trasparente.
- Usando le forbici curve, tagliare delicatamente intorno all'area tracciata, facendo attenzione a non tagliare le membrane embrionali o i vasi sanguigni e scartare la parte superiore del guscio d'uovo.
- Posizionare alcune gocce di terreno salino o di coltura cellulare sulla membrana dello spazio aereo per bagnarla in modo che si stacchi facilmente. Usando una pinza fine, perforare con cura la membrana dello spazio aereo sopra la parte superiore dell'embrione, rimuoverla e individuare la testa dell'embrione di pulcino.
- Utilizzare una pinza fine per afferrare la membrana amnionica trasparente che circonda immediatamente l'embrione per posizionare la testa in modo che il tectum ottico possa essere iniettato con le cellule. Con una mano, utilizzare la pinza fine per trattenere l'amnion per mantenere la testa in posizione durante il processo di iniezione. Fare attenzione a non danneggiare i vasi sanguigni extraembrionali nella membrana corioallantoica sul tuorlo.
NOTA: La procedura di cui sopra richiede un po 'di pratica per essere in grado di afferrare in modo efficiente la membrana amnionica trasparente che circonda intimamente l'embrione, poiché è invisibile fino a quando non viene afferrata e tirata. Esercitati con una pinza fine per afferrare l'amnione chiaro che circonda immediatamente l'embrione. - Tenere ferma la testa afferrando la membrana amnionica con una pinza fine. Utilizzando una micropipetta di vetro e una picopompa pneumatica, iniettare circa 50.000 cellule in 5 μL di terreno di coltura cellulare adatto nel tectum ottico (5 μL sono circa 1/2 di una micropipetta riempita).
NOTA: Vedi Cretu et al.1 per un'immagine di un embrione E5 che è stato iniettato con cellule GBM mescolate con colorante. - Mettere alcune gocce di 50 mg/ml di ampicillina sopra l'embrione.
- Coprire il foro nella parte superiore delle uova con nastro trasparente e lasciare in umidificatore fino a E15 per la dissezione.
2. Dissezione delle regioni cerebrali dagli embrioni E15
NOTA: La dissezione dei cervelli E15 qui per la fissazione è simile a quella descritta nel passaggio 8.2 per le fette di cervello vive, ma la dissezione qui non deve essere eseguita in condizioni asettiche.
- Tagliare via il nastro intorno alla parte superiore del guscio, consentendo l'accesso all'embrione.
- Decapitare l'embrione al collo e posizionare la testa in un piatto da 10 cm con soluzione sterile di Tyrodes priva di calcio e magnesio (CMF Tyrode). Cfr . figura supplementare 2.
- Usando una pinza sottile, staccare la pelle che copre il cervello per rivelare la dura madre sottostante. Rimuovere le ossa del cranio in formazione sui lati sinistro e destro del cervello. Le ossa del cranio in formazione non coprono ancora la maggior parte del cervello.
- Usa delicatamente una pinza appuntita fine per strappare le meningi sovrastanti il centro del cervello e sbucciarla su ciascun lato per scoprire il cervello.
- Usa una pinza curva per raccogliere il cervello dal basso e tirarlo delicatamente fuori dalla sua cavità.
- Seziona il cervello nelle sue parti: proencefalo, tecta, cervelletto.
- Rimuovere la pia madre sovrastante dalla tecta usando una pinza fine. Si noti che la pia si separa facilmente dalla tecta, ma non dal proencefalo o dal cervelletto. Rimuovere la pia dal proencefalo toccandola o arrotolandola su un piccolo pezzo di carta da filtro.
- Metti le regioni cerebrali sezionate in un piccolo piatto.
- Posizionare le regioni cerebrali per la fissazione in un pozzetto a 24 pozzetti e fissare in paraformaldeide (PFA) al 2% in tampone di cacodilato di sodio 0,1 M. Lasciare agire per almeno 24 ore a 4 °C.
- Dopo la fissazione, sciacquare il tessuto in 3x PBS per almeno 1 ora prima di incorporarlo in agar.
3. Incorporare e affettare le regioni cerebrali raccolte
- Preriscaldare una soluzione di agar al 3,5% e saccarosio all'8% in PBS fino a fusione e mantenerla alla temperatura di fusione.
- Usando una pinza curva come paletta, raccogliere delicatamente il tectum ottico o la regione del proencefalo del cervello dell'embrione di pulcino e asciugare leggermente sulla carta da filtro per rimuovere il liquido in eccesso per garantire l'adesione dell'agar alla superficie esterna del cervello.
- Utilizzando una pipetta di trasferimento sterile, riempire lo stampo con una soluzione di agar.
NOTA: Un semplice stampo può essere realizzato formando un foglio di alluminio attorno a un oggetto di dimensioni appropriate, come piccoli blocchi rettangolari di affettatrice di tessuto vibrante in metallo. - Metti la regione del cervello in agar e lascia che l'agar si solidifichi completamente.
- Rimuovere il foglio di alluminio intorno al cervello incorporato e tagliare l'agar in eccesso intorno ai lati usando una lama di rasoio o un bisturi.
- Posizionare una goccia di colla cianoacrilica sul quadrato in acciaio inossidabile del vassoio di affettatura, posizionare il blocco di agarosio con il cervello sulla colla e lasciare che la colla si leghi per 1 minuto. Posizionare il vassoio nel mandrino dell'affettatrice di tessuto vibrante, stringere e riempire il vassoio con abbastanza PBS da coprire la parte superiore del blocco di agar.
- Tagliare le fette di cervello su un'affettatrice di tessuto vibrante con uno spessore di 350 μm usando una lama di rasoio d'acciaio.
- Mentre le fette di cervello vengono tagliate e galleggiano liberamente nel vassoio di affettatura, utilizzare una spatola per raccogliere e rimuovere le fette dal vassoio. Far scivolare delicatamente la sezione dalla spatola e in una capsula di Petri etichettata da 10 cm con PBS in modo che le sezioni fluttuino liberamente.
NOTA: L'agar che circonda la fetta di cervello può staccarsi se il cervello non è sufficientemente tamponato con carta da filtro per rimuovere il liquido in eccesso prima dell'incorporamento. Se ciò si verifica, il tessuto cerebrale può essere rimosso delicatamente dall'agar solidificato e reincorporato dopo una corretta asciugatura del liquido in eccesso.
4. Immunocolorazione delle fette di cervello con cellule tumorali
- Utilizzando uno stereomicroscopio dotato di epifluorescenza, schermare le singole fette nel piatto da 10 cm una per una per la presenza o l'assenza di cellule tumorali.
- Preparare una quantità sufficiente di soluzione salina tamponata fosfato con Triton X-100 allo 0,1% e siero di capra normale al 5% (PBSTG) (vedere Tabella 1) per l'immunocolorazione e sciacquare le fette da immunocolorare.
- Pozzetti di riempimento a metà in una piastra da 24 pozzetti che verrà utilizzata per colorare sezioni cerebrali con PBS.
- Tagliare gli angoli di agar intorno alle fette del cervello con il bordo di una spatola o di un bisturi e posizionarli delicatamente nei pozzetti contenenti PBS.
- Aspirare il PBS e sostituirlo con 350 μL di soluzione di colorazione anticorpale primaria in PBSTG (ad esempio, 2 μg/mL UJ127 in PBSTG).
- Incubare per 24 ore in una cella frigorifera agitando delicatamente in modo che la fetta si muova liberamente all'interno del pozzetto.
- Dopo 24 ore, rimuovere la soluzione anticorpale primaria e risciacquare 3 x 1 h con PBSTG in una cella frigorifera agitando.
- Al termine del risciacquo, incubare in 350 μL/pozzetto di soluzione di colorazione anticorpale secondaria in PBSTG (ad esempio, 1/200 di diluizione di biotina-GAM in PBSTG). Incubare per 20 ore in una cella frigorifera con agitazione.
NOTA: Se si rinuncia alla fase terziaria e si incuba con l'anticorpo secondario contenente fluorocromo in questa fase, coprire per proteggere dalla luce durante l'incubazione di tanto in tanto e quindi saltare al punto 4.11. - Rimuovere la soluzione anticorpale secondaria e risciacquare 3 x 1 h in PBSTG in una cella frigorifera agitando.
- Rimuovere il PBSTG e incubare nella soluzione terziaria (ad esempio, diluizione 1:250 di Alexa Fluor 647 streptavidina in PBSTG). Se lo si desidera, colorare i nuclei in questa fase aggiungendo 0,1 μg/ml di bisbenzimmide alla miscela. Incubare per 20 ore in una cella frigorifera con agitazione.
- Rimuovere la soluzione terziaria e risciacquare 3 x 1 h in PBSTG in una cella frigorifera agitando.
- Lasciare in PBS fino al momento del montaggio su vetrini da microscopio.
5. Montaggio di fette su vetrini da microscopio
- Prepara tanti vetrini da microscopio quante sono le sezioni da montare.
- Per ogni vetrino, posizionare una striscia di 50 mm di nastro elettrotecnico in vinile di 10 mil (254 μm) di spessore su un pezzo di parafilm.
- Utilizzando un foro quadrato di 1 cm x 1 cm, perforare un foro attraverso il centro del nastro isolante e del parafilm.
- Estrarre il nastro dal parafilm e posizionarlo sopra il vetrino del microscopio, lasciando spazio per il nastro adesivo sul vetrino.
- Utilizzando una micropipetta, posizionare una o due gocce di supporto di montaggio anti-sbiadimento nel foro quadrato al centro del nastro isolante.
- Utilizzare una spatola curva per sollevare la sezione dell'affettatrice di tessuto vibrante desiderata dal PBSTG e rimuovere accuratamente l'umidità con una salvietta da laboratorio pulita o un pezzo di carta da filtro.
- Toccare il bordo della fetta fino alla goccia di montante e utilizzare un'altra spatola per far scorrere delicatamente la sezione nel supporto di montaggio.
- Coprire la sezione con qualche altra goccia di montante e posizionare con attenzione un coprislip di 24 mm x 30 mm (# 1,5 spessore) sopra la sezione e il montante.
- Sigillare i bordi della copertina con lo smalto per unghie per tenerlo in posizione.
6. Microscopia confocale di fette cerebrali fisse
- Utilizzare la fluorescenza a campo largo per trovare tumori fluorescenti nella fetta di cervello montata utilizzando l'obiettivo appropriato e i set di filtri.
NOTA: Alcuni tumori possono essere abbastanza grandi e sono facilmente visibili con l'obiettivo 4x, mentre le singole cellule potrebbero richiedere l'obiettivo 10x. - Passare alla microscopia confocale utilizzando una lente obiettiva, uno o più laser, dimensioni stenopeiche, e le impostazioni del rilevatore. Per seguire questo protocollo, utilizzare un obiettivo 20x (apertura numerica [N.A.] = 0,75) per l'imaging di routine e un obiettivo olio 60x (N.A. = 1,40) per l'imaging ad alta risoluzione.
- Impostare i limiti superiore e inferiore dell'asse z e la dimensione del passo (per la situazione specifica secondo le istruzioni del produttore del microscopio confocale) per l'acquisizione di sezioni ottiche. Acquisisci uno z-stack di sezioni ottiche.
- Utilizzare il software del microscopio confocale per creare un rendering del volume 3D del tumore, secondo le istruzioni del produttore.
7. Preparazione sferoidale
- Creazione di lastre di poli(2-idrossietilmetacrilato) (poli-HEMA)
- Creare una soluzione di 10 μg/mL di poli-HEMA in etanolo al 95% e rivestire piastre di Petri da 35 mm (o piastre di coltura cellulare) con 1 mL di questa soluzione.
- Lasciare che i piatti si adagino su un bilanciere scoperto durante la notte a temperatura ambiente per sviluppare un rivestimento della superficie del piatto.
NOTA: Il solvente evaporerà e lascerà un rivestimento traslucido sul piatto, che potrebbe apparire irregolare, ma ciò non influirà sulla capacità di produrre sferoidi cellulari. - Dopo l'essiccazione, sterilizzare i piatti aperti sotto la luce UV in un armadio di biosicurezza per 1 ora. Sostituire i coperchi dopo la sterilizzazione. I piatti rivestiti sono ora pronti per l'uso.
- Colorazione fluorescente DiD per microscopia time-lapse
NOTA: Questa sezione è per la colorazione di singole cellule con colorante fluorescente DiD da utilizzare per produrre sferoidi, che ottimizza la visualizzazione della motilità cellulare per l'imaging time-lapse dal vivo.- Sospendi le cellule in un terreno di coltura privo di siero.
- Aggiungere 5 μL di stock di DiD/mL di sospensione cellulare e mescolare delicatamente mediante pipettaggio. Incubare per 20 min a 37 °C.
- Centrifugare la sospensione cellulare marcata a 800 × g a 5 °C per 5 minuti.
- Aspirare il surnatante e risospendere le cellule in mezzi caldi per risciacquare.
- Ripetere questa centrifugazione e risciacquare altre due volte.
NOTA: se lo si desidera, saltare al passaggio 7.3.6 per creare sferoidi immediatamente dopo questo processo.
- Fare sferoidi cellulari
- Riscaldare la soluzione di tripsina/acido etilendiamminotetraacetico (EDTA) allo 0,25% a 37 °C.
- GSC di coltura in terreni GSC17 (tabella 1) e cellule gloima maligne (MG) U-118 in terreno di coltura U-118 (cfr. tabella 1). Utilizzare un piatto confluente di 10 cm per la preparazione di una piastra di sferoidi da 35 mm. Aggiungere i fattori di crescita bFGF (concentrazione finale di 10 ng/ml) e TGF-α (concentrazione finale di 20 ng/ml) alle GSC inizialmente e poi ogni 3 giorni.
NOTA: Le cellule utilizzate nel presente studio erano GSC15-2/K72 verdi, GSC16-4/K72 verdi, U-118/L1LE/mCherry2x rosse e U-118/1879/mCherry2x rosse. Una piastra di sferoidi dovrebbe essere sufficiente per due piastre a 6 pozzetti di fette di cervello su inserti di membrana. - Risciacquare le cellule sulle piastre con PBS sterile e posizionare 1 mL di soluzione di tripsina su una capsula da 10 cm. Mettere nell'incubatore di coltura cellulare per 2-3 minuti fino a quando le cellule iniziano a staccarsi.
- Inattivare la tripsina aggiungendo 10 ml di terreno di coltura appropriato contenente siero nel piatto da 10 cm e staccare le cellule pipettando su e giù. Inserire la sospensione cellulare in una provetta conica da centrifuga da 15 ml.
- Pellettare le celle mediante centrifugazione a 800 × g per 5 minuti a 4 °C.
- Aspirare il mezzo dal pellet cellulare e risospendere le cellule in 10 ml di supporto.
- Posizionare 2 mL di sospensione cellulare su ciascun piatto rivestito in poliHEMA da 35 mm e aggiungere altri 2 ml di supporto appropriato per ottenere 4 mL di supporto totale per piatto. Se utilizzi le GSC, aggiungi i fattori di crescita.
- Incubare le cellule in un incubatore cellulare fino a quando gli aggregati raggiungono una dimensione di 100-200 μm, che potrebbe essere di 1-2 giorni a seconda della densità delle cellule che sono state placcate.
8. Dissezione del cervello dell'embrione di pulcino vivo e affettatrice di tessuto vibrante
- Preparazione per la dissezione
- Preparare una piastra di inserto a membrana di poliestere a 6 pozzetti con 1 ml di terreno di coltura a fetta di cervello (vedere Tabella 1) sotto l'inserto della membrana.
- Sterilizzare l'area di lavoro e gli strumenti con etanolo al 70%.
- Posizionare la piastra di inserimento a 6 pozzetti sul ghiaccio mentre è in corso la dissezione.
- Preparare 100 ml di mezzo vibrante per affettare l'affettatrice di tessuto (Tabella 1) e metterlo sul ghiaccio.
- Introdurre un flaconcino di agarosio a basso contenuto di fusione al 4% in PBS a bagnomaria fino a quando non si scioglie in un liquido (circa 50 °C).
- Riempire la vasca dell'affettatrice di tessuto vibrante con ghiaccio.
- Dissezione asettica del cervello dell'embrione di pulcino E14/15
- Usando un candelabro per uova, tracciare lungo il perimetro della sacca d'aria sopra l'embrione E14 o E15 con una matita e coprire l'area delineata con nastro trasparente.
- Usando forbici curve o sottili, tagliare delicatamente intorno all'area tracciata, facendo attenzione a non tagliare la membrana embrionale o i vasi sanguigni e scartare il pezzo superiore del guscio.
- Utilizzando una pinza curva, rimuovere la membrana dello spazio aereo sopra la parte superiore dell'embrione e individuare la testa dell'embrione di pulcino.
- Decapitare l'embrione e posizionare la testa in un piatto da 10 cm con soluzione fredda sterile di CMF. Cfr . figura supplementare 2.
- Usando una pinza fine sterile, staccare la pelle che copre il cervello per rivelare la dura madre sottostante. Rimuovere le ossa del cranio in formazione sui lati sinistro e destro del cervello. Le ossa del cranio in formazione non coprono ancora la maggior parte del cervello.
- Usa delicatamente una pinza appuntita fine (# 5 o # 55) per strappare le meningi che sovrastano il centro del cervello e sbucciarlo su ciascun lato per scoprire il cervello.
- Usando una pinza curva fine, raccogli il cervello dalla parte anteriore inferiore e tiralo delicatamente fuori dalla sua cavità.
- Seziona il cervello nelle sue tre parti principali: proencefalo, mesencefalo (ottica tecta), cervelletto. Consultare un atlante dello sviluppo dei pulcini, se necessario.
- Rimuovere la pia madre sovrastante dalla tecta usando una pinza fine.
NOTA: La pia si separa facilmente dalla tecta, ma non dal proencefalo o dal cervelletto. La pia può essere rimossa dal proencefalo toccandola o arrotolandola su una garza sterile. - Posizionare le regioni cerebrali sezionate in un piccolo piatto sterile sul ghiaccio.
- Incorporare e affettare il cervello
- Usando una pinza curva come paletta, raccogliere delicatamente il tectum ottico o la regione del proencefalo e asciugare leggermente sulla garza sterile per rimuovere il liquido in eccesso per garantire l'adesione dell'agarosio alla superficie esterna del cervello.
- Utilizzando una pipetta di trasferimento sterile, riempire lo stampo con agarosio a basso punto di fusione. Preparare uno stampo semplice formando un foglio di alluminio attorno a un oggetto di dimensioni appropriate. Il piccolo blocco rettangolare di affettatrice di tessuto vibrante in metallo viene abitualmente utilizzato come oggetto. Cfr. figura supplementare 3.
- Posizionare rapidamente la regione del cervello in agarosio e lasciarla solidificare (circa 4-5 minuti) sul ghiaccio.
NOTA: Il cervello può affondare sul fondo dello stampo prima che l'agarosio si indurisca. Se ciò si verifica, attendere 1 minuto per lasciare che l'agar inizi a tramontare, quindi posizionare la regione del cervello nell'agarosio. Prova a sospendere la regione del cervello direttamente nel mezzo dell'agarosio. - Rimuovere il foglio di alluminio da intorno al cervello incorporato nell'agarosio solidificato e tagliare l'agarosio in eccesso intorno ai lati usando una lama di rasoio sterile o un bisturi.
- Posizionare una goccia di colla cianoacrilica sul quadrato in acciaio inossidabile del piatto / vassoio di affettatura, posizionare il blocco di agarosio con il cervello e lasciare che la colla si leviga per 1 minuto. Posizionare il piatto/vassoio nel mandrino dell'affettatrice di tessuto vibrante, stringere e riempire il vassoio con un mezzo di taglio per coprire la parte superiore del blocco di agarosio.
- Tagliare le sezioni su un'affettatrice di tessuto vibrante a 250-350 μm usando un coltello zaffiro, che è stato segnalato per causare meno danni ai tessuti vivi rispetto a una lama di rasoio d'acciaio.
- Mentre le fette di cervello vengono tagliate e galleggiano liberamente nel vassoio di affettatura, utilizzare una spatola sterile per raccogliere e rimuovere la fetta dal vassoio. Far scorrere delicatamente la sezione dalla spatola e su un inserto di membrana usando un'altra spatola sterile.
NOTA: Normalmente, due o tre fette di cervello possono essere posizionate su ciascun inserto di membrana, se lo si desidera. L'agarosio che circonda la fetta di cervello può staccarsi se il cervello non è sufficientemente tamponato con una garza sterile per rimuovere il liquido in eccesso. Se ciò si verifica ancora dopo una sufficiente tamponatura prima dell'incorporazione, raccogliere delicatamente la fetta senza l'agarosio circostante e farla scorrere sull'inserto della membrana. - Posizionare la piastra a 6 pozzetti di fette di cervello sugli inserti di membrana nell'incubatore di coltura cellulare a 37 °C e al 5% di CO2.
- Il giorno dopo la placcatura, utilizzare una pipetta Pasteur sterile per aspirare il fluido da sotto l'inserto (ci sono spazi vuoti nei lati degli inserti per consentire a una pipetta di accedere al supporto sottostante). Aggiungere 1 mL di mezzo fresco per fette a ciascun pozzetto sotto l'inserto della membrana. Continua a cambiare media a giorni alterni in seguito.
- Attendi qualche giorno affinché le fette di cervello si attacchino saldamente agli inserti della membrana e sembrino appiattirsi un po '. Questo è un segno che le fette sono vitali e pronte per l'introduzione delle celle GBM.
9. Introduzione delle cellule GBM sulle fette di cervello
- Metodo sferoidale
- Dopo che gli sferoidi cellulari hanno raggiunto 150-200 μm di dimensione, utilizzare una micropipetta da 20 μL impostata su 5 μL per rimuovere da uno a più sferoidi dal loro piatto di coltura. Cfr. figura supplementare 3.
- Espellere delicatamente il supporto con sferoidi sulla fetta di cervello desiderata.
NOTA: gli sferoidi cellulari devono essere visibili nella punta della pipetta. L'uso di una punta per pipetta trasparente li renderà più facili da vedere nella punta. Se lo sferoide cade dalla fetta di cervello quando il liquido viene rilasciato, è necessario utilizzare una ciglia sterilizzata con etanolo incollata a un sottile bastoncino applicatore di legno per spingere delicatamente lo sferoide sulla fetta di cervello. - Lasciare che gli sferoidi si coltivino sulle fette del cervello per 2-5 giorni.
NOTA: La limitazione qui sembra essere l'eventuale degradazione dei vasi sanguigni e delle cellule cerebrali nella fetta. I vasi sanguigni degradati appariranno come palline discontinue nella fetta quando macchiati per la laminina.
- Metodo di punzonatura bioptica
- Lasciare che le fette di cervello si attacchino sembrando appiattirsi sull'inserto della membrana (potrebbero essere necessari 2-5 giorni in coltura).
- Scongelare la matrice cellulare sul ghiaccio.
- Nell'armadio di biosicurezza della coltura cellulare, collegare un punch sterile per biopsia di 1 mm di diametro a un tubo aspiratore a vuoto.
- Toccare delicatamente la fetta di cervello con il punzone bioptico per creare un foro di 1 mm al centro della fetta di cervello.
NOTA: Il tessuto nel punch bioptico verrà aspirato nel punch dal vuoto. - Preparare una sospensione di matrice cellulare tripsinizzando un piatto di cellule confluenti del 60% -70% di 10 cm e riassumendo in 10 ml di supporto; quindi, mescolare 1 mL di quella sospensione con 100 μL di matrice.
- Utilizzando una micropipetta da 20 μL, posizionare 1 μL della miscela di matrice cellulare in ciascun foro delle fette di cervello.
- Dopo aver terminato il posizionamento della miscela cellulare, posizionare il piatto di fette di cervello con cellule incorporate nell'incubatore e consentire alla matrice di solidificarsi e alle cellule di invadere potenzialmente la fetta di cervello circostante.
10. Microscopia time-lapse a fluorescenza a largo campo
- Posizionare il nastro rimovibile attorno al bordo della piastra a 6 pozzetti per impedire l'evaporazione del fluido, lasciando un piccolo spazio su un lato per lo scambio di gas.
- Posizionare le piastre in una camera di coltura personalizzata su uno stadio automatizzato regolabile su un microscopio a epifluorescenza invertita.
NOTA: La camera è stata mantenuta in condizioni atmosferiche del 5% di CO2 e del 95% di aria utilizzando un controller di iniezione di gas e la temperatura è stata mantenuta a 37 °C con un regolatore di temperatura dell'aria calda e un inserto a temperatura controllata. Vedi Fotos et al.18 per i dettagli del sistema utilizzato qui. - Utilizzando un apposito software di controllo del microscopio, creare un programma di acquisizione time-lapse che raccolga immagini fluorescenti delle aree di interesse una volta ogni 10 minuti per 20 ore.
NOTA: Se nelle cellule viene utilizzata un'etichetta fluorescente verde (ad esempio, proteina fluorescente verde [GFP]), utilizzare la quantità minima di luce di eccitazione blu richiesta per visualizzare le cellule per prevenire la fototossicità. Le etichette rosse (ad esempio, mCherry) e rosso lontano (ad esempio, DiD) non sembrano avere questo potenziale problema a causa dell'eccitazione a lunghezza d'onda più lunga.
11. Immunocolorazione delle fette di cervello dopo microscopia time-lapse
NOTA: Questo protocollo di immunocolorazione è ottimizzato per colorare i vasi sanguigni con laminina e nuclei con bisbenzimide. Utilizzare anticorpi appropriati per la/e molecola/e desiderata/e di interesse.
- Aspirare il fluido da sotto l'inserto della membrana utilizzando una pipetta Pasteur e posizionare 1 mL di PFA al 2% in tampone cacodilato di sodio 0,1 M sotto l'inserto e 1 mL sopra l'inserto per coprire la fetta di cervello. Lasciare fissare le fette per una notte a 4 °C.
- Rimuovere il fissativo da sotto l'inserto della membrana e qualsiasi fissativo rimasto sulla fetta di cervello (il fissativo tende a fuoriuscire attraverso l'inserto della membrana nel pozzo sottostante).
- Rimuovere l'inserto con fette dal pozzetto da 35 mm e metterli in un piatto di plastica più grande.
- Preparare una piastra da 24 pozzetti aggiungendo 350 μL di PBS in tanti pozzetti quante sono le fette di cervello (una fetta / pozzetto durante l'immunocolorazione).
- Utilizzando una spatola sottile, rimuovere delicatamente l'agarosio da intorno alla fetta di cervello senza staccare la fetta di cervello dall'inserto della membrana. Assicurati che l'agarosio si stacchi facilmente dal bordo esterno della fetta del cervello. In caso contrario, lasciare l'agarosio attaccato alla fetta di cervello.
- Usando un bisturi affilato, tagliare la membrana intorno alla fetta del cervello fino a quando la fetta con la membrana sottostante è libera dal resto dell'inserto. Raccogli la membrana con la fetta di cervello attaccata, usando una pinza fine per afferrare la membrana e posizionala in un pozzetto della piastra a 24 pozzetti in PBS.
- Risciacquare le fette 3 volte con PBS nell'arco di 1 ora nella cella frigorifera con agitazione o oscillazione costante e delicata, in modo che la fetta si muova all'interno del pozzetto.
- Durante il risciacquo, preparare la soluzione anticorpale primaria.
- Diluire l'anti-laminina a 2 μg/mL in PBSTG (vedere Tabella 1).
- Rimuovere il PBS dai pozzetti e incubare durante la notte in soluzione anticorpale primaria in una cella frigorifera con delicata agitazione.
- Dopo almeno 20 ore di incubazione, aspirare la soluzione anticorpale primaria e sciacquare le sezioni 3x per 1 ora in PBSTG.
- Durante il risciacquo, preparare la soluzione anticorpale secondaria.
- Diluire fluorescente-GAM con il fluorocromo specifico necessario per una diluizione 1:200 in PBSTG insieme a una concentrazione di 0,1 μg/mL di bisbenzimmide.
- Rimuovere PBSTG e incubare durante la notte in una stanza fredda con agitazione nella soluzione di anticorpi secondari fluorescenti.
- Rimuovere l'anticorpo secondario e sciacquare 2 volte nell'arco di 1 ora in PBSTG e 1 volta in 1 ora in PBS.
- Lasciare in PBS fino al momento del montaggio su vetrini da microscopio (sezione 5) e visualizzare.
Risultati
Qui sono presentate più figure per mostrare alcuni risultati rappresentativi ottenuti eseguendo iniezioni in vivo nel tectum ottico (Figura 1 e Figura 2), coltivando fette di cervello vive e valutando la loro vitalità (Figura 3), creando colture di fette di cervello ex vivo e impiantando cellule marcate fluorescentmente usando il metodo del punch bioptico (Figura 4), generando sferoidi cellulari coltivando cellule su poli-HEMA (Figura 5 ), creando co-colture di fette di cervello ex vivo con sferoidi cellulari e registrando il comportamento cellulare invasivo utilizzando la microscopia time-lapse confocale 4D (Figura 6) e analizzando il comportamento delle cellule invasive dagli sferoidi rispetto ai vasi sanguigni in preparazioni fisse di fette cerebrali (Figura 7 e Figura 8). Questi risultati non sono affatto esaustivi, ma piuttosto forniscono buoni esempi di ciò che può essere ottenuto utilizzando il cervello dell'embrione di pulcino come modello di xenotrapianto per la ricerca sul GBM umano.
La figura 1 mostra alcuni risultati rappresentativi dei tumori che si sono formati nel tectum ottico in vivo dopo l'iniezione di GSC che esprimono GFP. Le GSC si attaccano alla superficie ventricolare e formano tumori invasivi nella parete cerebrale. Le GSC risiedono chiaramente vicino ai vasi sanguigni e sembrano migrare lungo di essi. I filmati dei rendering rotanti del volume 3D di fette fisse e immunocolorate di tumori GSC in vivo sono riportati in Video supplementare S1, Video supplementare S2, Video supplementare S3 e Video supplementare S4. In questo esperimento, sono stati utilizzati quattro colori per identificare cinque caratteristiche (GSC verdi, nuclei bianchi, vasi sanguigni bianchi, integrina alfa-6 blu e Sox2 rossa o nestina rossa).
La Figura 2 mostra alcuni risultati rappresentativi dei tumori che si sono formati nel tectum ottico in vivo dopo l'iniezione di GSC che esprimono GFP mescolate con cellule U-118/L1LE2 che esprimono mCherry a causa della trasduzione del vettore retrovirale. Questi esperimenti hanno rivelato che quando questi tumori si formavano da una sospensione a cellule miste, l'ordinamento avveniva in modo tale che le GSC risiedessero nella periferia o al centro, mentre le cellule U-118 comprendevano un nucleo interno o una corteccia esterna, a seconda della specifica linea GSC.
La figura 3 mostra i risultati di vitalità delle colture di fette di cervello ex vivo . Dopo 1 settimana in coltura, la fissazione e l'immunocolorazione per la laminina hanno rivelato molti vasi sanguigni intatti e l'espressione di Sox2, entrambi utilizzati qui per dimostrare la vitalità della fetta di cervello. Ciò ha dimostrato che le fette di cervello dell'embrione di pulcino potrebbero essere coltivate su inserti di membrana per circa 2 settimane e rimanere vitali con vasi sanguigni dall'aspetto normale e l'espressione del fattore di trascrizione.
La Figura 4 mostra i risultati dell'introduzione di "tappi" di cellule rosse U-118 / L1LE / mCherry (mescolate con matrice) in fette di cervello ex vivo dopo aver creato cavità nelle fette usando il metodo del punch bioptico. Le cellule U-118 hanno chiaramente invaso il tessuto cerebrale, a volte estesamente, e spesso lungo i vasi sanguigni. Tuttavia, l'invasione cellulare non era uniforme intorno alla circonferenza delle cellule introdotte. I vasi sanguigni a volte apparivano anche danneggiati o assenti in alcune fette, presumibilmente a causa del trauma aggiunto del metodo del punch o del periodo di tempo in coltura. Ciò ha dimostrato che il metodo del punch bioptico / tappo cellulare potrebbe essere utilizzato per introdurre cellule GBM in posizioni specifiche in una fetta di cervello ex vivo coltivata, dopo di che le cellule invadono la fetta di cervello.
La Figura 5 mostra sferoidi vivi in coltura e diversi esempi di fluorescenza a largo campo di sferoidi vivi di cellule GBM introdotti su fette di cervello ex vivo per esperimenti time-lapse. I filmati dell'invasione cellulare dagli sferoidi nella fetta del cervello sono riportati nel video supplementare S5 e nel video supplementare S6 . Ciò ha dimostrato che gli sferoidi cellulari sono un altro metodo efficace per introdurre cellule GBM o GSC su posizioni specifiche di una fetta di cervello ex vivo e il comportamento delle cellule invasive può essere monitorato mediante microscopia a fluorescenza a campo largo, sebbene la risoluzione delle singole cellule possa essere scarsa.
La Figura 6 mostra immagini statiche di esperimenti time-lapse confocali di GSC16-4/GFP e U-118/L1LE/mCherry cell invasion live in fette di cervello. Le immagini confocali z-stack sono state acquisite ogni 10 minuti per un periodo di 20 ore in un esperimento time-lapse multipunto. I filmati dell'invasione cellulare dagli sferoidi alle fette di cervello prese come z-stack confocali nel tempo sono presentati in Video supplementare S7, Video supplementare S8, Video supplementare S9, Video supplementare S10 e Video supplementare S11. Questo esperimento ha rivelato che l'imaging time-lapse confocale era superiore alla fluorescenza a largo campo per tracciare il comportamento invasivo delle singole cellule. Le cellule U-118/L1LE erano notevolmente più invasive delle GSC in queste condizioni. Ciò è evidente anche nelle immagini statiche, con le GSC posizionate più centralmente e le celle U-118 più disperse.
La Figura 7 mostra diversi esempi di preparazioni ex vivo di fette cerebrali/sferoidi, in cui due diversi sferoidi marcati separatamente (Sferoidi U-118/L1LE/mCherry e sferoidi GSC16-4/GFP) sono stati posizionati su fette di cervello, cresciuti per diversi giorni, e successivamente fissati, immunocolorati per la laminina e ripresi mediante sezionamento ottico su un microscopio confocale. Ciò ha rivelato che entrambi i tipi di cellule hanno invaso la fetta di cervello e hanno viaggiato lungo i vasi sanguigni. Quando i diversi tipi di sferoidi erano abbastanza vicini da entrare in contatto tra loro, sembrava esserci poca o nessuna invasione di un tipo di cellula nello sferoide dell'altro tipo di cellula, e gli sferoidi rimanevano segregati.
La Figura 8 mostra diversi esempi di preparazioni ex vivo di sferoidi / sferoidi di tipo cerebrale in cui sferoidi di "tipo a cellule miste" generati in coltura utilizzando due tipi di cellule marcate in modo diverso (U-118 / L1LE / mCherry miscelati con GSC16-4 / GFP) sono stati posizionati su fette di cervello, cresciuti per diversi giorni e successivamente fissati, immunocolorati per la laminina e ripresi mediante sezionamento ottico su un microscopio confocale. Ciò ha rivelato che le cellule rosse U-118 / L1LE / mCherry migravano fuori dagli sferoidi e si disperdevano molto più evidentemente delle cellule verdi GSC16-4 / GFP, che tendevano a rimanere in grumi vicino al centro degli sferoidi. Inoltre, anche le cellule U-118 / L1LE / mCherry sono state colorate con DiD in modo che le due etichette separate (mCherry e DiD) potessero essere confrontate direttamente nei preparati fissi ex vivo . L'etichetta DiD poteva ancora essere rilevata, anche in singole cellule che avevano invaso la fetta di cervello; Tuttavia, questo era come puncta intracellulare.
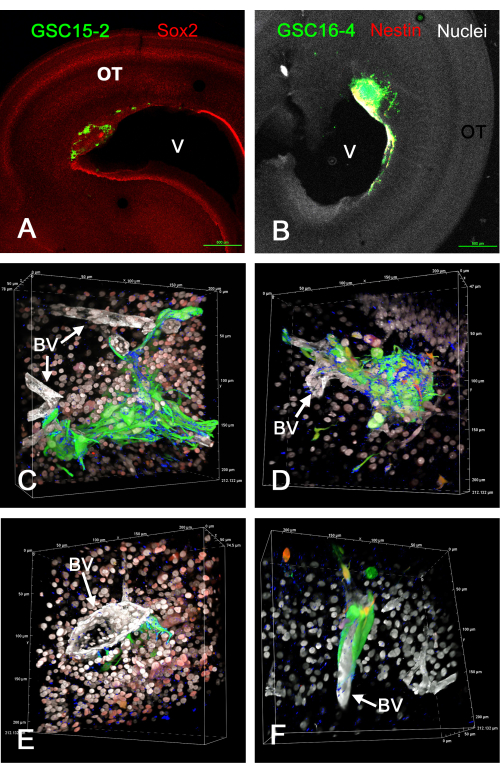
Figura 1: Tumori a E15 derivanti dall'iniezione di GSC nel tectum ottico E5 in vivo. Le GSC sono verdi a causa dell'espressione GFP. Le celle GSC15-2 sono mostrate nei pannelli A, C ed E, mentre le celle GSC16-4 sono mostrate nei pannelli B, D e F. (A) Vista a basso ingrandimento del tectum ottico con un tumore vicino al ventricolo (V). La colorazione Sox2 è mostrata in rosso, che colora la maggior parte dei nuclei delle cellule OT. (B) Immagine simile a A ma con celle GSC16-4 che sono anche colorate per la nestina in rosso, che possono apparire gialle o bianche nell'immagine a causa della miscelazione dei colori e dell'esposizione dell'immagine. I nuclei OT appaiono bianchi a causa della controcolorazione con bisbenzimide. (C-F) Diverse prospettive di rendering del volume generati da z-stack utilizzando un obiettivo di immersione in olio 60x. I nuclei cellulari appaiono bianchi a causa della colorazione con bisbenzimmide e alcuni appaiono rossi nei pannelli C ed E a causa dell'immunocolorazione per Sox2. La colorazione rossa nei pannelli D e F deriva dalla colorazione per la nestina. Si noti che a causa di "Alpha Blending" per i rendering di volume nel software del microscopio confocale, i colori non si fondono come farebbero usando una proiezione di massima intensità e il colore più prevalente predomina e oscura il colore meno intenso. I vasi sanguigni sono macchiati di bianco a causa dell'immunocolorazione per la laminina. La colorazione dell'integrina alfa-6 del marcatore GSC è mostrata in blu e appare punteggiata sulle superfici GSC. Le scale micron vengono visualizzate lungo i bordi dei rendering del volume. I video delle rotazioni dei rendering del volume nei pannelli C-F sono presentati in Video supplementare S1, Video supplementare S2, Video supplementare S3 e Video supplementare S4. Barre di scala = 500 μm (A,B). Abbreviazioni: GSCs = cellule staminali del glioblastoma; OT = tectum ottico; GFP = proteina fluorescente verde; BV = vaso sanguigno. Fare clic qui per visualizzare una versione ingrandita di questa figura.
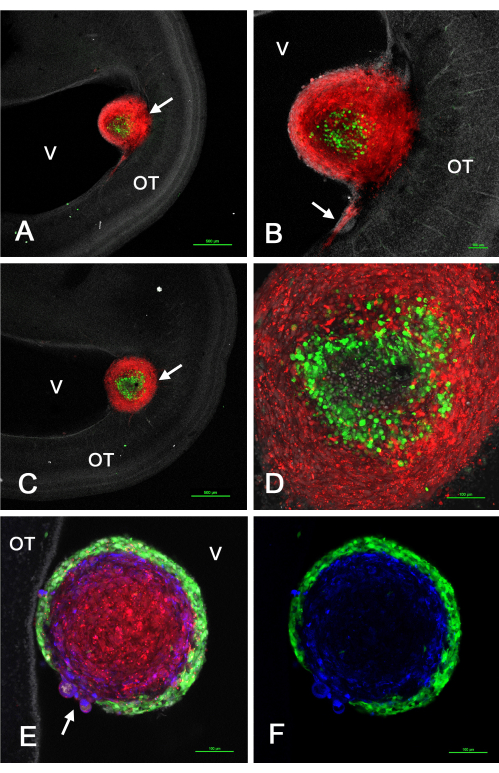
Figura 2: Tumori a E15 derivanti da una miscela di GSC e cellule U-118 GBM iniettate nel tectum ottico E5. Le GSC sono verdi a causa dell'espressione di GFP e le cellule U-118 / L1LE sono rosse a causa dell'espressione di mCherry. GSC15-2 sono mostrati nei pannelli A-D e GSC16-4 sono mostrati nei pannelli E e F. (A) Piano z singolo confocale a basso ingrandimento di un tumore a cellule miste (freccia) vicino al ventricolo. I nuclei sono controcolorati di bianco con bisbenzimmide. (B) Maggiore ingrandimento (obiettivo 10x) del tumore mostrato in A con invasione di cellule rosse U-118 nell'OT vicino alla superficie ventricolare. (C) Un piano leggermente diverso di sezione ottica da quello in A che mostra il tumore (freccia) incorporato più in profondità nella parete OT. (D) Proiezione massima (obiettivo 20x) di più piani z del tumore in C che mostrano i dettagli delle cellule ordinate all'interno del tumore. (E) Singola immagine z-plane (obiettivo 20x) di un tumore misto con cellule GSC16-4, che mostra che lo smistamento all'interno del tumore si è verificato in un modello opposto alle cellule GSC15-2, con le GSC verdi che creano una corteccia sottile e uniforme che circonda le cellule rosse U-118. L'area di attacco del tumore alla parete OT non è mostrata in questo piano z. Si noti l'area del tumore in cui vi è una discontinuità della corteccia GSC con cellule U-118 / L1LE che sporgono attraverso (freccia). L'immunocolorazione per L1CAM è mostrata in blu. (F) Stessa immagine di E, ma mostra solo le GSC verdi e la colorazione L1CAM blu. Barre della scala = 500 μm (A,C), 100 μm (B,D,E,F). Abbreviazioni: GSCs = cellule staminali del glioblastoma; OT = tectum ottico; GFP = proteina fluorescente verde; V = ventricolo. Fare clic qui per visualizzare una versione ingrandita di questa figura.
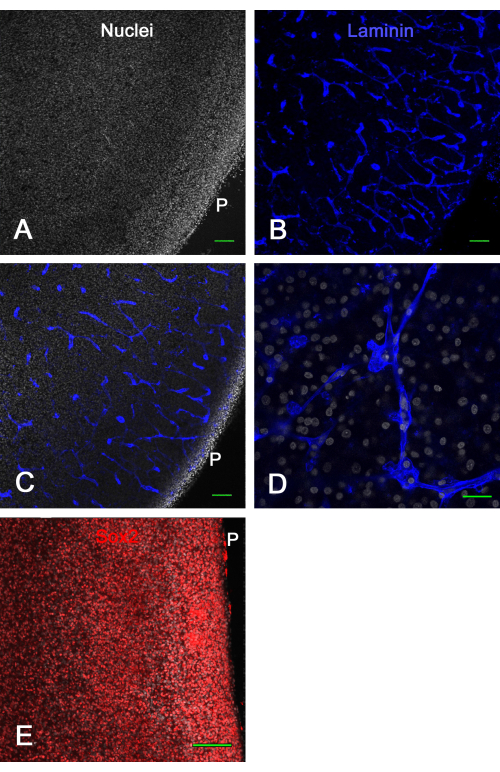
Figura 3: Viabilità delle fette di tectum ottico ex vivo dopo 1 settimana in coltura. Le fette di tectum ottico E14 sono state coltivate su inserti di membrana per 1 settimana e poi fissate e immunocolorate. In A e B sono mostrate immagini confocali (obiettivo 10x) di una fetta di cervello colorata per nuclei con bisbenzimmide (A) e immunocolorata per laminina (B), che mostra chiaramente vasi sanguigni normali e intatti sezionati otticamente in varie configurazioni in virtù della colorazione della laminina. (C) Un'immagine confocale simile a quella mostrata nei pannelli A e B in cui i nuclei e la colorazione della laminina sono entrambi visibili. (D) Un'immagine confocale ad alto ingrandimento (obiettivo petrolifero 60x) che mostra i dettagli della colorazione nucleare e della laminina. (E) Immagine di proiezione massima dello z-stack confocale (obiettivo 20x) della fetta di cervello colorata per il fattore di trascrizione Sox2 in rosso e nuclei totali con bisbenzimmide in bianco. Si noti che la maggior parte dei nuclei mostra colorazione Sox2, come mostrato in vivo (vedi Figura 1). Barre di scala = 100 μm (A,B,C,E), 25 μm (D). Abbreviazione: P = superficie piale. Fare clic qui per visualizzare una versione ingrandita di questa figura.
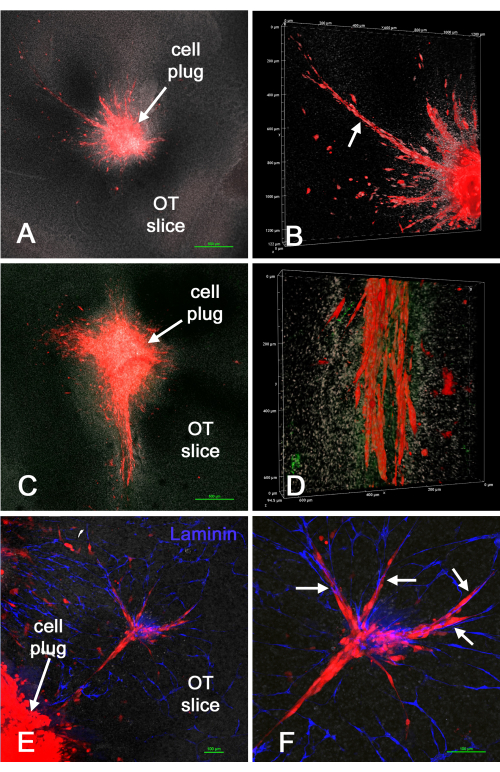
Figura 4: U-118/mCherry cells placed in an ex vivo brain slice through the biopsy punch method. Le cavità sono state create in fette di cervello usando un pugno bioptico da 1 mm, e quindi le cellule rosse U-118 / L1LE / mCherry mescolate con la matrice sono state impiantate come un "tappo". Dopo diversi giorni, le fette di cervello sono state fissate, immunocolorate per la laminina e montate su vetrini per l'analisi al microscopio confocale. I pannelli A e C mostrano immagini a basso ingrandimento, confocali, a singolo piano z (obiettivo 4x) del "tumore" risultante e delle cellule circostanti che hanno invaso la fetta di cervello. (B) Un rendering del volume di uno z-stack dalla preparazione nel pannello A ad un ingrandimento maggiore (obiettivo 20x), che mostra un'estesa invasione di celle U-118 (freccia). (D) L'immagine mostra un rendering del volume simile della parte inferiore delle celle ampiamente invasori mostrate nel pannello C. La colorazione della laminina è mostrata in verde, ma non sono evidenti vasi sanguigni chiari. (E) L'immagine mostra parte di un tappo cellulare e un gruppo di cellule che hanno invaso la fetta di cervello, insieme alla colorazione della laminina per i vasi sanguigni in blu. (F) Un maggiore ingrandimento delle cellule invasori mostrate nel pannello E, e le cellule possono essere chiaramente viste allineate lungo i vasi sanguigni (frecce). Tutti i pannelli mostrano una controcolorazione nucleare bianca con bisbenzimmide. Barre della scala = 500 μm (A,C), 100 μm (E,F). La scala per i pannelli B e D si trova lungo gli assi di rendering del volume. Abbreviazione: OT = ottica tectum. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 5: Sferoidi di cellule vive in coltura e immagini di fluorescenza ad ampio campo di cellule GBM vive in fette di cervello ex vivo. Nei pannelli A e B sono mostrate immagini a contrasto di fase (usando un obiettivo 10x su un microscopio invertito) di cellule GBM U-118/L1LE (A) e GSC (B) che crescono come sferoidi (frecce). Sullo sfondo del pannello A è mostrata l'irregolarità fuori fuoco del rivestimento poly-HEMA che può verificarsi sul piatto di coltura cellulare. Nei pannelli C-F sono mostrate immagini a fluorescenza a largo campo di sferoidi cellulari U-118/L1LE e cellule invasori (frecce) durante un esperimento time-lapse per monitorare il comportamento vivo dell'invasione nelle fette ex vivo (utilizzando un obiettivo 20x su un sistema di microscopio time-lapse personalizzato18). Nei pannelli C ed E, le cellule sono colorate con il colorante a membrana fluorescente rosso lontano DiD, e nei pannelli D e F, le cellule vengono visualizzate tramite la loro espressione mCherry rossa. Barre della scala = 100 μm. I video degli esperimenti time-lapse a fluorescenza a campo larghi mostrati nei pannelli C e D si trovano rispettivamente nel video supplementare S5 e nel video supplementare S6. Abbreviazioni: GBM = glioblastoma; GSC = cellule staminali GBM; S = sferoide. Fare clic qui per visualizzare una versione ingrandita di questa figura.
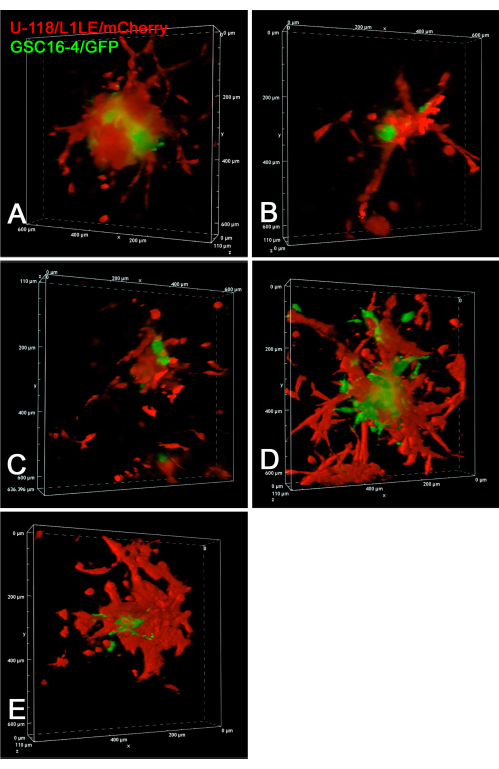
Figura 6: Immagini di rendering del volume del time-lapse 4D confocale di GSC e cellule GBM vive. In tutti i pannelli sono mostrate le immagini finali di cinque diversi attecchimenti sferoidi a cellule miste su fette di cervello separate. Per i pannelli A-E, sono state acquisite immagini confocali z-stack a passi di 10 μm ogni 10 minuti per un periodo di 20 ore. I preparativi includevano fette di cervello con sferoidi a cellule miste impiantati di cellule rosse U-118 / L1LE / mCherry e cellule GSC16-4 / GFP verdi. Le immagini confocali sono state scattate mentre le fette di cervello sono state coltivate su inserti di membrana in un piatto di coltura cellulare di plastica a 6 pozzetti utilizzando un obiettivo extra-lunga distanza di lavoro (ELWD) 20x (0,45 NA), che ha fornito la distanza di lavoro aggiuntiva necessaria. I rendering del volume sono stati generati utilizzando il software per microscopio confocale "Alpha Blending", che fornisce un apparente effetto 3D. I video time-lapse di questi rendering del volume confocale nel tempo sono presentati in Video supplementare S7, Video supplementare S8, Video supplementare S9, Video supplementare S10 e Video supplementare S11. Abbreviazioni: GBM = glioblastoma; GSC = cellule staminali GBM; GFP = proteina fluorescente verde; NA = apertura numerica. Fare clic qui per visualizzare una versione ingrandita di questa figura.
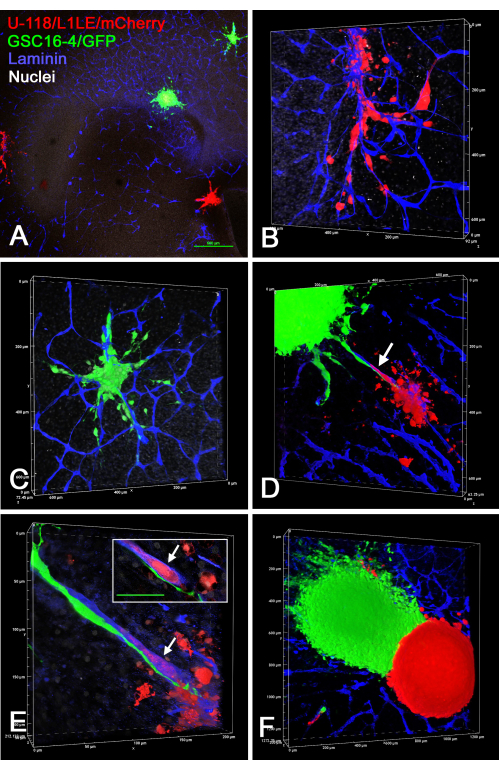
Figura 7: Immagini confocali di fette cerebrali fisse con cellule GBM invasive da sferoidi di diversi tipi di cellule. Gli sferoidi verdi erano composti da cellule GSC16-4/GFP e gli sferoidi rossi erano composti da cellule U-118/L1LE/mCherry. Nei pannelli A-F sono mostrate diverse viste di fette di cervello, su cui sono stati coltivati più sferoidi rossi e verdi per diversi giorni prima della fissazione e dell'immunocolorazione per la laminina (blu). I pannelli A-C sono della stessa fetta OT in cui A è stato preso con un obiettivo 4x, e i pannelli B e C sono rendering di volume di ingrandimento più elevato (obiettivo 20x) di cellule che hanno invaso la fetta di cervello da due degli sferoidi mostrati nel pannello A. Entrambi i tipi di cellule hanno chiaramente invaso il tessuto lungo i vasi sanguigni. Il pannello D mostra un rendering del volume (obiettivo 20x) di una diversa fetta di cervello in cui due diversi sferoidi erano situati vicini e le cellule di entrambi sono viste migrare lungo lo stesso vaso sanguigno che si trova tra di loro (freccia). Il pannello E è un rendering del volume ad alto ingrandimento (obiettivo dell'olio 60x) che rivela che le cellule verdi stanno migrando lungo la superficie esterna del vaso sanguigno, mentre i globuli rossi stanno migrando all'interno del vaso sanguigno (freccia). L'inserto mostra una singola sezione ottica sul piano z, dove il globulo rosso è chiaramente circondato dalla colorazione blu del vaso sanguigno (freccia) e la cellula verde è chiaramente al di fuori del vaso sanguigno. Barra della scala nel riquadro = 50 μm. Il pannello F mostra un rendering del volume (obiettivo 10x) di una fetta del proencefalo con due sferoidi di colore diverso ravvicinati. Pochissima, se non nessuna, invasione cellulare si è verificata da uno sferoide all'altro, e un confine netto esisteva tra di loro. I pannelli A, B, C ed E mostrano anche una controcolorazione nucleare bianca con bisbenzimmide. Barra di scala = 500 μm (A). Le scale per i pannelli B-F si trovano lungo gli assi di rendering del volume. Abbreviazioni: GBM = glioblastoma; GSC = cellule staminali GBM; GFP = proteina fluorescente verde; OT = tectum ottico. Fare clic qui per visualizzare una versione ingrandita di questa figura.
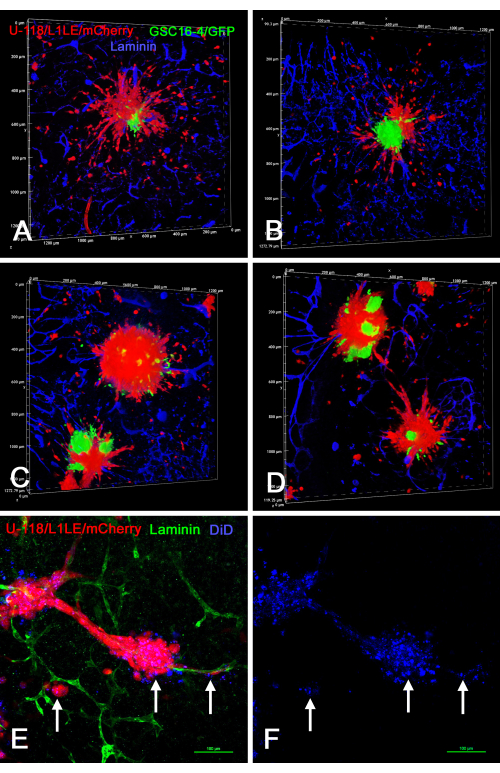
Figura 8: Immagini confocali di fette cerebrali fisse con cellule GBM invasive da sferoidi a cellule miste e sferoidi marcati con DiD. I pannelli A-D mostrano i rendering del volume di fette di cervello che contenevano sferoidi a cellule miste composti da cellule GSC16-4 / GFP verdi e cellule rosse U-118 / L1LE / mCherry. Numerose cellule rosse dell'U-118 si dispersero dagli sferoidi e invasero la fetta di cervello in tutte le direzioni, mentre le GSC verdi non si dispersero e rimasero nelle posizioni centrali degli sferoidi. I pannelli E e F mostrano una preparazione a fette ex vivo con sferoidi rossi U-118 / L1LE / mCherry etichettati anche con colorante a membrana rosso lontano DiD (mostrato come blu). Dopo la fissazione, la fetta è stata immunocolorata per la laminina in verde. L'etichetta DiD era visibile nei globuli rossi come colorazione puntata (frecce) ed era visibile anche nelle cellule che si erano disperse dagli sferoidi lungo i vasi sanguigni. La controcolorazione nucleare con bisbenzimmide non è mostrata in questa figura in modo che l'altra colorazione sia più chiaramente visibile. Barre della scala = 100 μm (E,F). Abbreviazioni: GBM = glioblastoma; GSC = cellule staminali GBM; GFP = proteina fluorescente verde. Fare clic qui per visualizzare una versione ingrandita di questa figura.
| Mezzo/soluzione | Composizione | ||
| Mezzi di comunicazione dell'SGC | Miscela 1:1 di DMEM/F12, siero bovino fetale all'1% (FBS), tampone HEPES 15 mM, 2 mM di L-glutammina, 100 μg/mL di penicillina-streptomicina (penna/streptococco), 2% di B27 senza vitamina A e 2,5 μg/mL di eparina. | ||
| Supporti GBM | DMEM (alto glucosio), 10% FBS, penna / streptococco e 2 mM L-glutammina. | ||
| Buffer di fissazione | 2% PFA in tampone cacodilato di sodio 0,1 M | ||
| Supporto di incorporamento | 3,5% agar e 8% saccarosio in PBS | ||
| PBSTG | 0,1% Triton X-100 + 5% siero di capra normale (NGS) in PBS | ||
| U-118 MG terreno di coltura cellulare | DMEM + 10% FBS + penna/streptococco + L-glutammina | ||
| Brain Slice Culture Media | 50% MEM + 25% HBSS + 25% siero di cavallo + B27 + penna/streptococco + L-glut + 15 mM tampone HEPES | ||
| Affettatrice vibrante di tessuto Mezzi di affettatura | Media 199 + penna/streptococco + tampone HEPES 15 mM | ||
Tabella 1: Composizione dei supporti e dei buffer utilizzati in questo protocollo.
Figura supplementare 1: Iniezione nel tectum ottico E5. (A) Dopo che un foro è stato praticato nel guscio d'uovo sopra lo spazio aereo e la membrana dello spazio aereo è stata bagnata con soluzione salina o media, la membrana viene rimossa con una pinza fine. (B) Per iniettare cellule nel tectum ottico, l'amnione viene pizzicato e tenuto con una pinza fine per posizionare la testa in modo che il tectum ottico sia accessibile. Quindi la micropipetta viene inserita nel tectum ottico e le cellule vengono iniettate in pressione. (C) Dopo l'iniezione di cellule, alcune gocce di soluzione di ampicillina vengono aggiunte sopra l'embrione usando una siringa e un ago sottile. Clicca qui per scaricare questo file.
Figura supplementare 2: Dissezione delle regioni cerebrali E15. (A) Dopo la decapitazione, la testa dell'embrione E15 viene posta in un piatto con soluzione sterile di CMF. (B) La pelle sovrastante il cervello viene quindi rimossa usando una pinza sottile. (C) Le due ossa del cranio vengono quindi rimosse dalla sovrastante i due emisferi del proencefalo (FB). (D) La dura del tessuto connettivo viene quindi delicatamente rimossa dal circondare il proencefalo (FB), il tectum ottico e il cervelletto. (E) L'intero cervello viene quindi rimosso dalla testa estraendolo delicatamente dalla cavità cerebrale da sotto usando una pinza curva. (F ) Viene mostrata la vista dorsale dell'intero cervello rimosso con proencefalo (FB), tectum ottico (OT) e cervelletto (CB). (G ) Il cervello isolato viene quindi sezionato negli emisferi del proencefalo (FB), del tectum ottico (OT) e del cervelletto (CB) usando forbici sottili. (H ) La delicata pia del tessuto connettivo viene quindi facilmente rimossa dagli emisferi del tectum ottico (OT) utilizzando una pinza fine. Clicca qui per scaricare questo file.
Figura supplementare 3: Incorporamento e affettamento del tectum ottico E15 e posizionamento degli sferoidi cellulari. (A) Un emisfero del tectum ottico è immerso nell'agarosio a basso punto di fusione usando una pinza curva. (B) Dopo che l'agarosio si è indurito sul ghiaccio, il blocco contenente il tectum ottico viene rifilato e incollato al piedistallo in acciaio inossidabile nel piatto/vassoio di affettatura. (C) Dopo che la colla si è asciugata, il piatto/vassoio per affettare viene inserito nel mandrino dell'affettatrice di tessuto vibrante e riempito con mezzi di affettatura a freddo. Le fette vengono quindi tagliate con il coltello zaffiro dal blocco di tessuto sommerso. Le fette tagliate galleggeranno nel piatto / vassoio e possono essere rimosse usando una spatola. (D) Le fette tagliate vengono rimosse dal piatto/vassoio e posizionate direttamente sugli inserti di membrana con il terreno di coltura sottostante in una piastra multipozzetto. (E) Dopo che gli sferoidi cellulari sono cresciuti su piatti rivestiti di poli-HEMA, uno sferoide viene rimosso dal piatto in una quantità minima di mezzi utilizzando un micropipettatore da 20 μL. (F) Lo sferoide isolato viene quindi posizionato direttamente sulla fetta di cervello nel mezzo minimo. (G) Se lo sferoide cade dalla fetta di cervello a causa del flusso del mezzo, allora può essere spinto indietro sulla fetta di cervello usando una ciglia incollata a un bastoncino applicatore di legno. Clicca qui per scaricare questo file.
Video supplementare S1: Video del rendering ad alto volume di ingrandimento di un piccolo tumore GSC15-2 a E15. Le GSC sono verdi a causa dell'espressione GFP. Il video corrisponde alla Figura 1C e mostra le celle GSC15-2. Il video mostra la rotazione di un rendering del volume generato da uno z-stack utilizzando un obiettivo di immersione in olio 60x. I nuclei cellulari appaiono bianchi a causa della colorazione delle bisbenzimmide, e alcuni appaiono rossi a causa dell'immunocolorazione per Sox2. Si noti che a causa di "Alpha Blending" per i rendering di volume nel software del microscopio confocale, i colori non si fondono come farebbero usando una proiezione di massima intensità e il colore più intenso predomina e oscura il colore meno intenso. I vasi sanguigni sono macchiati di bianco a causa dell'immunocolorazione per la laminina. La colorazione dell'integrina alfa-6 del marcatore GSC è mostrata in blu e appare punteggiata sulle superfici GSC verdi. Le scale di micron vengono visualizzate lungo i bordi del rendering del volume. Clicca qui per scaricare questo video.
Video supplementare S2: Video del rendering ad alto volume di ingrandimento di un piccolo tumore GSC16-4 a E15. Le GSC sono verdi a causa dell'espressione GFP. Il video corrisponde alla Figura 1D e mostra le celle GSC16-4. Il video mostra la rotazione di un rendering del volume generato da uno z-stack utilizzando un obiettivo di immersione in olio 60x. I nuclei cellulari appaiono bianchi a causa della colorazione delle bisbenzimmidi e alcune GSC appaiono rosse a causa dell'immunocolorazione per la nestina. Si noti che a causa di "Alpha Blending" per i rendering di volume nel software del microscopio confocale, i colori non si fondono come farebbero usando una proiezione di massima intensità e il colore più intenso predomina e oscura il colore meno intenso. I vasi sanguigni sono macchiati di bianco a causa dell'immunocolorazione per la laminina. La colorazione dell'integrina alfa-6 del marcatore GSC è mostrata in blu e appare punteggiata sulle superfici GSC verdi. Le scale di micron vengono visualizzate lungo i bordi del rendering del volume. Clicca qui per scaricare questo video.
Video supplementare S3: Video di rendering ad alto volume di ingrandimento di un piccolo tumore GSC15-2 a E15. Le GSC sono verdi a causa dell'espressione GFP. Il video corrisponde alla Figura 1E e mostra le celle GSC15-2. Il video mostra la rotazione di un rendering del volume generato da uno z-stack utilizzando un obiettivo di immersione in olio 60x. I nuclei cellulari appaiono bianchi a causa della colorazione delle bisbenzimmide, e alcuni appaiono rossi a causa dell'immunocolorazione per Sox2. Si noti che a causa di "Alpha Blending" per i rendering di volume nel software del microscopio confocale, i colori non si fondono come farebbero usando una proiezione di massima intensità e il colore più intenso predomina e oscura il colore meno intenso. I vasi sanguigni sono macchiati di bianco a causa dell'immunocolorazione per la laminina. La colorazione dell'integrina alfa-6 del marcatore GSC è mostrata in blu e appare punteggiata sulle superfici GSC verdi. Le scale di micron vengono visualizzate lungo i bordi del rendering del volume. Clicca qui per scaricare questo video.
Video supplementare S4: Video di rendering ad alto volume di ingrandimento di piccoli tumori GSC16-4 a E15. Le GSC sono verdi a causa dell'espressione GFP. Il video corrisponde alla Figura 1F e mostra le celle GSC16-4. Il video mostra la rotazione di un rendering del volume generato da uno z-stack utilizzando un obiettivo di immersione in olio 60x. I nuclei cellulari appaiono bianchi a causa della colorazione delle bisbenzimmide, e alcuni appaiono rossi a causa dell'immunocolorazione per la nestina. Si noti che a causa di "Alpha Blending" per i rendering di volume nel software del microscopio confocale, i colori non si fondono come farebbero usando una proiezione di massima intensità e il colore più intenso predomina e oscura il colore meno intenso. I vasi sanguigni sono macchiati di bianco a causa dell'immunocolorazione per la laminina. La colorazione dell'integrina alfa-6 del marcatore GSC è mostrata in blu e appare punteggiata sulle superfici GSC verdi. Le scale di micron vengono visualizzate lungo i bordi del rendering del volume. Clicca qui per scaricare questo video.
Video supplementare S5: Video di cellule GBM vive in una fetta di cervello ex vivo . Il video corrisponde alla Figura 5C e mostra immagini di fluorescenza a campo largo di sferoidi cellulari U-118/L1LE e cellule invasori durante un esperimento time-lapse per monitorare il comportamento vivo dell'invasione nella fetta ex vivo (utilizzando un obiettivo 20x su un sistema di microscopio time-lapse personalizzato). Le cellule U-118/L1LE sono state colorate con il colorante a membrana fluorescente rosso lontano DiD. Le immagini sono state acquisite con una fotocamera monocromatica. Clicca qui per scaricare questo video.
Video supplementare S6: Video di cellule GBM vive in una fetta di cervello ex vivo . Il video corrisponde alla Figura 5D e mostra immagini di fluorescenza ad ampio campo di sferoidi cellulari U-118/L1LE e cellule invasori durante un esperimento time-lapse per monitorare il comportamento dal vivo dell'invasione nella fetta ex vivo (utilizzando un obiettivo 20x su un sistema di microscopio time-lapse personalizzato). Le cellule sono state riprodotte tramite la loro espressione mCherry rossa. Le immagini sono state acquisite con una fotocamera monocromatica. Clicca qui per scaricare questo video.
Video supplementare S7: Video di rendering del volume immagini di time-lapse 4D confocale di GSC e celle GBM live. Il video corrisponde alla Figura 6A. Le immagini confocali z-stack sono state acquisite a passi di 10 μm ogni 10 minuti per un periodo di 20 ore. La preparazione era di una fetta di cervello con sferoidi a cellule miste impiantati di cellule rosse U-118 / L1LE / mCherry e cellule GSC16-4 / GFP verdi. Le immagini confocali sono state scattate mentre la fetta di cervello è stata coltivata su un inserto di membrana in un piatto di coltura cellulare di plastica a 6 pozzetti utilizzando una lente obiettivo ELWD 20x (0,45 NA), che ha fornito la distanza di lavoro aggiuntiva necessaria. Il rendering del volume è stato generato utilizzando il software per microscopio confocale "Alpha Blending", che fornisce un apparente effetto 3D. Le scale di micron vengono visualizzate lungo i bordi del rendering del volume. Il video viene osservato al meglio trascinando manualmente il cursore di avanzamento del video nel lettore video avanti e indietro per osservare il movimento della cella piuttosto che consentire al lettore video di procedere alla sua normale bassa velocità. Clicca qui per scaricare questo video.
Video supplementare S8: Video di rendering del volume immagini di time-lapse 4D confocale di GSC live e celle GBM. Il video corrisponde alla Figura 6B. Le immagini confocali z-stack sono state acquisite a passi di 10 μm ogni 10 minuti per un periodo di 20 ore. La preparazione era di una fetta di cervello con sferoidi a cellule miste impiantati di cellule rosse U-118 / L1LE / mCherry e cellule GSC16-4 / GFP verdi. Le immagini confocali sono state scattate mentre la fetta di cervello è stata coltivata su un inserto di membrana in un piatto di coltura cellulare di plastica a 6 pozzetti utilizzando una lente obiettivo ELWD 20x (0,45 NA), che ha fornito la distanza di lavoro aggiuntiva necessaria. Il rendering del volume è stato generato utilizzando il software per microscopio confocale "Alpha Blending", che fornisce un apparente effetto 3D. Le scale di micron vengono visualizzate lungo i bordi del rendering del volume. Il video viene osservato al meglio trascinando manualmente il cursore di avanzamento del video nel lettore video avanti e indietro per osservare il movimento della cella piuttosto che consentire al lettore video di procedere alla sua normale bassa velocità. Clicca qui per scaricare questo video.
Video supplementare S9: Video del volume di rendering delle immagini di time-lapse 4D confocale di GSC live e celle GBM. Il video corrisponde alla Figura 6C. Le immagini confocali z-stack sono state acquisite a passi di 10 μm ogni 10 minuti per un periodo di 20 ore. La preparazione era di una fetta di cervello con sferoidi a cellule miste impiantati di cellule rosse U-118 / L1LE / mCherry e cellule GSC16-4 / GFP verdi. Le immagini confocali sono state scattate mentre la fetta di cervello è stata coltivata su un inserto di membrana in un piatto di coltura cellulare di plastica a 6 pozzetti utilizzando una lente obiettivo ELWD 20x (0,45 NA), che ha fornito la distanza di lavoro aggiuntiva necessaria. Il rendering del volume è stato generato utilizzando il software per microscopio confocale "Alpha Blending", che fornisce un apparente effetto 3D. Le scale di micron vengono visualizzate lungo i bordi del rendering del volume. Il video viene osservato al meglio trascinando manualmente il cursore di avanzamento del video nel lettore video avanti e indietro per osservare il movimento della cella piuttosto che consentire al lettore video di procedere alla sua normale bassa velocità. Clicca qui per scaricare questo video.
Video supplementare S10: Video del volume di rendering immagini di time-lapse 4D confocale di GSC live e celle GBM. Il video corrisponde alla Figura 6D. Le immagini confocali z-stack sono state acquisite a passi di 10 μm ogni 10 minuti per un periodo di 20 ore. La preparazione era di una fetta di cervello con sferoidi a cellule miste impiantati di cellule rosse U-118 / L1LE / mCherry e cellule GSC16-4 / GFP verdi. Le immagini confocali sono state scattate mentre la fetta di cervello è stata coltivata su un inserto di membrana in un piatto di coltura cellulare di plastica a 6 pozzetti utilizzando una lente obiettivo ELWD 20x (0,45 NA), che ha fornito la distanza di lavoro aggiuntiva necessaria. Il rendering del volume è stato generato utilizzando il software per microscopio confocale "Alpha Blending", che fornisce un apparente effetto 3D. Le scale di micron vengono visualizzate lungo i bordi del rendering del volume. Il video viene osservato al meglio trascinando manualmente il cursore di avanzamento del video nel lettore video avanti e indietro per osservare il movimento della cella piuttosto che consentire al lettore video di procedere alla sua normale bassa velocità. Clicca qui per scaricare questo video.
Video supplementare S11: Video del volume di rendering immagini di time-lapse 4D confocale di GSC live e celle GBM. Il video corrisponde alla Figura 6E. Le immagini confocali z-stack sono state acquisite a passi di 10 μm ogni 10 minuti per un periodo di 20 ore. la preparazione includeva una fetta di cervello con sferoidi a cellule miste impiantati di cellule rosse U-118 / L1LE / mCherry e cellule GSC16-4 / GFP verdi. Le immagini confocali sono state scattate mentre la fetta di cervello è stata coltivata su un inserto di membrana in un piatto di coltura cellulare di plastica a 6 pozzetti utilizzando una lente obiettivo ELWD 20x (0,45 NA), che ha fornito la distanza di lavoro aggiuntiva necessaria. Il rendering del volume è stato generato utilizzando il software per microscopio confocale "Alpha Blending", che fornisce un apparente effetto 3D. Le scale di micron vengono visualizzate lungo i bordi del rendering del volume. Il video viene osservato al meglio trascinando manualmente il cursore di avanzamento del video nel lettore video avanti e indietro per osservare il movimento della cella piuttosto che consentire al lettore video di procedere alla sua normale bassa velocità. Clicca qui per scaricare questo video.
Discussione
I passaggi critici nel protocollo per l'iniezione di cellule nel ventricolo del mesencefalo (tectum ottico) includono non danneggiare i vasi sanguigni nella membrana corioallantoica nell'uovo o circondare l'embrione prima e durante l'iniezione, sebbene la membrana amnionica immediatamente circostante l'embrione possa essere delicatamente tirata e tenuta per posizionare la testa quando si iniettano le cellule nel mesencefalo. L'amnione è relativamente duro e può essere tirato con una pinza sottile per posizionare la testa e tenerla ferma con una mano, per l'iniezione di cellule con l'altra mano nel tectum ottico, che è la grande struttura rotonda nel mezzo del cervello. Generalmente, la vitalità degli embrioni iniettati varia dal 25% al 75%, a seconda di fattori sconosciuti, e praticamente ogni embrione che sopravvive contiene almeno un piccolo tumore nel tectum ottico. I passaggi critici nella generazione di fette di cervello vitali includono la cancellazione del tessuto di liquido in eccesso in modo che l'agarosio aderisca al cervello durante il taglio e per mantenere il tessuto e le fette fredde fino a quando non vengono posizionate sull'inserto della membrana. Poiché diversi tipi di cellule formano sferoidi in modo diverso (in velocità e dimensioni), la densità cellulare placcata su piastre di poli-HEMA e il periodo di tempo prima della raccolta degli sferoidi dovrebbero essere ottimizzati per ciascun tipo di cellula.
Il lavoro qui non è stato oggetto di uno studio longitudinale formale della vitalità delle fette di cervello. Yang et al. hanno usato colture di fette di cervello di embrione di pulcino simili a quelle utilizzate qui e hanno mostrato una buona vitalità delle fette per almeno 7 giorni16. Il lavoro precedente ha dimostrato che quando il tessuto OT è stato mantenuto in mezzi subottimali, molti nuclei picnotici sono apparsi nel tessuto, che non si sono verificati nelle fette nel lavoro qui. Inoltre, quando le fette degenerano in condizioni non ottimali, i vasi sanguigni si frammentano e appaiono come file di sfere positive alla laminina (non mostrate). Pertanto, sebbene la vitalità qui non sia stata verificata con metodi come l'elettrofisiologia o l'espressione attiva della caspasi-3, nessuno degli indicatori di morte cellulare che sono stati osservati in condizioni di coltura non ottimali è apparso qui.
L'OT è stato focalizzato per esperimenti di tumore cerebrale in vivo perché è la regione più facilmente iniettabile con il ventricolo più grande. A E5, che è l'ultimo giorno in cui l'embrione è abbastanza piccolo da rimanere accessibile sopra il tuorlo, le iniezioni devono essere fatte in un ventricolo, poiché tutte le regioni del cervello non sono altro che una sottile zona ventricolare. Tuttavia, queste iniezioni provocano con successo tumori incorporati con cellule che invadono il parenchima cerebrale. A volte, i tumori risultanti si trovano nel proencefalo o nel cervelletto, ma questo non è comune. Fette ex vivo di tectum ottico E15 sono state utilizzate principalmente per esperimenti qui, in modo che i risultati della co-coltura ex vivo possano essere correlati con gli esperimenti di iniezione in vivo . Tuttavia, anche le fette del proencefalo sono adatte e hanno una superficie più ampia e un ventricolo molto sottile rispetto al tectum ottico, il che potrebbe rendere il proencefalo più adatto per co-colture ex vivo che non sono correlate con iniezioni in vivo .
È stato dimostrato qui che le iniezioni in vivo, seguite dalla fissazione dei tessuti, dal sezionamento vibrante dell'affettatrice tissutale e dall'immunocolorazione per la laminina e altri marcatori, hanno portato a immagini ad alta risoluzione delle cellule GBM e delle GSC nel tessuto cerebrale in prossimità dei vasi sanguigni. La capacità di determinare le interrelazioni tra cellule tumorali e vasi sanguigni è stata notevolmente facilitata dalla creazione di rendering di volumi 3D da z-stack di sezioni ottiche confocali utilizzando il software confocale e le istruzioni del produttore. Era possibile l'imaging time-lapse utilizzando la microscopia a fluorescenza a largo campo di cellule marcate con GFP, mCherry e DiD; Tuttavia, le cellule in migrazione che si trovavano in prossimità degli sferoidi altamente fluorescenti erano talvolta oscurate dal "bagliore" dello sferoide. Questo effetto indesiderato può essere in qualche modo ridotto al minimo regolando attentamente i tempi di esposizione per la raccolta di immagini a campo ampio. L'imaging time-lapse utilizzando z-stack confocali nel tempo (4D) ha eliminato il bagliore fuori fuoco dagli sferoidi e ha portato a cellule migratorie nettamente definite con uno sfondo scuro. Questo non è stato descritto nel protocollo, ma è stato eseguito in modo simile all'imaging time-lapse a campo ampio, che è stato eseguito mentre le fette di cervello erano sugli inserti della membrana trasparente in una piastra di plastica a 6 pozzetti. Sebbene l'imaging time-lapse confocale produca immagini marcatamente più chiare delle singole cellule e del loro comportamento, un esperimento time-lapse multi-punto che raccoglie pile z di 10 piani z / punto, a intervalli di 10 minuti su un periodo di 20 ore, è un uso estensivo dei galvanometri a testa di scansione. Poiché ciò potrebbe ridurre significativamente la durata della vita dei galvanometri, questo metodo viene utilizzato con giudizio.
Sebbene il sistema embrionale di pulcino sia molto adatto sia per l'iniezione in vivo che per gli esperimenti di co-coltura ex vivo che studiano il comportamento delle cellule GBM, ci sono diverse limitazioni a questo sistema modello. Come con qualsiasi sistema di xenotrapianto, l'ambiente in cui vengono impiantate le cellule umane non è il cervello umano, ma il comportamento delle cellule GBM sembra imitarlo nei modelli di roditori e nei pazienti umani. Dopo aver eseguito esperimenti di iniezione in vivo su E5, i tumori sono normalmente autorizzati a formarsi per 10 giorni, fino a E15. Questo chiaramente non è abbastanza tempo per studiare tutti gli aspetti della tumorigenesi e dell'invasione cellulare. Tuttavia, è stato dimostrato qui che i tumori solidi si formano nel parenchima cerebrale, le cellule interagiscono e si riorganizzano all'interno del tumore e si verificano significative invasioni cerebrali sia lungo i vasi sanguigni che diffusamente in questo periodo di tempo relativamente breve. Un'altra limitazione al sistema embrionale di pulcino in vivo è che non è adatto per farmaci o altri trattamenti a causa del grande tuorlo e del sistema circolatorio extraembrionale che opera durante lo sviluppo dell'embrione di pulcino. I trattamenti topici con farmaci liquidi si tradurrebbero in una concentrazione altamente variabile e sconosciuta nel cervello a causa della diffusione lontano dall'embrione nella massa di tuorlo molto più grande. Allo stesso modo, l'iniezione endovenosa di farmaci nel delicatissimo sistema circolatorio extraembrionale fuoriuscirebbe o diffonderebbe dai vasi sanguigni e provocherebbe anche concentrazioni sconosciute nel cervello. Questo è uno dei motivi principali per cui è stato adottato il metodo di coltura a fetta ex vivo , in modo che non solo il comportamento cellulare potesse essere osservato e monitorato tramite microscopia time-lapse, ma anche in modo che i trattamenti che hanno avuto successo nell'alterare il comportamento delle cellule GBM in un piatto4 potessero essere testati in un ambiente di tessuto cerebrale più rilevante.
Lo sviluppo del sistema modello di tumore cerebrale ortotopico dell'embrione di pulcino è visto come un'aggiunta significativa ai sistemi e agli strumenti disponibili per lo studio della formazione del tumore GBM e del comportamento cellulare invasivo. È probabile che le uova di gallina fecondate siano prontamente disponibili nella maggior parte delle aree, sono poco costose rispetto ai roditori, non ci sono costi di cura degli animali, gli embrioni sono molto resistenti e resistenti alle infezioni (cioè, la maggior parte del lavoro viene svolto su un banco), gli embrioni sono altamente manipolabili e possono essere coltivati in coltura senza guscio19 e gli embrioni di pulcino non sono considerati animali vertebrati e quindi non richiedono l'approvazione IACUC dalle linee guida NIH (requisiti istituzionali può variare). Pertanto, questi molteplici vantaggi rendono il sistema embrionale di pulcino molto attraente se si limitano le loro domande ed esperimenti a quelli che rientrano nei suoi limiti. Diversi studi sulle cellule GBM sono stati eseguiti da altri utilizzando l'embrione di pulcino, ma questi hanno utilizzato quasi esclusivamente la membrana corioallantoica (CAM) dell'embrione 20,21,22,23,24,25,26,27,28,29 e gemma degli arti 30, e non il cervello. C'è stato anche un rapporto che ha impiantato il medulloblastoma nel cervello del pulcino su E231. Indubbiamente, l'uso dell'embrione di pulcino come sistema modello di xenotrapianto ortotopico, come descritto qui, dovrebbe produrre risultati molto più significativi per la biologia tumorale del GBM umano rispetto agli studi che utilizzano la CAM.
Sebbene questi studi abbiano appena iniziato a utilizzare pienamente il sistema modello di tumore cerebrale dell'embrione di pulcino per studi sulle cellule GBM umane e sul comportamento delle GSC, si spera che altri estenderanno gli usi e troveranno ulteriori potenziali applicazioni. Si potrebbe immaginare che non solo questo sistema scoprirà i meccanismi che regolano la formazione del tumore GBM e il comportamento cellulare, ma consentirà anche test preclinici di farmaci e sostanze specifici su cellule di pazienti specifici. Ad esempio, se le colture di fette di cervello sono state impostate in anticipo, le cellule tumorali, i pezzi di resezioni tumorali chirurgiche o gli organoidi GBM32 derivati dal paziente potrebbero essere collocati direttamente in co-coltura ex vivo e vari trattamenti potrebbero essere valutati in pochi giorni. Allo stesso modo, le cellule dissociate del paziente potrebbero essere iniettate direttamente nei mesencefalo E5 in ovo per valutare la loro capacità di formare tumori e invadere il parenchima cerebrale. Pertanto, si spera che le descrizioni dei metodi e dei risultati rappresentativi qui facilitino e incoraggino un maggiore uso di questo sistema altamente sottoutilizzato per la ricerca sul cancro al cervello.
Divulgazioni
Nessuno degli autori ha conflitti di interesse.
Riconoscimenti
Questo lavoro è stato finanziato in parte da una sovvenzione al DSG del National Cancer Institute (R03CA227312) e da una generosa sovvenzione della Lisa Dean Moseley Foundation. I campioni vivi di GBM sono stati ottenuti con il consenso del paziente attraverso il Tissue Procurement Center dell'Helen F. Graham Cancer Center and Research Institute. Il finanziamento ad A.R. è stato fornito dal National Center for Research Resources e dal National Center for Advancing Translational Sciences, National Institutes of Health (UL1TR003107). Le borse di ricerca universitarie estive a N.P., A.L., Z.W. e K.S. sono state fornite dal programma di ricerca universitaria dell'Università del Delaware.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 cm x 1 cm square hole paper punch | Birabira | N/A | |
| 1 mm biopsy punch pen | Robbins Instruments | 20335 | |
| 6 well insert plate (Corning Transwell) | Millipore Sigma | CLS3450 | |
| 9" Disposable Pasteur Pipets | Fisher Scientific | 13-678-20C | |
| 15 mL centrifuge tubes | Fisher Scientific | 05-539-12 | |
| 24 well plate | Corning Costar | 3526 | |
| 50 mL centrifuge tubes | Fisher Scientific | 05-539-9 | |
| Agar | Fisher BioReagents | BP1423-500 | for embedding fixed brains |
| Alexafluor 488-conjugated GAM IgG | Jackson Immunoresearch | 115-605-146 | |
| Alexafluor 647-conjugated GAM IgG | Jackson Immunoresearch | 115-545-146 | |
| Aluminum foil | ReynoldsWrap | N/A | |
| Ampicillin | Sigma Aldrich | A-9518 | |
| anti-integrin alpha-6 monoclonal antibody GOH3 | Santa Cruz Biotechnology | sc-19622 | |
| anti-L1CAM monoclonal antibody UJ127 | Santa Cruz Biotechnology | sc-53386 | |
| anti-laminin monoclonal antibody | Developmental Studies Hybridoma Bank | 3H11 | |
| anti-nestin monoclonal antibody 10c2 | Santa Cruz Biotechnology | sc-23927 | |
| anti-Sox2 monoclonal antibody E-4 | Santa Cruz Biotechnology | sc-365823 | |
| B27 supplement without vitamin A | GIBCO | 17504-044 | |
| bisbenzimide (Hoechst 33258) | Sigma-Aldrich | B2883 | nuclear stain |
| Cell culture incubator | Forma | standard humidified CO2 incubator | |
| Centrifuge | Beckman Coulter | ||
| Confocal microscope | Nikon Instruments | C2si+ | With custom-made cell incubator chamber |
| Confocal microscope objective lenses | Nikon Instruments | Plan Apo lenses, except S Plan Fluor ELWD 20x 0.45 NA objective lens for confocal time-lapse imaging | |
| Confocal microscope software | Nikon Instruments | NIS Elements | Version 5.2 |
| Curved foreceps | World Precision Intruments | 504478 | |
| Curved scissors | Fine Science Tools | ||
| Curved spatula | Fisher Scientific | 14-375-20 | |
| Cyanoacrylate glue | Krazy Glue | KG-585 12R | |
| D-Glucose | Millipore Sigma | G8270 | |
| DiD far red fluorescent dye | Invitrogen | V22887 | Vybrant DiD |
| DMEM | Sigma Aldrich | D5671 | |
| DMEM/F12 | Sigma Aldrich | D8437 | |
| DMSO | Sigma Aldrich | D4540 | |
| Dulbecco's Phosphate buffered saline (PBS) | Sigma Aldrich | P5493-1L | |
| egg incubator | Humidaire | ||
| electrical tape (10 mil thick/254 µm) | Scotch | N/A | |
| Ethanol 200 proof | Decon Laboratories | 2701 | |
| Fast green FCF dye | Avocado Research Chemicals | 16520 | |
| FBS | Gemini Bio-products | 900-108 | |
| filter paper | Fisher Scientific | ||
| Gauze | Dynarex | 3353 | |
| Glass Capillaries for microinjection | World Precision Instruments | TW100-4 | |
| Glycerol | Fisher BioReagents | BP228-1 | for mounting media |
| GSCs (human glioblastoma stem cells) | Not applicable | Isolated from patient GBM specimens in Galileo laboratory in GSC media and then transduced with a GFP encoding lentiviral vector. Cells used were between passage 10 and 30. | |
| Hanks Balanced Salt Solution (HBSS) | Corning | 21-020-CV | |
| Hemacytometer | Hausser scientific | ||
| Heparin | Fisher Scientific | BP2524-100 | |
| HEPES buffer | Sigma Aldrich | H0887 | |
| Horse Serum (HI) | Gibco | 26050-088 | |
| Human FGF-2 | BioVision | 4037-1000 | |
| Human TGF-α | BioVision | 4339-1000 | |
| Inverted phase contrast microscope | Nikon Instruments | TMS | for routine viewing of cultured cells |
| KCl | Fisher Scientific | BP366 | |
| KH2PO4 | Fisher Scientific | P284 | |
| Laboratory film | Parafilm | ||
| Labquake Shaker | LabIndustries | T400-110 | |
| L-Glut:Pen:Strep | Gemini Bio-products | 400-110 | |
| Low-melt agarose | Fisher Scientific | BP1360 | for embedding live brains |
| Matrix | Corning Matrigel | 354234 | |
| Medium 199 | GIBCO | 11150-059 | |
| MEM | Corning | 10-010-CV | |
| Metal vibratome block | |||
| Micropipette tips (20, 200, 1,000 µL) | Fisherbrand | ||
| Micropipettors (20, 200, 1,000 µL) | Gilson | ||
| Microscope Coverglass (no. 1.5 thickness) | Fisherbrand | 12544A | |
| NaCl | Fisher Scientific | S271 | |
| NaH2PO4 + H2O | Fisher Scientific | S369 | |
| NaHCO3 | Fisher Scientific | BP328 | |
| Normal goat serum | Millipore Sigma | 526-M | |
| N-propyl gallate | Sigma Aldrich | P3130 | for mounting media |
| Parafilm | Parafilm | ||
| Paraformaldehyde | Electron Microscopy Sciences | 15710 | |
| PBS | Sigma Aldrich | P5493-1L | |
| Pencil | |||
| Plain Microscope slides | Fisherbrand | 12-550-A3 | |
| Plastic 35 mm Petri dish | Becton Dickinson | 351008 | |
| pneumatic picopump | World Precision Intruments | PV830 | |
| Poly(2-hydroxyethyl methacrylate) (poly-HEMA) | Sigma Aldrich | P-3932 | |
| razor blade- double edge | PACE | for cutting fixed brain slices | |
| sapphire knife | Delaware Diamond Knives | for cutting live brain slices | |
| Scalpel | TruMed | 1001 | |
| Sodium cacodylate buffer 0.2 M pH 7.4 | Electron Microscopy Sciences | 11652 | |
| Specimen chamber for vibratome | custom-made | ||
| Stereo Dissecting Microscope | Nikon Instruments | SMZ1500 | Equipped with epifluorescence |
| straight foreceps | World Precision Intruments | 500233 | |
| straight scissors | Fine Science Tools | ||
| Sucrose | Mallinckrodt | 7723 | |
| Time-lapse fluorescence microscope (widefield fluorescence) | Nikon Instruments | TE2000-E | With custom-made cell incubator chamber (see Fotos et al., 2006) |
| Tissue culture dish polystyrene 100 mm | Thermo Fisher Scientific | 130182 | for cell culturing |
| Tissue culture dish polystyrene 60 mm | Becton Dickinson | 353004 | for cell culturing |
| Transfer pipette | American Central Scientific Co. | FFP011 | |
| Transparent tape | Scotch | ||
| Triton X-100 | Sigma Aldrich | T-8787 | |
| Trypsin (0.25%) + 2.21 mM EDTA | Corning | 25-053-CI | |
| U-118 MG human GBM cell line | ATCC | HTB-15 | Cells were transduced with a lentiviral vector encoding the entire ectodomain sequence of the L1CAM adhesion protein and then with lentiviral vector pUltra-hot encoding mCherry. Passage numbers are unknown. |
| Vacuum pump | Cole-Parmer | EW-07532-40 | "Air Cadet" |
| Vibrating tissue slicer | Vibratome | 3000 | for cutting live and fixed brain slices |
Riferimenti
- Cretu, A., Fotos, J. S., Little, B. W., Galileo, D. S. Human and rat glioma growth, invasion, and vascularization in a novel chick embryo brain tumor model. Clinical & Experimental Metastasis. 22 (3), 225-236 (2005).
- Yang, M., et al. L1 stimulation of human glioma cell motility correlates with FAK activation. Journal of Neuro-Oncology. 105 (1), 27-44 (2011).
- Mohanan, V., Temburni, M. K., Kappes, J. C., Galileo, D. S. L1CAM stimulates glioma cell motility and proliferation through the fibroblast growth factor receptor. Clinical & Experimental Metastasis. 30 (4), 507-520 (2013).
- Anderson, H. J., Galileo, D. S. Small-molecule inhibitors of FGFR, integrins and FAK selectively decrease L1CAM-stimulated glioblastoma cell motility and proliferation. Cellular Oncology. 39 (3), 229-242 (2016).
- Pace, K. R., Dutt, R., Galileo, D. S. Exosomal L1CAM stimulates glioblastoma cell motility, proliferation, and invasiveness. International Journal of Molecular Sciences. 20 (16), 3982(2019).
- Stoppini, L., Buchs, P. A., Muller, D. A simple method for organotypic cultures of nervous tissue. Journal of Neuroscience Methods. 37 (2), 173-182 (1991).
- Ohnishi, T., Matsumura, H., Izumoto, S., Hiraga, S., Hayakawa, T. A novel model of glioma cell invasion using organotypic brain slice culture. Cancer Research. 58 (14), 2935-2940 (1998).
- Humpel, C. Organotypic brain slice cultures: A review. Neuroscience. 305, 86-98 (2015).
- Aaberg-Jessen, C., et al. Invasion of primary glioma- and cell line-derived spheroids implanted into corticostriatal slice cultures. International Journal of Clinical and Experimental Pathology. 6 (4), 546-560 (2013).
- Matsumura, H., Ohnishi, T., Kanemura, Y., Maruno, M., Yoshimine, T. Quantitative analysis of glioma cell invasion by confocal laser scanning microscopy in a novel brain slice model. Biochemical and Biophysical Research Communications. 269 (2), 513-520 (2000).
- Ren, B., et al. Invasion and anti-invasion research of glioma cells in an improved model of organotypic brain slice culture. Tumori. 101 (4), 390-397 (2015).
- Fayzullin, A., et al. Time-lapse phenotyping of invasive glioma cells ex vivo reveals subtype-specific movement patterns guided by tumor core signaling. Experimental Cell Research. 349 (2), 199-213 (2016).
- Jensen, S. S., et al. Establishment and characterization of a tumor stem cell-based glioblastoma invasion model. PloS One. 11 (7), e0158746(2016).
- Marques-Torrejon, M. A., Gangoso, E., Pollard, S. M. Modelling glioblastoma tumour-host cell interactions using adult brain organotypic slice co-culture. Disease Models & Mechanisms. 11 (2), 031435(2018).
- Tamura, R., et al. Visualization of spatiotemporal dynamics of human glioma stem cell invasion. Molecular Brain. 12 (1), 45(2019).
- Yang, C., et al. Organotypic slice culture based on in ovo electroporation for chicken embryonic central nervous system. Journal of Cellular and Molecular Medicine. 23 (3), 1813-1826 (2019).
- Murrell, W., et al. Expansion of multipotent stem cells from the adult human brain. PloS One. 8 (8), e71334(2013).
- Fotos, J. S., et al. Automated time-lapse microscopy and high-resolution tracking of cell migration. Cytotechnology. 51 (1), 7-19 (2006).
- Tufan, A. C., Akdogan, I., Adiguzel, E. Shell-less culture of the chick embryo as a model system in the study of developmental neurobiology. Neuroanatomy. 3 (1), 8-11 (2004).
- Shoin, K., et al. Chick embryo assay as chemosensitivity test for malignant glioma. Japanese Journal of Cancer Research. 82 (10), 1165-1170 (1991).
- Hagedorn, M., et al. Accessing key steps of human tumor progression in vivo by using an avian embryo model. Proceedings of the National Academy of Sciences. 102 (5), 1643-1648 (2005).
- Balciūniene, N., et al. Histology of human glioblastoma transplanted on chicken chorioallantoic membrane. Medicina. 45 (2), 123-131 (2009).
- De Magalhães, N., et al. Applications of a new In vivo tumor spheroid based shell-less chorioallantoic membrane 3-D model in bioengineering research. Journal of Biomedical Science and Engineering. 3 (1), 20-26 (2010).
- Szmidt, M., et al. Morphology of human glioblastoma model cultured in ovo. Journal of Veterinary Research. 56 (2), 261-266 (2012).
- Jaworski, S., et al. Comparison of tumor morphology and structure from U87 and U118 glioma cells cultured on chicken embryo chorioallantoic membrane. Journal of Veterinary Research. 57 (4), 593-598 (2013).
- Yuan, Y. J., Xu, K., Wu, W., Luo, Q., Yu, J. L. Application of the chick embryo chorioallantoic membrane in neurosurgery disease. International Journal of Medical Sciences. 11 (12), 1275-1281 (2014).
- Urbańska, K., et al. The effect of silver nanoparticles (AgNPs) on proliferation and apoptosis of in ovo cultured glioblastoma multiforme (GBM) cells. Nanoscale Research Letters. 10, 98(2015).
- DeBord, L. C., et al. The chick chorioallantoic membrane (CAM) as a versatile patient-derived xenograft (PDX) platform for precision medicine and preclinical research. American Journal of Cancer Research. 8 (8), 1642-1660 (2018).
- Han, J. M., Jung, H. J. Synergistic anticancer effect of a combination of berbamine and arcyriaflavin A against glioblastoma stem-like cells. Molecules. 27 (22), 7968(2022).
- Ruiz-Ontañon, P., et al. Cellular plasticity confers migratory and invasive advantages to a population of glioblastoma-initiating cells that infiltrate peritumoral tissue. Stem Cells. 31 (6), 1075-1085 (2013).
- Cage, T. A., et al. Distinct patterns of human medulloblastoma dissemination in the developing chick embryo nervous system. Clinical & Experimental Metastasis. 29 (4), 371-380 (2012).
- Darrigues, E., et al. Biobanked glioblastoma patient-derived organoids as a precision medicine model to study inhibition of invasion. International Journal of Molecular Sciences. 22 (19), 10720(2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon