このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
遺伝子発現解析のための胚ゼブラフィッシュとcDNA合成からのRNA分離
要約
高品質の単離は、インタクトなRNAは、多くの実験室のプロトコルで必須の工程である。ここで、我々は全体のゼブラフィッシュの胚と遺伝子発現のマイクロアレイ解析を含む様々な実験手順で、後続のアプリケーションのためのcDNA合成からのRNA抽出を示しています。
要約
多くの重要かつ複雑な実験手順は、高品質でインタクトなRNAの入力が必要です。劣化したサンプルや不純物の存在は、下流の実験的なアプリケーションでは悲惨な結果につながることができます。それは、優れたサンプルを確保するために多数の保護及び品質管理チェックと固体のテクニックを使用するために最も重要なこと、それゆえです。ここで、我々詳細商用RNA単離キットを用いてDNAと不純物の痕跡を除去するために市販の化学変性剤とそれに続くクリーンアップを使用して全体のゼブラフィッシュの胚からトータルRNAを分離するためのプロトコル。としてRNAは、直接RNA鋳型から合成されたcDNA産物を使用するほとんどのプロトコルはアッセイの遺伝子の発現は比較的不安定で、RNアーゼによる切断を容易に発生しやすくなります。我々は、詳細手順を市販のキットを使用して、より安定したcDNA産物にRNAに変換する。これらの手順を通して、サンプルは劣化や汚染がないことを保証するために数多くの品質管理チェックがあります。これらのプロトコルの最終製品は、マイクロアレイ解析、RT - PCRまたは長期保存に適しているcDNAである。
プロトコル
パート1:TRIzol試薬を用いてトータルRNAの抽出
RNAを扱うときには、RNaseのない環境で動作するように非常に重要です。このようなRNAの手順でのみ使用するために予約されたピペットを持つと手順を開始する前に除染装置試薬(例えば、RNアーゼアウェイ)で作業領域をスプレーのような単純な予防策は非常に有用です。
- 1.5mlマイクロチューブにRNAプール50ゼブラフィッシュの胚の十分な量を達成し、ピペットでできるだけ多くの水として削除する。
- すぐに胚を含んだマイクロ遠心チューブにTRIzol試薬1の250μlを加える。 TRIzol試薬はフェノールが含まれており、唯一のヒュームフードの下で使用する必要があります。
- 組織が十分に中断されるまで、ペレット乳棒(約20ストローク)で胚を溶解し、ホモジナイズする。
- 細胞が十分に均質化されている場合、1mlの総体積と等しくなるように750μlのTRIzol試薬を加える。
- 核タンパク質複合体の完全な解離を許可するには、室温で5分間均質化した試料をインキュベートする。この段階では、サンプルはフラッシュが液体窒素で凍結し、-80℃またはプロトコルを継続することができます保存することもできます。
- クロロホルム0.2 mlを追加し続けると混合するために15秒間チューブをロックする。
- 4℃で15分間12,000 xgで遠心分離後、室温で2分間サンプルをインキュベートし、℃の
- 混合物は、下の赤色のフェノールクロロホルム相、中間相、そして無色の上部の水相に分離します。上部の水相は、RNAが含まれています。最上層は、TRIZOLの初期体積の約60%(約0.6 ml)を含むべきである。相間の層のいずれかを転送しないように注意しながら1 mlのピペットを使用して新しい微量遠心管に上部の水層を移す。
- RNAは0.5 mlのイソプロパノールを加える沈殿する。
- サンプルは室温で10分間座って、その後4℃で10分間、12,000 xgでサンプルを遠心分離することができます℃をRNAは、チューブの底にゲル状のペレットを形成します。
- ペレットが剥がれないように、ピペットで上清を除去し、1mlの75%エタノールでペレットを洗浄する。 4℃で5分間7,500 × gで穏やかに転倒し、遠心して、サンプルを混ぜる℃に
- 遠心分離後、ピペットでエタノールを除去し、10分間反転しながら、サンプルが乾燥することができます。
- RNaseフリー水100μlを添加してペレットを再懸濁し、10分間55℃でサンプルをインキュベートする。それは、RNAの水分補給を支援するためにインキュベーション中に頻繁に渦を指することをお勧めします。必要に応じて、ナノドロップND - 1000分光光度計を使用して、サンプルの質と量を確認することができますか、直接次の手順に進みます。
パート2:キアゲンRNeasyミニキットを使用してRNAクリーンアップ
キアゲン社RNeasyミニキット2は、他のメソッドから単離したRNAサンプルから汚染物質や不純物を除去するのに便利です。この手順では、β-メルカプトエタノール、エタノール、およびヌクレアーゼフリー水はキットに含まれるコンポーネントに加えて必要とされる。オプションのDNase処理は、この手順の実行中に推奨されます。
- キットの初回使用時に、バッファのRPEは、Buffer RPEの1ボリュームに100%エタノールの4倍量を加えて調製する必要があります。
- 手続きの日に、のBuffer RLTは新鮮に準備する必要があります。必要とされるのBuffer RLTの合計金額を計算する。 350μlの量は、サンプルごとに必要とされる。のBuffer RLTを準備するには、バッファの各1 mlのβ-メルカプトエタノール10μlを加える。これは、ヒュームフードの下で行われる必要があります。
- 各サンプルにのBuffer RLTおよび100%エタノール250μlの350μlを加える。数回上下にピペッティングしてよく混ぜる。
- 室温で1分間、8,000 xgで2 mlのコレクションチューブと遠心分離機内に配置されRNeasyカラムに、約700μlのあるべきサンプルを、転送する。
- 遠心分離した後、フロースルーを捨てて、RNeasyスピンカラムにBuffer RW1 700μlを加える。
- 室温で1分間、8,000 × gでサンプルを遠心し、フロースルーを捨てる。
- キアゲンのRNaseフリーDNaseキットによるDNase処理:DNase処理はオプションですが強く、最高品質のサンプルをお勧めします。
- 最初にDNase処理のキットを受け取るときには、DNアーゼは550μlヌクレアーゼフリー水を添加してのDNase Iストック溶液を準備しておく必要があります。反転によって静かに混和し、ボルテックスしないでください。ストック溶液の10μlを含む単回使用のバイアルに分注し。は、最大9ヶ月まで-20℃でストック溶液を保存することができます。融解したアリコートを必要に応じて6週間に4℃で保存することができますが、解凍後にアリコートを再凍結しないでください。
- のDNase Iインキュベーションミックスを準備するには10μlのDNase IをsにRDD 70μlのBufferを加えるサンプルあたりトクソリューション。静かに転倒し、遠心簡単にチューブ残液の形態チューブの側面を収集することによりミックス。直接RNeasyスピンカラムに80μlののDNase Iインキュベーションミックスを追加。室温で30分間DNase処理をインキュベートする。
- DNase処理後に室温で1分間8000 × gでスピンカラムや遠心に350μlのBuffer RW1を追加します。
- 遠心分離後にフロースルーを捨てて、スピンカラムに500μlのBuffer RPEを追加。室温で5分間サンプルをインキュベートする。
- 室温で1分間、8,000 × gでサンプルを遠心し、フロースルーを捨てる。
- 遠心分離後にスピンカラムに500μlの75%エタノールを加える。
- 8000 × gで遠心操作を繰り返しますが、室温で2分間。
- 遠心分離後にフロースルーを捨てて、列が5分間乾燥させます。
- すぐにエタノールの痕跡を介して任意の左を削除するには、室温で5分間8000 × gで遠心。
- 遠心分離が完了したら新しい1.5 mlのコレクションチューブにスピンカラムを移し、10μlのヌクレアーゼフリー水を加える。 1分間インキュベートし、次いで室温で1分間10,000 × gで遠心。 RNAはフロースルーになります。
- スピンカラムにヌクレアーゼフリー水のさらに10μlを加え、室温で1分間インキュベートする。室温で1分間、10,000 × gでサンプルを遠心分離します。
- 遠心分離後にスピンカラムを削除し、破棄する。 RNAはフロースルーになります。
- 製造元の指示に従って光度計ND - 1000分光光度計を用いてRNAの質と量を確認してください。
- さらに、変性ゲルを実行するか、RNAの完全性をチェックするためにAgilent 2100バイオアナライザを使用してください。
- cDNA合成を行うことができるようになるまでサンプルを-80℃で保存してください。
パート3:インビトロジェンのSuperScriptファーストストランドシステムを使用してcDNAを合成
RNAが分解につながるアーゼによる切断を容易に発生しやすくなります。その結果、多くの遺伝子発現のプロトコルは、直接RNAから合成され、より安定したcDNA産物を使用するために開発されている。ここでは詳細インビトロジェンのSuperScriptファーストストランド合成システム3を使用して第一鎖cDNAの合成を。それぞれのcDNA合成反応は、5μgのRNAを収容することができます。
- 手続きのミックスを開始する前に、手短に各コンポーネントを遠心してください。
- 表1に示すように滅菌0.5 mlのマイクロ遠心チューブにRNA /プライマー混合物を準備します。
- ℃で5分間65℃でサンプルをインキュベートする。
- 直ちに10分間氷スラリーにサンプルを移す。
- サンプルは、表2に示す成分とマスターミックスを調製冷却されている間。ボリュームは、1反応あたりに表示されます。反応の合計数に応じてボリュームを調整します。
- 冷却後、各RNA /プライマー混合物に、手順5で調製したマスターミックスの9μlを加える。穏やかに混合し、短時間の遠心分離によって収集。各サンプルの合計量は19μLにする必要があります。
- サーマルサイクラーで2分間42℃でサンプルをインキュベートする。
- 、各チューブにのSuperScript II RTを1μl(50ユニット)を追加ミックス、および42℃でインキュベートサーマルサイクラーで60分間。
- 70で反応を終了して℃で15分間、4℃の寒さのサンプルクラーは、手順8および9のためにプログラムすることができます。
- サンプルは4 ° Cに達した後、氷浴に反応を転送する。
- 氷の上で各試験管にRNase Hを1μlを追加し、37℃で20分間インキュベート℃に総量は、現在21μlです。あなたのサンプルにRNase Hの加算は、RNAテンプレートを低下させ、唯一の一本鎖cDNA産物を残す。
パート4:cDNAの単離と降水量
- 1.5 mlの位相ロックゲルチューブは、cDNA産物の単離に使用されます。 2分間、12,000 xgで遠心分離によって1.5 mlの位相ロックゲルチューブを準備します。ゲルは、すべてのチューブの底になっているはず。
- 、(トリス飽和、pHを8.0にバッファリング)81.5μlのフェノール24:1クロロホルムイソアミルアルコールの81.5μL、および位相ロックゲルチューブにサンプル(21μl)を追加します。穏やかに転倒を数回混ぜる。
- 室温で5分間、12,000 xgでサンプルを遠心分離します。
- cDNAは、上部の水相にする必要があります。新しい1.5 mlのマイクロ遠心チューブに上層を移す。総量は20μlの前後となるはずです。
- 3 M NaOAc、pHが5.2の1X量を加え、穏やかに混ぜる。上部の水相の体積は20μlに等しいだった場合例えば、、あなたは3 M NaOAc、pHが5.2の2μlを追加する必要があります。
- 5 mg / mlのグリコーゲンの7μlを加え、穏やかに混合する。
- イソプロパノールの1X量を加え、穏やかに混ぜる。一般的にこれは、イソプロパノールの約30μlです。
- サンプルは、部屋のtemperaturでインキュベートすることができますその後10分間eとは、室温で20分間12,000 xgで遠心。
- 小さなcDNAのペレットは、チューブの底に形成すべきである。注意深くピペットで上清を取り除く。
- 70%エタノールと反転して混合液500μlを追加。
- 5分間12,000 xgでサンプルを遠心分離します。
- 遠心分離後にピペットで上清を除去し、70%エタノールで洗浄し、遠心分離を(ステップ10と11)を繰り返します。
- 遠心分離後にピペットで上清を取り除く。管の両側に残っている液体がある場合、それは簡単にチューブを回転させて収集することができます。余剰な溶液を取り除き、ペレットを室温で5分間乾燥させます。
- ペレットを溶解するヌクレアーゼフリー水20μlを添加する。ペレットの水分補給のための5分間55℃でサンプルを置きます。
- cDNA産物の量と質は、製造元の指示に従って、光度計ND - 1000分光光度計を用いてチェックされます。 cDNAは-20℃で保存することができます
代表的な結果
RNAのクリーンアップの後、高品質のトータルRNAの約15μgのは50ゼブラフィッシュの胚から期待できる。トータルRNAの品質、分光光度計、ゲル電気泳動、およびAgilent 2100バイオアナライザを評価するために使用することができます。光度計ND - 1000分光光度計は280nm(図1)の吸光度と比較して260nmの吸光度を評価するために使用することができます。 280分の260の比率は、汚染物質の存在を明らかにし、可能な限り劣化の証拠を与えることができます。 260 / 1.9から2.0までの280比率は許容範囲とみなされます。さらに、それは非常にRNAの変性ゲルはRNAの完全性を評価するために実行することをお勧めします。 RNA鎖は小さい鎖はゲルさらに下に移行して、そのサイズに基づいて分離します。非分解サンプルでは、2つの強力なバンドとの塗抹標本として表示されます。塗抹標本は、サイズの異なるmRNAの鎖と28Sと18SのrRNAのに対応するバンドの人口の結果である。 28Sのバンドは、18Sのバンド(図2)などの約二倍強にする必要があります。また、トータルRNAの完全性は、Agilent 2100バイオアナライザを用いて評価することができる。 28Sと18SのrRNAを表す2つの明確なピークは(図3)期待されています。 28Sのピークはそれぞれ、約2:1のピーク下の面積と18Sのピークよりも大きくする必要があります。二つの異なるピークが低下した試料では観察されません。劣化を示すRNAサンプルは、その後の実験的な手続きを経て実施されるべきではない。
分光光度分析は、cDNA産物の品質(図4)を評価するために使用することができます。 280分の260は、1.8前後となるはずです。トータルRNAの5μgの入力を有するcDNA反応が日常的に我々の研究室ではcDNA産物の1〜2μgのをもたらす。
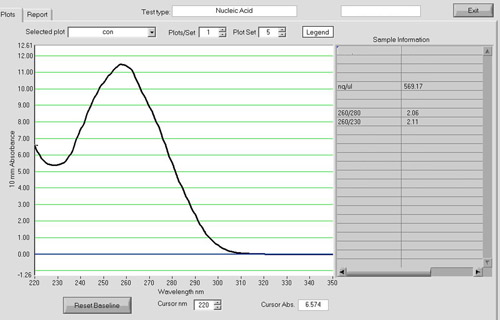
図1。 RNAサンプルの光度計のスペクトル。RNAのクリーンアップの後、RNAの量と質は、光度計ND - 1000分光光度計を用いて評価することができる。高品質のトータルRNAの約15μgのは、日常的に1.9から2.0前後280分の260比で50ゼブラフィッシュ胚から得られたれる。
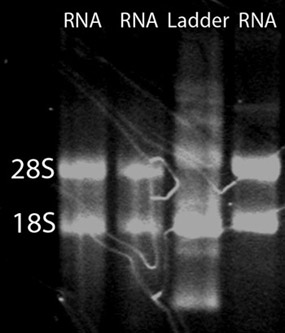
図2 RNAの変性ゲル。トータルRNAの分離の整合性を評価するために、RNAの変性ゲルを実行することができます。 28Sと18SのrRNAに対応する2つの明るいバンドとスミアが期待されている。 28Sのバンドは、18Sのバンドの約2倍強にする必要があります。
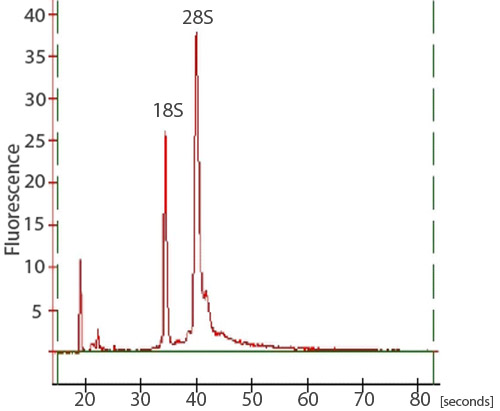
図3。バイオアナライザによるRNAサンプルのトレース。トータルRNAの分離の整合性を評価するために、サンプルも、Agilent 2100バイオアナライザで分析することができる。 28Sと18SのrRNAのに対応する2つのシャープなピークが、、目に見えるはずです。 28Sのピークはそれぞれ、約2:1のピーク下の面積と18Sのピークよりも大きくする必要があります。
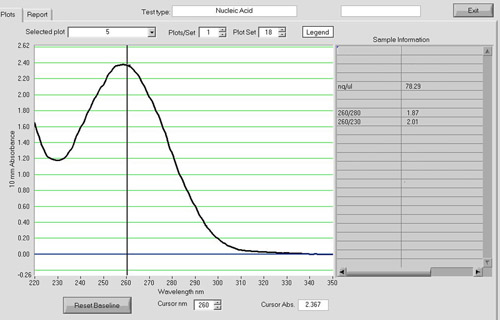
図4。 cDNAサンプルの光度計のスペクトル。光度分析光度計ND - 1000とは、cDNA産物の量と質を評価するために使用することができます。 280分の260の比は、1.8前後となるはずです。トータルRNAの5 UGの入力を有するcDNA反応が日常的に我々の研究室のcDNA 1-2 UGをもたらす。
表1は、。部品cDNA合成のステップ2でRNA /プライマー混合物について追加する。
| コンポーネント | ボリューム |
| トータルRNA(最大5μgの) | 10μlの |
| の10mM dNTPミックス | 1μL |
| オリゴ(dT)12-18(0.5μg/μLの) | 1μL |
| DEPC処理水 | μL のn |
| 合計ボリューム | 10μlの |
cDNA合成のステップ5の表2。反応混合物。記載されているボリュームは、反応毎にあります。反応の総数については、各成分の量を増やします。
| コンポーネント | ボリューム |
| 10X RTバッファー | 2μlの |
| 25 mMのMgCl2を | 4μlの |
| 0.1 mMのDTT | 2μlの |
| RNAseOUT | 1μL |
Access restricted. Please log in or start a trial to view this content.
ディスカッション
RNA分子の脆弱性は、このプロトコルを通じて忘れてはならない最も重要な考慮事項です。 RNaseフリーである滅菌装置は、常に研究室のRNaseフリーゾーンで使用する必要があります。 RNAのみの手続きに使用する実験室の一部を確保しておくと便利です。この領域は頻繁にアウェイなどRNaseなどRNアーゼ除去製品、を散布してください。 RNAサンプルは常に手袋を使用して処理し、劣化を防ぐため?...
Access restricted. Please log in or start a trial to view this content.
開示事項
The authors have nothing to disclose.
資料
| Name | Company | Catalog Number | Comments |
| Glycogen (5 mg/ml) | Ambion | 9510 | |
| NanoDrop ND-100 | Thermo Fisher Scientific, Inc. | ND-1000 | |
| Pellet Pestles with Microfuge Tube | VWR international | KT749520-0090 | |
| Phase Lock Gel Light, 1.5 ml | Eppendorf | 2302800 | |
| Phenol, Tris-saturated | Roche Group | 3117944001 | |
| RNAse Away | VWR international | 17810-491 | |
| RNAse-Free DNase Set | Qiagen | 79254 | |
| RNEasy Mini Kit | Qiagen | 74104 | |
| SuperScript® First-Strand Synthesis System for RT-PCR | Invitrogen | 11904-018 | |
| TRIzol | Invitrogen | 15596-026 |
参考文献
- TRIzol Reagent Manual. , (2007).
- RNEasy Mini Handbook. , 4th ed, (2006).
- SuperScript First-Strand Synthesis System for RT-PCR. , Version E. (2003).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved