È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Isolamento RNA da embrionali Sintesi Zebrafish e cDNA per l'analisi di espressione genica
In questo articolo
Riepilogo
L'isolamento di alta qualità, intatto RNA è un passo essenziale in molti protocolli di laboratorio. In questo studio dimostriamo estrazione di RNA da embrioni di zebrafish tutto e sintesi del DNA per la successiva applicazione in diverse procedure sperimentali inclusa l'analisi microarray di espressione genica.
Abstract
Molte procedure di laboratorio importanti e complesse richiedono un input di alta qualità, l'RNA intatto. Un campione degradato o la presenza di impurità può portare a risultati disastrosi in valle applicazioni sperimentali. E 'quindi, di estrema importanza di utilizzare tecniche solido con numerose garanzie e controlli di qualità per assicurare un campione superiore. Qui, abbiamo dettaglio un protocollo per isolare RNA totale da embrioni di zebrafish tutto utilizzando una sostanza chimica denaturante disponibile in commercio e successiva pulizia per rimuovere le tracce di DNA e le impurità con un kit di isolamento commerciale RNA. Poiché l'RNA è relativamente instabile e facilmente soggetta a scissione da RNasi, più saggio di protocolli di espressione genica utilizzando un prodotto che è direttamente cDNA sintetizzato da un modello di RNA. Noi dettaglio una procedura per convertire l'RNA nel prodotto più stabile cDNA utilizzando un kit disponibile in commercio. In tutto queste procedure ci sono numerosi controlli di qualità per garantire che il campione non è degradato o contaminato. Il prodotto finale di questi protocolli è cDNA che è adatto per l'analisi microarray, RT-PCR o conservazione a lungo termine.
Protocollo
Parte 1: Estrazione di RNA totale usando il reagente TRIzol
Quando si lavora con l'RNA è molto importante lavorare in un ambiente che sia privo di RNasi. Semplici precauzioni come avere pipette riservati per l'utilizzo solo con le procedure di RNA e spruzzare l'area di lavoro con un reagente decontaminante (ad esempio, RNAse Away) prima di iniziare la procedura sono molto utili.
- Per ottenere una sufficiente quantità di RNA piscina 50 embrioni di pesce zebra in una provetta da 1,5 ml microcentrifuga e rimuovere l'acqua il più possibile con una pipetta.
- Immediatamente si aggiungano 250 microlitri di reagente TRIzol 1 alla provetta da microcentrifuga contenente gli embrioni. Reagente TRIzol contiene fenolo e dovrebbe essere usato solo sotto una cappa aspirante.
- Lyse e omogeneizzare gli embrioni con un pestello di pellet (circa 20 colpi) fino a quando il tessuto è sufficientemente perturbato.
- Quando le cellule sono sufficientemente omogeneo, aggiungere 750 microlitri di reagente TRIzol pari a un volume totale di 1 ml.
- Per consentire completa dissociazione dei complessi nucleoproteina, incubare campioni omogeneizzati per 5 minuti a temperatura ambiente. In questa fase, il campione può essere istantaneamente congelati in azoto liquido e conservati a -80 ° C o il protocollo può essere continuato.
- Per continuare ad aggiungere 0,2 ml di cloroformio e rock il tubo per 15 secondi per mescolare.
- Incubare il campione per 2 minuti a temperatura ambiente e poi centrifugare a 12000 xg per 15 minuti a 4 ° C.
- La miscela si separa in basso un rosso fenolo-cloroformio fase, un interfase, e una fase superiore acquosa incolore. La fase superiore acquosa contenente l'RNA. Lo strato superiore dovrebbe comprendere di circa il 60% del volume iniziale di TRIzol (circa 0,6 ml). Trasferire lo strato superiore acquoso in una provetta per microcentrifuga con una nuova pipetta da 1 ml, facendo attenzione a non trasferire dello strato di interfase.
- Per precipitare l'RNA aggiungere 0,5 ml di isopropanolo.
- Lasciare il campione a temperatura ambiente per 10 minuti, quindi si centrifuga il campione a 12.000 xg per 10 minuti a 4 ° C. L'RNA si forma un gel simile a pellet sul fondo della provetta.
- Senza disturbare il pellet, rimuovere il surnatante con una pipetta e lavare il pellet in 1 ml di etanolo al 75%. Mescolare il campione capovolgendo delicatamente e centrifugare a 7500 xg per 5 minuti a 4 ° C.
- Dopo la centrifugazione, rimuovere l'etanolo con una pipetta e lasciare campione asciutto mentre invertita per 10 minuti.
- Risospendere il pellet aggiungendo 100 ml di acqua senza RNasi e incubare il campione a 55 ° C per 10 minuti. Si raccomanda di dito vortice di frequente durante l'incubazione per aiutare nella reidratazione RNA. Opzionalmente, è possibile verificare la qualità e la quantità del campione utilizzando un NanoDrop ND-1000 spettrofotometro o direttamente passare alla fase successiva.
Parte 2: RNA di pulizia con il Qiagen RNeasy Mini Kit
Il Qiagen RNeasy Mini Kit 2 è utile nella rimozione di contaminanti e le impurità da un campione di RNA isolato da altri metodi. Per questa procedura, β-mercaptoetanolo, etanolo ed acqua priva di nucleasi sono necessari in aggiunta ai componenti inclusi nel kit. Un trattamento opzionale DNAsi è consigliato durante questa procedura.
- Al primo utilizzo del kit, Buffer RPE dovranno essere preparati con l'aggiunta di 4 volumi di etanolo 100% per 1 volume di Buffer RPE.
- Il giorno della procedura, buffer RLT dovrà essere preparato fresco. Calcolare la quantità totale di RLT tampone che saranno necessarie. Un volume di 350 microlitri è necessaria per campione. Per preparare RLT tampone, aggiungere 10 ml di β-mercaptoetanolo per ogni 1 ml di tampone. Questo dovrebbe essere fatto sotto la cappa.
- Per ogni campione aggiungere 350 ml di RLT tampone e 250 ml di etanolo al 100%. Mescolare bene pipettando su e giù parecchie volte.
- Trasferire il campione, che dovrebbe essere di circa 700 microlitri, in una colonna RNeasy che è posto in un tubo di raccolta da 2 ml e centrifugare a 8000 xg per 1 minuto a temperatura ambiente.
- Dopo centrifugazione, scartare il flusso attraverso e aggiungere 700 ml di tampone RW1 a una colonna di spin RNeasy.
- Centrifugare il campione a 8000 xg per 1 minuto a temperatura ambiente e poi scarta il flusso attraverso.
- Trattamento con DNAsi Qiagen RNasi-free DNAse Kit: DNAsi trattamento è facoltativo, ma è altamente consigliato per i campioni di più alta qualità.
- La prima volta che riceve il kit di trattamento DNAsi, è necessario preparare il brodo DNAsi I soluzione con l'aggiunta di 550 microlitri acqua priva di nucleasi alla DNAsi. Mescolare delicatamente per inversione e non vortice. Aliquota in fiale monouso contenenti 10 ml di soluzione di riserva. È possibile memorizzare soluzione di riserva a -20 ° C per un massimo di 9 mesi. Le aliquote scongelato può essere conservato a 4 ° C per 6 settimane, se necessario, ma non ricongelare le aliquote dopo lo scongelamento.
- Per preparare l'incubazione DNAsi impasto aggiungere 70 microlitri Buffer RDD a 10 microlitri DNAsi I ssoluzione tac per campione. Mescolare delicatamente per inversione e provetta leggermente per raccogliere il residuo forma liquida i lati del tubo. Aggiungere 80 microlitri DNAsi mix di incubazione io direttamente alla colonna di spin RNeasy. Incubare il trattamento DNAsi per 30 minuti a temperatura ambiente.
- Dopo il trattamento DNAsi aggiungere 350 microlitri RW1 Buffer alla colonna di girare e centrifugare a 8000 xg per 1 minuto a temperatura ambiente.
- Dopo la centrifugazione eliminare il flusso attraverso e aggiungere 500 microlitri RPE Buffer alla colonna. Incubare campione per 5 minuti a temperatura ambiente.
- Centrifugare campione a 8000 xg per 1 minuto a temperatura ambiente ed eliminare il flusso attraverso.
- Dopo la centrifugazione aggiungere 500 microlitri di etanolo al 75% alla colonna di spin.
- Ripetere centrifugazione a 8.000 xg, ma per 2 minuti a temperatura ambiente.
- Dopo la centrifugazione eliminare il flusso attraverso la colonna e permettere ad asciugare per 5 minuti.
- Immediatamente centrifugare a 8.000 xg per 5 minuti a temperatura ambiente di togliere qualunque sinistra su tracce di etanolo.
- Dopo la centrifugazione è il trasferimento completo della colonna girare in un nuovo tubo di 1,5 ml di raccolta e aggiungere 10 microlitri acqua priva di nucleasi. Incubare per 1 minuto, quindi si centrifuga a 10.000 xg per 1 minuto a temperatura ambiente. L'RNA sarà il flusso attraverso.
- Aggiungere un altro 10 microlitri di acqua priva di nucleasi alla colonna di spin e incubare a temperatura ambiente per 1 minuto. Centrifugare campione a 10.000 xg per 1 minuto a temperatura ambiente.
- Dopo la centrifugazione rimuovere ed eliminare la colonna. L'RNA sarà il flusso attraverso.
- Controllare la quantità e la qualità del RNA utilizzando un NanoDrop ND-1000 spettrofotometro seguendo le istruzioni del produttore.
- Inoltre, eseguire un gel denaturante o utilizzare un Agilent 2100 Bioanalyzer per verificare l'integrità del RNA.
- I campioni devono essere conservati a -80 ° C fino a quando la sintesi del DNA può essere eseguita.
Parte 3: Sintesi cDNA utilizzando il Invitrogen SuperScript First-Strand sistema
RNA è facilmente soggetta a scissione da RNasi che porta al degrado. Di conseguenza molti protocolli di espressione genica sono stati sviluppati per utilizzare un prodotto più stabile del DNA che è stato sintetizzato direttamente dal RNA. Qui la sintesi dettaglio del primo filamento di cDNA utilizzando il Invitrogen SuperScript First-Strand sistema di sintesi 3. Ogni reazione di sintesi del DNA in grado di ospitare fino a 5 mg di RNA.
- Prima di iniziare la procedura di mix e centrifugare brevemente ciascun componente.
- Preparare RNA / Primer miscela in una provetta sterile 0,5 ml microcentrifuga come indicato nella Tabella 1.
- Incubare il campione a 65 ° C per 5 minuti.
- Immediatamente il trasferimento del campione di liquami ghiaccio per 10 minuti.
- Mentre il campione è di raffreddamento preparare un master mix con i componenti descritti nella tabella 2. I volumi sono elencati per 1 reazione. Regolare i volumi di conseguenza per il numero totale di reazioni.
- Dopo il raffreddamento, aggiungere 9 ml di master mix preparato al passaggio 5 per ogni RNA / miscela primer. Mescolare delicatamente e raccogliere per centrifugazione breve. Il volume totale di ogni campione dovrebbe essere 19 microlitri.
- Incubare campione a 42 ° C per 2 minuti in un termociclatore.
- Aggiungere 1 ml (50 unità) di SuperScript II RT di ogni tubo, mixare e incubare a 42 ° C per 60 minuti in un termociclatore.
- Terminare la reazione a 70 ° C per 15 minuti e poi campione freddo a 4 ° C. Un termociclatore può essere programmato per i punti 8 e 9.
- Dopo che il campione ha raggiunto 4 ° C, trasferire la reazione di un bagno di ghiaccio.
- Sul ghiaccio aggiungere 1 ml di RNAsi H a ciascuna provetta ed incubare per 20 minuti a 37 ° C. Il volume totale è ora di 21 microlitri. L'aggiunta di RNAsi H per il campione si degradano il modello di RNA e lasciare solo il prodotto singolo filamento cDNA.
Parte 4: Isolamento cDNA e precipitazioni
- Una fase da 1,5 ml tubo blocco di gel sarà utilizzato per l'isolamento del prodotto cDNA. Preparare una fase da 1,5 ml tubo blocco di gel per centrifugazione a 12.000 xg per 2 minuti. Il gel dovrebbe essere tutti sul fondo della provetta.
- Aggiungi 81,5 fenolo microlitri (Tris-saturi, tamponata a pH 8,0), 81,5 ml di alcool isoamilico 24:1 cloroformio, e il campione (21 microlitri) nel tubo gel fase di blocco. Mescolare più volte per inversione dolce.
- Centrifugare il campione a 12.000 xg per 5 minuti a temperatura ambiente.
- Il cDNA deve essere nella fase superiore acquosa. Trasferire la fase superiore in una provetta da microcentrifuga pulita 1,5 ml. Il volume totale dovrebbe essere di circa 20 l.
- Aggiungi 1X volume di 3 M NaOAc, pH 5.2 e mescolare delicatamente. Per esempio, se il volume della fase acquosa superiore era pari a 20 l, è necessario aggiungere 2 l di 3 M NaOAc, pH 5.2.
- Aggiungere 7 ml di 5 mg / ml di glicogeno e mescolare delicatamente.
- Aggiungi 1X volume di isopropanolo e mescolare delicatamente. Generalmente si tratta di circa 30 ml di isopropanolo.
- Lasciare campione incubare a Temperatura ambientee per 10 minuti e poi centrifugare a 12000 xg per 20 minuti a temperatura ambiente.
- Un piccolo pellet cDNA deve formare sul fondo della provetta. Rimuovere con cautela il surnatante con una pipetta.
- Aggiungere 500 ml di etanolo al 70% e mescolare per inversione.
- Centrifugare il campione a 12.000 xg per 5 minuti.
- Dopo la centrifugazione eliminare il surnatante con una pipetta e ripetere il lavaggio etanolo al 70% e centrifugazione (punti 10 e 11).
- Dopo la centrifugazione eliminare il surnatante con una pipetta. Se vi è il liquido restante ai lati del tubo, può essere raccolto con una breve rotazione del tubo. Rimuovere il liquido in eccesso e lasciare asciugare il pellet a temperatura ambiente per 5 minuti.
- Aggiungere 20 ml di acqua priva di nucleasi per reidratare il pellet. Mettere il campione a 55 ° C per 5 minuti per la reidratazione del pellet.
- La quantità e la qualità del prodotto cDNA viene poi controllato con un NanoDrop ND-1000 spettrofotometro seguendo le istruzioni del produttore. Il cDNA possono essere conservati a -20 ° C.
Rappresentante Risultati
Dopo la pulizia RNA, circa 15 mg di RNA totale di alta qualità si può aspettare da 50 embrioni di zebrafish. Per valutare la qualità totale RNA, uno spettrofotometro, elettroforesi su gel, e un Agilent 2100 Bioanalyzer può essere utilizzato. Un NanoDrop ND-1000 spettrofotometro può essere usato per valutare l'assorbanza a 260 nm rispetto al assorbanza a 280 nm (Figura 1). La A 260 / A 280 rapporto può rivelare la presenza di contaminanti e dare evidenza di possibile degrado. Un A 260 / A 280 rapporto di 1,9-2,0 è considerato accettabile. Inoltre, è altamente raccomandato che un gel di denaturazione RNA da eseguire per valutare l'integrità dell'RNA. Filamenti di RNA separerà sulla base delle loro dimensioni, con i più piccoli filamenti di migrazione più in basso il gel. Un campione non degradati apparirà come uno striscio con due fasce forti. Lo striscio è il risultato di una popolazione di diversi filoni mRNA dimensioni e le bande corrispondono a 28S e 18S rRNA. La band 28S dovrebbe essere circa due volte più intenso come la band 18S (Figura 2). In alternativa, totale integrità dell'RNA può essere valutata utilizzando un Agilent 2100 Bioanalyzer. Due picchi distinti che rappresenta il rRNA 28S e 18S è previsto (Figura 3). Il picco 28S dovrebbe essere maggiore del 18S picco con l'area sotto i picchi di circa 2:1, rispettivamente. Due picchi distinti non saranno osservati nei campioni degradati. Campioni di RNA espone degrado non deve essere effettuata attraverso successive procedure sperimentali.
Analisi spettrofotometriche possono essere usati per valutare la qualità del prodotto cDNA (Figura 4). La A 260 / A 280 dovrebbe essere intorno a 1,8. Reazioni cDNA con un ingresso di 5 mg di RNA totale di routine resa mcg 1-2 di cDNA prodotto nel nostro laboratorio.
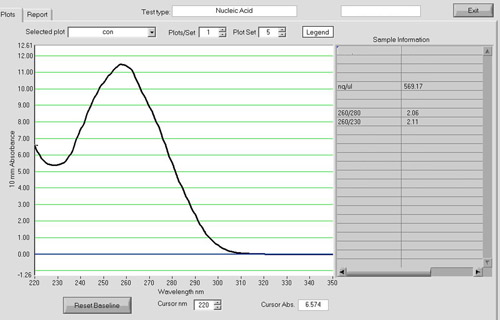
Figura 1. Uno spettro NanoDrop di campioni di RNA. Dopo la pulizia RNA, la quantità di RNA e la qualità può essere valutata utilizzando un NanoDrop ND-1000 spettrofotometro. Circa 15 mg di RNA di alta qualità totale è di routine dato da 50 embrioni di zebrafish con un A 260 / 280 Un rapporto intorno 1,9-2,0.
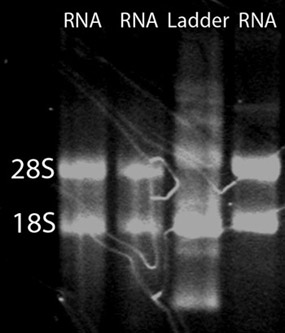
Figura 2. Un gel denaturazione dell'RNA. Per valutare l'integrità del totale isolamento di RNA, un gel denaturazione RNA può essere eseguito. Un striscio con due bande luminose corrispondenti a 28S e 18S rRNA è previsto. La band 28S dovrebbe essere di circa due volte più intenso come la band 18S.
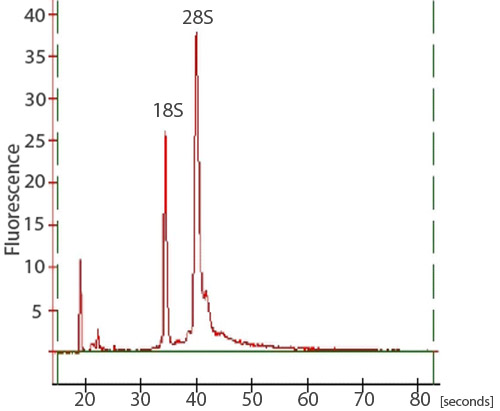
Figura 3. Una traccia di un campione di RNA da un Bioanalyzer. Per valutare l'integrità del totale isolamento dell'RNA, i campioni possono anche essere analizzati su un Agilent 2100 Bioanalyzer. Due cime aguzze, corrispondente al rRNA 28S e 18S, dovrebbe essere visibile. Il picco 28S dovrebbe essere maggiore del 18S picco con l'area sotto i picchi di circa 2:1, rispettivamente.
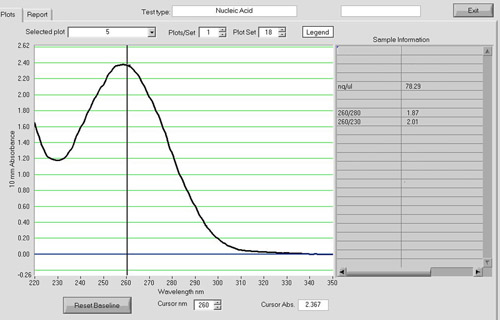
Figura 4. Uno spettro NanoDrop di un campione di cDNA. Analisi spettrofotometrica con un NanoDrop ND-1000 può essere utilizzato per valutare la quantità e la qualità del prodotto cDNA. La A 260 / A 280 rapporto dovrebbe essere intorno a 1,8. Reazioni cDNA con un ingresso di 5 ug di RNA totale di routine resa 1-2 ug di cDNA nel nostro laboratorio.
Tabella 1. Componenti da aggiungere per RNA / miscele di fondo al punto 2 della sintesi del DNA.
| Componente | Volume |
| RNA totale (fino a 5 mg) | 10 microlitri |
| 10 mM dNTP mix | 1 ml |
| Oligo (dT) 12-18 (0,5 mg / mL) | 1 ml |
| DEPC trattati con acqua | n microlitri |
| Volume totale | 10 microlitri |
Tabella 2. Miscela di reazione per il punto 5 della sintesi del DNA. I volumi indicati si intendono per reazione. Aumentare i volumi di ogni componente per il numero totale di reazioni.
| Componente | Volume |
| RT buffer 10X | 2 microlitri |
| 25 mM MgCl2 | 4 microlitri |
| 0,1 mM DTT | 2 microlitri |
| RNAseOUT | 1 ml |
Access restricted. Please log in or start a trial to view this content.
Discussione
La fragilità della molecola di RNA è la considerazione più importante che dovrebbe essere ricordato in tutto questo protocollo. Materiale sterile che viene RNasi-free deve essere sempre utilizzato in un RNAse zona libera del laboratorio. È utile riservare una parte del laboratorio da utilizzare per le procedure di RNA solo. Questa zona deve essere frequentemente spruzzato con un prodotto RNAse rimozione, come RNAse Lontano. I campioni di RNA devono essere sempre trattati con i guanti e tenuta in ghiaccio per evitare...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
The authors have nothing to disclose.
Materiali
| Name | Company | Catalog Number | Comments |
| Glycogen (5 mg/ml) | Ambion | 9510 | |
| NanoDrop ND-100 | Thermo Fisher Scientific, Inc. | ND-1000 | |
| Pellet Pestles with Microfuge Tube | VWR international | KT749520-0090 | |
| Phase Lock Gel Light, 1.5 ml | Eppendorf | 2302800 | |
| Phenol, Tris-saturated | Roche Group | 3117944001 | |
| RNAse Away | VWR international | 17810-491 | |
| RNAse-Free DNase Set | Qiagen | 79254 | |
| RNEasy Mini Kit | Qiagen | 74104 | |
| SuperScript® First-Strand Synthesis System for RT-PCR | Invitrogen | 11904-018 | |
| TRIzol | Invitrogen | 15596-026 |
Riferimenti
- TRIzol Reagent Manual. , (2007).
- RNEasy Mini Handbook. , 4th ed, (2006).
- SuperScript First-Strand Synthesis System for RT-PCR. , Version E. (2003).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon