É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Isolamento de RNA Síntese Zebrafish e cDNA embrionárias para Análise de Expressão Gênica
Resumo
O isolamento de alta qualidade, RNA intacto é um passo essencial para muitos protocolos laboratoriais. Aqui, demonstramos extração de RNA a partir de embriões zebrafish todo e síntese de cDNA para posterior aplicação em vários procedimentos experimentais, incluindo análise de microarray gene de expressão.
Resumo
Muitos procedimentos laboratoriais importantes e complexas exigem um trabalho de alta qualidade, RNA intacto. Uma amostra degradada ou a presença de impurezas pode levar a resultados desastrosos em downstream aplicações experimentais. É, portanto, de extrema importância o uso de técnicas sólidas com inúmeras salvaguardas e verificações de controle de qualidade para garantir uma amostra superior. Neste documento, detalhamos um protocolo para isolar RNA total a partir de embriões zebrafish inteiro usando um desnaturante químico comercialmente disponível e limpeza após a remoção de vestígios de DNA e impurezas através de um kit de isolamento comercial RNA. Como RNA é relativamente instável e facilmente propenso a clivagem pela RNAses, a maioria dos protocolos de expressão gênica de ensaio utilizando um produto que está diretamente cDNA sintetizado a partir de um modelo de RNA. Detalhamos um procedimento para converter RNA no produto mais estável cDNA usando um kit disponível comercialmente. Ao longo destes procedimentos, há inúmeros testes de qualidade de controlo para garantir que a amostra não é degradada ou contaminada. O produto final destes protocolos é cDNA que é adequado para análise de microarray, RT-PCR ou armazenamento de longo prazo.
Protocolo
Parte 1: Extração de RNA total utilizando reagente Trizol
Quando se trabalha com RNA é muito importante trabalhar em um ambiente que está livre de RNAses. Precauções simples, como ter pipetas reservados para uso apenas com os procedimentos de RNA e pulverizar a área de trabalho com um reagente descontaminante (por exemplo, RNAse Fora) antes de iniciar o procedimento são muito úteis.
- Para atingir uma quantidade suficiente de RNA piscina de 50 embriões zebrafish em um microtubo de 1,5 ml e remover tanta água quanto possível com uma pipeta.
- Imediatamente adicione 250 mL de reagente Trizol 1 para o tubo de microcentrífuga contendo os embriões. Reagente Trizol contém fenol e só deve ser utilizado sob uma coifa.
- Lyse e homogeneizar os embriões com um pilão pellet (aproximadamente 20 cursos) até que o tecido é suficientemente perturbada.
- Quando as células são suficientemente homogeneizada, adicionar 750 mL de reagente TRIzol para igualar um volume total de 1 ml.
- Para permitir a dissociação completa dos complexos de nucleoproteínas, incubar amostras homogeneizadas por 5 minutos em temperatura ambiente. Nesta fase, a amostra pode ser flash congelado em nitrogênio líquido e armazenadas a -80 ° C ou o protocolo pode ser continuado.
- Para continuar a adicionar 0,2 ml de clorofórmio e agitar o tubo por 15 segundos para misturar.
- Incubar a amostra por 2 minutos em temperatura ambiente e, em seguida, centrifugar a 12.000 xg por 15 minutos a 4 ° C.
- A mistura será separada em uma fase inferior fenol-clorofórmio vermelho, uma interfase, e uma fase superior aquosa incolor. A fase aquosa superior contém o RNA. A camada superior deve incluir cerca de 60% do volume inicial de TRIzol (aproximadamente 0,6 ml). Transferir a camada superior aquosa para um tubo de microcentrífuga novo usando uma pipeta de 1 ml com cuidado para não transferir qualquer da camada de interfase.
- Para precipitar o RNA adicionar 0,5 ml de isopropanol.
- Permitir que a amostra para sentar-se à temperatura ambiente por 10 minutos e, em seguida, centrifugar a amostra a 12.000 xg por 10 minutos a 4 ° C. RNA formará uma bolinha de gel na parte inferior do tubo.
- Sem perturbar o sedimento, remover o sobrenadante com uma pipeta e lavar o sedimento em 1 ml de etanol 75%. Misturar a amostra por inversão e centrifugar a 7500 xg por 5 minutos a 4 ° C.
- Após a centrifugação, remover o etanol com uma pipeta e permitir a amostra seca ao mesmo tempo invertido por 10 minutos.
- Ressuspender o sedimento, adicionando 100 ul de água livre de RNAse e incubar a amostra a 55 ° C por 10 minutos. Recomenda-se a dedo vortex freqüentemente durante a incubação para ajudar na reidratação RNA. Opcionalmente, você pode verificar a qualidade e quantidade de amostra usando um ND-1000 NanoDrop espectrofotômetro ou diretamente continuar para a próxima etapa.
Parte 2: Limpeza de RNA usando o Qiagen RNEasy Mini Kit
A Qiagen RNEasy Mini Kit 2 é útil na remoção de contaminantes e impurezas de uma amostra de RNA isolado de outros métodos. Para este procedimento, β-mercaptoetanol, etanol e água livre de nuclease são necessários além dos componentes incluídos no kit. Um tratamento DNAse opcional é recomendado durante este procedimento.
- Após a primeira utilização do kit, tampão RPE terá de ser preparado por adição de 4 volumes de etanol 100% para um volume de RPE Buffer.
- No dia do procedimento, tampão RLT terá de ser preparada. Calcule a quantidade total de tampão RLT que serão necessários. Um volume de 350 mL é necessário por amostra. Para preparar RLT Buffer, adicione 10 ml de β-mercaptoetanol para cada 1 ml de buffer. Isto deve ser feito sob a coifa.
- Para cada amostra adicionar 350 uL de tampão RLT e 250 mL de etanol a 100%. Misture bem, pipetando cima e para baixo várias vezes.
- Transferir a amostra, que deve ser de aproximadamente 700 mL, em uma coluna RNEasy que é colocado em um tubo de coleta de 2 ml e centrifugar a 8.000 xg por 1 minuto à temperatura ambiente.
- Após centrifugação, descartar a fluir através de e adicione 700 mL de tampão RW1 a uma coluna de spin RNEasy.
- Centrifugar a amostra a 8.000 xg por 1 minuto à temperatura ambiente e, em seguida, descartar a fluir.
- DNAse tratamento com RNAse Qiagen-Free DNAse Kit: DNAse tratamento é opcional, mas é altamente recomendado para as amostras da mais alta qualidade.
- Quando você receber o kit de tratamento DNAse, você vai precisar para preparar a solução estoque DNAse I, adicionando água 550 mL nuclease-free para a DNAse. Misture delicadamente por inversão e não vortex. Alíquotas em frascos de utilização única contendo 10 ml da solução estoque. Você pode armazenar a solução estoque a -20 ° C por até 9 meses. As alíquotas descongeladas podem ser armazenadas a 4 ° C durante 6 semanas, se necessário, mas não voltar a congelar as alíquotas após o descongelamento.
- Para preparar a mistura de incubação DNAse eu adicionar 70 mL de buffer RDD para 10 ml DNAse I stock solução por amostra. Misture por inversão suave e tubo de centrifugação rapidamente para recolher o líquido residual dos lados do tubo. Adicionar 80 mL DNAse mix de incubação eu diretamente para a coluna de spin RNEasy. Incubar o tratamento DNAse por 30 minutos em temperatura ambiente.
- Tratamento DNAse seguir adicionar 350 mL de buffer RW1 para a coluna de spin e centrifugar a 8.000 xg por 1 minuto à temperatura ambiente.
- Após a centrifugação descartar a fluir através de e adicionar 500 mL de buffer RPE para a coluna de spin. Incubar amostra por 5 minutos em temperatura ambiente.
- Centrifugar a 8.000 xg amostra por 1 minuto à temperatura ambiente e descarte a fluir.
- Após a centrifugação adicionar 500 mL de etanol 75% para a coluna de spin.
- Repetir a centrifugação a 8.000 xg, mas por 2 minutos em temperatura ambiente.
- Após a centrifugação descartar a fluir através de e permitir que a coluna para secar por 5 minutos.
- Imediatamente centrífuga a 8.000 xg por 5 minutos em temperatura ambiente para remover qualquer sobra de vestígios de etanol.
- Após a centrifugação é a transferência completa da coluna de spin em um novo tubo de coleta de 1,5 ml de água e adicionar 10 mL livre de nuclease. Incubar por 1 minuto e então centrifugar a 10.000 xg por 1 minuto à temperatura ambiente. O RNA será no fluir.
- Adicionar outro mL 10 de água livre de nuclease para a coluna girar e incubar a temperatura ambiente por 1 minuto. Centrífuga de amostra a 10.000 xg por 1 minuto à temperatura ambiente.
- Após a centrifugação remover e descartar a coluna spin. RNA será no fluir.
- Verificar a quantidade ea qualidade do RNA usando um espectrofotômetro ND-1000 NanoDrop seguindo as instruções do fabricante.
- Além disso, executar um gel desnaturante ou usar um Agilent 2100 Bioanalyzer para verificar a integridade do RNA.
- Amostras devem ser armazenadas a -80 ° C até a síntese de cDNA podem ser executadas.
Parte 3: Síntese de cDNA usando a Invitrogen SuperScript Sistema First-Strand
RNA é facilmente suscetível à clivagem pela RNAses levando à degradação. Como resultado protocolos expressão muitos gene têm sido desenvolvidos para usar um produto mais estável cDNA que foi diretamente sintetizado a partir do RNA. Aqui síntese detalhamos da vertente do primeiro cDNA usando a Invitrogen SuperScript Sistema de Síntese First Strand-3. Cada reação de síntese de cDNA pode acomodar até 5 mg de RNA.
- Antes de iniciar o procedimento de mistura e brevemente centrífuga cada componente.
- Prepare RNA / mistura de primers num tubo de microcentrífuga estéril de 0,5 ml, conforme descrito na Tabela 1.
- Incubar a amostra a 65 ° C por 5 minutos.
- Imediatamente para transferir a pasta de gelo por 10 minutos.
- Enquanto a amostra está esfriando preparar uma mistura principal com os componentes descritos na Tabela 2. Os volumes são listados por 1 reação. Ajustar volumes em conformidade, para o número total de reações.
- Após o resfriamento, adicionar 9 mL da mistura principal preparada na etapa 5 para cada RNA / mistura primer. Misture delicadamente e recolher por centrifugação breve. O volume total de cada amostra deve ser de 19 mL.
- Incubar amostra a 42 ° C por 2 minutos em um termociclador.
- Adicionar 1 ml (50 unidades) de SuperScript II RT para cada tubo, misturar e incubar a 42 ° C por 60 minutos em um termociclador.
- Finalizar a reação a 70 ° C por 15 minutos e depois relaxar amostra a 4 ° C. A termociclador pode ser programado para as etapas 8 e 9.
- Depois de o provete ter atingido 4 ° C, transferir a reação a um banho de gelo.
- No gelo adicionar 1 ml de RNAse H a cada tubo e incubar por 20 minutos a 37 ° C. O volume total é agora com 21 mL. Além de RNAse H para a amostra irá degradar o modelo de RNA e deixar apenas o produto único fio cDNA.
Parte 4: Isolamento de cDNA e Precipitação
- Um tubo de 1,5 ml de fase gel bloqueio será utilizado no isolamento do produto cDNA. Prepare um tubo de 1,5 ml de fase gel bloqueio por centrifugação a 12.000 xg por 2 minutos. O gel deve ser todos no fundo do tubo.
- Adicionar 81,5 mL de fenol (Tris-saturadas, tamponado a pH 8,0), 81,5 mL de álcool isoamílico 24:1 clorofórmio, e da amostra (21 mL) para o tubo de gel fase de bloqueio. Mix várias vezes por inversão suave.
- Centrifugar a amostra a 12.000 xg por 5 minutos em temperatura ambiente.
- O cDNA deve ser na fase aquosa superior. Transferir a fase superior para um tubo limpo de microcentrífuga de 1,5 ml. O volume total deve ser cerca de 20 mL.
- Adicionar 1X volume de 3 M NaOAc, pH 5,2 e misture delicadamente. Por exemplo, se o volume da fase aquosa superior foi igual a 20 l, você terá que adicionar 2 l da 3 M NaOAc, pH 5.2.
- Adicionar 7 mL de 5 glicogênio mg / ml e misture delicadamente.
- Adicionar 1X volume de isopropanol e misture delicadamente. Geralmente isto é cerca de 30 mL de isopropanol.
- Deixar a amostra para incubar a temperatur salae por 10 minutos e, em seguida, centrifugar a 12.000 xg por 20 minutos em temperatura ambiente.
- A pelota pequena cDNA deve formar no fundo do tubo. Remova cuidadosamente o sobrenadante com uma pipeta.
- Adicionar 500 mL de etanol 70% e misturar por inversão.
- Centrifugar a amostra a 12.000 xg por 5 minutos.
- Após a centrifugação remover o sobrenadante com uma pipeta e repita a lavagem de etanol 70% e centrifugação (etapas 10 e 11).
- Após a centrifugação remover o sobrenadante com uma pipeta. Se houver líquido restante nas laterais do tubo, que podem ser reunidos por breves instantes, girando o tubo. Remova qualquer excesso de líquido e permitir que o pellet secar à temperatura ambiente por 5 minutos.
- Adicionar 20 l de água livre de nuclease para reidratar o sedimento. Colocar a amostra a 55 ° C por 5 minutos para reidratação do sedimento.
- A quantidade ea qualidade do produto cDNA é então verificada usando um espectrofotômetro ND-1000 NanoDrop seguindo as instruções do fabricante. O cDNA pode ser armazenado a -20 ° C.
Resultados representante
Após a limpeza RNA, cerca de 15 mg de RNA total de alta qualidade pode ser esperado de 50 embriões zebrafish. Para avaliar a qualidade do RNA total, um espectrofotômetro, eletroforese em gel, e um Agilent 2100 Bioanalyzer pode ser usado. A ND-1000 NanoDrop espectrofotômetro pode ser usado para avaliar a absorbância a 260 nm, em comparação com a absorvância a 280 nm (Figura 1). O A 260 / A 280 relação pode revelar a presença de contaminantes e dar indícios de degradação possível. Um A 260 / A 280 de 1,9-2,0 é considerada aceitável. Além disso, é altamente recomendável que um gel desnaturação RNA ser executado para avaliar a integridade do RNA. RNA vai separar em função do seu tamanho, com fios menores migrando mais para baixo do gel. A amostra não-degradada aparecerá como uma mancha com duas bandas fortes. O esfregaço é resultado de uma população de diferentes vertentes mRNA tamanho e as bandas correspondem a 28S e 18S rRNA. A banda 28S deve ser cerca de duas vezes tão intensa como a banda 18S (Figura 2). Alternativamente, a integridade do RNA total pode ser avaliada através de um Agilent 2100 Bioanalyzer. Dois picos distintos representando o rRNA 28S e 18S é esperado (Figura 3). O pico 28S deve ser maior do que o pico de 18S com a área sob os picos de aproximadamente 2:1, respectivamente. Dois picos distintos não será observada em amostras degradadas. Amostras de RNA exibindo degradação não deve ser feita através de procedimentos experimentais subseqüentes.
Análise espectrofotométrica pode ser usado para avaliar a qualidade do produto cDNA (Figura 4). O A 260 / A 280 deve ser em torno de 1,8. reações cDNA com uma entrada de 5 mg de RNA total rotineiramente rendimento mg 1-2 de cDNA do produto em nosso laboratório.
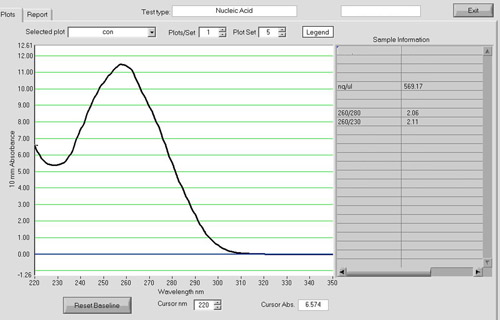
Figura 1. Um espectro NanoDrop de amostra de RNA. Após a limpeza RNA, RNA quantidade e qualidade pode ser avaliada através de um ND-1000 NanoDrop espectrofotômetro. Aproximadamente 15 mg de alta qualidade total RNA é rotineiramente rendeu a partir de 50 embriões zebrafish com um A 260 / A 280 em torno de relação de 1,9-2,0.
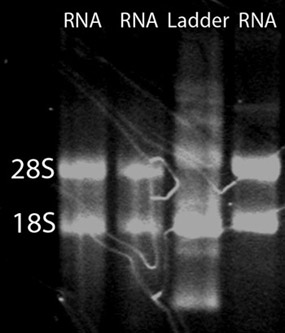
Figura 2. Um gel desnaturação RNA. Para avaliar a integridade do isolamento do RNA total, um gel desnaturação RNA pode ser executado. Uma mancha com duas faixas brilhantes correspondente a 28S e 18S rRNA é esperado. A banda 28S deve ser de aproximadamente duas vezes mais intensa que a banda 18S.
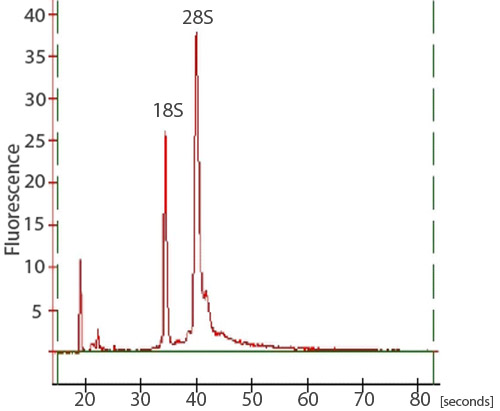
Figura 3. Um traço de uma amostra de RNA a partir de um Bioanalyzer. Para avaliar a integridade do isolamento do RNA total, as amostras também podem ser analisados em um Agilent 2100 Bioanalyzer. Dois picos, correspondentes aos 28S e 18S rRNA, deve ser visível. O pico 28S deve ser maior do que o pico de 18S com a área sob os picos de aproximadamente 2:1, respectivamente.
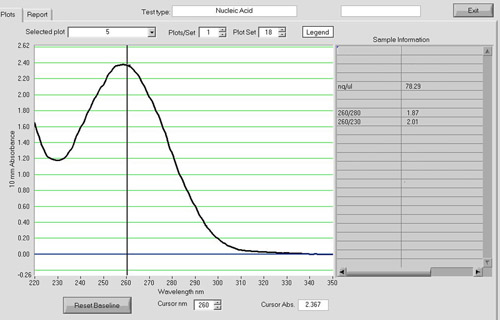
Figura 4. Um espectro NanoDrop de uma amostra de cDNA. Análise espectrofotométrica com um NanoDrop ND-1000 pode ser usado para avaliar a quantidade ea qualidade do produto cDNA. O A 260 / A 280 relação deve ser em torno de 1,8. reações cDNA com uma entrada de 5 ug de RNA total rotineiramente rendimento 1-2 ug de cDNA em nosso laboratório.
Tabela 1. Componentes para adicionar para RNA / misturas de primer no passo 2 da síntese de cDNA.
| Componente | Volume |
| Total de RNA (até 5 mg) | 10 ml |
| 10 mM dNTP mix | 1 ml |
| Oligo (dT) 12-18 (0,5 mg / mL) | 1 ml |
| DEPC tratados com água | mL n |
| Volume total | 10 ml |
Mesa mistura de reação 2. Para a etapa 5 da síntese de cDNA. Volumes indicados são por reação. Aumentar os volumes de cada componente para o número total de reações.
| Componente | Volume |
| Tampão RT 10X | 2 l |
| 25 mM MgCl2 | 4 mL |
| 0,1 mM DTT | 2 l |
| RNAseOUT | 1 ml |
Access restricted. Please log in or start a trial to view this content.
Discussão
A fragilidade da molécula de RNA é o aspecto mais crítico que deve ser lembrado ao longo deste protocolo. Equipamento estéril que é RNAse-free deve ser sempre usada em uma zona livre de RNAse do laboratório. É útil reservar uma parte do laboratório a ser utilizado para procedimentos de RNA apenas. Esta área deve ser freqüentemente pulverizados com um produto de remoção de RNAse, como RNAse Away. As amostras de RNA devem ser sempre manuseadas com luvas e mantidos em gelo para evitar a degradação.
Access restricted. Please log in or start a trial to view this content.
Divulgações
The authors have nothing to disclose.
Materiais
| Name | Company | Catalog Number | Comments |
| Glycogen (5 mg/ml) | Ambion | 9510 | |
| NanoDrop ND-100 | Thermo Fisher Scientific, Inc. | ND-1000 | |
| Pellet Pestles with Microfuge Tube | VWR international | KT749520-0090 | |
| Phase Lock Gel Light, 1.5 ml | Eppendorf | 2302800 | |
| Phenol, Tris-saturated | Roche Group | 3117944001 | |
| RNAse Away | VWR international | 17810-491 | |
| RNAse-Free DNase Set | Qiagen | 79254 | |
| RNEasy Mini Kit | Qiagen | 74104 | |
| SuperScript® First-Strand Synthesis System for RT-PCR | Invitrogen | 11904-018 | |
| TRIzol | Invitrogen | 15596-026 |
Referências
- TRIzol Reagent Manual. , (2007).
- RNEasy Mini Handbook. , 4th ed, (2006).
- SuperScript First-Strand Synthesis System for RT-PCR. , Version E. (2003).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados