Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
RNA Isolation aus embryonalen Zebrafisch und cDNA-Synthese für die Genexpressionsanalyse
In diesem Artikel
Zusammenfassung
Die Isolierung von hoher Qualität, ist intakte RNA ein wesentlicher Schritt in vielen Labor-Protokolle. Hier zeigen wir, die RNA-Extraktion aus ganzen Zebrafisch-Embryos und cDNA-Synthese für die spätere Anwendung in verschiedenen experimentellen Verfahren, einschließlich Genexpression Microarray-Analyse.
Zusammenfassung
Viele wichtige und komplexe Labor-Verfahren erfordern eine Eingabe von hoher Qualität, intakte RNA. A abgebaut Probe oder das Vorhandensein von Verunreinigungen können zu katastrophalen Ergebnissen in nachgelagerten experimentelle Anwendungen führen. Es ist daher von größter Bedeutung, solide Technik mit zahlreichen Sicherheitsvorkehrungen und Qualitätskontrollen zu verwenden, um ein überlegenes Probe zu gewährleisten. Hier berichten wir ausführlich ein Protokoll zur Gesamt-RNA aus ganzen Zebrafisch-Embryos mit einem handelsüblichen chemischen Denaturierungsmittel und anschließender Bereinigung um Spuren von DNA und Verunreinigungen unter Verwendung eines handelsüblichen RNA Isolation Kit entfernen zu isolieren. Als RNA ist relativ instabil und leicht anfällig für Spaltung durch RNAsen, mit den meisten Protokollen Assay Genexpression einer cDNA-Produkt, das direkt von einer RNA-Matrize synthetisiert wird. Wir Detail ein Verfahren zur RNA in die stabilere cDNA-Produkt mit einem kommerziell erhältlichen Kit zu konvertieren. Während diese Verfahren gibt es zahlreiche Qualitätskontrollen, um sicherzustellen, dass die Probe nicht abgebaut wird oder verschmutzt. Das Endprodukt dieser Protokolle cDNA, die für Microarray-Analyse, RT-PCR oder langfristige Lagerung ist.
Protokoll
Teil 1: Extraktion von Gesamt-RNA mit Trizolreagenz
Beim Arbeiten mit RNA ist es sehr wichtig, um in einem Umfeld, das frei von RNasen ist die Arbeit. Einfache Vorsichtsmaßnahmen wie mit reservierten Pipetten für die Verwendung nur mit RNA Verfahren und Besprühen der Arbeitsbereich mit einem Dekontaminationsmittel Reagenz (zB RNAse Away) vor Beginn des Verfahrens sind sehr hilfreich.
- Um eine ausreichende Menge an RNA Becken 50 Zebrafischembryonen in einem 1,5 ml Mikrozentrifugenröhrchen erreichen und entfernen Sie so viel Wasser wie möglich mit einer Pipette.
- Sofort im 250 ul Trizolreagenz 1 zum Mikrozentrifugenröhrchen mit den Embryonen. Trizolreagenz enthält Phenol und sollte nur unter einer Abzugshaube verwendet werden.
- Lyse und Homogenisierung der Embryonen mit einer Pellet-Stößel (ca. 20 Schläge), bis das Gewebe ausreichend gestört wird.
- Wenn die Zellen ausreichend homogenisiert, fügen Sie 750 ul Trizolreagenz auf ein Gesamtvolumen von 1 ml entspricht.
- Um zuzulassen, dass eine vollständige Dissoziation des Nukleoproteinkomplexe inkubieren homogenisierten Proben für 5 Minuten bei Raumtemperatur. In diesem Stadium kann die Probe in flüssigem Stickstoff eingefroren und bei -80 ° C oder das Protokoll fortgesetzt werden kann.
- Um weiterhin 0,2 ml Chloroform und Rock die Röhre für 15 Sekunden zu mischen.
- Inkubieren Sie die Probe für 2 Minuten bei Raumtemperatur und anschließend Zentrifuge bei 12.000 xg für 15 Minuten bei 4 ° C.
- Die Mischung wird in eine untere rote Phenol-Chloroform-Phase, eine Interphase und eine farblose obere wässrige Phase zu trennen. Die obere wässrige Phase enthält die RNA. Die oberste Schicht sollte etwa 60% des ursprünglichen Volumens des TRIzol (ca. 0,6 ml) enthalten. Übertragen Sie die obere wässrige Schicht in ein neues Mikrozentrifugenröhrchen mit einer 1 ml Pipette achten, dass keine der Interphase-Schicht übertragen.
- Zur Fällung der RNA mit 0,5 ml Isopropanol.
- Lassen Sie die Probe bei Raumtemperatur für 10 Minuten sitzen und dann Zentrifuge die Probe bei 12.000 xg für 10 Minuten bei 4 ° C. RNA wird eine gelartige Pellet am Boden des Röhrchens zu bilden.
- Ohne das Pellet, den Überstand mit einer Pipette und waschen das Pellet in 1 ml 75% Ethanol. Mischen Sie die Probe durch vorsichtiges Schwenken und Zentrifuge bei 7.500 xg für 5 Minuten bei 4 ° C.
- Nach Zentrifugation, entfernen Sie das Ethanol mit einer Pipette und ermöglichen Probe zu trocknen, während für 10 Minuten umgedreht.
- Das Pellet durch Zugabe von 100 ul RNAse-freiem Wasser und Inkubation der Probe bei 55 ° C für 10 Minuten. Es wird empfohlen, Wirbel häufig Finger während der Inkubation in RNA Rehydratation Hilfe. Optional können Sie überprüfen Probe qualitativ und quantitativ mit Hilfe eines NanoDrop ND-1000 Spektrophotometer oder direkt mit dem nächsten Schritt fortfahren.
Teil 2: RNA Cleanup mit dem Qiagen RNeasy Mini Kit
Die Qiagen RNeasy Mini Kit 2 ist nützlich bei der Beseitigung von Schadstoffen und Verunreinigungen aus einem RNA-Probe von anderen Methoden isoliert. Für dieses Verfahren sind β-Mercaptoethanol, Ethanol und Nuklease-freies Wasser zusätzlich zu den Komponenten im Kit enthalten nötig. Eine optionale DNAse Behandlung ist bei diesem Verfahren empfohlen.
- Bei der ersten Verwendung des Kits wird Buffer RPE müssen durch Zugabe von 4 Volumen 100% Ethanol zu 1 Volumen von Buffer RPE vorbereitet werden.
- Am Tag des Verfahrens, wird Buffer RLT müssen frisch zubereitet werden. Berechnen Sie den Gesamtbetrag von Buffer RLT, die benötigt werden. Ein Volumen von 350 ul pro Probe benötigt. Zur Vorbereitung Buffer RLT, mit 10 ul von β-Mercaptoethanol für jeden 1 ml Puffer. Dies sollte unter dem Abzug durchgeführt werden.
- Zu jeder Probe hinzugefügt 350 ul Puffer RLT und 250 ul 100% Ethanol. Gut mischen durch Auf-und Abpipettieren mehrmals.
- Übertragen Sie die Probe, die etwa 700 ul sein sollte, in eine RNeasy Säule in ein 2 ml Sammel-Tube und zentrifugieren bei 8.000 xg für 1 Minute bei Raumtemperatur befindet.
- Nach Zentrifugieren, entsorgen Sie die Durchströmung und fügen 700 ul Puffer RW1 eine RNeasy Spin-Säule.
- Zentrifugieren Sie die Probe bei 8.000 xg für 1 Minute bei Raumtemperatur und dann verwerfen Sie die Durchströmung.
- DNAse Behandlung mit Qiagen RNase-freie DNase Kit: DNAse Behandlung ist optional, ist jedoch für die höchste Qualität Proben empfohlen.
- Beim ersten erhalten die DNAse Behandlung Kit, müssen Sie die DNAse I-Stammlösung durch Zugabe von 550 ul Nuklease-freies Wasser auf die DNAse vorzubereiten. Vorsichtig mischen durch Invertieren und nicht vortexen. Aliquot in Durchstechflaschen zur einmaligen Anwendung mit 10 ul der Stammlösung. Sie können die Stammlösung bei -20 ° C lagern bis zu 9 Monate. Die aufgetauten Aliquots bei 4 ° C für 6 Wochen bei Bedarf gespeichert werden, aber nicht wieder einfrieren die Aliquots nach dem Auftauen.
- Zur Vorbereitung der DNAse I Inkubation Mischung hinzufügen 70 ul Puffer RDD bis 10 ul DNAse I stock-Lösung pro Probe. Mix durch vorsichtiges Schwenken und Zentrifugenröhrchen kurz zu sammeln Restflüssigkeit bilden die Seiten des Rohres. Add 80 ul DNAse I Inkubation Mix direkt auf die RNeasy spin column. Inkubieren Sie die DNAse Behandlung für 30 Minuten bei Raumtemperatur.
- Nach DNAse Behandlung add 350 ul Puffer RW1 auf die Spin-Säule und zentrifugieren bei 8.000 xg für 1 Minute bei Raumtemperatur.
- Nach der Zentrifugation verwerfen die Durchströmung und fügen 500 ul Puffer RPE auf die Spin-Säule. Inkubieren Sie die Probe für 5 Minuten bei Raumtemperatur.
- Centrifuge Probe bei 8.000 xg für 1 Minute bei Raumtemperatur und entsorgen Sie die Durchströmung.
- Nach Zentrifugation add 500 ul 75% igem Ethanol auf die Spin-Säule.
- Wiederholen Sie die Zentrifugation bei 8.000 xg, aber für 2 Minuten bei Raumtemperatur.
- Nach der Zentrifugation verwerfen die Durchströmung und damit die Spalte auch für 5 Minuten trocknen lassen.
- Unmittelbar bei 8.000 xg zentrifugieren Sie für 5 Minuten bei Raumtemperatur zu jeder übrig Spuren von Ethanol zu entfernen.
- Nach der Zentrifugation ist die vollständige Übertragung der Spin-Säule in ein neues 1,5 ml Sammel-Tube und füge 10 pl Nuklease-freies Wasser. Inkubation für 1 Minute und dann Zentrifuge bei 10.000 xg für 1 Minute bei Raumtemperatur. Die RNA wird in den Fluss durch sein.
- Add another 10 pl Nuklease-freies Wasser auf die Spin-Säule und Inkubation bei Raumtemperatur für 1 Minute. Centrifuge Probe bei 10.000 xg für 1 Minute bei Raumtemperatur.
- Nach der Zentrifugation zu entfernen und entsorgen Sie die Spin-Säule. RNA wird in den Fluss durch sein.
- Prüfen Sie die Qualität und Quantität der RNA mit einem NanoDrop ND-1000 Spektrophotometer folgenden Anweisungen des Herstellers.
- Darüber hinaus führen Sie einen denaturierenden Gel oder verwenden Sie ein Agilent 2100 Bioanalyzer, um die Integrität der RNA zu überprüfen.
- Die Proben sollten bei -80 ° C gelagert werden, bis die cDNA-Synthese durchgeführt werden kann.
Teil 3: cDNA-Synthese mit dem Invitrogen SuperScript First-Strand-System
RNA ist leicht anfällig für Spaltung durch RNAsen führt zu Abbau. Als Ergebnis vieler Genexpression Protokolle wurden entwickelt, um eine stabilere cDNA-Produkt, das direkt aus der RNA synthetisiert verwenden. Hier haben wir ausführlich Synthese der ersten cDNA mit dem Invitrogen SuperScript Erststrangsynthese System 3. Jede cDNA-Synthese-Reaktion kann bis zu 5 ug RNA.
- Vor Beginn des Verfahrens zu mischen und kurz zentrifugieren jede Komponente.
- Bereiten RNA / Primer-Gemisch in ein steriles 0,5 ml Mikrozentrifugenröhrchen, wie in Tabelle 1 dargestellt.
- Inkubieren Sie die Probe bei 65 ° C für 5 Minuten.
- Sofortüberweisung Probe Eisbrei für 10 Minuten.
- Während der Probe wird die Kühlung bereiten einen Master-Mix mit den Komponenten in Tabelle 2 dargestellt. Die Bände sind pro 1 Reaktion aufgeführt. Lautstärkeanpassung entsprechend der Gesamtzahl der Reaktionen.
- Nach dem Abkühlen 9 ul des Master-Mix in Schritt 5, um jede RNA / Primer-Mischung vorbereitet. Vorsichtig mischen und sammeln durch kurze Zentrifugation. Das Gesamtvolumen jeder Probe sollte 19 ul werden.
- Inkubieren Sie die Probe bei 42 ° C für 2 Minuten in einem Thermocycler.
- Fügen Sie 1 ul (50 Einheiten) Superscript II RT in jedes Röhrchen, mischen und inkubieren bei 42 ° C für 60 Minuten in einem Thermocycler.
- Beenden Sie die Reaktion bei 70 ° C für 15 Minuten und dann Chill Probe auf 4 ° C. Ein Thermocycler für die Schritte 8 und 9 programmiert werden.
- Nachdem die Probe 4 ° C erreicht hat, übertragen Sie die Reaktion auf ein Eisbad.
- Auf Eis hinzufügen 1 ul RNAse H in jedes Röhrchen und inkubieren für 20 Minuten bei 37 ° C. Das Gesamtvolumen beträgt jetzt 21 ul. Die Zugabe von RNAse H Ihrer Probe verschlechtern die RNA-Vorlage und lassen Sie nur den Einzelstrang cDNA-Produkt.
Teil 4: cDNA-Isolierung und Niederschlag
- Ein 1,5 ml Phase Lock Gel Rohr wird in die Isolierung der cDNA-Produkt verwendet werden. Bereiten Sie eine 1,5-ml-Phase Lock Gel Röhrchen durch Zentrifugation bei 12.000 xg für 2 Minuten. Das Gel sollte allen an der Unterseite des Rohres werden.
- Add 81,5 ul Phenol (Tris-gesättigt, gepuffert auf pH 8,0), 81,5 ul von 24:1 Chloroform Isoamylalkohol, und die Probe (21 ul) in die Phase Lock Gel Tube. Mix mehrfach durch vorsichtiges Schwenken.
- Zentrifugieren Sie die Probe bei 12.000 xg für 5 Minuten bei Raumtemperatur.
- Die cDNA sollte in der oberen wässrigen Phase. Oberphase in ein sauberes 1,5 ml Mikrozentrifugenröhrchen. Das Gesamtvolumen wird auf rund 20 ul werden.
- Add 1X Volumen 3 M NaOAc, pH 5,2 und vorsichtig mischen. Zum Beispiel, wenn das Volumen der oberen wässrigen Phase wurde in Höhe von 20 ul, müssen Sie 2 ul 3 M NaOAc, pH 5,2 hinzu.
- Add 7 ul von 5 mg / ml Glykogen und vorsichtig mischen.
- Add 1X Volumen Isopropanol und vorsichtig mischen. Im Allgemeinen ist dies etwa 30 ul Isopropanol.
- Lassen Probe bei Zimmertemperatur Temperatur inkubierene für 10 Minuten und dann bei 12.000 xg zentrifugieren für 20 Minuten bei Raumtemperatur.
- Eine kleine cDNA Pellet sollte auf dem Boden der Röhre bilden. Entfernen Sie vorsichtig den Überstand mit einer Pipette.
- Add 500 ul 70% Ethanol und mischen durch Umdrehen.
- Zentrifugieren Sie die Probe bei 12.000 xg für 5 Minuten.
- Nach der Zentrifugation den Überstand mit einer Pipette und wiederholen Sie die 70% Ethanol waschen und Zentrifugation (Schritte 10 und 11).
- Nach der Zentrifugation den Überstand mit einer Pipette. Wenn es verbliebene Flüssigkeit auf den Seiten des Rohres kann durch kurzes Drehen der Röhre gesammelt werden. Entfernen Sie überschüssige Flüssigkeit und lassen Sie das Pellet bei Raumtemperatur für 5 Minuten trocknen lassen.
- Add 20 ul Nuklease-freies Wasser, um das Pellet zu rehydrieren. Legen Sie die Probe bei 55 ° C für 5 Minuten für die Rehydrierung des Pellets.
- Die Quantität und Qualität der cDNA-Produkt wird dann überprüft, wobei ein NanoDrop ND-1000 Spektrophotometer folgenden Anweisungen des Herstellers. Die cDNA kann bei -20 ° C gelagert werden
Repräsentative Ergebnisse
Nach RNA-Aufreinigung, können etwa 15 ug von hoher Qualität Gesamt-RNA von 50 Zebrafischembryonen erwarten. Zur Beurteilung der Gesamt-RNA Qualität, einem Spektralphotometer, Gelelektrophorese und einem Agilent 2100 Bioanalyzer verwendet werden kann. Ein NanoDrop ND-1000 Spektralphotometer kann verwendet werden, um die Absorption bei 260 nm zu beurteilen im Vergleich zu der Absorption bei 280 nm (Abbildung 1). Die A 260 / A 280-Verhältnis offenbaren kann das Vorhandensein von Schadstoffen und geben Hinweise auf mögliche Abbau. Ein A 260 / A 280-Verhältnis von 1,9-2,0 wird als akzeptabel angesehen. Darüber hinaus ist es dringend empfohlen, dass eine RNA-Denaturierung Gellauf auf RNA-Integrität zu beurteilen. RNA-Stränge werden auf Grund ihrer Größe zu trennen, mit kleineren Stränge Migration weiter unten in der Gel. Ein nicht-degradierten Probe wird als ein Abstrich mit zwei starken Banden erscheinen. Der Abstrich ist ein Ergebnis einer Population von unterschiedlich großen mRNA-Stränge und die Bands, die 28S und 18S rRNA entsprechen. Die 28S-Band sollte etwa doppelt so intensiv wie die 18S-Band (Abbildung 2). Alternativ kann der Gesamt-RNA Integrität beurteilt mit Hilfe eines Agilent 2100 Bioanalyzer werden. Zwei unterschiedliche Peaks für 28S und 18S rRNA zu erwarten ist (Abbildung 3). Die 28S-Peak sollte größer sein als die 18S-Peak mit der Fläche unter den Gipfeln von etwa 2:1, jeweils. Zwei deutliche Spitzen nicht in abgebauten Proben beobachtet werden. RNA Proben mit Abbau sollte nicht durch nachträgliche experimentellen Verfahren durchgeführt werden.
Spektrophotometrische Analyse kann verwendet werden, um die Qualität der cDNA-Produkt (Abbildung 4) zu beurteilen. Die A 260 / A 280 sollte bei 1,8. cDNA-Reaktionen mit einem Eingang von 5 ug Gesamt-RNA routinemäßig Ertrag 1-2 ug von cDNA-Produkt in unserem Labor.
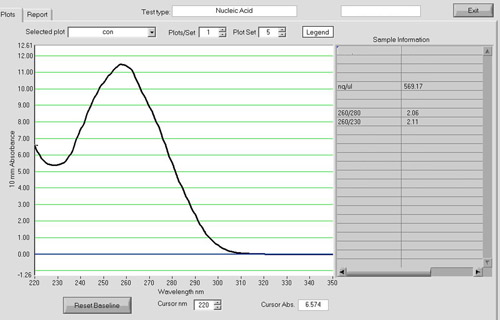
Abbildung 1. Ein NanoDrop Spektrum von RNA-Probe. Nach RNA-Aufreinigung, können RNA-Menge und Qualität anhand einer NanoDrop ND-1000-Spektrophotometer werden. Etwa 15 ug von hoher Qualität Gesamt-RNA wird routinemäßig von 50 Zebrafischembryonen mit einem A 260 / A 280-Verhältnis etwa 1,9-2,0 ergab.
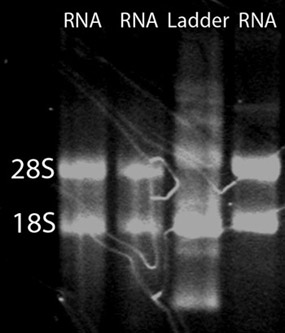
Abbildung 2. Ein RNA Denaturierung Gel. Für die Beurteilung der Integrität der Gesamt-RNA isoliert, kann eine RNA Denaturierung Gel lief. Ein Abstrich mit zwei hellen Streifen entsprechend 28S und 18S rRNA erwartet. Die 28S-Band sollte etwa doppelt so intensiv wie die 18S-Band sein.
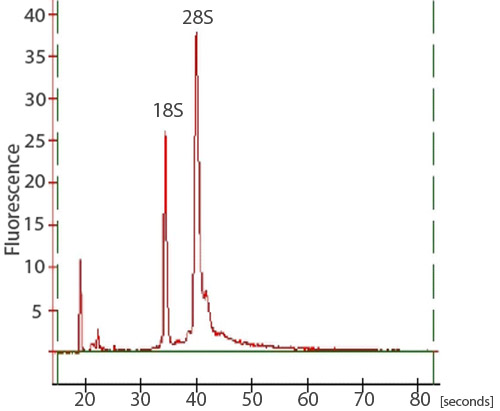
Abbildung 3. Eine Spur von einer RNA-Probe aus einem Bioanalyzer. Um die Integrität der Gesamt-RNA isoliert zu beurteilen, so können Proben auch auf einem Agilent 2100 Bioanalyzer analysiert werden. Zwei scharfe Spitzen, entsprechend der 28S und 18S rRNA, sollten sichtbar sein. Die 28S-Peak sollte größer sein als die 18S-Peak mit der Fläche unter den Gipfeln von etwa 2:1, jeweils.
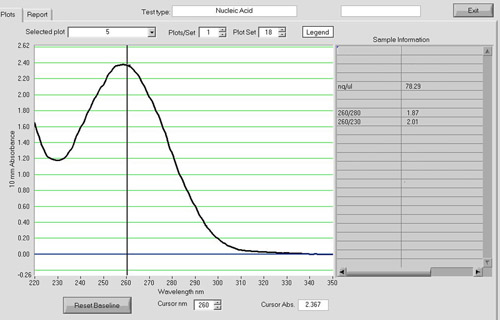
Abbildung 4. Ein NanoDrop Spektrum einer cDNA-Probe. Spektrophotometrische Analyse mit einem NanoDrop ND-1000 kann verwendet werden, um die Quantität und Qualität der cDNA-Produkt zu beurteilen. Die A 260 / A 280-Verhältnis sollte bei 1,8. cDNA-Reaktionen mit einem Eingang von 5 ug Gesamt-RNA routinemäßig Ertrag 1-2 ug cDNA in unserem Labor.
Tabelle 1. Komponenten für RNA / Primer-Mischungen bei Schritt 2 der cDNA-Synthese hinzuzufügen.
| Komponente | Volumen |
| Gesamt-RNA (bis zu 5 ug) | 10 pl |
| 10 mM dNTP-Mix | 1 ul |
| Oligo (dT) 12-18 (0,5 ug / ul) | 1 ul |
| DEPC-behandeltem Wasser | n ul |
| Gesamtvolumen | 10 pl |
Tabelle 2. Reaktionsgemisch Schritt 5 von cDNA-Synthese. Volumes aufgelistet sind pro Reaktion. Erhöhen Mengen jeder Komponente für die Gesamtzahl der Reaktionen.
| Komponente | Volumen |
| 10x RT-Puffer | 2 ul |
| 25 mM MgCl2 | 4 ul |
| 0,1 mM DTT | 2 ul |
| RNAseOUT | 1 ul |
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die Fragilität des RNA-Moleküls ist die kritische Betrachtung, die in diesem Protokoll darf nicht vergessen werden. Sterile Geräte, RNAse-frei ist, sollte immer in einem RNAse-freien Zone des Labors eingesetzt werden. Es ist hilfreich, ein Teil des Labors für RNA Verfahren nur verwendet werden, behalten. Dieser Bereich sollte häufig mit einer RNAse Entfernen Produkt, wie RNase AWAY gesprüht werden. Die RNA-Proben sollten immer mit Handschuhen angefasst werden und auf Eis gehalten, um den Abbau zu verhindern.
<...Access restricted. Please log in or start a trial to view this content.
Offenlegungen
The authors have nothing to disclose.
Materialien
| Name | Company | Catalog Number | Comments |
| Glycogen (5 mg/ml) | Ambion | 9510 | |
| NanoDrop ND-100 | Thermo Fisher Scientific, Inc. | ND-1000 | |
| Pellet Pestles with Microfuge Tube | VWR international | KT749520-0090 | |
| Phase Lock Gel Light, 1.5 ml | Eppendorf | 2302800 | |
| Phenol, Tris-saturated | Roche Group | 3117944001 | |
| RNAse Away | VWR international | 17810-491 | |
| RNAse-Free DNase Set | Qiagen | 79254 | |
| RNEasy Mini Kit | Qiagen | 74104 | |
| SuperScript® First-Strand Synthesis System for RT-PCR | Invitrogen | 11904-018 | |
| TRIzol | Invitrogen | 15596-026 |
Referenzen
- TRIzol Reagent Manual. , (2007).
- RNEasy Mini Handbook. , 4th ed, (2006).
- SuperScript First-Strand Synthesis System for RT-PCR. , Version E. (2003).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten