このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
カスタムmicroRNAのマイクロアレイの実験を行う
要約
カスタムmicroRNAのマイクロアレイの実験を行うの簡単な手順が説明されています。手順は、ハイブリダイゼーションシグナルを定量化し、分析、マイクロアレイのスキャン、マイクロアレイにサンプルをハイブリダイズ、RNA、標識RNAとリファレンスのDNAを単離することがあります。
要約
マイクロRNA(miRNA)は、広く真核生物1で表されている〜22ヌクレオチド(nt)長いRNA分子の大家族です。複雑なゲノムは主に転写後に標的遺伝子の膨大な数の2、3の発現を阻害するmiRNAは、の少なくとも数百人をエンコードする。 miRNAは生物学的プロセス1の広い範囲を制御します。さらに、変更されたmiRNAの発現は、癌などのヒトの疾患と関連している、とmiRNAは病気と予後4、5のためのバイオマーカーとして機能することができる。それは多くの異なる条件下での発現とmiRNAの機能を理解することが重要です。
リアルタイムPCR、マイクロアレイ、およびディープシーケンシング:三つの主要なアプローチは、プロファイルのmiRNAの発現に用いられてきた。 miRNAのマイクロアレイの技術は、一般的に安価、高スループットという利点があり、実験と解析の手順のほとんどは、カーすることができますほとんどの大学、医学校および関連病院での分子生物学の実験室でIED。ここでは、カスタムのmiRNAマイクロアレイ実験を行う方法を説明します。 miRNAのプローブセットは、miRNAのマイクロアレイを製造するガラススライド上に印刷されます。 RNAは、小さなRNA種を保存する方法または試薬を用いて単離し、そしてその後、蛍光色素で標識されている。コントロールとして、miRNAのサブセットに対応する基準DNAオリゴヌクレオチドはまた、異なる蛍光色素で標識されています。リファレンスDNAは、スライドとハイブリダイゼーションの品質を実証するのに役立つだろうし、また、データの正規化のために使用されます。 RNAとDNAを混合し、データベースにおけるmiRNAのほとんどのプローブを含むマイクロアレイスライドにハイブリダイズさせる。洗浄後、スライド、画像、および定量化の個々のスポットの強度を得るためにスキャンされます。これらの生信号をさらに処理し、対応するmiRNAの発現データとして解析されます。 Microarrayのスライドは、マイクロアレイのコストを削減し、マイクロアレイ実験の一貫性を高めるために除去し、再生成することができます。同じ原理と手順は、カスタムマイクロアレイ実験の他のタイプに適用可能である。
プロトコル
1。カスタムmiRNAのマイクロアレイの印刷
- マイクロアレイは、大学や企業ではマイクロアレイの中核施設のサービスを使用してスライド印刷。マイクロアレイ製造の品質は、マイクロアレイ実験の成功のための最も重要な要因の一つです。可能であればいくつかのサービスをお試しください。理想的には、一度に50から100までのスライドを印刷して、そしてすべてのスライドがうまく分離された、個々のスポットで、同一になります。
- マイクロアレイのサポートについては、我々はGAPS IIコートスライドを使用してください。
- miRNAのプローブでは、nCodeの複数種のマイクロアレイプローブがV2に設定miRNAを使用してください。
- 10μMで、3 × SSC内のすべてのオリゴヌクレオチドを溶解し、四重にスライド上に印刷してください。 GAAPS IIスライドの指示に従って紫外線を照射して、スライドを固定。印刷されたプローブによるエリアとサイドをマークするためにダイヤモンドペンでスライドにラベルを付けます。 22℃で保存する
2。サンプルの準備
- 任意のメソッドまたは試薬は、限り、それは、small RNAを保持するように、RNAを単離するために使用することができます。我々は一般的に260nmのと280nmの測定値が測定される、RNA調製物の十分な量と質で、トータルRNAを抽出するためにトリゾール(Invitrogen)を使用してください。
- RNAラベリング、5' - PCU - DY547 - 3の0.5μgのトータルRNAのミックス〜25μgの1'で6 ×緩衝液(50mMのHEPES、pH7.8の20 mMのMgCl 2、10μg/ mlのBSA、10% DMSO)〜10 mMのDTTとATP用10 × T4 RNAリガーゼ1バッファの〜0.02から0.03最終容量を補充〜20単位のT4 RNAリガーゼ1を、含んでいる。
- 2〜24時間、冷蔵庫内の氷の上で進めてラベリングできるように。 0.3 M酢酸ナトリウムとエタノールの3つのボリュームを持つRNAを沈殿させる。効率的なライゲーションと降水量の場合は、上の2 mg / mlのでトータルRNAを溶解し、〜20μlので合計ライゲーションのボリュームを維持する。
- 少ないRNAが使用可能な場合は、入力RNAおよび5' - PCU - DY547 - 3の量は、"スケールダウンすることができます。
- RNAは非常に希釈されている場合は、降水を支援するために、酵母のtRNAなどの担体を加える。
- UlysisのAlexa Fluor ®ハイブリダイゼーションプロセスの制御として647核酸ラベリングキット6を用いて哺乳動物のmiRNAのサブセットに対応する基準DNAオリゴヌクレオチドの合計1μgのに組み合わせるとラベル。 CentriSepカラムを用いて標識DNAを精製し、-20℃で暗所で保存
3。マイクロアレイハイブリダイゼーション
- 初めてのスライドを使用するには、3 × SSC、0.1%SDS、37℃で30〜60分間、0.2%BSA℃のフィルタリングソリューションでプレハイブリダイズスライド沈める水を数回で、その後、イソプロパノールでそれらを。 22℃5分間100gでスライドアダプタ℃で遠心分離機を用いてスライドを乾燥させる
注:我々は、マイクロアレイのハイブリダイゼーションを行うためにコーニングマイクロアレイのハイブリダイゼーションチャンバーとLifterslipsのエリーサイエンティフィック社のmSeriesを使用する。
- liをリンス空気中で乾燥前に水でfterslips、その後エタノールで、。マイクロアレイのハイブリダイゼーションチャンバーの基地内のスライド、そしてスライドの上にlifterslipを置きます。 lifterslipは、スライドに接触すると白いストリップで、プローブの領域をカバーします。
- 暗闇の中で、、標識RNAを沈殿をスピンダウン。 70%エタノールで1回それを洗って、そして空気がペレットを乾燥させます。ペレットは、赤でなければなりません。
- 400mMののNa 2 HPO 4 pH 7.0の、0.8%BSA、5%SDS、精製の1/15-1/100μlの12%ホルムアミド6、RNAサンプルごとに標識された参照DNA(2.4)を含むハイブリダイゼーション溶液を準備します。追加された参照DNAの量が異なるオリゴヌクレオチドを標識しているかによって異なってきます(2.4)。 100以上存在する場合は、以下の希釈が必要です。
- ハイブリダイゼーション溶液の約60μlを加えRNAペレットを溶解する。
- コーニングマイクロアレイのハイブリダイゼーションチャンバーベースの加湿各ウェルに水20μlを添加する。
- tを追加彼のスライドに標識されたRNAとリファレンスのDNAの混合物。細いピペットチップを使用して、そっとlifterslipの端に触れ、そして解決策は、吸引によってlifterslipとスライドの間にスペースを入力することができます。
- ベース上でハイブリダイゼーションチャンバーカバーを取り付け、その後、金属製のクリップでチャンバーを密閉する。
- コーニングからチャンバーのカセットに付属のビニール袋の内側全体チャンバーを置く。
- 接液ペーパータオルを容器の内側チャンバーカセット(s)を置き、ラップで容器を覆い、約24時間、37℃で湿度の高い、細胞培養インキュベーター内に配置。これらの予防措置(3.6、3.9、3.10)ハイブリダイゼーション溶液は、インキュベーション中に乾燥しないようにしてください。
4。ポストハイブリダイゼーション処理
- チャンバーカセット一つ一つを分解。 22で2 × SSCに浸し、スライドとそのlifterslip℃のlifterslipは自然にスライドして減少していきます。 0.8 × SSCに入れてください。洗う二回、その後、0.8 × SSC中でスライドを0.4 × SSC、合計で1〜2分で三回。スライドアダプター付き遠心機を使用してスライドを乾燥させる。水でlifterslipsを洗って、後で再利用のために保存。
- 任意の適切なスキャナ、例えば、パーキンエルマーScanArray 5000またはMolecular Devices社軸索GenePix 4000Bマイクロアレイスキャナーを使用してスライドをスキャンします。対応する画像ファイルを取得するために近い547 nmと647 nmの波長でスキャンする。
- 入力、4.2からイメージファイル)内のピクセル強度を定量化するなどBlueFuseなどのプログラムを使用してください。今後の検討からマイクロアレイ上の異常な箇所を除外するためのプログラムと個々のスポットを点検。これらの異常なスポットは、通常、ハイブリダイゼーション後の取り扱い不良スライドの印刷またはスライドから生じる。
- データ分析とプレゼンテーションのためのそのようなジーンスプリングやExcelなどのプログラムを使用してください。マイクロアレイデータの正規化のための一般的な考慮事項は、このホワイトペーパーの範囲を超えている、適用する。
- 十分な信号がAFを取得するとTER 4.3)、再利用7マイクロアレイスライドを取り除く。 10-20分間、62℃染色皿に予め温めておいた、1 mMのNaOHおよび0.1 × SSC ° Cにして浸漬、水にスライドをすすぎます。一度インキュベーションを繰り返します。 22℃まで穏やかに振盪しながら60分間、水で数回スライド℃を洗う遠心分離機を使用してスライド、および22℃で保存して乾燥させる
- 再生されたスライドを使用するには、それは再びそれらを事前にハイブリダイズさせる必要はありません。単にイソプロパノールに続いて水で洗って、右ハイブリダイゼーション前にスライドを乾燥させる。
5。代表的な結果:
我々は主に、すなわち、数千のサンプルでグローバルなmiRNA発現をプロファイルするために多くの異なる条件の下でゼブラフィッシュからヒト試料に単離されたRNAを説明する手順に従っている。 図1に非常に正確で強力なハイブリダイゼーションシグナルを示すために、マイクロアレイのスキャンした画像を示していますスライド。ピアソンcorrelati技術は、マイクロアレイのハイブリダイゼーションの複製との間の係数に優れた再現性を示す、〜0.99 7です。
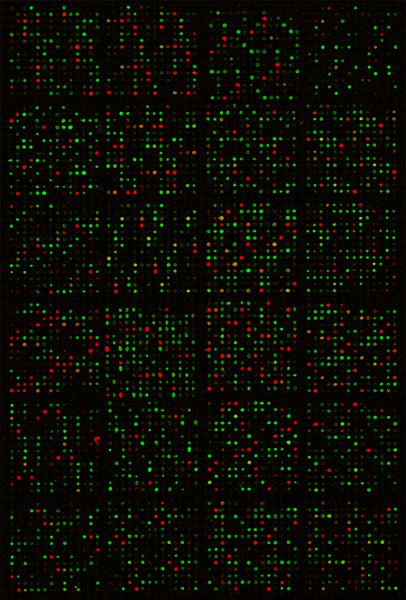
ハイブリダイゼーション後のスキャンされたmiRNAのマイクロアレイのスライドの1コンポジット画像を図 。黄色の斑点が同じプローブにDNAとRNAの両方でのハイブリダイゼーションからいる間に赤い斑点は、リファレンスのDNA、DY547標識RNAサンプルからの緑のスポットでハイブリダイゼーションに起因する。
ディスカッション
深いシーケンシング技術の最近の進歩にもかかわらず、マイクロアレイは、DNAとRNAのハイスループット分析のための実行可能な選択肢です。ディープシーケンシングに比べて、マイクロアレイ実験には安上がりだし、典型的な分子生物学の研究室では、柔軟性を可能にすると時間の節約、社内での実験とデータ分析のほとんどを実行できます。将来的には、マイクロアレイの可能性は適して?...
開示事項
利害の衝突は宣言されません。
謝辞
作業は、薬物乱用センター(P50 DA 011806)の国立研究所と国防総省の米国陸軍省(W81XWH - 07 - 1〜0183)によって部分的にサポートされていました。
資料
| Name | Company | Catalog Number | Comments |
| アイテム | ベンダ | カタログ番号 | コメント |
|---|---|---|---|
| マイクロアレイプローブセットV2をmiRNAの複数種をnCodeの | インビトロジェン | MIRMPS201 | miRBaseリリース9.0(2006年10月)に基づいて設計されています。それは、〜1140変更されていない、ワーム、ハエ、ゼブラフィッシュ、マウス、ラット、及びヒトmiRNAのプローブとして、34〜44 ntの長いオリゴヌクレオチド、およびそのようなsnoRNAsのような内部統制のプローブの数が含まれています。 miRNAのプローブは、それ故に成熟miRNAに相補的な配列、〜44ヌクレオチドのサイズのダブレットです。分析のために一つは関心のある特定のゲノム(s)からのmiRNAに焦点を当てることができます。 |
| トリゾール | インビトロジェン | 15596018 | トータルRNAサンプルは、このようなmRNAの解析のようなダウンストリームアプリケーションのための迅速かつ容易に準備すると、定量化し、適しているものの、我々はまた、標識のための豊かな、small RNA画分を使用している。 |
| T4 RNAリガーゼ1 | ニューイングランドBiolABS | M0204L | |
| UlysisのAlexa Fluor ® 647核酸標識キット | インビトロジェン | U21660 | このキットまたは類似の製品も同様に実験的なRNAサンプルやコントロールRNAを(代わりに、コントロールDNAの)ラベルを付けるために使用することができます。 |
| 5' - PCU - DY547 - 3' | Dharmacon | カスタムは行わ | small RNAの画分を同様に連結することにより標識することができる。 |
| CentriSep列 | プリンストンセパレーション | CS - 901 | |
| GAPSIIコートスライド | コーニング | 40004 | スライドの他のタイプも使用されることがあります。 |
| マイクロアレイハイブリダイゼーションチャンバー | コーニング | 2551または40080 | ハイブリダイゼーションチャンバーとカバースリップの他の種類も動作するはずです。市販のハイブリダイゼーションのマシンを使用すると、約2時間、、大幅に例えばハイブリダイゼーションの時間を短縮することができます。 |
| Lifterslips | 目ermo /エリー科学 | 25X60I - M5439 - 001 - LS | |
| BlueFuse | BlueGenome | ||
| ジーンスプリング | アジレント |
参考文献
- Ambros, V. The functions of animal microRNAs. Nature. 431, 350-355 (2004).
- Friedman, R. C., Farh, K. K., Burge, C. B., Bartel, D. P. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res. 19, 92-105 (2009).
- Chekulaeva, M., Filipowicz, W. Mechanisms of miRNA-mediated post-transcriptional regulation in animal cells. Curr. Opin. Cell Biol. 21, 452-460 (2009).
- Farazi, T. A., Spitzer, J. I., Morozov, P., Tuschl, T. miRNAs in human cancer. J. Pathol. 223, 102-115 (2011).
- Small, E. M., Olson, E. N. Pervasive roles of microRNAs in cardiovascular biology. Nature. 469, 336-342 (2011).
- Thomson, J. M., Parker, J., Perou, C. M., Hammond, S. M. A custom microarray platform for analysis of microRNA gene expression. Nat. Methods. 1, 47-53 (2004).
- Zhang, X., Xu, W., Tan, J., Zeng, Y. Stripping custom microRNA microarrays and the lessons learned about probe:slide interactions. Anal. Biochem. 386, 222-227 (2009).
- Griffiths-Jones, S., Saini, H. K., van Dongen, S., Enright, A. J. miRBase: tools for microRNA genomics. Nucl. Acids. Res. 36, D154-D158 (2008).
- Landgraf, P. A mammalian microRNA expression atlas based on small RNA library sequencing. Cell. 129, 1401-1414 (2007).
- Chiang, H. R. Mammalian microRNAs: experimental evaluation of novel and previously annotated genes. Genes. Dev. 24, 992-1009 (2010).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved