JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
사용자 정의 MicroRNA의 Microarray 실험을 수행
요약
사용자 정의 microRNA의 microarray 실험을 수행 간단한 절차 설명합니다. 단계, RNA, RNA 라벨링 및 참조 DNA를 분리 microarrays에 샘플을 hybridizing, microarrays를 검색, quantifying 및 하이브리드화 신호를 분석 등이 있습니다.
초록
microRNAs는 (miRNAs) 널리 eukaryotes 1 표현됩니다 ~ 22 세포핵 (NT) 긴 RNA 분자의 큰 가족입니다. 복잡한 genomes는 주로 사후 transcriptionally 대상 유전자 2, 3의 방대한 숫자의 표현을 억제 miRNAs 중 적어도 수백, 인코딩. miRNAs는 생물 학적 과정 1 광범위한 제어합니다. 또한, 변경 miRNA 발현이 같은 암 등 인간의 질병와 관련된되었으며, miRNAs는 질병 및 예후 (豫后) 4, 5에 대한 생체 역할을 수 있습니다. 그것은 표현과 여러 가지 조건에서 miRNAs의 기능을 이해하기 위해, 따라서, 중요합니다.
실시간 PCR, microarray, 깊이 시퀀싱 : 세 가지 주요 방법은 프로필 miRNA 발현에 고용되었습니다. miRNA의 microarray의 기술은 일반적으로 저렴 높은 처리량되는 장점을 가지고 있으며, 실험 및 분석 단계의 대부분은 카 수 있습니다대부분의 대학, 의과 대학 및 관련 병원에서 분자 생물학 연구실에서 지휘를 맡는. 여기, 우리는 사용자 정의 miRNA의 microarray 실험을 수행하는 방법을 설명합니다. miRNA 프로브 세트는 miRNA의 microarrays를 만들어 유리 슬라이드에 인쇄됩니다. RNA는 작은 RNA 수종을 보존하는 방법이나 시약을 사용하여 절연하고 형광 염료와 함께 표시됩니다. 컨트롤로서, miRNAs의 하위 집합에 해당 참조의 DNA oligonucleotides는 다른 형광 염료로 분류됩니다. 참조 DNA는 슬라이드와 하이브 리다이 제이션의 품질을 증명하기 위해 제공되며, 또한 데이터 정규화에 사용됩니다. RNA와 DNA가 혼합하여 데이터베이스에있는 miRNAs 대부분의 프로브를 포함하는 microarray 슬라이드에 hybridized입니다. 세탁 후, 슬라이드 이미지를 얻기 위해 스캔하고, 개별 명소의 농도는 계량입니다. 이러한 원시 신호는 추가로 처리하고 해당 miRNAs의 표현 데이터로 분석됩니다. Microarray 슬라이드는 옷을 벗긴 채 microarrays의 비용을 절감하고 microarray 실험의 일관성을 향상시키기 위해 생성하실 수 있습니다. 동일한 원칙과 절차를 정의 microarray 실험의 다른 유형에 적용됩니다.
프로토콜
1. 사용자 정의 miRNA의 microarrays의 인쇄
- microarray는 대학이나 회사에서 microarray 핵심 시설 서비스를 이용 슬라이드 인쇄합니다. microarray 제조의 품질은 microarray 실험의 성공을 위해 가장 중요한 요소 중 하나입니다. 가능하면 몇 가지 서비스를보십시오. 이상적으로, 하나는 한 번에 50-100 슬라이드를 인쇄 것이며, 모든 슬라이드가 잘 구분하여, 각각의 장소와 동일 보입니다.
- microarray 지원, 우리는 격차 II 코팅 슬라이드를 사용합니다.
- miRNA 프로브 위해, 우리는 NCode 멀티 종 (species)이 Microarray 프로브가 V2 세트 miRNA 사용합니다.
- 10 μm의 3 X SSC에있는 모든 oligonucleotides를 해산하고 quadruply 슬라이드 그들을 인쇄할 수 있습니다. GAAPS II 슬라이드에 대한 지침에 따라 자외선 조사에 의해 슬라이드를 수정. 인쇄 프로브와 함께 지역과 측면을 표시하는 다이아몬드 펜으로 슬라이드 레이블. 22 스토어 ° C.
2. 샘플 준비
- 모든 방법또는 시약이 한 그것이 작은 RNAs를 유지로 RNA를 분리하는 데 사용할 수 있습니다. 우리는 일반적으로 260nm와 280nm 판독하여 측정 RNA의 준비 만족 수량 및 품질과 함께, 총 RNA를 추출하기 위해 Trizol (Invitrogen)을 사용합니다.
- RNA 라벨, 1 X 버퍼 (50 MM의 HEPES, pH를 7.8, 20 MM MgCl 2, 10 μg / ML BSA, 10 % 5' - pCU - DY547 - 3 '6 0.5 μg과 함께 총 RNA의 믹스 ~ 25 μg을 위해 DMSO) 포함 ~ 20 대 T4 RNA ~ 10 밀리미터 DTT와 ATP에 대한 10 X T4 RNA Ligase 한 버퍼의 ~ 0.02-0.03 최종 볼륨 보충 Ligase 1.
- 2-24시간의 냉장고 안에 얼음 진행 라벨 수 있습니다. 0.3 M의 아세트산 나트륨과 에탄올 3 권와 RNA를 침전. 효율적인 결합과 강수량 들어, 이상이 MG / ML에서 총 RNA를 해산하고, ~ 20 μl의 총 내고 볼륨을 유지.
- 적은 RNA를 사용할 경우, 입력 RNA 및 5'가 - pCU - DY547 - 3 '는 크기를 줄일 수 있습니다의 금액입니다.
- RNA가 매우 묽은 경우, 강수량을 도울 효모 tRNA 등 통신 사업자를 추가합니다.
- Ulysis 알렉사 형석 하이브 리다이 제이션 프로세스 제어로 647 핵산 라벨링 키트 6을 사용 포유 동물 miRNAs의 하위 집합에 해당 참조의 DNA oligonucleotides 총 1 μg의 결합 및 레이블. CentriSep 칼럼을 사용하여 표시된 DNA를 정화하고 -20 ° C.에 어두운에 저장
3. Microarray의 하이브 리다이 제이션
- 처음으로 슬라이드를 사용하려면, 37 3 X SSC, 0.1 % SDS, 30-60 분 0.2 % BSA의 필터링 솔루션에 슬라이드를 미리 잡종 ° C. 잠수함 물이 몇 번에서 다음 이소프로판올에. 22 5 분 100g에서 슬라이드 어댑터 ° C.있는 원심 분리기를 사용하여 슬라이드를 건조
참고 : 우리는 microarray의 하이브 리다이 제이션을 수행하기 위해 코닝 microarray의 하이브 리다이 제이션 챔버와 Lifterslips의 에리 과학의 mSeries을 사용합니다.
- 리 씻어공기 건조하기 전에 물에 fterslips 다음 에탄올로. microarray의 하이브 리다이 제이션 챔버 기지 내부에 슬라이드와 슬라이드의 위에 lifterslip 놓으십시오. lifterslip은 슬라이드 연락은 흰색 스트립과 탐사 영역을 커버합니다.
- 어둠 속에서, 레이블 RNA를 시켰던을 돌린다. 70 % 에탄올로 한 번 씻고, 그리고 공기는 펠렛을 건조. 펠렛은 붉은해야합니다.
- 400 MM 나 2 HPO 4 산도 7.0, 0.8 % BSA 5 % SDS, 정화의 1/15-1/100 μl와 12 % 포름 아미드 6, RNA 샘플마다 라벨 참조 DNA (2.4)이있는 하이브 리다이 제이션 솔루션을 준비합니다. 추가 참조 DNA의 양은 다른 oligonucleotides는 (2.4) 얼마나 많은 레이블 아르에 따라 달라집니다. 100 개 이상있다면 덜 희석이 필요합니다.
- 하이브 리다이 제이션 솔루션 ~ 60 μl로 잘 RNA 펠렛를 해산.
- 코닝 microarray의 하이브 리다이 제이션 챔버 자료의 가습 우물의 물을 각각 20 μl를 추가합니다.
- t 추가슬라이드에 표시 RNA 및 참고 DNA의 그 혼합물. 얇은 pipet 팁을 사용하여 부드럽게 lifterslip의 가장자리를 터치하고, 솔루션은 흡입을 통해 lifterslip 및 슬라이드 사이의 공간을 입력할 수 있습니다.
- 하이브 리다이 제이션 챔버가베이스 커버를 통해 장소, 그때 금속 클립과 챔버를 봉인.
- 코닝에서 챔버 카세트와 함께 제공되는 비닐 봉투 안에 전체 챔버 놔.
- 침수 종이 타월로 용기 내부 챔버 카세트 (들)을 장소, 플라스틱 포장과 용기를 커버하고 37 안에 자리 ° ~ 24 시간 동안 C 습기, 휴대 문화 인큐베이터. 이러한주의 사항 (3.6, 3.9, 3.10)은 하이브 리다이 제이션 솔루션이 부화하는 동안 밖으로 건조하지 않도록.
4. 포스트 하이브 리다이 제이션 처리
- 챔버 카세트를 하나씩 분해. 22 2 X SSC에 잠수함 슬라이드와 lifterslip ° C. lifterslip 자연 슬라이드 떨어질 것입니다. 0.8 X SSC에 놓으십시오. 빨래두 번, 그리고 0.8 X SSC에있는 슬라이드 0.4 X SSC, 총 1-2 분 안에 세 번. 슬라이드 어댑터와 원심 분리기를 사용하여 슬라이드를 건조시킵니다. 물로 lifterslips을 씻고 나중에 다시 사용하기 위해 저장할 수 있습니다.
- 모든 적합한 스캐너, 예를 들어, Perkin 엘머 ScanArray 5000 또는 분자 장치 축삭 GenePix 4000B microarray 스캐너와 슬라이드를 검사합니다. 해당 이미지 파일을 얻기 위해 파장 547 nm의 가까이에와 647 nm의에서 스캔.
- 입력, 4.2에서 이미지 파일)에서 픽셀 농도를 정할 같은 BlueFuse 같은 프로그램을 사용합니다. 추가 고려 대상에서 microarrays에 대한 비정상적인 명소를 제외하는 프로그램을 각각의 장소를 검사합니다. 이러한 비정상적인 명소는 일반적으로 가난한 슬라이드 인쇄하거나 슬라이드 하이브 리다이 제이션 후 처리에서 발생.
- 데이터 분석 및 프레 젠 테이션 등 GeneSpring 및 Excel과 같은 프로그램을 사용합니다. microarray 데이터 정상화를위한 일반적인 고려 사항은이 문서의 범위를 넘어있는, 적용됩니다.
- 일단 만족스러운 신호는 AF를 얻을 수 있습니다ter 4.3), 재사용 7 microarray 슬라이드를 스트립. 10~20분를위한 62의 얼룩 접시에 미리 예열, 1 ㎜ NaOH 및 0.1 X SSC ° C에서 다음 젖어, 물속에 슬라이드를 씻어. 일단 부화를 반복합니다. 22까지 부드럽게 흔들어 60 분 동안 물에 몇 번 슬라이드 ° C. 씻으십시오 원심 분리기를 사용하여 슬라이드, 22에서 저장 ° C. 건조
- 재생 슬라이드를 사용하려면, 다시 그들을 미리 잡종 필요가 없습니다. 간단히 이소프로판올 다음 물 속에 그들을 씻고, 바로 하이브 리다이 제이션 전에 슬라이드를 건조.
5. 대표 결과 :
우리는 대부분 샘플 수천 글로벌 miRNA 발현을 프로필에 설명된 절차를 따라했습니다 즉, 여러 조건 하에서 인간의 표본으로 zebrafish에서 격리 RNAs가. 그림 1은 매우 정확하고 강력한 하이브 리다이 제이션 신호를 증명하기 위해 microarray의 스캔 이미지를 보여줍니다 슬라이드. 피어슨 correlati기술 사이의 계수에 대한 microarray의 하이브 리다이 제이션의 복제 우수한 재현성을 나타내는 ~ 0.99 7, 수 있습니다.
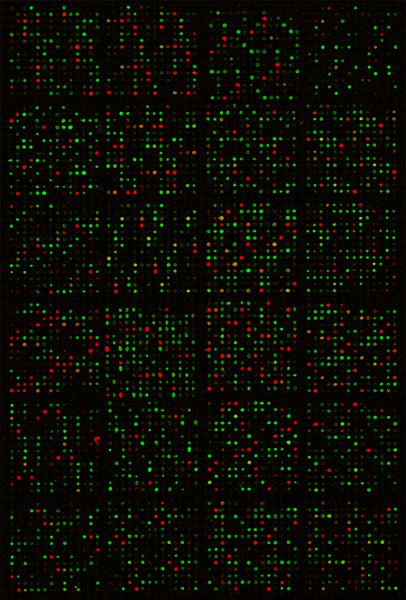
하이브 리다이 제이션 후 스캔 miRNA의 microarray 슬라이드 1 복합 이미지를 그림. 노란색 반점이 동일한 프로브에 DNA와 RNA 모두에 의해 hybridizations에서 동안 레드 명소 참조 DNA, DY547 - 라벨 RNA 샘플에서 녹색 관광 명소로 hybridizations에서 결과.
토론
깊은 시퀀싱 기술의 최근 발전에도 불구하고, microarray는 DNA와 RNA의 높은 처리량 분석을위한 가능한 선택 남아있다. 깊은 시퀀싱에 비해 microarray 실험은 저렴하고 있으며, 전형적인 분자 생물학 연구소의 실험과 유연성을 허용하고 시간을 절약할 수 데이터 분석 사내의 대부분을 수행할 수 있습니다. 앞으로, microarrays 가능성이 적합 집중적으로 유전자, 예를 들어 세트를 심문하러 있으며, 전부 또?...
공개
관심 없음 충돌 선언하지 않습니다.
감사의 말
작품은 약물 남용 센터 국립 연구소 (P50 DA 011806)와 국방의 미국 육군과 (W81XWH - 07 - 1-0183)에 의해 부분적으로 지원되었다.
자료
| Name | Company | Catalog Number | Comments |
| 항목 | 공급 업체 | 카탈로그 번호 | 댓글 |
|---|---|---|---|
| NCode 멀티 종 Microarray 프로브 세트 V2를 miRNA | Invitrogen | MIRMPS201 | miRBase 릴리스 9.0 (2006 년 10 월)에 따라 설계되었습니다. 그것은 1140 ~ 수정되지 않은, 웜, 비행, zebrafish, 마우스, 쥐, 인간 miRNAs을 위해 프로브로 34-44 NT 긴 oligonucleotides, 그리고 snoRNAs 등 내부 통제 프로브의 숫자가 포함되어 있습니다. miRNA 프로브는 따라서 성숙 miRNAs ~ 44 NT의 크기 보완 시퀀스 doublets 있습니다. 분석을 위해 하나가 관심의 특정 게놈 (S)에서 miRNAs에 초점을 맞출 수 있습니다. |
| Trizol | Invitrogen | 15596018 | 총 RNA 샘플 빠르고 준비하고 수치 쉽고 같은 mRNA 분석과 같은 다운 스트림 애플 리케이션에 적합하지만 우리는 또한 사용, 라벨에 대한 작은 RNA 분획을 농축 있습니다. |
| T4 RNA Ligase 일 | 뉴 잉글랜드 Biol복근 | M0204L | |
| Ulysis 알렉사 형석 647 핵산 라벨링 키트 | Invitrogen | U21660 | 이 키트 또는 유사한 제품뿐만 아니라 실험적인 RNA 샘플 또는 컨트롤 RNA를 (대신 제어 DNA의) 라벨을 사용하실 수 있습니다. |
| 5' - pCU - DY547 - 3 ' | Dharmacon | 사용자가 만든 | 작은 RNA 분획은 비슷하게 내고으로 분류하실 수 있습니다. |
| CentriSep 열 | 프린스턴 분판 | CS - 901 | |
| GAPSII 코팅 슬라이드 | 코닝 | 40,004 | 슬라이드의 다른 유형도 사용할 수 있습니다. |
| Microarray의 하이브 리다이 제이션 챔버 | 코닝 | 2551 또는 40080 | 하이브 리다이 제이션 실 및 coverslips 다른 종류도 작동합니다. 상용 하이브 리다이 제이션 기계를 사용하면 ~ 2 시간, 크게 예 하이브 리다이 제이션 시간을 줄일 수 있습니다. |
| Lifterslips | 일ermo / 에리 과학 | 25X60I - M5439 - 001 - LS | |
| BlueFuse | BlueGenome | ||
| GeneSpring | 애질런트 |
참고문헌
- Ambros, V. The functions of animal microRNAs. Nature. 431, 350-355 (2004).
- Friedman, R. C., Farh, K. K., Burge, C. B., Bartel, D. P. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res. 19, 92-105 (2009).
- Chekulaeva, M., Filipowicz, W. Mechanisms of miRNA-mediated post-transcriptional regulation in animal cells. Curr. Opin. Cell Biol. 21, 452-460 (2009).
- Farazi, T. A., Spitzer, J. I., Morozov, P., Tuschl, T. miRNAs in human cancer. J. Pathol. 223, 102-115 (2011).
- Small, E. M., Olson, E. N. Pervasive roles of microRNAs in cardiovascular biology. Nature. 469, 336-342 (2011).
- Thomson, J. M., Parker, J., Perou, C. M., Hammond, S. M. A custom microarray platform for analysis of microRNA gene expression. Nat. Methods. 1, 47-53 (2004).
- Zhang, X., Xu, W., Tan, J., Zeng, Y. Stripping custom microRNA microarrays and the lessons learned about probe:slide interactions. Anal. Biochem. 386, 222-227 (2009).
- Griffiths-Jones, S., Saini, H. K., van Dongen, S., Enright, A. J. miRBase: tools for microRNA genomics. Nucl. Acids. Res. 36, D154-D158 (2008).
- Landgraf, P. A mammalian microRNA expression atlas based on small RNA library sequencing. Cell. 129, 1401-1414 (2007).
- Chiang, H. R. Mammalian microRNAs: experimental evaluation of novel and previously annotated genes. Genes. Dev. 24, 992-1009 (2010).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유