Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Performing Benutzerdefinierte MicroRNA Microarray-Experimenten
In diesem Artikel
Zusammenfassung
Ein einfaches Verfahren zur Durchführung benutzerdefinierte microRNA Microarray-Experimenten beschrieben. Die Schritte umfassen Isolierung von RNA, die Kennzeichnung RNA und DNA-, Hybridisierung der Proben auf Microarrays, Scannen der Microarrays, Quantifizierung und Analyse Hybridisierungssignale.
Zusammenfassung
microRNAs (miRNAs) sind eine große Familie von ~ 22 Nukleotiden (nt) lange RNA-Moleküle, die weit in Eukaryoten 1 sind ausgedrückt. Komplexer Genome kodieren mindestens Hunderte von miRNAs, die in erster Linie hemmt die Expression einer Vielzahl von Zielgenen posttranskriptional 2, 3. miRNAs Steuerung einer breiten Palette von biologischen Prozessen 1. Darüber hinaus hat sich verändert miRNA-Expression mit menschlichen Krankheiten wie Krebs in Verbindung gebracht worden, und miRNAs können als Biomarker für Krankheiten und die Prognose 4, 5 dienen. Es ist daher wichtig, um die Expression und Funktion von miRNAs unter vielen verschiedenen Bedingungen zu verstehen.
Real-time PCR, Microarray und deep sequencing: Drei große Ansätze zum Profil miRNA-Expression eingesetzt. Die Technik der miRNA-Microarray hat den Vorteil, dass High-Throughput, in der Regel weniger teuer, und die meisten der experimentellen und Analyseschritte können carr werdenin einem molekularbiologischen Labor an den meisten Universitäten, medizinischen Fakultäten und die damit verbundenen Krankenhäusern IED. Hier beschreiben wir ein Verfahren zur Durchführung benutzerdefinierte miRNA Microarray-Experimenten. Ein miRNA-Sonde gesetzt werden auf Glasträger gedruckt werden, um miRNA-Mikroarrays herzustellen. RNA isoliert mit einer Methode oder Reagenz, dass kleine RNA-Spezies konserviert und anschließend mit einem Fluoreszenz-Farbstoff markiert. Als Kontrolle werden Referenz-DNA-Oligonukleotide entsprechend einer Untergruppe von miRNAs auch mit einer anderen Fluoreszenz-Farbstoff markiert. Die Referenz-DNA wird dazu dienen, die Qualität der Folie und Hybridisierung zu demonstrieren und auch für Daten Normalisierung verwendet werden. Die RNA und DNA werden gemischt und hybridisierte mit einer Microarray-Folie mit Sonden für die meisten der miRNAs in der Datenbank. Nach dem Waschen wird die Folie gescannt, um Bilder zu erhalten, und Intensitäten der einzelnen Spots quantifiziert. Diese Roh-Signale werden weiter verarbeitet und analysiert werden, wie der Ausdruck von Daten der entsprechenden miRNAs. MicroArray Folien können entfernt und regeneriert werden, um die Kosten von Microarrays zu reduzieren und die Konsistenz der Microarray-Experimente zu verbessern. Die gleichen Grundsätze und Verfahren sind für andere Arten von benutzerdefinierten Microarray-Experimenten.
Protokoll
1. Drucken von benutzerdefinierten miRNA-Mikroarrays
- Drucken Sie die Microarray-Slides mit Hilfe der Microarray-core facility services an einer Universität oder eines Unternehmens. Die Qualität der Microarray-Herstellung ist einer der wichtigsten Faktoren für den Erfolg eines Mikroarray-Experiment. Probieren Sie ein paar Dienste, wenn möglich. Idealerweise würde man 50-100 Dias zu einem Zeitpunkt, zu drucken und alle Folien werden identisch aussehen, mit gut voneinander getrennt, die einzelnen Spots.
- Für Microarray-Unterstützung, verwenden wir die GAPS II beschichtete Objektträger.
- Für miRNA Sonden, verwenden wir die NCode Multi-Species miRNA Microarray Probe Set V2.
- Lösen Sie alle Oligonukleotide in 3 x SSC bei 10 uM und vierfach drucken sie auf den Folien. Befestigen Sie die Folien durch UV-Bestrahlung nach den Anweisungen für die GAAPS II rutscht. Beschriften Sie die Folie mit einem Diamant-Stift, um die Gegend und die Seite der gedruckten Sonden zu markieren. Lagerung bei 22 ° C.
2. Probenvorbereitung
- Jede Methodeoder Reagenz kann verwendet werden, um RNA zu isolieren, solange es kleine RNAs bewahrt. Wir verwenden in der Regel Trizol (Invitrogen) zur Gesamt-RNA-Extrakt, mit befriedigender Qualität und Quantität der RNA-Präparationen, wie von A 260nm und 280nm A gemessene.
- Für die RNA-Kennzeichnung, Mix ~ 25 ug Gesamt-RNA mit 0,5 ug 5'-PCU-DY547-3 '6 in 1 x Puffer (50 mM HEPES, pH 7,8, 20 mM MgCl 2, 10 pg / ml BSA, 10% DMSO) mit ~ 20 Einheiten T4-RNA-Ligase 1, mit ~ 10 mM DTT und ~ 0,02-0,03 Endvolumen des 10 x T4-RNA-Ligase 1 Puffer für ATP ergänzt.
- Lassen Sie die Kennzeichnung auf Eis im Kühlschrank für 2-24 Stunden ablaufen. Niederschlag RNA mit 0,3 M Natriumacetat und 3 Volumen Ethanol. Für eine effiziente Ligation und Niederschlag, löst Gesamt-RNA von über 2 mg / ml, und halten Sie die gesamte Ligation Volumen bei ~ 20 pl.
- Wenn weniger RNA ist verfügbar, die Menge der Eingangs-RNA und 5'-PCU-DY547-3 'kann verkleinert werden.
- Wenn die RNA sehr verdünnt ist, fügen Träger, wie Hefe-tRNA an der Niederschläge zu unterstützen.
- Kombinieren und Etikett in insgesamt 1 pg von DNA-Oligonukleotiden entspricht einer Teilmenge von Säugetieren miRNAs mit dem Ulysis Alexa Fluor 647 Nucleic Acid Labeling Kit 6 als Kontrolle für die Hybridisierung Prozess. Purify der markierten DNA mit einem Centrisep Spalte und speichern in Dunkeln bei -20 ° C.
3. Microarray-Hybridisierung
- So verwenden Sie die Folien zum ersten Mal, vor hybridisieren die Folien in einer gefilterten Lösung von 3 x SSC, 0,1% SDS, 0,2% BSA für 30-60 min bei 37 ° C. Tauchen sie in Wasser ein paar Mal, dann in Isopropanol. Trocknen Sie die Folien mit einer Zentrifuge mit einem Dia-Adapter bei 100 g für 5 min bei 22 ° C.
Hinweis: Wir verwenden Corning Microarray-Hybridisierung Kammern und Erie Scientific M-Serie von LifterSlips auf Microarray-Hybridisierung durchzuführen.
- Spülen Sie den lifterslips in Wasser, dann in Ethanol, vor dem Trocknen an der Luft. Setzen Sie ein Bild in einem Microarray-Hybridisierung Kammer Basis und eine lifterslip am oberen Rand der Folie. Die lifterslip deckt die Sonde Bereich, mit seinen weißen Streifen Kontaktierung der Folie.
- In den dunklen, Spin-down der ausgefallene, markierte RNA. Waschen Sie es einmal mit 70% Ethanol, und an der Luft trocknen des Pellets. Das Pellet sollte rötlich.
- Bereiten Sie eine Hybridisierungslösung mit 400 mM Na 2 HPO 4, pH 7,0, 0,8% BSA, 5% SDS, 12% Formamid 6 mit 1/15-1/100 ul des gereinigten, markierten Referenz-DNA (2,4) pro RNA-Probe. Die Menge der zugesetzten Referenz-DNA hängt von vielen verschiedenen Oligonukleotiden gekennzeichnet sind (2,4). Wenn es mehr als 100 sind, dann weniger Verdünnung erforderlich.
- Lösen Sie das RNA-Pellet gut mit ~ 60 ul der Hybridisierungslösung.
- Fügen Sie 20 ul Wasser zu jedem der Befeuchtung Brunnen in der Corning Microarray-Hybridisierung Kammer Basis.
- Add ter Mischung aus markierter RNA und DNA-auf die Folie. Verwenden Sie einen dünnen Pipettenspitze, sanft berühren den Rand des lifterslip und lassen Sie die Lösung, um den Raum zwischen den lifterslip und schieben durch Saugen geben.
- Legen Sie die Hybridisierung Kammer über die Basis abdecken und versiegeln Sie die Kammer mit Metallklammern.
- Legen Sie die ganze Kammer in der Tüte, die mit der Kammer Kassette aus Corning kommt.
- Legen Sie die Kassette Kammer (n) in einen Behälter mit benetzten Papiertuch; Abdeckung der Behälter mit Plastikfolie und legen Sie sie in einem 37 ° C feucht, Zellkulturbrutschrank für ~ 24 Stunden. Diese Vorsichtsmaßnahmen (3,6, 3,9, 3,10) sicherzustellen, dass die Hybridisierungslösung nicht austrocknet während der Inkubation.
4. Post-Hybridisierung Verarbeitung
- Demontieren der Kammer Kassetten eins nach dem anderen. Tauchen Sie ein Dia und seine lifterslip in 2 x SSC bei 22 ° C. Die lifterslip natürlich fallen die Folie. Legen Sie es in 0,8 x SSC. Waschendie Folien in 0,8 x SSC zweimal, dann dreimal in 0,4 x SSC, 1-2 min insgesamt. Trocknen Sie die Folien mit einer Zentrifuge mit einem Dia-Adapter. Waschen Sie die LifterSlips mit Wasser und sparen für die Wiederverwendung später.
- Scannen Sie die Folien mit einem geeigneten Scanner, z. B. der Perkin Elmer ScanArray 5000 oder Molecular Devices Axon GenePix 4000B Mikroarray-Scanner. Scan bei Wellenlängen nahe 547 nm und 647 nm zu den entsprechenden Bild-Dateien zu erhalten.
- Verwenden Sie Programme wie BlueFuse zu Pixelintensitäten in der Eingabe-, Bild-Dateien von 4,2) zu quantifizieren. Überprüfen Sie die einzelnen Spots mit dem Programm auf ungewöhnliche Flecken auf der Microarrays von der weiteren Betrachtung ausschließen. Diese abnormen Stellen entstehen in der Regel aus armen slide Druck oder Dia-Handling nach der Hybridisierung.
- Verwenden Sie Programme wie GeneSpring und Excel für die Datenanalyse und-präsentation. Allgemeine Überlegungen für Microarray-Daten Normalisierung gelten, die über den Rahmen dieses Papiers.
- Nach zufriedenstellenden Signale af erhaltenter 4,3), Streifen der Microarray-Slides zur Wiederverwendung 7. Spülen Sie die Folien in Wasser, dann tauchen in vorgewärmten, 1 mM NaOH und 0,1 x SSC in einer Färbung Schale bei 62 ° C für 10-20 Minuten. Wiederholen Sie die Inkubationszeit einmal. Waschen Sie die Slides ein paar Mal im Wasser für bis zu 60 Minuten unter leichtem Schütteln bei 22 ° C. Trocknen Sie die Folien mit einer Zentrifuge, und speichern Sie bei 22 ° C.
- Zur Verwendung des regenerierten gleitet, ist es nicht notwendig, vor hybridisieren sie wieder. Einfach waschen Sie sie in Wasser durch Isopropanol folgte, und trocknen Sie die Dias rechts vor der Hybridisierung.
5. Repräsentative Ergebnisse:
Wir haben vor allem die beschriebenen Verfahren gefolgt, um globale miRNA-Expression in Tausenden von Proben-Profil, dh RNAs vom Zebrafisch auf den menschlichen Proben isoliert unter vielen verschiedenen Bedingungen. Abbildung 1 zeigt das gescannte Bild eines Mikroarrays, sehr präzise und starke Hybridisierungssignale auf den Beweis gleiten. Die Pearson correlatiauf Koeffizienten zwischen technischen Replikate von Microarray-Hybridisierung sind ~ 0,99 7 zeigt eine hervorragende Reproduzierbarkeit.
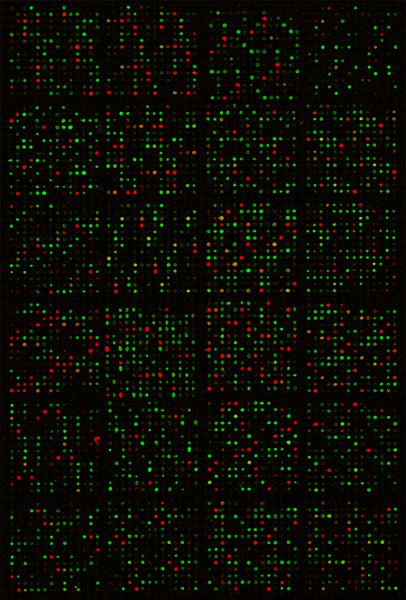
Abbildung 1 Composite Bild einer gescannten miRNA Microarray-Rutsche nach der Hybridisierung. Rote Flecken resultierte aus Hybridisierungen von der Referenz-DNA, grünen Flecken aus dem DY547-markierte RNA-Probe, während die gelben Flecken wurden von Hybridisierungen sowohl von der DNA und RNA, die gleichen Sonden.
Diskussion
Trotz der jüngsten Fortschritte in der Deep-Sequencing-Technologien, bleibt Microarray eine gute Wahl für High-Throughput-Analyse von DNA und RNA. Im Vergleich zum tiefen Sequenzierung sind Microarray-Experimenten billiger, und ein typischer Molekularbiologie-Labor können die meisten der Experimente und Datenanalyse in-Haus, das für Flexibilität erlaubt und spart Zeit durchzuführen. In der Zukunft, Microarrays wahrscheinlich gut geeignet, um intensiv zu befragen Gruppen von Genen, z. B. sind, können alle oder ein...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Arbeit wurde teilweise durch National Institute of Drug Abuse Center (P50 DA 011806) und United States Army Department of Defense (W81XWH-07-1 bis 0183) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Items | Verkäufer | Katalog-Nummer | Kommentare |
|---|---|---|---|
| NCode Multi-Species miRNA Microarray Probe Set V2 | Invitrogen | MIRMPS201 | Entwickelt auf Basis der miRBase Release 9.0 (Oktober 2006). Es enthält ~ 1.140 unverändert, 34-44 nt lange Oligonukleotide als Sonden für Wurm, Fliege, Zebrafisch, Maus, Ratte und Mensch miRNAs, und eine Reihe von internen Kontroll-Sonden wie snoRNAs. Die miRNA-Sonden werden Dubletten der Sequenzen komplementär zu reifen miRNAs, damit die Größe ~ 44 nt. Für die Analyse kann man auf miRNAs aus einer bestimmten Erbgut (s) von Interesse zu konzentrieren. |
| Trizol | Invitrogen | 15596018 | Wir haben auch bereichert, kleine RNA-Fraktion für die Kennzeichnung, obwohl der Gesamt-RNA-Proben schneller und einfacher zu erstellen und zu quantifizieren und für nachgelagerte Anwendungen wie mRNA-Analyse sind. |
| T4 RNA Ligase 1 | New England Biolabs | M0204L | |
| Ulysis Alexa Fluor 647 Nucleic Acid Labeling Kit | Invitrogen | U21660 | Dieses Kit oder ähnliche Produkte können verwendet werden, um experimentelle RNA-Proben oder ein Kontroll-RNA (anstelle von Kontroll-DNA) sowie Label werden. |
| 5'-PCU-DY547-3 ' | Dharmacon | Custom made | Kleine RNA-Fraktion kann in ähnlicher Weise durch Ligation bezeichnet werden. |
| Centrisep Spalten | Princeton Separations | CS-901 | |
| GAPSII beschichteten Objektträger | Corning | 40004 | Andere Arten von Folien können ebenfalls verwendet werden. |
| Microarray-Hybridisierung Kammern | Corning | 2551 oder 40080 | Andere Arten von Hybridisierung Kammern und Deckgläser sollten auch funktionieren. Mit kommerziell erhältlichen Hybridisierung Maschinen können Hybridisierung Zeit deutlich zu reduzieren, zB nach ~ 2 Stunden. |
| LifterSlips | ThErmo / Erie Scientific | 25X60I-M5439-001-LS | |
| BlueFuse | BlueGenome | ||
| GeneSpring | Agilent |
Referenzen
- Ambros, V. The functions of animal microRNAs. Nature. 431, 350-355 (2004).
- Friedman, R. C., Farh, K. K., Burge, C. B., Bartel, D. P. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res. 19, 92-105 (2009).
- Chekulaeva, M., Filipowicz, W. Mechanisms of miRNA-mediated post-transcriptional regulation in animal cells. Curr. Opin. Cell Biol. 21, 452-460 (2009).
- Farazi, T. A., Spitzer, J. I., Morozov, P., Tuschl, T. miRNAs in human cancer. J. Pathol. 223, 102-115 (2011).
- Small, E. M., Olson, E. N. Pervasive roles of microRNAs in cardiovascular biology. Nature. 469, 336-342 (2011).
- Thomson, J. M., Parker, J., Perou, C. M., Hammond, S. M. A custom microarray platform for analysis of microRNA gene expression. Nat. Methods. 1, 47-53 (2004).
- Zhang, X., Xu, W., Tan, J., Zeng, Y. Stripping custom microRNA microarrays and the lessons learned about probe:slide interactions. Anal. Biochem. 386, 222-227 (2009).
- Griffiths-Jones, S., Saini, H. K., van Dongen, S., Enright, A. J. miRBase: tools for microRNA genomics. Nucl. Acids. Res. 36, D154-D158 (2008).
- Landgraf, P. A mammalian microRNA expression atlas based on small RNA library sequencing. Cell. 129, 1401-1414 (2007).
- Chiang, H. R. Mammalian microRNAs: experimental evaluation of novel and previously annotated genes. Genes. Dev. 24, 992-1009 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten