このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
リボソーム結合した新生ポリペプチドの分離
要約
mRNA上の翻訳停止部位を同定する技術が記載されている。この手順は、変性ゲル電気泳動を用いた初期のチェーンの大きさの分析に続いて標的mRNAのin vitro翻訳、イン時にリボソーム上で蓄積新生ポリペプチドの分離に基づいています。
要約
翻訳伸長率は、非均一である。二次構造、コドン使用頻度をmRNAと1を参照して 、関連するタンパク質はレビューのためにメッセージにリボソームの動きを変更することがmRNAを。しかし、現在広く同義コドンの使用率が不均一な翻訳伸長率が1の主な原因であることを受け入れている。同義コドンは、同一の周波数で使用されていません。バイアスは、他の2よりも頻繁に使用されるいくつかのコドンを同義コドンの使用に存在しています。コドンバイアスは、生物と同様に2,3 -特定の組織である。また、コドンの使用頻度は、同族のtRNA 4の濃度に直接比例します。したがって、頻繁に使用されるコドンは、さらに頻繁なコドンが速くまれなものよりも翻訳されることを意味し、対応するtRNAの高い多数を持っています。したがって、使用頻度の低いコドン(潜在的な休止サイト)に富んでmRNA上の領域は、原則としてミサにリボソームの動きが遅くなりますそれぞれのサイズ5-8の新生ペプチドのGEの原因と蓄積。これらのポーズのサイトはレビューのために機能的なタンパク質の発現に及ぼす影響は、mRNAの安定性とタンパク質の折りたたみを持っている9を参照することができます。確かに、そのようなポーズサイトの緩和は、mRNAにリボソームの動きを変更することができ、その後、共同翻訳( インビボ )タンパク質のフォールディング1,7,10,11の効率に影響を与える可能性があることが示された。最終的には翻訳伸長中のmRNAに沿ってリボソームの移動に関するコドン使用頻度/ tRNAのコンテンツの影響に包括的な洞察を得ることが不可欠であるタンパク質合成のプロセスに結合されている細胞で、 生体内でのタンパク質の折り畳み過程を理解するために、 。
ここでは、様々な無細胞系6-8に翻訳与えられたmRNAの主要な翻訳停止部位を見つけるために使用することができる簡単なテクニックについて説明します。この手順は、新生ポリペプチドaccumulatiの分離に基づいています標的mRNAのin vitro翻訳中のリボソーム上でNG。根拠は、低周波のコドンでは、対応するサイズの新生ペプチドの増加量のリボソーム結果の滞留時間の増加が。in vitroで転写されたmRNA には放射性標識アミノ酸の存在下でin vitroでの翻訳反応に使用されていることです。新生鎖の検出を可能にします。リボソーム結合した新生ポリペプチド複合体を単離するために翻訳反応は、遠心分離に続いて、30%グリセロール溶液の上に階層化されています。ポリソームペレットの新生ポリペプチドはさらにリボヌクレアーゼで処理し、SDS PAGEで解決されます。このテクニックは、潜在的に任意のタンパク質で使用され、リボソームのmRNAに沿って動き、主要な休止部位の検出の分析を可能にすることができます。さらに、このプロトコルは、リボソームの動きを変えることができるので、潜在的にも目を変えることができる要素や条件を研究するために適応させることができます電子機能/タンパク質のコンフォメーション。
プロトコル
1。 DNAテンプレートの調製とin vitro転写
- 興味のある遺伝子はT7および/または例えばSP6転写プロモーターの下に複製されます。
- in vitro転写用テンプレートDNAは、ORFの終止コドンおよび/ またはmRNAの3 '末端の下流を切断し、適切な制限酵素で直線化されています。一つは、アガロースゲル電気泳動で制限酵素消化製品を実行することにより、プラスミドDNAの完全な線形化を確認する必要があります。
- 線状プラスミドは 、in vitro 転写反応に使用されます。テンプレートDNAの異なる濃度は 、in vitro転写のために必要な最適なDNA濃度を識別するためにテストすることができます。一般的には、AmbionのMEGAscript高利回り転写キット(Ambion社/ライフテクノロジーズ、ニューヨーク州グランドアイランド)で、1μgの直鎖状DNA、mRNAの40から60μgを得られます。in vitro転写メーカーの指示(Ambion社/ライフテクノロジーズ、グランドIslan後に行われますD、NY)。
- in vitro転写で以下の mRNAは、製造元の指示(AmbionのMEGAscript高利回り転写キット)によると塩化リチウム沈殿により精製されています。
- mRNAの整合性は、さらに、アクリルアミドまたはアガロースゲル上で電気泳動によって確認されています。
(2)in vitroのセルフリー翻訳
以下の手順に従ってくださいR abbit R eticulocyte溶解、RRL(プロメガ、マディソン、WI)を用いたin vitro翻訳のために:
- メーカーの指示に続くin vitro翻訳反応で 100μlを準備します。簡単に言えば、ヌクレアーゼフリーの水に2μlのアミノ酸混合物を引いたメチオニン(1mM)を、RNase阻害剤(Invitrogen社/ライフテクノロジーズ、グランドアイランド、NY)、放射性の20μCiの[35 S] -メチオニン(MPのバイオメディカルの10単位を追加ソロン、オハイオ州)、ウサギ網状赤血球ライセート(プロメガ社、マジソン、WI)の70μlの。
- 目をインキュベート暖かい翻訳反応を事前に5分間30℃eの反応。 5分間30℃での翻訳反応とインキュベートするmRNAを2μgのを追加します。
- 氷の上で翻訳反応を置くことによって反応を停止します。
3。 in vitro翻訳反応でから新生ポリペプチドの分離
- リボソーム(ポリソーム)にバインドされた新生ポリペプチドを単離するためには、翻訳反応は、100mM KCl、10mMのMgCI 2を含む10 mM Tris-HCl緩衝液のpH 7.6の30%グリセロール4.5 mlの上に重ね、100,000×gで遠心分離し4℃で1時間TLA-110ローター(ベックマン·コールター社、ブレア、カリフォルニア州)で
- 上清をさらに慎重に削除されます。
- 新生ポリペプチドを含有するポリソームペレットを0.5 mg / mlのリボヌクレアーゼ(Invitrogen社/ライフテクノロジーズ、ニューヨーク州グランドアイランド)とインキュベートを含む、1 mMのTris-HCl緩衝液pH 7.6の少量(10-15μL)に再懸濁している37℃で30分間で順番にペプチジル-tRNAをエステル結合の加水分解を高めるために、水酸化ナトリウムを10mMの最終濃度まで添加し、インキュベーションをさらに30分間継続されます。
in vitroで翻訳反応を発現することなく組み立てられたと同じ条件(記載されたように新生ペプチドの単離に続いて)の下で行われ、主要なコントロールとして機能するでしょう。密度勾配遠心分離抽出を施す前にピューロマイシンによる翻訳反応(s)の治療は、追加のコントロールとして機能することができる。
4。トリス - トリシンSDS PAGE上での新生ポリペプチドを解決する
- 孤立した新生ポリペプチドは、トリス - トリシンSDS-PAGEで分離し、分析されます。ゲルにロードされた材料の量は、タンパク質の標識、タンパク質翻訳の効率や他の多くのパラメータの効率に依存しますのでご注意ください。ロードされた材料の量は、BESを可能にするために経験的に調整する必要がありますトンの可視化と個々の新生ポリペプチド鎖の分離。
- 電気泳動ゲルは固定された後、真空ゲル染色を使用して、オートラジオグラフィーにかけ乾燥させた。新生ポリペプチドの分布はphosphoimagingを用いて観察しています。
5。代表的な結果
ウシγ-Bクリスタリンは、同様にE.のように、ウサギ網状赤血球ライセートで翻訳された大腸菌 S30抽出システム(プロメガ社、マジソン、WI)新生ポリペプチド鎖の分離に続いて、 図1は、リボソーム結合した新生鎖の分離に必要な手順を示しています。本研究での目的は、tRNAの翻訳すなわちレパートリーの環境を(( 大腸菌 )生物間のコドンバイアスの違いに起因するシステムを実質的哺乳類で異なる(RRL)と原核生物であることが知られて)変更すると、可能であれば、テストすることでしたmRNAに沿ってリボソームの動きを変更して、翻訳の一時停止部位の分布に影響を与える。変更リボソームの動きは翻訳中に蓄積新生ポリペプチド、並びにそれらの相対強度の大きさの分布の変化につながるはずだ。バンドの相対強度は一時停止の期間を反映しています。新生ポリペプチドは単離され、16.5%T、6%のTris-CトリシンSDS PAGE(図2)で解決されました。 γ-Bクリスタリンは20 kDaの蛋白質である。 RRLとE.非同一の長さと強度の新生ポリペプチドの観察大腸菌のシステムは、これらの2つのシステムのmRNAに沿ってリボソームの動きが異なる翻訳速度を、以下のことを示しています。たとえば、我々はE. 17.7 kDaの顕著なバンドが観察さRRLシステムの大腸菌ライセートとしない、それはE.に翻訳されるmRNAの3'-末端領域(ガンマBクリスタリンのエンコーディングのC末端領域)に追加の翻訳停止部位が存在することを示唆している大腸菌システム。我々は注意して、γ-Bクリスタリン港タンデムのArgコドン(AGG 1哺乳類のシステムで頻繁にありますが、非常にまれであることが知られてゆっくりとEに変換されます53、AGA 154) 大腸菌、E.に蓄積小説新生ペプチド大腸菌のシステム(〜17.7 kDaの図2.2)は、おそらくこれらのタンデム稀なコドンで停止リボソームによって引き起こされます。全くバンドがmRNAの不在( 図2.3)で観察されず、そのピューロマイシン治療がポリソーム( 図2.3)からの新生鎖の大部分を削除することができることに注意してください。ピューロマイシン(> 15分)による長期治療は、すべての新生鎖(データは示さず)を削除します。これは観測されたポリペプチド産物ではなくポリソームでcosedimenting mRNPsよりリボソーム関連した新生鎖であることを確認します。
上記のように、コドンバイアスは、生物だけでなく、組織固有のものです。代表的な例は、ここで説明する、明らかにするtRNA /コドンの翻訳すなわちレパートリーの環境を変更することを示します。バイアスは、mRNAに沿ってリボソームの変化した動きにつながる可能性があります。明らかに、この単純なテクニックだけでmRNAに沿って並進一時停止部位の同定を可能にするだけでなく、異なるシステム間での翻訳停止部位の迅速な比較を可能にすることはできません。
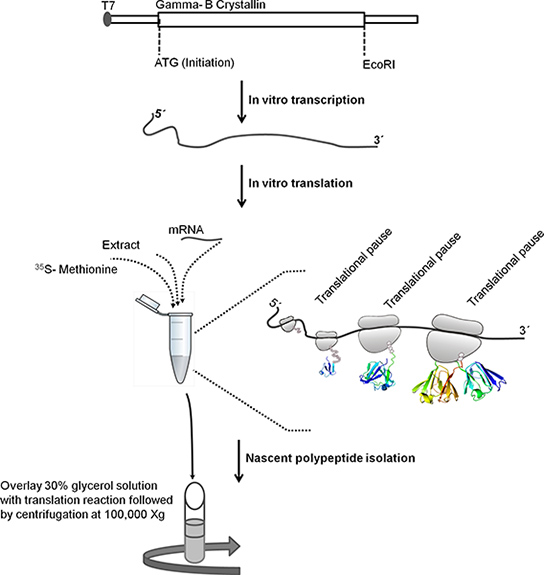
図1。リボソーム結合した新生ポリペプチドを単離に必要な手順を説明する回路図は、ウシ γ-BクリスタリンのORF配列(528塩基対)がpBSKSで下流にT7プロモーターのクローニングした+。 in vitro転写用テンプレートDNAをEcoRIで処理することにより線形化されました。線形化されたテンプレートは、in vitro転写のために使用されていました。 DNAテンプレートのT7プロモーター下流に、γ-Bクリスタリン遺伝子を転写するT7 RNAポリメラーゼによって認識されている。 mRNAは、塩化リチウム沈澱法を用いて精製されています。精製したmRNAは、in vitro翻訳のために使用されます。以下インキュベーション、翻訳反応は、30%グリセロール溶液の上に重層し、4℃で1時間遠心分離されるリボソーム結合した新生ポリペプチドを単離するための100,000 x gで°C。
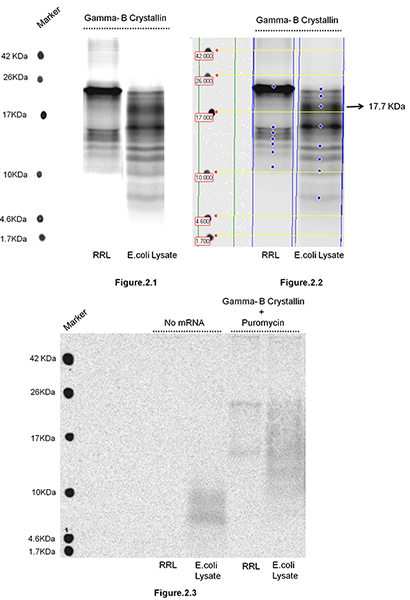
図2。ウサギ網状赤血球ライセート(RRL)とE.に翻訳ウシγ-Bクリスタリンの新生ポリペプチドの分離大腸菌ライセートを 2μgのmRNAは、RRL または E を含有する100μlの反応混合した5分20μCiの[35 S] -メチオニン、30℃でインキュベートしたメーカーの指示に従って大腸菌 S30抽出システム(Promega、Madison、WI)を。以下のインキュベーションは、翻訳反応は、100mM KCl、10mMのMgCI 2を含む10 mM Tris-HCl緩衝液のpH 7.6で30%グリセロール4.5 mlの上に階層化し、で4℃、100,000×gで1時間遠心分離したTLA-110ローター(ベックマン·コールター社、ブレア、カリフォルニア州)。隔離されたポリリボソームペレットresuでした(Invitrogen社/ライフテクノロジーズ、ニューヨーク州グランドアイランド)のNaOH(などのプロトコルのセクションで説明)で処理、続いては0.5 mg / mlのリボヌクレアーゼを含む、1 mMのトリス-HCl pH 7.6の20μlにspended。新生ポリペプチドは、16.5パーセントT、6%のTris-CトリシンSDS PAGEゲル上で解決されました。ゲルを乾燥し、オートラジオグラフィー用に公開されました。暴露後のゲルは、GEヘルスケアの台風イメージングスキャナを使用してスキャンしました。 図2.1は、翻訳反応は、2つの異なるシステム(RRLと大腸菌 )で行われた後、分離し、解決する新生ポリペプチドゲルを示しています。 図2.2は、ゲルがイメージクワントTLにより解析した、v2005ソフトウェア、2つのシステムに蓄積新生ポリペプチドの分子量と強度を簡単に分析し、比較することができることを示すための例としてここで使用する図2.3は、コントロールの2つのセットを示しています。翻訳後、SDS PAGEで分離し、解決する新生ポリペプチドを反応が組み立てられた反応は、遠心分離に供した前に、mRNAおよび/または後にすることなく翻訳反応は、ピューロマイシン(5分、最終濃度1 mM)で処理した。
ディスカッション
再現可能な結果を得るためには、in vitro転写·翻訳反応に使用されるコンポーネントの品質と濃度が非常に重要です。現在の研究では、慎重に処理した場合、再現性の高いデータを提供する市販のキット及び抽出物を使用しています。ただし、必要であれば、翻訳、有能な抽出物は、自分の選択した細胞から調製することができる。 mRNAの品質は翻訳に影響を与えることがないの?...
開示事項
利害の衝突が宣言されません。
謝辞
この作品は、ヒューマン·フロンティア·サイエンス·プログラム助成RGP0024によって資金を供給された。
資料
| Name | Company | Catalog Number | Comments |
| 試薬/キットの名前 | 会社 | カタログ番号 | |
| MEGAscript T7高収率転写キット | Ambionの | AM1333 | |
| リボヌクレアーゼ阻害剤 | インビトロジェン | 15518012 | |
| トランス[35 S] -ラベル | MPのバイオメディカル | 0151006 | |
| リボヌクレアーゼ | インビトロジェン | 12091 | |
| ウサギ網状赤血球ライセートシステム、ヌクレアーゼは、治療 | プロメガ | L4960 | |
| リニアテンプレートの大腸菌 S30抽出システム | プロメガ | L1030 | |
| 遠心分離 | ベックマン·コールター | オプティマTLX超遠心 | |
| 貯蔵燐光体オートラジオグラフY | GEヘルスケア | 台風9410可変モードイメージャ | |
| 新生ポリペプチド分析用ソフトウェア | GEヘルスケア | 画像クワントTL、v2005 |
参考文献
- Komar, A. A. A pause for thought along the co-translational folding pathway. Trends Biochem. Sci. 34, 16-24 (2009).
- Sharp, P. M., Cowe, E., Higgins, D. G., Shields, D. C. Codon usage patterns in Escherichia coli, Bacillus subtilis, Saccharomyces cerevisiae, Schizosaccharomyces pombe, Drosophila melanogaster and Homo sapiens; a review of the considerable within-species diversity. Nucleic Acids Res. 16, 8207-8210 (1988).
- Dittmar, K. A., Goodenbour, J. M., Pan, T. Tissue-specific differences in human transfer RNA expression. PLoS Genet. 2, e221 (2006).
- Ikemura, T. Codon usage and tRNA content in unicellular and multicellular organisms. Mol. Biol. Evol. 2, 13-34 (1985).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. EMBO J. 7, 3559-3569 (1998).
- Krasheninnikov, I. A., Komar, A. A., Adzhubei, I. A. Nonuniform size distribution of nascent globin peptides, evidence for pause localization sites, and a cotranslational protein-folding model. J. Protein Chem. 10, 445-454 (1991).
- Komar, A. A., Lesnik, T., Reiss, C. Synonymous codon substitutions affect ribosome traffic and protein folding during in vitro translation. FEBS Lett. 462, 387-391 (1999).
- Komar, A. A., Jaenicke, R. Kinetics of translation of γ B crystallin and its circularly permutated variant in an in vitro cell-free system: possible relations to codon distribution and protein folding. FEBS Lett. 376, 195-198 (1995).
- Jha, S., Komar, A. A. Birth, life and death of nascent polypeptide chains. Biotechnol. J. 6, 623-640 (2011).
- Thanaraj, T. A., Argos, P. Ribosome-mediated translational pause and protein domain organization. Protein Sci. 5, 1594-1612 (1996).
- Kimchi-Sarfaty, C., Oh, J. M., Kim, I. W., Sauna, Z. E. A "silent" polymorphism in the MDR1 gene changes substrate specificity. Science. 315, 525-528 (2007).
- Schägger, H., von Jagow, G. Tricine-sodium dodecyl sulfate-polyacrylamide gel electrophoresis for the separation of proteins in the range from 1 to 100 kDa. Anal. Biochem. 166, 368-379 (1987).
- Shirole, N., Balasubramanian, S., Yanofsky, C., Cruz-Vera, L. Isolation of Translating Ribosomes Containing Peptidyl-tRNAs for Functional and Structural Analyses. J. Vis. Exp. (48), e2498 (2011).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324, 218-223 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved