Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Isolement du ribosome Bound polypeptides naissants
Dans cet article
Résumé
Une technique pour identifier les sites de pause translationnelle sur l'ARNm est décrite. Cette procédure est basée sur l'isolement des polypeptides naissants qui s'accumulent sur les ribosomes lors de la traduction in vitro d'un ARNm cible, suivie par l'analyse de la taille des chaînes naissantes en utilisant une électrophorèse sur gel dénaturant.
Résumé
Le taux d'allongement de translation n'est pas uniforme. ARNm structure secondaire, l'usage des codons et ARNm des protéines associées peuvent modifier le mouvement du ribosome sur le message pour revue, voir 1. Cependant, il est maintenant largement admis que l'usage des codons synonymes est la principale cause de non-uniformes des taux d'allongement de translation 1. Codons synonymes ne sont pas utilisés avec une fréquence identique. Un biais existe dans l'utilisation des codons synonymes avec certains codons utilisés plus fréquemment que d'autres 2. Codon biais est l'organisme ainsi que le tissu spécifique 2,3. En outre, la fréquence de l'usage des codons est directement proportionnelle à la concentration des ARNt apparenté 4. Ainsi, un codon fréquemment utilisé aura plus multitude de ARNt correspondants, ce qui implique en outre que d'un codon fréquentes seront traduits plus rapidement qu'une rares. Ainsi, les régions sur l'ARNm enrichi en codons rares (sites de pause potentiels) seront, en règle ralentir le mouvement du ribosome sur le message d'accumulation et de la cause des peptides naissants des tailles respectives 5-8. Ces sites de pause peut avoir un impact fonctionnel sur l'expression des protéines, stabilité de l'ARNm et le repliement des protéines pour revue, voir 9. En effet, il a été montré que l'atténuation de ces sites de pause peut altérer le mouvement du ribosome sur l'ARNm et par la suite peut affecter l'efficacité du co-traductionnelle repliement des protéines (in vivo) 1,7,10,11. Pour comprendre le processus de repliement des protéines in vivo, dans la cellule, qui est finalement couplé au processus de synthèse des protéines, il est essentiel d'obtenir un aperçu complet sur l'impact du codon d'utilisation / ARNt contenu sur le mouvement des ribosomes le long de l'ARNm en cours d'élongation translationnelle .
Nous décrivons ici une technique simple qui peut être utilisé pour localiser les principaux sites de pause de traduction pour un ARNm donné traduit en diverses des systèmes acellulaires 6-8. Cette procédure est basée sur l'isolement des polypeptides naissante accumulating sur les ribosomes lors de la traduction in vitro d'un ARNm cible. La logique est celle à basse fréquence des codons, l'augmentation du temps de séjour des résultats ribosomes dans des quantités accrues de peptides naissants des tailles correspondantes. Dans ARNm transcrit in vitro est utilisé pour des réactions de translation in vitro de la présence d'acides aminés marqués radioactivement pour permettre la détection des chaînes naissantes. Afin d'isoler les ribosomes liés naissantes complexes polypeptidiques la réaction de traduction est posée sur le dessus de la solution de glycérol à 30% suivie d'une centrifugation. Polypeptides naissants dans polysomique granulés sont traités avec la ribonucléase A et résolus par SDS-PAGE. Cette technique peut être éventuellement utilisées pour une protéine et permet l'analyse de mouvement le long du ribosome ARNm et la détection des sites de pause principaux. En outre, ce protocole peut être adapté pour étudier les facteurs et conditions qui peuvent altérer le mouvement du ribosome et donc potentiellement peut également modifier ee fonction / conformation de la protéine.
Protocole
1. Préparation Patron de l'ADN et la transcription in vitro
- Le gène d'intérêt est cloné dans T7 et / ou, par exemple SP6 promoteur transcriptionnel.
- Pour la transcription in vitro de la matrice d'ADN est linéarisé par une enzyme de restriction appropriée de coupe en aval du codon stop ORF et / ou de l'ARNm extrémité 3 '. Il faut vérifier linéarisation complète de l'ADN plasmidique en exécutant le produit de digestion de restriction à l'électrophorèse sur gel d'agarose.
- Le plasmide linéarisé est utilisé pour en réaction de transcription in vitro. Concentration différente de la matrice ADN peuvent être testés pour déterminer la concentration d'ADN optimale requise pour la transcription in vitro. En règle générale, avec Ambion Trousse MEGAscript à haut rendement de transcription (Ambion / Life Technologies, Grand Island, NY), 1 pg d'ADN linéarisé donne 40-60 ug d'ARNm. La transcription in vitro se fait suivant les instructions du fabricant (Ambion / Life Technologies, Grand Island, État de New York).
- Après transcription in vitro de l'ARNm est purifié par précipitation au chlorure de lithium selon les instructions du fabricant (Ambion de MEGAscript Kit Haut de transcription de rendement).
- l'intégrité ARNm est encore vérifiée par électrophorèse sur des gels d'acrylamide ou d'agarose.
2. Traduction cellulaire in vitro gratuit
Pour la traduction in vitro en utilisant R abbit R eticulocyte Lysate, RRL (Promega, Madison, WI), suivez les étapes ci-dessous:
- Préparer 100 pi dans l'enseignement de traduction in vitro la réaction constructeur suivante. En bref, dans la nucléase l'eau libre, ajouter 2 pl mélange d'acides aminés moins méthionine (1 mM), 10 unités d'inhibiteur de RNase (Invitrogen / Life Technologies, Grand Island, NY), 20 pCi de matières radioactives [35 S]-méthionine (MP Biomedicals, Solon, OH), 70 pi de lysat de réticulocytes de lapin (Promega, Madison, WI).
- Incuber eréaction e à 30 ° C pendant 5 min à l'avance au chaud la réaction de traduction. Ajouter 2 ug d'ARNm de la réaction de traduction et incuber à 30 ° C pendant 5 min.
- Arrêter la réaction en plaçant la réaction de traduction sur la glace.
3. Isolement de polypeptides naissants de réaction de traduction in vitro
- À isoler polypeptide naissant lié aux ribosomes (polysomes), réaction de traduction est couché au-dessus de 4,5 ml de glycérol 30% dans 10 mM de Tris-HCl de pH 7,6 tampon, contenant 100 mM, KCl 10 mM MgCI 2, et centrifugé à 100 000 xg dans un TLA-110 du rotor (Beckman Coulter, Inc, Brea, CA) pendant 1 h à 4 ° C.
- Le surnageant est encore soigneusement enlevées.
- Le culot contenant polysomique polypeptides naissants est remis en suspension dans un petit volume (10-15 pi) de 1 mM pH tampon Tris-HCl 7,6, contenant 0,5 mg / ml ribonucléase A, (Invitrogen / Life Technologies, Grand Island, NY) et incubées pendant 30 min à 37 ° C. Dans l'ordrepour améliorer l'hydrolyse de la liaison ester d'peptidyl-ARNt, NaOH est ajouté à une concentration finale de 10 mM, et l'incubation est poursuivie pendant 30 minutes supplémentaires.
Réaction de traduction in vitro assemblés sans ARNm et réalisée dans les mêmes conditions (suivie de l'isolement de peptides naissants comme cela a été décrit) servirait de contrôle majeur. Le traitement de la réaction de traduction (s) à la puromycine avant de soumettre l'extrait à une centrifugation en gradient de densité peut servir comme un contrôle supplémentaire.
4. Résoudre le polypeptide naissant sur Tris-tricine SDS-PAGE
- Les polypeptides isolés naissantes sont résolus et analysés sur Tris-tricine SDS-PAGE. S'il vous plaît noter que le montant de la matière chargée sur le gel dépend de l'efficacité de l'étiquetage des protéines, l'efficacité sur la traduction des protéines et de nombreux autres paramètres. La quantité de matière chargée doit être ajustée de façon empirique pour permettre aux besvisualisation t et la séparation des différents naissantes chaînes polypeptidiques.
- Après les gels d'électrophorèse sont fixés, séché à l'aide de gel sous vide teinturier et soumis à une autoradiographie. La distribution des polypeptides naissants sont observées à l'aide phosphoimaging.
5. Les résultats représentatifs
Bovine Gamma-B cristallin a été traduit en lysat de réticulocytes de lapin ainsi que dans E. coli S30 Systèmes Extrait (Promega, Madison, WI), suivie par l'isolement des chaînes polypeptidiques naissantes. La figure 1 illustre les étapes impliquées dans l'isolement des ribosomes liés chaînes naissantes. Dans la présente étude, le but était de tester, en cas de changement de l'environnement du répertoire c.-à-traduction de ARNt (connu pour être sensiblement différente chez les mammifères (RRL) et procaryotes (E. coli) des systèmes en raison de différences dans les biais de codon entre les organismes) peut modifier le mouvement du ribosome le long d'ARNm et de la distribution des sites de pause de traduction.Altered mouvement ribosome devrait conduire à des changements dans la distribution des tailles des polypeptides naissants qui s'accumulent pendant la traduction, ainsi que leurs intensités relatives. Les intensités relatives des bandes de refléter la durée de la pause. Les polypeptides naissants ont été isolés et résolus sur les 16,5% T, 6% C Tris-tricine SDS-PAGE (Figure 2). Gamma-B cristallin est une protéine de 20 kDa. L'observation des polypeptides naissants de non-identiques longueurs et des intensités de RRL et E. coli systèmes indique que le mouvement le long des ribosomes ARNm dans ces deux systèmes suivants cinétique de traduction différents. Par exemple, nous avons observé une bande importante de 17,7 kDa dans E. lysat coli et non dans le système de RRL, ce qui suggère qu'il existe un site de pause additionnel de translation dans la région 3'-fin de l'ARNm (encodage région C-terminale de Gamma B cristallin), quand il est traduit en E. coli système. Nous notons que Gamma-B ports cristallin tandem Arg codons (AGG 153 et AGA 154) qui sont fréquentes dans les systèmes mammaliens, mais sont connus pour être extrêmement rare et lentement traduit dans E. coli. un nouveau peptide naissant s'accumulent dans E. système coli (~ 17,7 kDa Figure 2.2) est probablement causée par le ribosome en s'arrêtant à ces codons rares en tandem. S'il vous plaît noter que pas de bandes peuvent être observés en l'absence de l'ARNm (Figure 2.3) et que le traitement puromycine supprime la plupart des chaînes naissantes de polysomes (Figure 2.3). Un traitement prolongé à la puromycine (> 15 min) supprime toutes les chaînes naissantes (données non présentées). Cela confirme que les produits observés polypeptidiques sont associés des chaînes naissantes ribosome, plutôt que mRNP cosedimenting aux polysomes.
Comme mentionné ci-dessus, le biais de codon est l'organisme ainsi que des tissus spécifiques. L'exemple représentatif décrit ici, indique clairement que le changement de l'environnement du répertoire c.-à-traduction de ARNt / codonbiais peut conduire à des mouvements modifié du ribosome le long de l'ARNm. Évidemment, cette technique simple peut non seulement permettre l'identification des sites de pause de translation le long d'ARNm, mais permet également une comparaison rapide des sites de pause de traduction entre les différents systèmes.
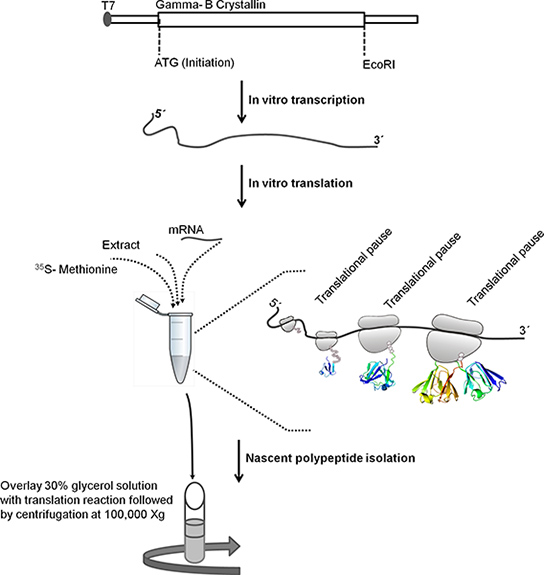
Figure 1. Schéma expliquant les étapes à suivre pour isoler les ribosomes liés polypeptides naissants. Bovine Gamma-B cristallin séquence ORF (528 paires de bases) a été cloné en aval du promoteur T7 dans pBSKS +. Pour la transcription in vitro de la matrice d'ADN est linéarisé en le traitant avec EcoRI. Modèle linéarisé a été utilisé pour la transcription in vitro. Promoteur T7 dans la matrice d'ADN est reconnue par l'ARN polymérase T7 qui transcrit l'aval Gamma-gène B cristallin. ARNm est purifié en utilisant la méthode de précipitation au chlorure de lithium. L'ARNm purifié est utilisé pour la traduction in vitro. Suivantd'incubation, la réaction est la traduction en couches sur le dessus de la solution de glycérol à 30% et centrifugé pendant 1 h à 4 ° C à 100.000 X g d'isoler ribosome liés polypeptides naissants.
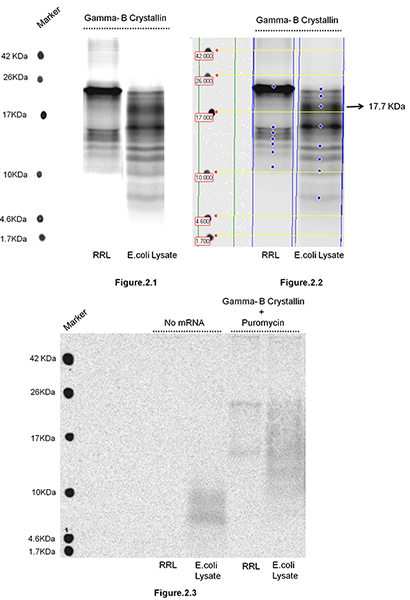
Figure 2. Isolement des bovins polypeptides cristallines Gamma-B naissantes traduites en lysat de réticulocytes de lapin (RRL) et E. Lysate coli. 2 mRNA pg a été mélangé dans 100 ul de réaction contenant RRL ou E. coli S30 Extrait System (Promega, Madison, WI) selon les instructions du fabricant avec 20 pCi de [35 S]-méthionine et incubées à 30 ° C pendant 5 min. Après l'incubation de la réaction de traduction a été superposé au-dessus de 4,5 ml de glycérol 30% dans 10 mM de pH tampon Tris-HCl 7,6, contenant 100 mM, KCl 10 mM MgCI 2, et centrifugé pendant 1 h à 4 ° C et 100.000 X g dans une TLA-rotor 110 (Beckman Coulter, Inc, Brea, CA). Le culot a été isolé polyribosome resuspended dans 20 pi de 1 mM de Tris-HCl pH 7,6, contenant 0,5 mg / ml ribonucléase A (Invitrogen / Life Technologies, Grand Island, NY), suivie par un traitement avec NaOH (comme décrit dans la section du protocole). Polypeptides naissants ont été résolus sur les 16,5% T, 6% C Tris-Tricine SDS-PAGE sur gel. Le gel a été séché et exposé pour autoradiographie. Suite à l'exposition du gel a été numérisé à l'aide du scanner de GE Healthcare Imaging Typhoon. La figure 2.1 montre le gel avec des polypeptides naissants isolés et résolus après des réactions de traduction fait dans deux systèmes différents (RRL et E. coli). Figure 2.2 Le gel a été analysée par l'image Quant TL ., logiciels v2005 et utilisé ici comme un exemple pour montrer que le poids moléculaire et l'intensité des polypeptides naissants qui s'accumulent dans deux systèmes peuvent être facilement analysé et comparé la figure 2.3 montre deux ensembles de contrôles: les polypeptides naissants isolés et résolus sur SDS-PAGE après la traduction réactions ont été assembléssans ARNm et / ou après les réactions de conversion ont été traitées avec la puromycine (5 min, concentration finale. 1 mM) avant la réaction a été soumis à une centrifugation.
Discussion
Pour obtenir des résultats reproductibles, la qualité et la concentration des composants utilisés pour la transcription in vitro et les réactions de traduction sont essentielles. Dans l'étude actuelle, nous avons utilisé des kits disponibles dans le commerce et les extraits qui fournissent des données hautement reproductibles, s'il est manipulé avec soin. Cependant, la traduction-compétentes extraits peuvent être préparés à partir de la cellule de son choix, si nécessaire. Qualit?...
Déclarations de divulgation
Pas de conflits d'intérêt déclarés.
Remerciements
Ce travail a été financé par Human Frontier Science Program de subvention RGP0024.
matériels
| Name | Company | Catalog Number | Comments |
| Nom de réactif / Kit | Entreprise | Numéro de catalogue | |
| MEGAscript T7 haute Kit de transcription rendement | Ambion | AM1333 | |
| Inhibiteur de la ribonucléase | Invitrogen | 15518012 | |
| Trans [35 S]-Label | MP Biomedicals | 0151006 | |
| Ribonucléase-A | Invitrogen | 12091 | |
| De réticulocytes de lapin Lysate système, nucléase traité | Promega | L4960 | |
| E. coli S30 Extrait système pour les modèles linéaires | Promega | L1030 | |
| Centrifugation | Beckman Coulter | Optima TLX Ultracentrifugeuse | |
| Luminophore de mémorisation autoradiographiey | GE Healthcare | Typhoon 9410 imageur mode variable | |
| Logiciel pour l'analyse du polypeptide naissant | GE Healthcare | Quant image TL, v2005 |
Références
- Komar, A. A. A pause for thought along the co-translational folding pathway. Trends Biochem. Sci. 34, 16-24 (2009).
- Sharp, P. M., Cowe, E., Higgins, D. G., Shields, D. C. Codon usage patterns in Escherichia coli, Bacillus subtilis, Saccharomyces cerevisiae, Schizosaccharomyces pombe, Drosophila melanogaster and Homo sapiens; a review of the considerable within-species diversity. Nucleic Acids Res. 16, 8207-8210 (1988).
- Dittmar, K. A., Goodenbour, J. M., Pan, T. Tissue-specific differences in human transfer RNA expression. PLoS Genet. 2, e221 (2006).
- Ikemura, T. Codon usage and tRNA content in unicellular and multicellular organisms. Mol. Biol. Evol. 2, 13-34 (1985).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. EMBO J. 7, 3559-3569 (1998).
- Krasheninnikov, I. A., Komar, A. A., Adzhubei, I. A. Nonuniform size distribution of nascent globin peptides, evidence for pause localization sites, and a cotranslational protein-folding model. J. Protein Chem. 10, 445-454 (1991).
- Komar, A. A., Lesnik, T., Reiss, C. Synonymous codon substitutions affect ribosome traffic and protein folding during in vitro translation. FEBS Lett. 462, 387-391 (1999).
- Komar, A. A., Jaenicke, R. Kinetics of translation of γ B crystallin and its circularly permutated variant in an in vitro cell-free system: possible relations to codon distribution and protein folding. FEBS Lett. 376, 195-198 (1995).
- Jha, S., Komar, A. A. Birth, life and death of nascent polypeptide chains. Biotechnol. J. 6, 623-640 (2011).
- Thanaraj, T. A., Argos, P. Ribosome-mediated translational pause and protein domain organization. Protein Sci. 5, 1594-1612 (1996).
- Kimchi-Sarfaty, C., Oh, J. M., Kim, I. W., Sauna, Z. E. A "silent" polymorphism in the MDR1 gene changes substrate specificity. Science. 315, 525-528 (2007).
- Schägger, H., von Jagow, G. Tricine-sodium dodecyl sulfate-polyacrylamide gel electrophoresis for the separation of proteins in the range from 1 to 100 kDa. Anal. Biochem. 166, 368-379 (1987).
- Shirole, N., Balasubramanian, S., Yanofsky, C., Cruz-Vera, L. Isolation of Translating Ribosomes Containing Peptidyl-tRNAs for Functional and Structural Analyses. J. Vis. Exp. (48), e2498 (2011).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324, 218-223 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon