É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Isolamento de Ribossomos ligados a polipeptídeos nascentes
Neste Artigo
Resumo
Uma técnica para identificar locais de pausa de translação sobre mRNA é descrito. Este procedimento é baseado no isolamento de polipéptidos nascentes que se acumulam nos ribossomas durante in vitro tradução de um ARNm alvo, seguido de análise de tamanho das cadeias nascentes utilizando uma electroforese em gel de desnaturação.
Resumo
A taxa de alongamento de translação é não-uniforme. mRNA estrutura secundária, uso de códon mRNA e proteínas associadas podem alterar movimento ribossomo na mensagem para revisão ver 1. No entanto, é agora amplamente aceito que o uso de codons sinônimos é a principal causa da não-uniformes taxas de alongamento de translação 1. Codons sinônimos não são utilizados com freqüência idêntica. Um viés existe no uso de codons sinônimos com alguns códons utilizados com mais freqüência do que os outros 2. Códon viés é organismo, assim como tecidos específicos 2,3. Além disso, a frequência de utilização de codões é directamente proporcional à concentração de tRNAs cognato 4. Assim, um codão frequentemente utilizadas terão maior multiplicidade de tRNAs correspondentes, o que implica ainda que um codão frequente será traduzida mais rápido do que um um pouco frequentes. Assim, as regiões de mRNA enriquecido em códons raros locais (pausa potenciais), regra geral diminuam o movimento ribossomo na message e causa acúmulo de peptídeos nascentes dos respectivos tamanhos 5-8. Estes sites de pausa pode ter impacto funcional sobre a expressão de proteínas, a estabilidade do mRNA e enovelamento de proteínas para revisão ver 9. Com efeito, foi demonstrado que a atenuação dos sítios de pausa tais pode alterar movimento ribossoma em ARNm e, subsequentemente, pode afectar a eficiência da co-translacional enrolamento de proteínas (in vivo) 1,7,10,11. Para compreender o processo de enrolamento de proteínas in vivo, na célula, que é finalmente acoplado ao processo de síntese de proteína que é essencial para obter uma percepção abrangentes para o impacto do codão de conteúdo de uso / tRNA sobre o movimento dos ribossomas ao longo do mRNA durante o alongamento translacional .
Descrevemos aqui uma técnica simples que pode ser usado para localizar locais de tradução importantes de pausa para uma dada mRNA traduzido em vários sistemas isentos de células de 6-8. Este procedimento é baseado no isolamento de polipéptidos nascente accumulating em ribossomas durante a tradução in vitro de um mRNA alvo. A razão é que a baixa frequência codões, o aumento no tempo de residência dos resultados ribossomas em quantidades aumentadas de péptidos nascentes dos tamanhos correspondentes. Em mRNA transcrito in vitro é utilizado para reacções de translação in vitro na presença de marcados radioactivamente aminoácidos para permitir a detecção das cadeias nascentes. A fim de isolar bound ribossomas nascentes complexos polipeptídicas a reacção de tradução é em camadas no topo de solução de glicerol a 30%, seguido por centrifugação. Polipéptidos nascente na polissomal pelete são ainda tratadas com ribonuclease A e resolvidos por SDS-PAGE. Esta técnica pode ser potencialmente usada para qualquer proteína e permite a análise de movimento ao longo do mRNA ao ribossoma e à detecção dos sítios de pausa principais. Além disso, este protocolo pode ser adaptado para estudar factores e condições que podem alterar o movimento ribossoma e assim potencialmente também podem alterar the função / conformação da proteína.
Protocolo
1. Preparação de ADN de modelo e transcrição in vitro
- O gene de interesse é clonado sob T7 e / ou, por exemplo SP6 promotor transcricional.
- Para transcrição in vitro do DNA molde é linearizado com uma enzima de restrição apropriada de corte a jusante do codão de paragem ORF e / ou mRNA extremidade 3 '. Uma das necessidades para verificar a linearização completa do DNA de plasmídeo, executando o produto da digestão de restrição em electroforese em gel de agarose.
- O plasmídeo linearizado é usado para reacção de transcrição in vitro. Concentração diferente de ADN molde pode ser testado para identificar a concentração de DNA óptima necessária para transcrição in vitro. Geralmente, com Kit Ambion da transcrição de alto rendimento MEGAscript (Ambion / Life Technologies, Grand Island, NY), 1 ug de DNA linearizado produz 40-60 ug de mRNA. Transcrição in vitro é efectuado de acordo com as instruções do fabricante (Ambion / Life Technologies, Grand Island, NY).
- Após transcrição in vitro do ARNm é purificado por precipitação com cloreto de lítio de acordo com as instruções do fabricante (Kit Ambion da transcrição MEGAscript de alto rendimento).
- integridade do mRNA é ainda verificado por electroforese em géis de acrilamida ou de agarose.
2. Tradução de células in vitro gratuita
Para a tradução in vitro utilizando R abbit R eticulocyte Lysate, RRL (Promega, Madison, WI) siga os passos abaixo:
- Preparar 100 ul na instrução tradução in vitro fabricante seguinte reacção de. Resumidamente, em água livre de nuclease adicionar 2 ul mistura de ácido amino menos metionina (1 mM), 10 unidades de RNase inibidor (Invitrogen / Life Technologies, Grand Island, NY), 20 uCi de radioactivos [35 S]-Metionina Biomedicals (MP, Solon, OH), 70 ul de reticulócitos de coelho Lysate (Promega, Madison, WI).
- Incubar ªreacção e, a 30 ° C durante 5 min de pré reacção a quente de tradução. Adicionar 2 ug de mRNA para a reacção de tradução e incubar a 30 ° C durante 5 min.
- Parar a reacção através da colocação da reacção de tradução em gelo.
3. Isolamento de polipéptidos a partir de nascentes in vitro reacção de tradução
- Para isolar polipéptido nascente ligado a ribossomas (polissoma), a reacção de tradução é colocado em camadas sobre 4,5 ml de glicerol 30% em 10 mM de tampão Tris-HCl pH 7,6, contendo 100 mM de KCl, 10 mM de MgCI 2, e centrifugado a 100.000 xg em um TLA-110 do rotor (Beckman Coulter, Inc, Brea, CA) durante 1 hora a 4 ° C.
- O sobrenadante é ainda mais cuidadosamente removido.
- O sedimento contendo polipéptidos polissomal nascentes é ressuspenso em um pequeno volume (10-15 uL) de 1 mM de tampão Tris-HCl pH 7,6, contendo 0,5 mg / ml ribonuclease A, (Invitrogen / Life Technologies, Grand Island, NY) e incubadas durante 30 min a 37 ° C. A fimpara aumentar a hidrólise do peptidil-tRNA éster ligação, NaOH é adicionado a uma concentração final de 10 mM, ea incubação é continuada durante mais 30 min.
In vitro reacção de tradução montado sem mRNA e executada sob as mesmas condições (seguido por isolamento de péptidos nascentes como foi descrito) serviria como um controlo principal. O tratamento da reacção de tradução (s) com puromicina antes de se submeter o extracto de centrifugação em gradiente de densidade pode servir como um controle adicional.
4. Resolvendo o Polipeptídeo Nascente em SDS-PAGE Tris-tricina
- Os polipéptidos isolados nascentes são resolvidos e analisados em Tris-tricina SDS-PAGE. Note que a quantidade do material carregado no gel será dependente da eficiência de rotulagem de proteína, a eficiência de tradução da proteína e os parâmetros de muitos outros. A quantidade de material carregado deve ser ajustado empiricamente para permitir que os besvisualização T e separação de individuais nascentes cadeias polipeptídicas.
- Depois de géis de electroforese são fixados, secou-se utilizando vácuo gel Dyer e submetido a autorradiografia. A distribuição de polipéptidos nascentes são observados utilizando phosphoimaging.
5. Os resultados representativos
Bovina Gamma-B cristalina foi traduzido em lisado de reticulócitos de coelho, bem como em E. coli S30 Extracto Systems (Promega, Madison, WI), seguido por isolamento de nascentes cadeias polipeptídicas. A Figura 1 ilustra os passos envolvidos no isolamento de ribossomas ligados cadeias nascentes. No presente estudo o objectivo foi testar, se a alteração no ambiente de repertório tradução isto é, de tRNAs (conhecido como sendo substancialmente diferente em mamíferos (RRL) e procarióticos (E. coli) sistemas devido a diferenças na polarização codão entre os organismos) pode alterar movimento ribossomo ao longo do mRNA e efetuar a distribuição dos locais de pausa de tradução.Movimento ribossoma Altered deve conduzir a alterações na distribuição de tamanhos de polipéptidos nascentes que se acumulam durante a tradução, bem como as suas intensidades relativas. Intensidades relativas das bandas função da duração da pausa. Os polipéptidos nascentes foram isolados e resolvidos em 16,5% T, 6% C Tris-tricina SDS-PAGE (Figura 2). Gama-B cristalina é uma proteína de 20 kDa. A observação de polipéptidos nascentes de não idênticos comprimentos e intensidades em RRL e E. coli sistemas indica que o movimento de ribossomas ao longo do mRNA em estes dois sistemas segue uma cinética de tradução diferentes. Por exemplo, observamos uma banda de destaque de 17,7 kDa em E. coli lisado e não no sistema RRL, sugerindo que há um local de pausa adicional translacional na região 3'-fim de ARNm (codificação região C-terminal da gama B cristalina), quando ela é traduzida em E. coli do sistema. Notamos que Gamma-B cristalina portos conjunto Arg códons (AGG 153 e AGA 154) que são frequentes em sistemas de mamíferos, mas são conhecidos por serem extremamente raro e lentamente traduzida em E. coli. um novo peptídeo nascente acumulando no E. sistema coli (~ 17,7 kDa Figura 2.2) é provavelmente causado pelo ribossomo pausa nesses códons raros em tandem. Note-se que nenhuma banda pode ser observado na ausência de mRNA (Figura 2.3) e que o tratamento puromicina remove a maioria das cadeias nascentes de polissomas (Figura 2.3). O tratamento prolongado com puromicina (> min 15) remove todas as cadeias de nascente (dados não mostrados). Isto confirma que os produtos observados são polipeptídeos ribossomas associados cadeias nascentes, ao invés de mRNPs cosedimenting com polissomos.
Como mencionado acima, a polarização codão é organismo, bem como o tecido específico. O exemplo representativo descrito aqui, indica claramente que a mudança do ambiente do repertório de tradução ou seja, de tRNAs / códonpolarização pode levar a circulação alterada do ribossoma ao longo do mRNA. Evidentemente, esta técnica simples não só pode permitir a identificação dos sítios de pausa de translação ao longo de ARNm, mas também permite a comparação rápida dos sítios de tradução de pausa entre sistemas diferentes.
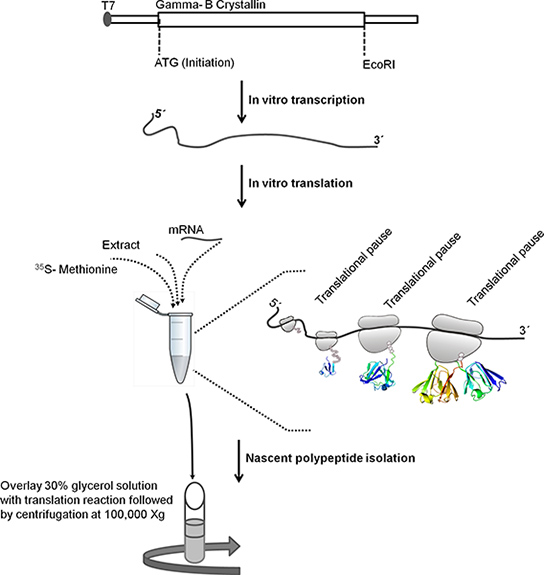
Figura 1. Esquemático explicando as etapas envolvidas no isolamento ribossomas ligados polipeptídeos nascentes. Bovina Gamma-B cristalina seqüência ORF (528 pares de bases) foi clonado a jusante do promotor T7 em pBSKS +. Para transcrição in vitro do DNA molde foi linearizado por tratamento com EcoRI. Modelo linearizado foi utilizado para transcrição in vitro. T7 promoter em molde de ADN é reconhecido por T7 RNA polimerase que transcreve a jusante Gamma B-crystallin gene. mRNA é purificado utilizando lítio método de precipitação de cloreto. O mRNA purificado é utilizado para tradução in vitro. Seguintede incubação, a reacção de tradução é em camadas no topo de solução de glicerol a 30% e centrifugado durante 1 hr a 4 ° C a 100.000 xg para isolar bound ribossomas polipéptidos nascentes.
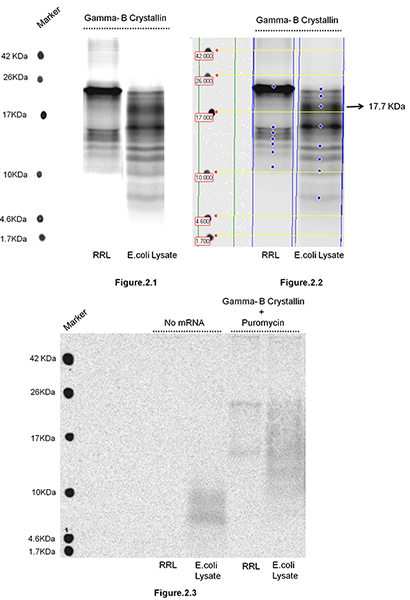
Figura 2. Isolamento de Bovinos polipeptídeos Gamma-B cristalina nascentes traduzidas em reticulócitos de coelho Lysate (RRL) e E. Lisado coli. 2 mRNA ug foi misturado em 100 ul de reacção contendo RRL ou E. coli S30 Extracto System (Promega, Madison, WI) de acordo com as instruções do fabricante, com 20 uCi [35 S]-metionina e incubadas a 30 ° C durante 5 min. Após a incubação a reacção de tradução foi derramado por cima de 4,5 ml de glicerol 30% em 10 mM de tampão Tris-HCl pH 7,6, contendo 100 mM de KCl, 10 mM de MgCI 2, e centrifugou-se durante 1 hora a 4 ° C e 100.000 X g em um TLA-110 do rotor (Beckman Coulter, Inc, Brea, CA). O sedimento foi isolado polyribosome resuspended em 20 uL de 1 mM de Tris-HCl pH 7,6, contendo 0,5 mg / ml ribonuclease A (Invitrogen / Life Technologies, Grand Island, NY), seguido por tratamento com NaOH (como descrito na secção de protocolo). Polipéptidos nascentes foram resolvidos em 16,5% T, 6% C Tris-Tricina gel SDS PAGE. O gel foi seco e exposto para autorradiografia. Após a exposição do gel foi digitalizada utilizando Scanner GE Healthcare Imaging da Typhoon. A Figura 2.1 mostra o gel com polipéptidos nascentes isoladas e resolvido depois reacções de tradução feito em dois sistemas diferentes (RRL e E. coli). Figura 2.2 O gel foi analisado por imagem Quant TL ., v2005 software e usado aqui como um exemplo para mostrar que o peso molecular ea intensidade dos polipéptidos nascentes que se acumulam em dois sistemas podem ser facilmente analisadas e comparadas Figura 2.3 mostra dois conjuntos de controlos: polipéptidos nascentes isolados e resolvidos em SDS-PAGE após a tradução As reacções foram montadassem ARNm e / ou após as reacções de tradução foram tratados com puromicina (5 min, conc final. 1 mM) antes da reacção foi submetida a centrifugação.
Discussão
Para obter resultados reprodutíveis, a qualidade ea concentração dos componentes utilizados para transcrição in vitro e reacções de tradução são críticos. No presente estudo, utilizamos kits disponíveis comercialmente e extratos que fornecem dados altamente reprodutíveis, se manuseados com cuidado. No entanto, a tradução-competentes extractos podem ser preparados a partir da célula da sua escolha, se necessário. Qualidade de ARNm pode afectar a tradução, por isso, é de extrema impor...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho foi financiado pelo Programa Ciência das Fronteiras Humanas concessão RGP0024.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome de reagente / Kit | Companhia | Número de catálogo | |
| MEGAscript T7 Kit de Transcrição de alto rendimento | Ambion | AM1333 | |
| Inibidor da ribonuclease | Invitrogen | 15518012 | |
| Trans [35 S]-Label | Biomedicals MP | 0151006 | |
| Ribonuclease-A | Invitrogen | 12091 | |
| Sistema de Lisado de reticulócitos de coelho, Nuclease Tratada | Promega | L4960 | |
| E. coli S30 Sistema de Extrato de Modelos Lineares | Promega | L1030 | |
| Centrifugação | Beckman Coulter | Optima ultracentrífuga TLX | |
| Armazenamento de fósforo autorradiografiay | GE Healthcare | Typhoon 9410 imager modo variável | |
| Software para análise polipeptídeo nascente | GE Healthcare | Imagem Quant TL, v2005 |
Referências
- Komar, A. A. A pause for thought along the co-translational folding pathway. Trends Biochem. Sci. 34, 16-24 (2009).
- Sharp, P. M., Cowe, E., Higgins, D. G., Shields, D. C. Codon usage patterns in Escherichia coli, Bacillus subtilis, Saccharomyces cerevisiae, Schizosaccharomyces pombe, Drosophila melanogaster and Homo sapiens; a review of the considerable within-species diversity. Nucleic Acids Res. 16, 8207-8210 (1988).
- Dittmar, K. A., Goodenbour, J. M., Pan, T. Tissue-specific differences in human transfer RNA expression. PLoS Genet. 2, e221 (2006).
- Ikemura, T. Codon usage and tRNA content in unicellular and multicellular organisms. Mol. Biol. Evol. 2, 13-34 (1985).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. EMBO J. 7, 3559-3569 (1998).
- Krasheninnikov, I. A., Komar, A. A., Adzhubei, I. A. Nonuniform size distribution of nascent globin peptides, evidence for pause localization sites, and a cotranslational protein-folding model. J. Protein Chem. 10, 445-454 (1991).
- Komar, A. A., Lesnik, T., Reiss, C. Synonymous codon substitutions affect ribosome traffic and protein folding during in vitro translation. FEBS Lett. 462, 387-391 (1999).
- Komar, A. A., Jaenicke, R. Kinetics of translation of γ B crystallin and its circularly permutated variant in an in vitro cell-free system: possible relations to codon distribution and protein folding. FEBS Lett. 376, 195-198 (1995).
- Jha, S., Komar, A. A. Birth, life and death of nascent polypeptide chains. Biotechnol. J. 6, 623-640 (2011).
- Thanaraj, T. A., Argos, P. Ribosome-mediated translational pause and protein domain organization. Protein Sci. 5, 1594-1612 (1996).
- Kimchi-Sarfaty, C., Oh, J. M., Kim, I. W., Sauna, Z. E. A "silent" polymorphism in the MDR1 gene changes substrate specificity. Science. 315, 525-528 (2007).
- Schägger, H., von Jagow, G. Tricine-sodium dodecyl sulfate-polyacrylamide gel electrophoresis for the separation of proteins in the range from 1 to 100 kDa. Anal. Biochem. 166, 368-379 (1987).
- Shirole, N., Balasubramanian, S., Yanofsky, C., Cruz-Vera, L. Isolation of Translating Ribosomes Containing Peptidyl-tRNAs for Functional and Structural Analyses. J. Vis. Exp. (48), e2498 (2011).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324, 218-223 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados