このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
水素交換質量分析法を用いてタンパク質のダイナミクスを分析する
要約
タンパク質の立体構造とダイナミクスは、タンパク質の構造と機能の関係を理解するための鍵となります。高分解能質量分析と組み合わせた水素の交換は、タンパク質のコンフォメーションのダイナミクスを研究だけでなく、接触インタフェースおよびアロステリック効果を含むタンパク質 - リガンドとタンパク質 - タンパク質相互作用を特徴付けるための汎用的な方法である。
要約
すべての細胞プロセスは、タンパク質の機能に依存します。所与のタンパク質の機能性は、そのユニークなアミノ酸配列の直接的な結果であるが、それは単一の定義された三次元配置またはより一般的には相互変換する立体配座のアンサンブル内にポリペプチド鎖の折り畳みによって実現される。タンパク質の立体構造とその機能との間の接続を調査すると、タンパク質は、タスクの彼らの多種多様を満たすことができる方法を完全に理解するために不可欠である。その機能的なサイクルを進行しながら、タンパク質が受けるコンホメーション変化を研究するための一つの可能性は、高分解能質量分析(HX-MS)と組み合わせた水素を1 H / 2 H交換である。 HX-MSは、 例えば結晶解析によって得られた構造情報に新たな次元を追加して汎用性の高い、堅牢な方法です。これは、小さいモルの結合は、フォールディング及びアンフォールディングタンパク質を研究するために使用されているeculeリガンド、タンパク質 - タンパク質相互作用、触媒作用、およびアロステリック酵素に連結コンホメーション変化。タンパク質の量は非常に限られているまたはタンパク質の結晶化が不可能な場合に加えて、HX-MSは、しばしば使用される。ここでは、HX-MSを用いたタンパク質のダイナミクスを研究するための一般的なプロトコルを提供し、どのように複雑では2つのタンパク質の相互作用界面を明らかにすることを例として説明します。
概要
タンパク質およびタンパク質複合体の結晶構造の数は、近年急速に増加した。彼らは、これらのタンパク質の構造組織の貴重なスナップショットを提示し、構造 - 機能分析のための基礎を提供する。しかし、タンパク質のダイナミクスとその機能のために不可欠な立体構造変化は、、めったにX線結晶構造解析によって明らかにされていません。クライオelectronmicroscopyは、一方で、異なる構造タンパク質およびタンパク質複合体を捕捉することができるが、一般的に二次構造レベル1にコンホメーション変化を下に解決できない。原子細部で溶液中のタンパク質のコンフォメーションダイナミックスは、NMRによって解決することができますが、この方法では、まだ比較的小さいサイズ(一般的には≤30 kDa)ののタンパク質に制限されて実験を妨げるタンパク質を高濃度(≥100μM)を、必要とされているオリゴマー化または凝集しやすいタンパク質2。一つの方法は、その高分解能X線結晶学およびクライオelectronmicroscopy間をブリッジすることができ、タンパク質サイズ又は濃度によって限定されないアミド水素1 H / 2質量分析(MS)と組み合わせたH-交換(HX)である。近年では、この方法は、タンパク質のダイナミクス、タンパク質のフォールディング、タンパク質の安定性およびコンフォメーション変化3-5の分析のための貴重な分析ツールを開発しました。この方法の分子的基礎は、タンパク質がD 2 O溶液中に置かれたときに重水素原子と交換するであろうタンパク質中の骨格アミド水素の不安定な性質である。経時的なタンパク質質量のその後の増加は、高分解能MSで測定する。
HXは温度のみに依存する短い非構造化ペプチド、触媒濃度-及び誘導のために隣接する残基のアミノ酸側鎖(OH、H 3 O +、すなわち pHが、 図3を参照)、ネコalyticと立体効果。固有の化学交換速度k CH ONこれらの効果は、エレガントな白ら 6によって定量化されたプログラムは、pHおよび温度に依存してポリペプチド内の各アミノ酸のK CHを算出する(礼儀Z.チャン)、提供されています。中性pH及び周囲温度でのk個のchが 10 1〜10 3秒-1のオーダーである。きつく折り畳まれたタンパク質の内部へのイオン-折り畳まれたタンパク質で、HXは、主に二次構造における水素結合とOHの水和のアクセス制限による軽微な程度に遅くなる大きさの2-9桁です。天然タンパク質中のHXはそのため部分的または全体的な展開、化学交換と式に従って天然の状態にリフォールディングを関与(1)とK OBS開口率kのOP、クロージング率kのCLと真性化学交換に依存して観測された為替レートRAテchの k個の式(2)に従って。
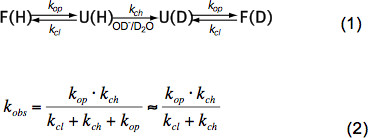
天然の状態条件下のk opは k個のchのよりもはるかに小さく、分母に無視することができる。 EX1とEX2と呼ばれる2の極端な為替制度があります。 k個のclが k chを (EX1)よりもはるかに小さい場合に観察された速度は、開口率と実質的に等しく、HXは、構造要素のアンフォールディングの直接の観察を可能にする。構造的要素の開始時に一度にすべてのアミドプロトン交換は、同位体ピーク7の二峰性の分布によって、MSに容易に観察可能であるような為替制度、。 k個のclが k chをよりはるかに大きい場合には、比例定数は折りたたみ展開均衡に等しいができる(EX2)が観察された速度は、k個のchのに比例する定数K uは k個のオペアンプ / K CL。同位体分布がほぼ同じままであるこれらの条件下では、多くの開閉イベントは、平均質量が徐々に増加をもたらす、重陽子のすべてのアミドプロトン交換前に必要である。 EX2体制はΔGUアンフォールディングの自由エネルギーと構造要素の、したがって安定性の決定を可能にする。ネイティブ状態の条件の下でEX2政権が最も一般的である。 pHとカオトロピック剤の添加の増加は、EX1に交換メカニズムをシフトすることができます。したがって、HX-MSは、熱力学ならびにタンパク質フォールディングの動力学的パラメーターおよびコンフォメーション変化を探索するために使用することができる。
HX上述したように本質的になる骨格アミド基の完全に露出したプロトン溶媒のpHおよび温度依存性の交換半減期は、生理的pH(pH7.6)中で5〜400ミリ秒、および30℃、しかし10分の間である> pHは2.9と0℃で> 2時間の平均は15時間( およその半減期との交流ポリペプチドの最初の骨格アミド結合、1〜2分間のプロトンを除く)C。そのような遅い交換条件下ではタイムアウトが組み込ま重陽子に含まれるすべての情報を失うことに、これらの条件下で活性であるプロテアーゼ( 例えばペプシン)を用いて試料を消化することができる。遅い交換の条件でペプシン消化の導入以来、完全長タンパク質の全体的なHX速度のみならず、解析することができますが、HXは特定の領域8,9に局在することができます。空間分解能は、現在、10〜30残基の間、一般的に消化され生成されたフラグメントの大きさに制限される。しかし、ペプシンによって起因する切断の非特異的な性質のために作成された重複フラグメントは、空間分解能の増加につながる可能性があります。さらに、いくつかの他のプロテアーゼしかしながら、はるかに効率的な10ペプシンより、急冷条件下で活性であることが判明した。さらにincrea空間分解能のそれは、例えば、電子捕獲解離(ECD)、電子移動解離(ETD)と、赤外多光子解離(IRMPD)11-13として重水素化パターンを保存方法によって、気相中のペプチドの断片化によって到達することができる。これらの技術は、衝突誘起解離(CID)によって観察された分子内プロトン移動(「スクランブル」)、最も一般的に使用される断片化法に空間分解能の損失を防ぐ。しかし、これらの方法は、すべての個々のペプチドについての最適化を必要とし、したがって、依然として非常に困難である。
HX-MSは、ウイルスキャプシドアセンブリ14-17を含むタンパク質-リガンドとタンパク質-タンパク質相互作用を分析するために使用されている。タンパク質がアンフォールディングおよびリフォールディングだけでなく、温度が誘発されるコンフォメーション変化は、7,18,19を調査した。リン酸化と、単一のアミノ酸変異に関連したコンホメーション変化16,20とnucleotアミド誘導変化は21,22を分析した。したがって、この方法は、アセンブリおよび分子機械の動力学を分析するために理想的に好適と思われる。そのメカニズム大将軍興味深い1つの魅力的な候補者は、Hsp90のシャペロン複合体である。
プロトコル
1。バッファーおよびタンパク質サンプルの調製
- H 2 Oのバッファを準備します。 H 2 OバッファとしてのHsp90標準緩衝液(40mMのHEPES / KOH、pH7.5で、50mMのKCl、5mMのMgCl 2、10%グリセロール)の使用。
注 :サンプルは、分析の前に透析した場合、H 2 Oのバッファとして透析緩衝液を使用しています。それは、D 2 Oのバッファのみ水素同位体中のH 2 Oバッファと異なっていることが不可欠です。 NH 4 CO 3又はNH 4 -アセテートまたはバッファーの構成要素のような揮発性のバッファが適切ではない! - 真空濃縮器を用いたHsp90標準緩衝液の凍結乾燥により、D 2 Oのバッファを準備します。 H 2 Oを完全に蒸発させた後、初期の全体積( 例えば、1〜850μlのD 2 Oの添加を必要とし、15%グリセロール緩衝液ミリリットル)に達するまでチューブに純粋なD 2 Oを加える。バッファの完全な蒸発を繰り返し、バッファ/塩COMを再溶解D 2 O 2X内ponents。
- クエンチバッファーを準備します(0.4M KH 2 PO 4 / H 3 PO 4 pHは2.2)。
注 :4 M塩酸グアニジンおよび0.5 Mトリス(2 -カルボキシエチル)ホスフィン(TCEP塩酸)を添加することができる非常に安定なタンパク質のペプシン消化物の効率を改善する。 - 100%の対照サンプル(6 M塩酸グアニジン、D 2 O)を調製する。完全に試料からH 2 Oを蒸発させ、10%グリセロールとの最初の合計体積( 例えば 、100μlのサンプルに到達するためにチューブにD 2 Oを追加6 M.の最終濃度に達するようにするHsp90アリコートに塩酸グアニジンを追加添加を必要とする90μlのD 2 O)の。バッファの完全な蒸発を繰り返しとD 2 Oのバッファ/塩成分を再溶解
注意 :サンプルの20〜100 pmolの各注射のために必要とされる。 HX-MS実験の日々のために100%のコントロールを持ってするのに十分なサンプルを準備します。 - 5μLのHsp90標準バッファに50ピコモルのHsp90を準備します。
注意 :サンプルの20〜100 pmolの量は、生データの各ポイントに必要です。反応中の試料の量は、理想的には1〜5μLです。これらの要件に合わせて濃度を調整します。任意の緩衝剤は、それが洗剤または揮発性成分を含んでいないとして使用することができる。
2。アルデヒド活性化ビーズ上に固定化ペプシンの調製
- 2ミリリットルの50mMクエン酸ナトリウム(pHは5)に80mgの新鮮なペプシンを溶解する。
- 、シアノ水素化ホウ素ナトリウム20mgを溶解する1ミリリットルの2MのNa 2 SO 4でのケア( 非常に毒性が強い !)で処理し、ペプシン溶液に加える。
- 穏やかに( 例えば、オーバーヘッドシェーカー)を攪拌しながら室温で10分間混合物をインキュベートする。
- 混合物に固定化されたアルデヒド基を有する600mgのビーズを添加し、室温で5〜10分間インキュベートする。
- SO 2のNa 2の2.2ミリリットルを追加します。4100μlのアリコート中(pHが5)3分毎に1時間以上、ゆっくりペプシンを塩に。そっと室温でオーバーヘッドシェーカーに追加間のサンプルをミックス。
- オーバーヘッドシェーカー中で一晩14〜16時/ 4℃でペプシンビーズをインキュベートする。
- 2時間室温で1ミリリットルの1Mエタノールアミンおよびインキュベーションを加えることにより反応を停止さ。
- 、500 rpmで50ミリリットルファルコンチューブ内のビーズをスピンダウンし、上清を廃棄し、0.1%ギ酸中でビーズを懸濁します。このステップの2倍を繰り返します。最後の遠心分離工程の後、上清およびビーズの推定体積を捨てる。 0.1%ギ酸の当量を添加し、4℃で保存し
3。アミド水素交換用カラムの作製
- 列をトラップすると、ペプシン列の2ミリメートルと1ミリメートルの内径のガードカラムを使用してください。
- ガードカラムの片側を外し、フィルターを取り外します。しっかり表示Funn梱包ネジ列の開放端の上にEL。塔の底部出口に空のシリンジ(5ミリリットル)を添付し、1/16インチのアダプターとチューブを使用してください。ガードカラムに気密それを修正してください。
- 漏斗の上にスラリービーズ材料を数滴を適用する。ガードカラムに漏斗でスラリーを吸引するシリンジのプランジャを引く。漏斗の上に、よりスラリービーズ材料を適用し、ガードカラムが完全にビーズ材料で満たされるまでの手順を続けます。漏斗を取り外し、開放端にフィルター、フィルターリングを配置。しっかりとガードカラムにカラム·スクリューキャップと反対側からシリンジを取り外します。カラム材料から乾燥を避けるためにプラグでガードカラムの両端を閉じる。
4。水素交換質量分析(HX-MS)のためのシステムの設定
- HPLCシステムでトラップカラム( 図1)を接続します。の流量を設定することにより、カラムを平衡化溶媒として0.1%ギ酸を0.4ミリリットル/分でポンプ。まだペプシン列や分析カラムを接続しないでください。
- 質量分析計を較正し、質量分析計のソースにHPLCの出口を接続する。
5。為替のダイナミックレンジの決定
- 超純溶媒A(水中0.1%ギ酸)、超純溶媒B(アセトニトリル中0.1%ギ酸)を調製し、レディーミクスト溶媒市販されている。 HPLCポンプをパージします。短い脱塩工程の後に段階勾配を使用してプログラムを選択することで、クロマトグラフィーおよび制御ソフトウェアにおける質量分析法を設定します。フルレングスのHsp90 5%に溶出するために脱塩/ LOADおよび90%の溶媒A/10%溶媒Bから段階的勾配から溶媒A/95%の溶媒を6ポートバルブを切り替えることで、その後1〜2分の脱塩工程を使用B.は、注入前/積載位置を脱塩するためにロードするために噴射弁および6ポートバルブを設定します。
ノートこの実験中にペプシンや分析カラムを使用しないでください。 - 1月10日μlのH 2 OバッファにHsp90の100〜200 pmolのを準備し、30℃で10分間インキュベート100μLまでのサンプル量を持参し、一定の期間( 例えば 10秒、100秒、千秒)のために、正確にインキュベートし、温度調整のD 2 Oのバッファを追加します。 100μlのクエンチ緩衝液を添加し、上下にピペッティングにより混和する。ハミルトンシリンジを有する噴射弁の噴射口に200μlのサンプルを注入する。ー·プログラムを開始して、インジェクトの位置に注入バルブを切り替える。 2分後に溶出するために脱塩/ LOADINGから6ポートバルブを切り替える。少なくとも3の時点でこれを繰り返します。
- 手順を繰り返しますが、5.2は以前の30℃で10分間インキュベートしたHsp90サンプルにSTI1の2〜3倍の過剰を追加
- 質量分析ソフトウェアでスペクトルのデコンボリューションにより全長タンパク質の質量を決定。 incorporatの数を計算し各実行( 例えば 、10秒、100秒、千秒)後に観察された質量との完全長Hsp90の分子量を比較することによって、重陽子を編
- 非存在と時間(x軸)対STI1(y軸)の存在下でのHsp90のため援用重陽子をプロットする。両方の曲線間の差が最大であるダイナミックレンジの時点を決定します。ペプチドレベルでのHsp90-STI1インターフェイスとダイナミクスを識別するとき、D 2 O中のインキュベーション時間のためにこの値を使用します。
6。 MS / MSスペクトルを用いて消化ペプチドの定量
- システムにペプシンカラムと分析カラムを接続します。
- グラデーションの種類及び質量分析法を選択することによって、制御ソフトウェアにおけるクロマトグラフィーおよび質量分析のためのパラメータを設定する。良いクロマトグラフの解像度を確保するために長い勾配( 例えば 90以上の分)を選択します。質量分析計でのMS / MSスペクトルを有効にします。
注:良い分解能HPLCおよび高い質量精度へのTiONは、このステップで最も高い優先度を持っている。 - 100μlのH 2 OバッファにHsp90の100〜200 pmolのを準備します。 100μlのクエンチ緩衝液を添加し、上下にピペッティングにより混和する。ハミルトンシリンジを有する噴射弁の噴射口に200μlのサンプルを注入する。ー·プログラムを開始して、インジェクトの位置に注入バルブを切り替える。 2分の位置に溶出するために脱塩/ LOADINGから6ポートバルブを切り替えた後。
注 :複数のタンパク質が、HX-MS( すなわち、タンパク質-タンパク質相互作用インターフェース)で分析する場合には各タンパク質のペプチドは、個別に決定されなければならない。 - 得られたペプチドのデータベース( すなわちマスコット)を検索することによって、Hsp90の消化性ペプチドを同定する。
注 :目的は、できるだけ多くの消化性ペプチドを決定することであると分析は、精製タンパク質を用いて行われているようにこれは、カスタム·データベースを使用することが可能である。サンプル純度意志H考慮すべきaveは。 - MS / MSせずに、実際のHX実験に使用される勾配で、この手順を繰り返します。 Hsp90はとSTI1は45%で、溶媒A/55%溶媒Bに90%の溶媒A/10%溶媒Bから10分間の勾配を使用してください
注意 :グラデーションは通常の間5〜15分間であり、高度にサンプルの複雑さおよびHXシステムの仕様に依存します。 - 6.5で使用するグラデーションで6.4で識別された消化性ペプチドの保持時間を決定し、1を含むリストを作成してください。)ペプチド配列2)のペプチド電荷状態と3。)保持時間。これはHX実験後に各ペプチドを識別するために使用されます。
注意 :小のm / zの違い、同一の電荷状態と同一の保持時間を有するペプチドは、曖昧さの原因となる可能性があることに注意してください。
7。タンパク質 - タンパク質相互作用界面の同定
- 制御softwarを勾配と質量分析法を設定E。のほとんどが、300〜1,500のm / zの間の質量の検出のために最適化されている質量分析法を読み込み溶媒A/55%溶媒Bでの45%と90%溶媒A/10%溶媒Bの10分間の直線勾配を使用しペプチドは、千メートル/ zの下になります。負荷位置、ロード/脱塩位置に6ポートバルブへの注入バルブを設定します。 0.4ミリリットル/分に装填ポンプの流量を設定する。
注意 :長さやグラデーションの種類は、サンプルに依存しており、最適化されなければならないことがある。長い勾配はクロマトグラフィの分解能を向上させることが、バック交換をする予定蛋白質に重陽子の取り込みを減少させる。選択された方法は、比較される全ての実験で同じである必要がある。 - 未交換の参考のために100μlのH 2 OバッファにHsp90の20〜100 pmolのを準備します。二回ピペット上下に、100μLの氷冷クエンチバッファーを添加し、HPLCの注入バルブに試料を注入する。すぐにクロマトグラフィー·プログラムとSWを開始INJECT位置に噴射弁かゆみ。 2分の位置に溶出するために脱塩/ LOADINGから6ポートバルブを切り替えた後。個別タンパク質の混合物のためのHsp90とSTI1のためにこれを行う。
- 1-5μLの容量でHsp90の20〜100 pmolのを準備します。 100μlにサンプル量を表示し、一定の期間のために、正確にインキュベート2 Oバッファに温度調整され、Dを追加します( 例:30秒; ノートを参照してコンフォメーションダイナミックスの場合)。氷のように冷たいクエンチ緩衝液100μl、ピペットアップを追加し、上下二回、迅速に、HPLCの注入バルブに200μLを注入。すぐにクロマトグラフィー·プログラムを開始し、噴射位置に注入バルブを切り替える。 2分の位置に溶出するために脱塩/ LOADINGから6ポートバルブを切り替えた後。相互作用するタンパク質の非存在下での各ペプチドへの重陽子の取り込みを決定するために、個々のタンパク質のためにこれを行います。
注意 :準拠した形のダイナミクスを研究するときmational変化、D 2 OバッファにHsp90の異なるインキュベーション時間で実験を繰り返す。 ( 例えば 、10秒、30秒、100秒、300秒、千秒など ) を対数的にインキュベーション時間を選択することで、広いタイムスケールをカバーしよう。 D 2 Oバッファとサンプル·バッファは、水素同位体を除いて、正確に同じでなければなりません。 - 100%のコントロールサンプル(20〜100ピコモル)を同量を用意し、100μlまでのサンプル量をもたらすためにD 2 Oバッファを追加します。氷のように冷たいクエンチ緩衝液100μl、ピペットアップを追加し、上下二回、迅速に、HPLCの注入バルブに200μLを注入。すぐにクロマトグラフィー·プログラムを開始し、噴射位置に注入バルブを切り替える。 2分の位置に溶出するために脱塩/ LOADINGから6ポートバルブを切り替えた後。
- 相互作用面を決定するため( 注 )束縛状態に平衡を移動し、インキュベートするSTI1の少なくとも2倍過剰のHsp90を混ぜる所望の温度での複合体形成は、平衡状態になるまで。温度調整Dの100μlにサンプル量を表示し、一定の期間のために、正確にインキュベート2 Oバッファ( 例えば 30秒)を追加します。氷のように冷たいクエンチ緩衝液100μl、ピペットアップを追加し、上下二回、迅速に、HPLCの注入バルブに注入。 2分の位置に溶出するために脱塩/ LOADINGから6ポートバルブを切り替えた後。 20〜100 STI1のピコモルとHsp90の過剰のこの実験を繰り返します。
注意 :絶対濃度は、相互作用の解離平衡定数に依存します。理想的には、D 2 Oへの希釈後のタンパク質の濃度が(下限濃縮タンパク質の> 85%に相当する)は、少なくとも10倍のK Dであるべきで過剰に添加した。 - 適切なソフトウェアで取得したデータを分析し、分析の各ペプチドを発見するために、ステップ6.5で決定された保持時間を使用しています。 CalculaTE未交換タンパク質(ステップ7.2)用およびHX実験(ステップ7.3)のための同位体分布の重心。
注意 :交換されたペプチドの同位体パターンは、ペプチドの電荷状態についての知識と重心の容易な決定を可能にする質量分析装置の高い質量精度の両方、彼らの未交換の対応が異なって見える場合があるが。 - 単独および結合パートナー過剰の標的タンパク質の重陽子の取り込みを比較してください。これはスプレッドシートプログラムで自動的に商用ソフトウェアにあるか、手動で行うことができます。 100%の対照サンプルの値は、各ペプチドの最大の交換を表し、O標識による逆交換し、実験中に失わD 2の量を決定するために使用することができる。
結果
Hsp90は、酵母およびHSP90シャペロンファミリーのメンバーの分子シャペロンである。複雑なATPアーゼサイクルを経由することにより、多くのタンパク質のクライアントの後期折りたたみステップを支援します。効率的な折りたたみのHsp70のクライアントからの移転と共同シャペロンSti1/Hopの相互作用を必要とする。 STI1は直接のHsp90に結合したHsp90のATPase活性の阻害によって結合クライアントを?...
ディスカッション
タンパク質に相互作用パートナーの結合は、必然的に結合部位に溶媒接近の変化を引き起こす。さらに、多くのタンパク質は、実際の結合界面以外の領域に影響を結合すると、動的な立体構造変化を受ける。 HX-MSは、これらの変更を監視するための堅牢な方法であり、他の方法もカバーすることはできませんタイムスケール上のタンパク質の立体構造変化を明らかにすることさえ可能です。 ...
開示事項
我々は、開示することは何もありません。
謝辞
私たちは、原稿にコメントをM·ボイセンに感謝します。このプロジェクトは、(MPMに1278/4-1 SFB638とMA、そして卓越したクラスター:CellNetworks EXC 1分の81)ドイツ学術振興によって資金を供給された。 CellNetworks:MPMは卓越性のクラスタの研究者でもある。
資料
| Name | Company | Catalog Number | Comments |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
参考文献
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5'-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O'Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved