É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Analisando Dynamics proteína utilizando hidrogênio câmbio Espectrometria de Massa
Neste Artigo
Resumo
Conformação da proteína e dinâmicas são a chave para a compreensão da relação entre estrutura e função de proteínas. Troca de hidrogênio acoplada a espectrometria de massa de alta resolução é um método versátil para estudar a dinâmica de conformação das proteínas, bem como a caracterização da proteína-ligante e interações proteína-proteína, incluindo interfaces de contato e efeitos alostéricos.
Resumo
Todos os processos celulares dependem da funcionalidade das proteínas. Embora a funcionalidade de uma dada proteína é a consequência directa da sua sequência de aminoácidos única, apenas é realizado pela dobragem da cadeia polipeptídica em um único arranjo tridimensional definido ou mais vulgarmente em um conjunto de conformações interconversão. Investigando a ligação entre a conformação da proteína e sua função é, portanto, essencial para um entendimento completo de como as proteínas são capazes de cumprir a sua grande variedade de tarefas. Uma possibilidade para estudar mudanças de conformação de uma proteína sofre, progredindo através do seu ciclo funcional é hidrogénio-1 H / 2 H-troca em combinação com espectrometria de massa de alta resolução (HX-MS). HX-MS é um método versátil e robusto que acrescenta uma nova dimensão às informações estruturais obtidas por cristalografia por exemplo. Ele é usado para estudar a proteína dobrando e desdobrando, a ligação da pequena molligantes ecule, interações proteína-proteína, mudanças conformacionais ligadas à catálise enzimática e allostery. Além disso, HX-MS é muitas vezes utilizado quando a quantidade de proteína é muito limitada, ou a cristalização da proteína não é viável. Aqui proporciona-se um protocolo geral para o estudo da dinâmica das proteínas com HX-MS e descrevem como um exemplo de como a interface para revelar a interacção de duas proteínas de um complexo.
Introdução
O número de estruturas de cristal de proteínas e complexos de proteína aumentou rapidamente nos últimos anos. Apresentam instantâneos inestimáveis da organização estrutural das proteínas e fornecer uma base para a análise da estrutura-função. No entanto, a dinâmica das proteínas e as mudanças conformacionais, que são essenciais para as suas funções, são raramente revelado por cristalografia de raios-X. Cryo-electronmicroscopy, por outro lado, é capaz de capturar os complexos de proteínas e de proteínas em diferentes conformações, mas geralmente não é possível resolver as alterações conformacionais para baixo para o nível de uma estrutura secundária. Dinâmica de conformação de proteínas em solução em detalhes atómicas só pode ser resolvido por RMN, mas este método é ainda limitado a proteínas de tamanhos relativamente pequenos (geralmente ≤ 30 kDa) e necessita de altas concentrações de proteínas (≥ 100 pM), o que dificulta as experiências com oligomerização ou agregação de proteínas propensas 2. Um método queé capaz de ponte entre a alta resolução de cristalografia de raios-X e crio-electronmicroscopy e que não é limitado pelo tamanho ou a concentração de proteína é de hidrogénio amida-1 H / 2 H-troca (HX) em combinação com espectrometria de massa (MS). Nos últimos anos, este método tem desenvolvido para um instrumento analítico valioso para a análise da dinâmica das proteínas, o dobramento de proteínas, a estabilidade da proteína e alterações conformacionais 3-5. A base molecular deste método é a natureza lábil do backbone hidrogénios de amida em proteínas, que irá trocar com átomos de deutério, quando a proteína é colocada em um D 2 O solução. O consequente aumento da massa de proteína ao longo do tempo é medido com alta resolução MS.
Em péptidos não estruturados curtas só HX depende da temperatura, da concentração do catalisador (OH -, H 3 O + por exemplo pH, ver Figura 3) e as cadeias laterais de resíduos de aminoácidos adjacentes, devido à indutivo, gatoefeitos alytic e estéricos. Estes efeitos sobre o ch intrínseca taxa de permuta química k foram elegantemente quantificada por Bai et al. 6 e um programa está disponível (por cortesia Z. Zhang), que calcula k ch para cada aminoácido num polipéptido dependente do pH e da temperatura. Em pH neutro e temperatura ambiente k ch é da ordem de 10 -10 1 3 seg -1. Em proteínas dobradas HX pode ser 2-9 ordens de grandeza mais lento, principalmente devido a pontes de hidrogénio na estrutura secundária e a um grau menor devido ao acesso limitado de hidratadas OH - iões para o interior de uma proteína fortemente dobrado. HX em proteínas nativas, portanto, implica desdobramento, por permuta química parcial ou global e redobrando para o estado nativo de acordo com a equação (1) e as taxas de câmbio observadas k obs dependem da taxa de abertura op k, os cl k taxa de fechamento ea troca química intrínseca rate k ch de acordo com a equação (2).
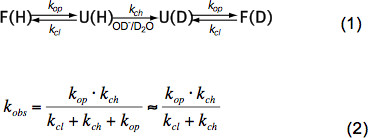
Sob condições de estado nativo k op é muito menor do k ch e pode ser negligenciada no denominador. Existem dois regimes cambiais extremos chamados EX1 e EX2. Se o cl k é muito menor do k ch (EX1) a taxa observada é praticamente igual à taxa de abertura e HX permite a observação imediata do desdobramento de um elemento estrutural. Tal regime cambial, onde todos os prótons amida troca de uma só vez aquando da abertura do elemento estrutural, é facilmente observáveis em MS por uma distribuição bimodal dos picos de isótopos 7. Se k cl é muito maior do que k ch (EX2) a taxa observada é proporcional à k ch em que a constante de proporcionalidade é igual aos equilíbrios-desdobramento dobráveis constante op K u = k K / cl. Sob estas condições, muitos eventos de abertura e fecho são necessárias antes de todos os protões amida troca de deutério, que conduz a um aumento gradual da massa média, enquanto a distribuição isotópica permanece praticamente a mesma. O regime EX2 permite a determinação da energia livre de desdobramento ÄG u e, por conseguinte, a estabilidade de um elemento estrutural. Sob condição de estado nativo do regime EX2 é mais comum. Aumento do pH e da adição de agentes caotrópicos pode mudar o mecanismo de troca de EX1. Portanto, HX-MS pode ser utilizada para explorar termodinâmico, bem como os parâmetros cinéticos de dobragem de proteína e alterações conformacionais.
Como mencionado acima HX é intrinsecamente pH e dependente da temperatura e meia-vida a troca de um protão exposta completamente solvente do grupo amida espinha dorsal é entre 5-400 ms a um pH fisiológico (pH 7,6) e 30 ° C, mas 10 min a> 15 horas, com uma média de> 2 horas a pH 2,9 e 0 °C (excepto para o protão da primeira estrutura de ligação amida de um polipeptídeo, que troca com uma meia-vida de aprox. 1-2 min). Sob tais condições lenta troca é possível para digerir a amostra usando proteases (por exemplo pepsina) que atuam nessas condições, sem perder todas as informações contidas nos dêuterons incorporadas. Desde a introdução de digestão péptica sob condições troca lenta, não apenas da cinética geral HX de proteínas de comprimento completo pode ser analisado, mas HX pode ser localizado em regiões específicas 8,9. A resolução espacial está limitado ao tamanho dos fragmentos gerados pépticas, que é, em geral, entre 10-30 resíduos. No entanto, fragmentos sobrepostos criados devido à natureza não específica da clivagem por pepsina pode levar a um aumento da resolução espacial. Além disso, várias outras proteases foram encontrados para ser activo em condições de têmpera, contudo, muito menos eficiente do que a pepsina 10. Além disso aumense de resolução espacial pode ser alcançado pela fragmentação de peptídeos em fase gasosa por meio de métodos que preservaram o padrão deuteração tais como a captura eletrônica de dissociação (ECD), transferência eletrônica de dissociação (ETD) e dissociação multiphoton infravermelho (IRMPD) 11-13. Estas técnicas de evitar a perda de resolução espacial devido à migração intramolecular de protões ("scrambling"), que é observada por dissociação induzida por colisão (CID) a técnica de fragmentação mais utilizada. No entanto, estes métodos requerem otimização para cada peptídeo individual e é, portanto, ainda bastante desafiador.
HX-MS foi utilizada para analisar interacções proteína-ligando e de proteína-proteína, incluindo a montagem da cápside viral 14-17. Proteína desdobramento e redobrando assim como temperatura provocadas mudanças conformacionais foram investigados 7,18,19. Fosforilação e conformacional relacionada com mutação único aminoácido muda 16,20 e nucleotmudanças ide-induzidas foram analisados 21,22. Portanto, esse método parece idealmente adequado para analisar a montagem e dinâmica de máquinas moleculares. Um candidato atraente, cujo mecanismo é de grande interesse geral, é o complexo chaperone Hsp90.
Protocolo
1. Preparação de Amortecedores e Proteína Amostras
- Prepare H2O tampão. Utilização Hsp90 tampão padrão (40 mM HEPES / KOH, pH 7,5, KCl 50 mM, MgCl2 5 mM, glicerol a 10%) na forma de H 2 O tampão.
Nota: Se a amostra foi dialisada antes da análise, usar o tampão de diálise como H2O tampão. É essencial que o O tampão D 2 difere do ó tampão H 2 apenas no isótopo de hidrogénio. Buffers voláteis como NH 4 componentes 4-acetato ou tampão CO 3 ou NH não são adequados! - Prepare D 2 O tampão por liofilização de solução tampão padrão de Hsp90 usando um concentrador de vácuo. Após completa evaporação da H2O adicionar puro D 2 O para o tubo para atingir o volume total inicial (por exemplo, 1 ml de tampão com 15% de glicerol requerem a adição de 850 ul de D 2 O). Repita a evaporação completa do buffer e dissolver o com buffer / salnentes em D 2 O 2x.
- Preparar tampão de têmpera (0,4 M de KH 2 PO 4 / H 3 PO 4 de pH 2,2).
Nota: Para melhorar a eficiência da digestão péptica de proteínas muito estáveis 4 M de cloridrato de guanidina e 0,5 M de Tris (2-carboxietil) fosfina (TCEP-HCl) podem ser adicionados. - Prepare a 100% da amostra de controlo (6 M de cloridrato de guanidina, D 2 O). Adicionar cloridrato de guanidina a uma alíquota de Hsp90 para atingir uma concentração final de 6 M. evaporar completamente H2O a partir da amostra e adicionar D 2 O para o tubo, para atingir o volume total inicial (por exemplo, amostra de 100 ul com 10% de glicerol requerem a adição de 90 ul de D 2 O). Repetir a evaporação completa do tampão e redissolver os componentes de tampão / sal em D 2 O.
Nota: 20-100 pmol de amostra são necessários para cada injeção. Prepare amostra suficiente para ter um controle de 100% para todos os dias de experimentos HX-MS. - Preparar 50 pmol Hsp90 em 5 ul de tampão de Hsp90 padrão.
Nota: Uma quantidade de 20-100 pmol de amostra é necessário para cada ponto de dados brutos. O volume da amostra na reacção é de 1-5 ul idealmente. Ajustar a concentração para atender a esses requisitos. Qualquer tampão pode ser utilizado, desde que ele não contém detergentes ou componentes voláteis.
2. Preparação de Imobilizado pepsina em Aldeído Beads ativado
- Dissolve-se 80 mg de pepsina fresco em citrato de sódio 50 mM de 2 ml (pH 5).
- Dissolver 20 mg de cianoborohidreto de sódio, manusear com cuidado (muito tóxico!) Em 1 ml de 2 M Na 2 SO 4 e adicionar a solução de pepsina.
- Incubar mistura durante 10 minutos à temperatura ambiente enquanto se agita suavemente (por exemplo, agitador de cima).
- Adicionar 600 mg de grânulos com grupos aldeído imobilizadas à mistura e incubar durante 5-10 min à temperatura ambiente.
- Adicionar 2,2 ml de 2 M Na 2 SO4 (pH 5) em 100 mL alíquotas a cada 3 min mais de uma hora de sal lentamente a pepsina. Misture suavemente a amostra entre as adições em um agitador vertical à temperatura ambiente.
- Incubar os grânulos de pepsina a 4 ° C durante 14-16 horas / dia para o outro num agitador vertical.
- Extingue-se a reacção por adição de 1 ml de etanolamina 1 M e incubação à temperatura ambiente durante 2 horas.
- Girar as pérolas num tubo falcon de 50 ml a 500 rpm, desprezar o sobrenadante e ressuspender as contas em ácido fórmico a 0,1%. Repita este passo 2x. Após o último passo de centrifugação, descartar o sobrenadante e estimativa do volume de esferas. Adicionar um volume equivalente de ácido fórmico 0,1% e armazenar a 4 ° C.
3. Preparação de Colunas para Amida de hidrogênio-troca
- Use colunas de guarda com um diâmetro interno de 1 mm para prendendo colunas e com 2 mm para colunas pepsina.
- Desapertar um lado da coluna de guarda e remover o filtro. Fortemente parafuso funn embalagemel para a extremidade aberta da coluna. Usar um adaptador de 1/16 de polegada e tubo para conectar uma seringa vazia (5 ml) para a saída de fundo da coluna. Certifique-se de corrigi-lo à prova de gás para a coluna de guarda.
- Aplique algumas gotas de material de cordão de lama no topo do funil. Puxar o êmbolo da seringa para sugar a lama através de um funil para a coluna de guarda. Aplicar mais material talão lama para o funil e continuar o processo até que a pré-coluna é completamente preenchida com material de cordão. Remover o funil e colocar filtro e filtro de anel na extremidade aberta. Aperte o parafuso da tampa de coluna na coluna de guarda e remover a seringa do outro lado. Feche ambas as extremidades das colunas de guarda com fichas para evitar a secagem do material da coluna.
4. Configuração do Sistema de Hidrogênio câmbio Espectrometria de Massa (HX-MS)
- Ligue a coluna armadilha com o sistema de HPLC (Figura 1). Equilibrar a coluna ajustando o caudal deUma bomba de 0,4 ml / min com 0,1% de ácido fórmico como solvente. Não ligue a coluna pepsina nem a coluna de análise ainda.
- Calibra-se o espectrómetro de massa e ligar a saída da HPLC para a fonte do espectrómetro de massa.
5. Determinação da Faixa Dinâmica de câmbio
- Prepare ultrapura solvente A (0,1% de ácido fórmico em água) e ultrapura solvente B (0,1% de ácido fórmico em acetonitrilo); solventes misturados prontos estão disponíveis comercialmente. Purificai as bombas de HPLC. Configure a cromatografia e métodos de espectrometria de massa no software de controle, escolhendo um programa com um gradiente passo depois de uma etapa de dessalinização curto. Para full-length Hsp90 utilizar um passo de dessalinização de 1-2 min seguido por comutação da válvula de 6 portas de dessalinizar / LOAD para eluir e um gradiente em degrau de 90% de solvente A/10% de solvente B a 5% de solvente A/95% de solvente B. Antes da injecção definir a válvula de injeção de carga e válvula de 6 portas para dessalinizar / posição de carregamento.
Nota: Não use a pepsina ou coluna analítica durante esta experiência. - Prepare 100-200 pmol de Hsp90 em 1-10 ul H2O tampão e incubar durante 10 min a 30 ° C. Adicionar temperatura ajustada D 2 O tampão para perfazer o volume da amostra até aos 100 ml e incubar exactamente, por um período definido de tempo (por exemplo 10 segundos, 100 segundos, 1000 seg). Adicionar 100 mL de tampão de têmpera e misture por pipetagem cima e para baixo. Injectar a amostra de 200 ul para o orifício de injecção da válvula de injecção com uma seringa Hamilton. Inicie o programa de cromatografia e trocar a válvula de injecção na posição INJECT. Após 2 min trocar a válvula de 6 portas de dessalinizar / CARREGAR para eluir. Repita isso por pelo menos três pontos de tempo.
- Repetir o passo 5.2, mas adicionar excesso de 2 a 3 vezes de STI1 para a Hsp90 amostra antes da incubação durante 10 min a 30 ° C.
- Determinar as massas de proteínas de comprimento completo por desconvolução de espectros no software de espectrometria de massa. Calcule o número de incorporandoed dêuterons comparando o peso molecular de Hsp90 de comprimento completo com a massa observada após cada ensaio (por exemplo, 10 segundos, 100 segundos, 1000 seg).
- Traçar os deuterões incorporadas para Hsp90 na ausência e presença de STI1 (eixo y) versus tempo (eixo x). Determinar o ponto de tempo no intervalo dinâmico, onde a diferença entre as duas curvas é máxima. Utilize este valor para o tempo de incubação em D 2 O na identificação de interface e dinâmica sobre os níveis de peptídeo Hsp90-STI1.
6. Determinação da péptica Peptídeos Usando MS / MS Spectra
- Ligue a coluna de pepsina e uma coluna analítica para o sistema.
- Configure os parâmetros para a cromatografia e espectrometria de massa no software de controle, escolhendo tipo de gradiente e método de espectrometria de massa. Escolha uma rampa longa (por exemplo, mais de 90 min) para garantir uma boa resolução cromatográfica. Ativar espectros MS / MS no espectrômetro de massa.
Nota: Boa resolução no HPLC e precisão de massa de alta tem maior prioridade nesta etapa. - Prepare 100-200 pmol de Hsp90 em 100 ul H2O tampão. Adicionar 100 mL de tampão de têmpera e misture por pipetagem cima e para baixo. Injectar a amostra de 200 ul para o orifício de injecção da válvula de injecção com uma seringa Hamilton. Inicie o programa de cromatografia e trocar a válvula de injecção na posição INJECT. Após 2 min trocar a válvula de 6 portas de dessalinizar / CARREGAR para eluir posição.
Nota: Se existir mais do que uma proteína deve ser analisada em HX-MS (isto é, as interfaces de interacção proteína-proteína) para cada um dos péptidos de proteínas tem de ser determinadas individualmente. - Identificar os péptidos pépticas de Hsp90, pesquisando uma base de dados (isto é, Mascot) para os péptidos resultantes.
Nota: É possível utilizar uma base de dados personalizada como o objectivo é determinar como muitos péptidos pépticas quanto possível e análise é feito com a proteína purificada. Amostra de pureza vontade have a ser levado em conta. - Repetir esta etapa sem MS / MS e com o gradiente que vai ser utilizado para a experiência real HX. Para a Hsp90 e STI1 utilizar um gradiente de 10 min desde 90% de solvente A/10% de solvente B a 45% de solvente A/55% de solvente B.
Nota: Os gradientes são normalmente entre 5-15 min e altamente dependem da complexidade da amostra e as especificações do sistema HX. - Tempo de retenção determinar os tempos de retenção dos péptidos identificados pépticas em 6.4, o gradiente utilizado em 6.5 e criar uma lista que compreende 1). Sequência do péptido 2.) Péptido estado de carga e 3.). Isto irá ser utilizado para identificar cada péptido após as experiências HX.
Nota: Esteja ciente de que os peptídeos com diferenças pequenas m / z, estado de carga idêntica e tempo de retenção idêntico pode ser uma fonte de ambigüidade.
7. Identificação de Interfaces de interação proteína-proteína
- Configure o gradiente eo método de espectrometria de massa no softwar controlee. Use um gradiente linear de 10 minutos a partir de 90% de solvente A/10% de solvente B a 45% de solvente A/55% solvente B. Coloque um método de espectrometria de massa que é otimizado para detecção de massas entre 300-1,500 m / z, embora a maior parte os péptidos serão superiores a 1000 m / z. Ajuste a válvula de injeção na posição de carga, a válvula de 6 portas em carga / dessalinização Position. Ajuste a velocidade de fluxo da bomba de carga de 0,4 ml / min.
Nota: A duração eo tipo de gradiente são dependentes da amostra e pode ter que ser otimizado. Gradientes mais longos melhorar a resolução da cromatografia, mas diminuir a incorporação de deutério em proteínas, devido ao back-troca. Os métodos escolhidos precisa ser a mesma para todas as experiências deve ser comparado. - Para a referência unexchanged preparar 20-100 pmol de Hsp90 em 100 ul H2O tampão. Adicionar 100 ul de tampão gelada têmpera, pipeta cima e para baixo duas vezes e injetar amostra na válvula de injeção do HPLC. Iniciar imediatamente o programa de cromatografia e swcoçar a válvula de injeção na posição INJECT. Após 2 min trocar a válvula de 6 portas de dessalinizar / CARREGAR para eluir posição. Faça isso por Hsp90 e STI1 individualmente e para a mistura de proteínas.
- Prepare 20-100 pmol de Hsp90 em um volume de 1-5 ul. Adicionar temperatura ajustada D 2 O tampão para trazer o volume de amostra até aos 100 ml e incubar exatamente por um período definido de tempo (por exemplo, 30 segundos, pois dinâmica conformacional ver nota). Adicionar 100 ul de tampão de têmpera gelada, pipeta-se para baixo e duas vezes rapidamente e injectar 200 ul para a válvula de injecção da HPLC. Iniciar imediatamente o programa de cromatografia e trocar a válvula de injecção na posição INJECT. Após 2 min trocar a válvula de 6 portas de dessalinizar / CARREGAR para eluir posição. Fazer isto para cada uma das proteínas individualmente, para determinar a incorporação de deutério em cada péptido na ausência da proteína que interage.
Nota: Ao estudar a dinâmica de conformational alterações, repita o experimento com diferentes tempos de incubação de Hsp90 em D 2 O buffer. Tentar cobrir uma ampla escala de tempo, escolhendo os tempos de incubação logaritmicamente (por exemplo, 10 segundos, 30 segundos, 100 segundos, 300 segundos, 1000 seg, etc.) D 2 O tampão tampão e amostra deve ser exatamente idênticos, exceto para o isótopo de hidrogênio. - Prepara-se uma quantidade igual de 100% da amostra de controlo (20-100 pmol) e adicionar D 2 O tampão para perfazer o volume da amostra a 100 ul. Adicionar 100 ul de tampão de têmpera gelada, pipeta-se para baixo e duas vezes rapidamente e injectar 200 ul para a válvula de injecção da HPLC. Iniciar imediatamente o programa de cromatografia e trocar a válvula de injecção na posição INJECT. Após 2 min trocar a válvula de 6 portas de dessalinizar / CARREGAR para eluir posição.
- Para determinar a superfície de interacção misturar Hsp90 com um excesso de, pelo menos, 2 vezes superior de STI1 para desviar o equilíbrio para o estado ligado (ver nota) e incubarà temperatura desejada até que a formação do complexo está em equilíbrio. Adicionar temperatura ajustada D 2 O tampão para perfazer o volume da amostra até aos 100 ml e incubar exactamente, por um período definido de tempo (por exemplo 30 segundos). Adicionar 100 ul de tampão de têmpera gelada, pipeta-se para baixo e duas vezes rapidamente e injectar na válvula de injecção da HPLC. Após 2 min trocar a válvula de 6 portas de dessalinizar / CARREGAR para eluir posição. Repita esta experiência com 20-100 pmol de STI1 e excesso de Hsp90.
Nota: As concentrações absolutas dependem da constante de equilíbrio de dissociação da interacção. Idealmente, após diluição em D 2 O a concentração da proteína em excesso adicionado deverá ser pelo menos 10 vezes a K D (correspondente a> 85% da proteína ligada inferior concentrado). - Analisar os dados adquiridos, com um software adequado e usar o tempo de retenção determinadas no passo 6.5 para encontrar cada péptido na análise. Calculate o centróide de distribuição de isótopos para a proteína unexchanged (passo 7.2) e para as experiências de HX (passo 7.3).
Nota: Embora os padrões de isótopos de peptídeos trocados parecer diferente dos seus homólogos unexchanged, tanto o conhecimento sobre o estado de carga do peptídeo ea exatidão elevada massa do espectrômetro de massa permite a fácil determinação dos centróides. - Compare incorporação de dêuterons de proteína alvo sozinho e com excesso de parceiro de ligação. Isso pode ser feito automaticamente com o software comercial ou manualmente com um programa de planilha. Os valores da amostra de controlo de 100% indicam a troca máxima para cada péptido e pode ser utilizado para determinar a quantidade de D 2 O rótulo perdido durante a experiência, devido à troca de volta.
Resultados
Hsp90 é uma chaperone molecular em leveduras e membro da família acompanhante Hsp90. Ao passar por um ciclo de ATPase complexo que auxilia etapas de dobramento final de muitos clientes de proteína. Dobrável eficiente requer transferência de clientes de Hsp70 e interação do Sti1/Hop co-acompanhante. STI1 se liga diretamente a Hsp90 e facilita o cliente a ligação através da inibição da atividade ATPase da Hsp90. Interação da Hsp90 com STI1 foi estudada recentemente usando HX-MS 23. Aqui apresentam...
Discussão
A ligação de um parceiro de interacção com uma proteína, inevitavelmente, provoca alterações na acessibilidade do solvente sobre o local de ligação. Além disso, muitas proteínas sofrem alterações conformacionais dinâmicas sobre a ligação, que afetam outras regiões do que a interface de união real. HX-MS é um método robusto para monitorar essas mudanças e ainda é capaz de revelar mudanças conformacionais em proteínas em escalas de tempo que outros métodos não pode cobrir.
Divulgações
Não temos nada a divulgar.
Agradecimentos
Agradecemos M. Boysen para comentários sobre o manuscrito. Este projecto foi financiado pelo Deutsche Forschungsgemeinschaft (SFB638 e MA 1278/4-1 a MPM, e Cluster de Excelência: CellNetworks EXC 81/1). MPM é investigador do Cluster de Excelência: CellNetworks.
Materiais
| Name | Company | Catalog Number | Comments |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
Referências
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5'-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O'Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados