È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analizzando la dinamica delle proteine che utilizzano l'idrogeno Scambio Spettrometria di Massa
In questo articolo
Riepilogo
Proteine conformazione e la dinamica sono la chiave per comprendere il rapporto tra struttura e funzione delle proteine. Scambio idrogeno accoppiata alla spettrometria di massa ad alta risoluzione è un metodo versatile per studiare le dinamiche conformazionali di proteine e caratterizzazione della proteina-ligando e proteina-proteina interazioni, comprese le interfacce di contatto ed effetti allosterici.
Abstract
Tutti i processi cellulari dipendono dalla funzionalità delle proteine. Sebbene la funzionalità di una data proteina è la diretta conseguenza della sua sequenza amminoacidica unico, si realizza solo dal ripiegamento della catena polipeptidica in un'unica disposizione tridimensionale definito o più comunemente in un insieme di conformazioni interconversione. Indagare il rapporto tra proteine conformazione e la sua funzione è quindi essenziale per una comprensione completa di come le proteine sono in grado di adempiere al loro grande varietà di compiti. Una possibilità per studiare i cambiamenti conformazionali una proteina subisce mentre procede attraverso il suo ciclo funzionale è idrogeno H-1/2 H-scambio in combinazione con spettrometria di massa ad alta risoluzione (HX-MS). HX-MS è un metodo versatile e robusto che aggiunge una nuova dimensione alle informazioni sulla struttura ottenuta per esempio cristallografia. E 'utilizzato per lo studio delle proteine piegatura e dispiegamento, legame di piccole molligandi ecule, interazioni proteina-proteina, cambiamenti conformazionali legati alla catalisi enzimatica, e allosteria. Inoltre, HX-MS è spesso utilizzato quando la quantità di proteina è molto limitata o cristallizzazione della proteina non è fattibile. Qui forniamo un protocollo generale per studiare la dinamica delle proteine con HX-MS e descriviamo un esempio di come rivelare l'interfaccia interazione di due proteine in un complesso.
Introduzione
Il numero di strutture cristalline di proteine e complessi proteici aumentato rapidamente negli ultimi anni. Essi presentano inestimabili istantanee della organizzazione strutturale di queste proteine e forniscono una base per l'analisi struttura-funzione. Tuttavia, la dinamica delle proteine e dei cambiamenti conformazionali, che sono essenziali per le loro funzioni, sono raramente rivelate da cristallografia a raggi X. Crio-electronmicroscopy, invece, è in grado di catturare proteina e proteina complessi in differenti conformazioni ma generalmente non possono risolvere cambiamenti conformazionali fino a livello di struttura secondaria 1. Dinamica conformazionale delle proteine in soluzione a dettagli atomiche possono essere risolti solo da NMR, ma questo metodo è ancora limitato alle proteine di relativamente piccole dimensioni (generalmente ≤ 30 kDa) e ha bisogno di alte concentrazioni di proteine (≥ 100 micron), che ostacola gli esperimenti con oligomerizzazione o aggregazione di proteine incline 2. Un metodo cheè in grado di colmare tra l'alta risoluzione cristallografia a raggi X e crio-electronmicroscopy e che non è limitata dalla dimensione proteine o concentrazione è ammide idrogeno-1 H / 2 H-scambio (HX) in combinazione con spettrometria di massa (MS). Negli ultimi anni questo metodo è sviluppata per uno strumento analitico che per l'analisi della dinamica delle proteine, folding, la stabilità della proteina e cambiamenti conformazionali 3-5. La base molecolare di questo metodo è la natura labile di backbone idrogeni ammidici in proteine, che si scambieranno con atomi di deuterio quando la proteina viene posto in un D 2 O soluzione. Il conseguente aumento della massa proteica nel tempo viene misurata con alta risoluzione MS.
In brevi peptidi strutturati HX dipende solo dalla temperatura, concentrazione di catalizzatore (OH -, H 3 O + pH, vedi Figura 3) e catene laterali di aminoacidi residui adiacenti a causa induttiva, gattoeffetti alytic e sterici. Questi effetti sul intrinseca scambio chimico tasso k ch sono state elegantemente quantificata Bai et al. 6 e un programma è disponibile (cortesia Z. Zhang), che calcola k ch per ogni amminoacido in un polipeptide dipende dal pH e temperatura. A pH neutro e temperature ambiente k ch è dell'ordine di 10 1 -10 3 sec -1. Nelle proteine ripiegate HX può essere 2-9 ordini di grandezza più lento dovuto principalmente legame idrogeno nella struttura secondaria e in misura minore a causa di accesso ristretto di idrati ioni OH - all'interno di una proteina strettamente ripiegata. HX in proteine native implica quindi dispiegarsi, a scambio chimico parziale o globale e ripiegamento allo stato nativo secondo l'equazione (1) e ai tassi di cambio osservati k obs dipendono op tasso di apertura k, il tasso di chiusura k cl e lo scambio chimico intrinseco raTE k ch secondo l'equazione (2).
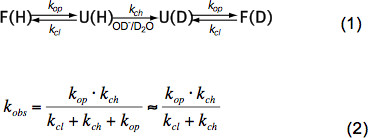
In condizioni di stato nativo k op è molto più piccolo di k ch e può essere trascurato nel denominatore. Ci sono due regimi di cambio estreme chiamati EX1 ed EX2. Se k cl è molto più piccolo k ch (EX1) il tasso osservato è praticamente uguale al tasso di apertura e HX permette l'osservazione immediato dello svolgimento di un elemento strutturale. Un tale regime di scambio, dove ogni scambio contemporaneamente protoni ammidici all'apertura dell'elemento strutturale, è facilmente osservabile in MS da una distribuzione bimodale dei picchi isotopici 7. Se k cl è molto maggiore di k ch (EX2) il tasso osservato è proporzionale k ch cui la costante di proporzionalità è uguale equilibri-piegatori dispiegarsi costante K u = k op / K cl. In queste condizioni, molti eventi di apertura e chiusura sono necessarie prima di effettuare cambi protoni ammidici per deuteroni, portando ad un graduale aumento della massa media mentre la distribuzione isotopica rimane sostanzialmente identico. Il regime EX2 consente la determinazione dell'energia libera di dispiegarsi ΔG u e quindi la stabilità di un elemento strutturale. In condizioni stato nativo il regime EX2 è più comune. Aumento del pH e aggiunta di agenti caotropici può spostare il meccanismo di scambio di EX1. Pertanto, HX-MS può essere utilizzato per esplorare termodinamico e parametri cinetici di folding e cambiamenti conformazionali.
Come accennato in precedenza HX è intrinsecamente pH e temperatura dipendente e lo scambio emivita di un protone completamente solvente esposta del gruppo ammidico backbone è tra 5-400 msec a pH fisiologico (pH 7,6) e 30 ° C, ma 10 min a> 15 ore con una media di> 2 ore a pH 2,9 e 0 °C (tranne per il protone del primo backbone ammide legame di un polipeptide, che scambia con un'emivita di ca. 1-2 min). In tali condizioni lento scambio è possibile digerire l'esempio utilizzando proteasi (ad esempio pepsina) che sono attivi in queste condizioni, con fuori perdere tutte le informazioni contenute nei deuteroni incorporati. Dopo l'introduzione di digestione peptica in condizioni scambio lento, non solo la cinetica complessiva HX di proteine integrali possono essere analizzati ma HX possono essere localizzati alle regioni specifiche 8,9. Risoluzione spaziale è attualmente limitata alle dimensioni dei frammenti generati peptiche, che è in generale tra 10-30 residui. Tuttavia, frammenti sovrapposti creati a causa della natura non specifica di clivaggio con la pepsina potrebbe portare ad un aumento della risoluzione spaziale. Inoltre, diverse altre proteasi sono risultati essere attiva in condizioni di tempra, tuttavia, molto meno efficiente rispetto pepsina 10. Ulteriore Increase di risoluzione spaziale può essere raggiunto con la frammentazione dei peptidi in fase gassosa con metodi che conservano il modello deuterazione come cattura di elettroni dissociazione (ECD), il trasferimento di elettroni dissociazione (ETD) e infrarossi multiphoton dissociazione (IRMPD) 11-13. Queste tecniche prevenire la perdita di risoluzione spaziale dovuta alla migrazione intramolecolare protone ("scrambling"), che si osserva per dissociazione indotta da collisione (CID) la frammentazione tecnica più comunemente usata. Tuttavia, questi metodi richiedono l'ottimizzazione per ogni singolo peptide ed è quindi ancora molto impegnativo.
HX-MS è stato utilizzato per analizzare proteina-ligando e proteina-proteina interazioni compresi l'assemblaggio del capside virale 14-17. Proteine dispiegamento e ripiegamento, nonché di temperatura indotti cambiamenti conformazionali sono stati studiati 7,18,19. Fosforilazione e singolo aminoacido mutazione legata conformazionale cambiamenti 16,20 e nucleotcambiamenti ide-indotti sono stati analizzati 21,22. Pertanto, questo metodo sembra idealmente adatto per analizzare il montaggio e dinamica di macchine molecolari. Un candidato attraente, il cui meccanismo è di grande interesse generale, è il complesso chaperone Hsp90.
Protocollo
1. Preparazione di tamponi e di proteine Campioni
- Preparare H 2 O buffer. Tampone standard uso Hsp90 (40 mM HEPES / KOH, pH 7.5, 50 mM KCl, 5 mM MgCl 2, 10% glicerolo) e H 2 O buffer.
Nota: Se il campione è stato dializzato prima dell'analisi, utilizzare il buffer di dialisi come H 2 O buffer. È essenziale che l'O tampone D 2 differisce dalla H 2 O tampone solo in isotopo dell'idrogeno. Tamponi volatili come NH 4 CO 3 o NH componenti 4 acetato o tampone non sono adatti! - Preparare D 2 O tampone mediante liofilizzazione di tampone standard Hsp90 usando un concentratore a vuoto. Dopo completa evaporazione di H 2 O aggiungere puro D 2 O al tubo per raggiungere il volume totale iniziale (ad esempio, 1 ml di tampone con 15% glicerolo richiede l'aggiunta di 850 microlitri D 2 O). Ripetere la completa evaporazione del buffer e ridisciogliere il com buffer / salecomponenti in D 2 O 2x.
- Preparare tampone di quench (0,4 M KH 2 PO 4 / H 3 PO 4 pH 2,2).
Nota: Per migliorare l'efficienza della peptica digest di proteine molto stabili possono essere aggiunti 4 M guanidina cloridrato e 0,5 M Tris (2-carbossietil) fosfina (TCEP-HCl). - Preparare campione di controllo di 100% (6 M guanidina cloridrato, D 2 O). Aggiungere guanidina cloridrato di un'aliquota Hsp90 per raggiungere una concentrazione finale di 6 M. completamente evaporare H 2 O dal campione e aggiungere D 2 O al tubo per raggiungere il volume totale iniziale (ad esempio 100 microlitri del campione con il 10% glicerolo richiedono l'aggiunta 90 microlitri di D 2 O). Ripetere la completa evaporazione del buffer e riportare in soluzione i componenti tampone / sale di D 2 O.
Nota: 20-100 pmol di campione sono necessari per ogni iniezione. Preparare abbastanza esempio per avere un controllo al 100% per ogni giorno di esperimenti HX-MS. - Preparare 50 pmol Hsp90 in 5 microlitri tampone standard Hsp90.
Nota: è necessaria una quantità di 20-100 pmol di campione per ciascun punto di dati grezzi. Il volume di campione nella reazione è 1-5 microlitri ideale. Regolare la concentrazione per soddisfare queste esigenze. Ogni buffer può essere utilizzato fintanto che non contiene detergenti o componenti volatili.
2. Preparazione di immobilizzato pepsina sulla aldeidi Beads Attivato
- Disciogliere 80 mg pepsina fresco in 2 ml di citrato di sodio 50 mM (pH 5).
- Sciogliere 20 mg di sodiocianoboroidruro, maneggiare con cura (molto tossico!) In 1 ml 2 M Na 2 SO 4 e aggiungere alla soluzione di pepsina.
- Incubare la miscela per 10 minuti a temperatura ambiente mentre delicatamente agitando (shaker es dall'alto).
- Aggiungere 600 mg perline con gruppi aldeidici immobilizzati alla miscela e incubare per 5-10 minuti a temperatura ambiente.
- Aggiungere 2.2 ml di 2 M Na 2 SO4 (pH 5) in aliquote di 100 microlitri ogni 3 min oltre un'ora di sale lentamente la pepsina. Mescolare delicatamente il campione tra le aggiunte in un agitatore in testa a temperatura ambiente.
- Incubare le perline pepsina a 4 ° C per 14-16 ore / notte in un agitatore in testa.
- Estinguere la reazione aggiungendo 1 ml di 1 M etanolammina e incubazione a temperatura ambiente per 2 ore.
- Spin giù le perline in un tubo falcon da 50 ml a 500 rpm, scartare il surnatante e risospendere le sfere nello 0,1% di acido formico. Ripetere questo passaggio 2x. Dopo l'ultima fase di centrifugazione, scartare il surnatante e il volume stima di perline. Aggiungere un volume equivalente di 0,1% di acido formico e conservare a 4 ° C.
3. Preparazione delle Colonne per Ammide Idrogeno-scambio
- Utilizzare le colonne di guardia con un diametro interno di 1 mm per intrappolare le colonne e di 2 mm per colonne pepsina.
- Svitare un lato della colonna di guardia e rimuovere il filtro. Avvitare funn imballaggioel sull'estremità aperta della colonna. Utilizzare un adattatore 1/16 di pollice e tubi per collegare una siringa vuota (5 ml) all'uscita inferiore della colonna. Assicurati di risolvere il problema a tenuta di gas alla colonna di guardia.
- Applicare alcune gocce di slurry materiale tallone sulla parte superiore dell'imbuto. Tirare lo stantuffo della siringa per aspirare il liquame attraverso l'imbuto nella colonna di guardia. Applicare più slurry materiale tallone sul imbuto e continuare la procedura fino a quando la colonna di guardia è completamente riempito con materiale tallone. Rimuovere l'imbuto e posizionare filtro e portafiltro sull'estremità aperta. Fissare la calotta colonna sulla colonna di guardia e rimuovere la siringa dall'altro lato. Chiudere entrambe le estremità delle colonne guardia con tappi per evitare l'essiccamento di materiale colonna.
4. Configurazione del sistema per l'idrogeno di Exchange Spettrometria di Massa (HX-MS)
- Collegare colonna trappola con il sistema HPLC (Figura 1). Equilibrare la colonna impostando la portata diUna pompa a 0,4 ml / min con 0,1% di acido formico come solvente. Non collegare la colonna pepsina, né la colonna analitica ancora.
- Calibrare lo spettrometro di massa e collegare l'uscita del HPLC alla sorgente dello spettrometro di massa.
5. Determinazione della gamma dinamica di scambio
- Preparare ultrapura solvente A (0,1% di acido formico in acqua) e ultrapura solvente B (0,1% di acido formico in acetonitrile); solventi premiscelati sono disponibili in commercio. Spurgare le pompe HPLC. Impostare la cromatografia e metodi di spettrometria di massa nel software di controllo, scegliendo un programma con un gradiente passo dopo passo è breve dissalazione. Per tutta lunghezza Hsp90 utilizzare un desalificazione 1-2 min passo seguito commutando la valvola a 6 porte da desalt / LOAD per eluire e un gradiente passo dal 90% di solvente A/10% di solvente B al 5% di solvente A/95% solvente B. Prima dell'iniezione impostare la valvola di iniezione di carico e la valvola a 6 porte per desalt / posizione di carico.
Appunto: Non utilizzare un pepsina o colonna analitica durante questo esperimento. - Preparare 100-200 pmol di Hsp90 in 1-10 ml H 2 O buffer e incubare per 10 min a 30 ° C. Aggiungi la temperatura regolata D 2 O tampone per portare il volume del campione fino a 100 ml e incubare esattamente per un determinato periodo di tempo (ad esempio 10 sec, 100 sec, 1.000 sec). Aggiungere 100 microlitri di buffer tempra e mescolare pipettando su e giù. Iniettare il campione 200 microlitri nella porta di iniezione della valvola di iniezione con una siringa Hamilton. Avviare il programma cromatografia e commutare la valvola di iniezione in posizione di iniezione. Dopo 2 min commutare la valvola a 6 porte da desalt / CARICAMENTO eluire. Ripetere questo per almeno tre punti temporali.
- Ripetere il punto 5.2 ma add 2-3x eccesso di Sti1 al campione Hsp90 prima dell'incubazione per 10 min a 30 ° C.
- Determinare le masse proteina intera per deconvoluzione degli spettri nel software spettrometria di massa. Calcolare il numero di IncorporatEd deuteroni confrontando il peso molecolare di full-length Hsp90 con la massa osservata dopo ogni corsa (ad esempio 10 sec, 100 sec, 1.000 sec).
- Tracciare i deuteroni incorporati per Hsp90 in assenza e presenza di Sti1 (asse y) in funzione del tempo (asse x). Determinare il tempo-punto della gamma dinamica in cui la differenza tra le due curve è massima. Utilizzare questo valore per il tempo di incubazione in D 2 O quando si identifica l'interfaccia Hsp90-Sti1 e le dinamiche a livello di peptide.
6. Determinazione del peptica Peptidi Utilizzando MS / MS Spectra
- Collegare la colonnina pepsina e la colonna analitica al sistema.
- Impostare i parametri per la cromatografia e spettrometria di massa nel software di controllo scegliendo il tipo di gradiente e metodo di spettrometria di massa. Scegli una lunga discesa (ad esempio, più di 90 min) per assicurare una buona risoluzione cromatografica. Attiva MS / MS spettri dello spettrometro di massa.
Nota: Buona risoluzionezione sul HPLC e l'alta accuratezza di massa hanno la massima priorità in questa fase. - Preparare 100-200 pmol di Hsp90 in 100 ml H 2 O buffer. Aggiungere 100 microlitri di buffer tempra e mescolare pipettando su e giù. Iniettare il campione 200 microlitri nella porta di iniezione della valvola di iniezione con una siringa Hamilton. Avviare il programma cromatografia e commutare la valvola di iniezione in posizione di iniezione. Dopo 2 min commutare la valvola a 6 porte da desalt / CARICAMENTO eluire posizione.
Nota: Se più di una proteina deve essere analizzato in HX-MS (ossia le interfacce di interazione proteina-proteina) i peptidi per ogni proteina devono essere determinati individualmente. - Identificare peptidi peptiche di Hsp90 con la ricerca di un database (cioè mascotte) per i peptidi risultanti.
Nota: E 'possibile utilizzare un database personalizzato come scopo è determinare quante peptidi peptiche possibile e analisi viene fatta con proteina purificata. Purezza campione have essere preso in considerazione. - Ripetere questo passo senza MS / MS e con il gradiente che verrà utilizzata per l'esperimento vero HX. Per Hsp90 e Sti1 usano un 10 min pendenza dal 90% di solvente A/10% di solvente B al 45% di solvente A/55% di solvente B.
Nota: Le sfumature sono normalmente compresi tra 5-15 min e molto dipenderà dalla complessità del campione e specifiche di sistema HX. - Determinare i tempi di ritenzione dei peptidi peptiche identificati nella 6.4 in sfumatura utilizzata in 6.5 e creare un elenco comprendente 1.) Sequenza peptidica 2.) Peptide stato di carica e 3.) Tempo di ritenzione. Questo verrà utilizzato per identificare ciascun peptide dopo esperimenti HX.
Nota: Tenere presente che i peptidi con differenze piccola m / z, stato di carica identica e tempo di ritenzione identico possono essere una fonte di ambiguità.
7. Identificazione delle proteina-proteina di interazione Interfacce
- Impostare il gradiente e metodo di spettrometria di massa nel softwar controlloe. Utilizzare un gradiente lineare a 10 min dal 90% di solvente A/10% di solvente B al 45% di solvente A/55% B. solvente Caricare un metodo di spettrometria di massa che è ottimizzato per il rilevamento delle masse tra 300-1,500 m / z, sebbene la maggior parte di i peptidi saranno inferiori a 1.000 m / z. Impostare la valvola di iniezione in posizione di carico, la valvola a 6 porte in CARICO / dissalazione posizione. Impostare la portata della pompa di carico a 0,4 ml / min.
Nota: La lunghezza e tipo di sfumatura sono dipendenti del campione e possono essere ottimizzati. Gradienti più lunghe migliorano la risoluzione della cromatografia, ma diminuiscono l'incorporazione di deutoni in proteine a causa di back-scambio. I metodi scelti devono essere la stessa per tutti gli esperimenti da confrontare. - Per il riferimento unexchanged preparare 20-100 pmol di Hsp90 in 100 ml H 2 O buffer. Aggiungere 100 microlitri di buffer quench ghiacciata, pipetta su e giù due volte e iniettare campione nella valvola di iniezione del HPLC. Avviare immediatamente il programma di cromatografia e swprudere la valvola di iniezione in posizione di iniezione. Dopo 2 min commutare la valvola a 6 porte da desalt / CARICAMENTO eluire posizione. Fate questo per Hsp90 e Sti1 individualmente e per la miscela di proteine.
- Preparare 20-100 pmol di Hsp90 in un volume di 1-5 ml. Aggiungi la temperatura regolata D 2 O tampone per portare il volume del campione fino a 100 ml e incubare esattamente per un determinato periodo di tempo (ad esempio 30 sec, per dinamica conformazionale vedi Nota). Aggiungere 100 ml di tampone quench ghiacciato, pipetta su e giù due volte e rapidamente iniettare 200 microlitri nella valvola di iniezione del HPLC. Avviare immediatamente il programma di cromatografia e commutare la valvola di iniezione in posizione di iniezione. Dopo 2 min commutare la valvola a 6 porte da desalt / CARICAMENTO eluire posizione. Fate questo per ogni proteina singolarmente, per determinare l'incorporazione deuterone in ciascun peptide in assenza della proteina interagente.
Nota: Quando si studiano le dinamiche di conformational modifiche, ripetere l'esperimento con diversi tempi di incubazione di Hsp90 in D 2 O buffer. Cercare di coprire un ampio lasso di tempo, scegliendo i tempi di incubazione logaritmica (ad esempio 10 sec, 30 sec, 100 sec, 300 sec, 1.000 sec, ecc.) D 2 O tampone e campione buffer deve essere esattamente identico tranne per l'isotopo dell'idrogeno. - Preparare una pari quantità di campione di controllo 100% (20-100 pmol) e aggiungere D 2 O tampone per portare il volume del campione fino a 100 microlitri. Aggiungere 100 ml di tampone quench ghiacciato, pipetta su e giù due volte e rapidamente iniettare 200 microlitri nella valvola di iniezione del HPLC. Avviare immediatamente il programma di cromatografia e commutare la valvola di iniezione in posizione di iniezione. Dopo 2 min commutare la valvola a 6 porte da desalt / CARICAMENTO eluire posizione.
- Per determinare la superficie di interazione mescolare Hsp90 con un eccesso di almeno 2 volte di Sti1 di spostare l'equilibrio verso lo stato legato (vedi nota) e incubarealla temperatura desiderata fino alla formazione del complesso è in equilibrio. Aggiungere temperatura regolata D 2 O tampone per portare il volume del campione fino a 100 microlitri e incubare esattamente per un determinato periodo di tempo (ad esempio 30 secondi). Aggiungere 100 microlitri di tampone quench ghiacciata, pipetta su e giù due volte e iniettare nella valvola di iniezione del HPLC rapidamente. Dopo 2 min commutare la valvola a 6 porte da desalt / CARICAMENTO eluire posizione. Ripetere questo esperimento con 20-100 pmol di Sti1 ed eccesso di Hsp90.
Nota: Le concentrazioni assolute dipendono dalla costante di equilibrio di dissociazione dell'interazione. Idealmente, dopo diluizione in D 2 O la concentrazione della proteina aggiunta in eccesso deve essere almeno 10 volte il K D (corrispondente a> 85% delle proteine inferiore concentrato vincolato). - Analizzare i dati acquisiti con software adatto e utilizzare i tempi di ritenzione determinati nella fase 6.5 di trovare ogni peptide nell'analisi. CalculaTE il baricentro della distribuzione isotopo per la proteina unexchanged (passo 7.2) e per gli esperimenti HX (passo 7.3).
Nota: Anche se i modelli di isotopi di peptidi scambiati aspetto diverso dalle loro controparti unexchanged, sia la conoscenza sullo stato di carica del peptide e l'elevata accuratezza di massa dello spettrometro di massa permette una facile determinazione dei centroidi. - Confronta incorporazione di deutoni di proteine bersaglio solo e con eccesso di partner di legame. Questo può essere fatto automaticamente con software commerciale o manualmente con un programma di foglio. I valori dei campioni di controllo 100% indicano il cambio massimo per ciascun peptide e possono essere utilizzati per determinare la quantità di D 2 O etichetta perso durante l'esperimento a causa di scambio indietro.
Risultati
Hsp90 è un chaperon molecolare nel lievito e membro della famiglia chaperone Hsp90. Passando attraverso un ciclo di ATPasi complesso si assiste fasi tardive di piegatura di molti clienti di proteine. Pieghevole efficiente richiede il trasferimento di clienti da Hsp70 e l'interazione del Sti1/Hop co-chaperone. Sti1 lega direttamente alla Hsp90 e facilita client vincolante inibizione della ATPasi attività di Hsp90. L'interazione di Hsp90 con Sti1 è stata recentemente studiata utilizzando HX-MS 23. Pr...
Discussione
Il legame di un partner di interazione di una proteina provoca inevitabilmente variazioni di accessibilità al solvente sul sito di legame. Inoltre, molte proteine subiscono cambiamenti conformazionali dinamici su vincolante, che colpiscono altre regioni oltre l'interfaccia reale di rilegatura. HX-MS è un metodo affidabile per monitorare questi cambiamenti ed è ancora capace di rivelare cambiamenti conformazionali nelle proteine in tempi che gli altri metodi non possono coprire.
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Ringraziamo M. Boysen per i commenti sul manoscritto. Questo progetto è stato finanziato dalla Deutsche Forschungsgemeinschaft (SFB638 e MA 1278/4-1 di MPM e Cluster di Eccellenza: CellNetworks EXC 81/1). MPM è ricercatore del Cluster di Eccellenza: CellNetworks.
Materiali
| Name | Company | Catalog Number | Comments |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
Riferimenti
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5'-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O'Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon