Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Анализируя Динамика белков с использованием водорода валютный масс-спектрометрии
В этой статье
Резюме
Конформации белка и динамика являются ключевыми для понимания взаимосвязи между структурой и функции белка. Обмен водорода в сочетании с масс-спектрометрии высокого разрешения является универсальным методом для изучения конформационной динамики белков, а также характеризующие белок-лиганд и белок-белковых взаимодействий, в том числе контактных интерфейсов и аллостерических эффектов.
Аннотация
Все клеточные процессы зависят от функциональности белков. Хотя функциональность данного белка является прямым следствием его уникальной аминокислотной последовательности, он реализуется только по сворачивания полипептидной цепи в единую определенную трехмерную договоренности или чаще в ансамбль взаимопревращени конформации. Исследуя связь между конформации белка и его функции Поэтому для полного понимания того, как белки в состоянии выполнить их большое разнообразие задач. Одной из возможностей для изучения конформационных изменений белка претерпевает в то время как прогрессирует через его функционального цикла представляет собой водород-1Н / 2 Н-обменного в сочетании с масс-спектрометрии высокого разрешения (HX-MS). HX-МС является универсальным и надежный метод, который добавляет новое измерение к структурной информации, полученной, например, кристаллографии. Он используется для изучения белок складывания и раскладывания, связывание небольшой мольecule лиганды, белок-белковые взаимодействия, конформационные изменения, связанные с ферментативного катализа и allostery. Кроме того, HX-MS часто используется, когда количество белка очень ограничено или кристаллизации белка не представляется возможным. Здесь мы предлагаем общий протокол для изучения динамики белков с HX-МС и описать в качестве примера, как показывают интерфейс взаимодействия двух белков в комплексе.
Введение
Количество кристаллических структур белков и белковых комплексов резко увеличилось в последние годы. Они представляют бесценные снимки структурной организации этих белков и обеспечить основу для структуры и функции анализа. Тем не менее, динамика белков и конформационных изменений, которые необходимы для их функций, которые редко показал с помощью рентгеновской кристаллографии. Крио-Электронная микроскопия, с другой стороны, имеет возможность захвата белка и белковых комплексов в различных конформаций, но в целом не может решить конформационные изменения до среднего уровня структуры 1. Конформационная динамика белков в растворе на атомном деталей может быть решен только с помощью ЯМР, но этот метод по-прежнему ограничен белков относительно небольших размеров (обычно ≤ 30 кДа) и нуждается в высокой концентрации белков (≥ 100 мкм), что затрудняет эксперименты с олигомеризации или агрегации, склонные белков 2. Одним из методов,способна преодолеть между высоким разрешением рентгеновской кристаллографии и крио-Электронная микроскопия и который не ограничен размером белка или концентрации водорода является амид-1Н / 2 Н-обменного (HX) в сочетании с масс-спектрометрией (MS). В последние годы этот метод разработан для ценного аналитического инструмента для анализа динамики белков, сворачивания белка, стабильности белка и конформационных изменений 3-5. Молекулярные основы этого метода является лабильным характер системообразующих амидных атомов водорода в белках, которые будут обмениваться с атомами дейтерия, когда белок помещается в D 2 O раствора. Последующее увеличение массы белка с течением времени измеряется с высоким разрешением MS.
В коротких неструктурированных пептидов HX зависит только от температуры, концентрации катализатора (ОН -, Н 3 О + т.е. рН, см. рисунок 3) и боковых цепей аминокислот из соседних остатков из-за индуктивной, кошкиalytic и пространственные эффекты. Эти эффекты на внутренней химической обменный курс К ч были элегантно количественно Бай и др.. 6 и программа доступна (любезность З. Чжан), который вычисляет К ч для каждой аминокислоты в полипептид в зависимости от рН и температуры. При нейтральном рН и температуре окружающей среды К ч составляет порядка 10 1 -10 3 с -1. В свернутых белков HX может быть 2-9 порядков медленнее в основном за счет водородных связей в вторичной структуры и в меньшей степени за счет ограниченного доступа гидратированных ОН - ионов к внутренней части плотно уложенного белка. HX в нативных белков поэтому подразумевает частичную или глобальной разворачивается, химический обмен и повторной укладки в нативном состоянии в соответствии с уравнением (1) и наблюдаемые курсы обмена K набл зависеть от темпов открытия К ор, скорость закрытия К кл и внутренней химического обмена раТе К ч в соответствии с уравнением (2).
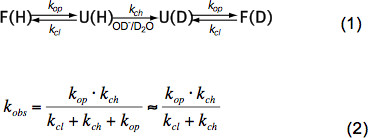
Под носителями государственных условиях к оп намного меньше, чем К ч и ими можно пренебречь в знаменателе. Есть два крайних режимов обмена называемые EX1 и EX2. Если к кл намного меньше, чем К ч (EX1) наблюдаемая скорость практически равна скорости открытия и HX позволяет сразу же наблюдение разворачивание структурного элемента. Такой обмен режим, где все амидные протоны обмен сразу при открытии структурного элемента, легко наблюдаемая в MS бимодальным распределением изотопов пиков 7. Если к кл намного больше, чем К ч (EX2) наблюдаемая скорость пропорциональна К ч причем коэффициент пропорциональности равен складных-разворачивания равновесий постоянная К U = K оп К / кл. В этих условиях многие открытия и закрытия мероприятия необходимы, прежде всего обмен амидные протонов для дейтронов, что приводит к постепенному увеличению средней массы в то время как изотопный распределение остается примерно такой же. Режим EX2 позволяет определить свободной энергии разворачивается ΔG и и поэтому стабильность структурного элемента. Под родной государственной состоянии режим EX2 является наиболее распространенным. Повышение рН и добавлением хаотропных агентов можно сдвинуть обменный механизм EX1. Таким образом, HX-MS может быть использован для изучения термодинамических а также кинетические параметры складывания белка и конформационных изменений.
Как упоминалось выше HX внутренне рН и температура зависят и обмен полураспада полностью открытой растворителя протона магистральной амидной группы находится между 5-400 мс при физиологическом значении рН (рН 7,6) и 30 ° С, но 10 мин до> 15 час при среднем значении> 2 ч при рН 2,9 и 0 °С (для протона первого магистральной амидной связи полипептида, который обменивается с периодом полураспада ок. 1-2 мин исключением). В таких медленно обменивающихся условиях можно переварить примера с использованием протеазы (например пепсин), которые активны в этих условиях, с нашими потерять всю информацию, содержащуюся в объединенных дейтронов. С момента введения язвенной пищеварения при медленных условиях обмена, не только общие HX кинетика полноразмерные белки могут быть проанализированы, но HX могут быть локализованы в конкретных регионах 8,9. Пространственное разрешение в настоящее время ограничивается размером пептической фрагментов, полученных, которая является в целом 10-30 остатков. Тем не менее, перекрывающиеся фрагменты, созданные за счет неспецифического характера расщепления пепсином может привести к увеличению пространственного разрешения. Кроме того, несколько других протеаз, оказались активными при условиях закалки, однако, значительно менее эффективен, чем пепсина 10. Кроме того увеличилисьсебе пространственного разрешения может быть достигнуто путем фрагментации пептидов в газовой фазе методами, которые сохранились дейтерирование шаблон например захвата электронов диссоциации (ECD), перенос электрона диссоциации (ETD) и инфракрасного многофотонная диссоциация (ИКМФД) 11-13. Эти методы предотвращения потери пространственного разрешения в связи с внутримолекулярной миграции протонов ("скремблирования"), которое наблюдается на столкновительное диссоциации (CID) наиболее часто используется методика фрагментации. Однако эти методы требуют оптимизации для каждого отдельного пептида и, таким образом, все еще довольно сложной задачей.
HX-MS была использована для анализа белок-лиганд и белок-белковых взаимодействий в том числе вирусного капсида сборки 14-17. Белок разворачивается и повторной укладки, а также температуры, вызванные конформационные изменения были исследованы 7,18,19. Фосфорилирования и один аминокислотный мутация связанных конформационные изменения 16,20 и nucleotязь-индуцированные изменения были проанализированы 21,22. Таким образом, этот метод кажется идеально подходит для анализа сборки и динамики молекулярных машин. Один привлекательным кандидатом, механизм которого имеет большое общий интерес, является компаньонка комплекс Hsp90.
протокол
1. Подготовка буферов и Белки Образцы
- Подготовка H 2 O буфер. Использование Hsp90 стандартный буфер (40 мМ HEPES / KOH, рН 7,5, 50 мМ KCl, 5 мМ MgCl 2, 10% глицерина) в H 2 O буфер.
Примечание: Если образец подвергают диализу перед анализом, использовать буфер диализа как Н 2 О буфера. Очень важно, чтобы уплотнительное буфера D 2 отличается от вывода буфера H 2 только в изотопа водорода. Летучие буферы, такие как NH 4 CO 3 или NH 4-ацетат или буферные компоненты не подходят! - Подготовка D 2 O буфер путем лиофилизации Hsp90 стандартного буфера с помощью вакуумного концентратора. После полного испарения H 2 O добавлять чистый D 2 O в пробирку, чтобы достичь первоначального общего объема (например, 1 мл буфера с 15% глицерина требует добавления 850 мкл D 2 O). Повторите полное испарение буфера и повторного растворения буфер / соль комкомпоненты в D 2 O 2x.
- Подготовка закалки буфер (0,4 М KH 2 PO 4 / H 3 PO 4 рН 2,2).
Примечание: Чтобы повысить эффективность язвенной дайджест очень стабильными белками 4 М гуанидин гидрохлорид и 0,5 М трис (2-карбоксиэтил) фосфин (ТСЕР-HCl) могут быть добавлены. - Подготовьте 100% контрольного образца (6 М гуанидина гидрохлорид, D 2 O). Добавить гидрохлорид гуанидина к аликвоте Hsp90, чтобы достичь конечной концентрации 6 М Полностью испаряется H 2 O из образца и добавить D 2 O в пробирку, чтобы достичь первоначального общего объема (например, 100 мкл образца с 10% глицерина требует добавления 90 мкл D 2 O). Повторите полное испарение буфера и растворяться компоненты буфера / соли в D 2 O.
Примечание: 20-100 пмоль образца требуются для каждой инъекции. Подготовьте достаточно образец иметь контроль на каждый день HX-MS экспериментов 100%. - Подготовка 50 пмоль Hsp90 в 5 мкл Hsp90 стандартного буфера.
Примечание: количество 20-100 пмоль образца требуется для каждой точки исходных данных. Объем образца в реакции, 1-5 мкл идеально. Отрегулируйте концентрацию, чтобы соответствовать этим требованиям. Любой буфер может быть использован тех пор, пока он не содержит моющие средства или летучие компоненты.
2. Получения иммобилизованных Пепсин на альдегидов Активированный бисер
- Растворите 80 мг свежего пепсин в 2 мл 50 мМ цитрата натрия (рН 5).
- Растворите 20 мг натрийцианборгидрида, обращаться с осторожностью (очень токсичного!) В 1 мл 2 М Na 2 SO 4 и добавить в пепсина решения.
- Инкубировать смесь в течение 10 мин при комнатной температуре при перемешивании осторожно (например, пленка шейкер).
- Добавить 600 мг шарики с иммобилизованными альдегидных групп к смеси и инкубируют в течение 5-10 мин при комнатной температуре.
- Добавить 2,2 мл 2 М Na 2 SO4 (рН 5) в 100 мкл аликвоты через каждые 3 мин в течение одного часа медленно соль из пепсин. Аккуратно перемешайте пробу между дополнений в накладных шейкере при комнатной температуре.
- Инкубируйте пепсина бусы при 4 ° С в течение 14-16 ч / ночь в накладных шейкере.
- Реакцию останавливают добавлением 1 мл из 1 М этаноламина и инкубации при комнатной температуре в течение 2 часов.
- Спином вниз бисером в 50 мл трубки сокола при 500 оборотах в минуту, отбросить супернатант и ресуспендируют бисером в 0,1% муравьиной кислоты. Повторите этот шаг 2x. После последнего центрифугирования, отбросить супернатант и объем оценку бусины. Добавить эквивалентный объем 0,1% муравьиной кислоты и хранить при 4 ° С.
3. Подготовка столбцов для Амид водород-обмен
- Используйте столбцы охраны с внутренним диаметром 1 мм для улавливания столбцы и 2 мм для колонок пепсина.
- Отвинтите одну сторону защитной колонкой и снимите фильтр. Плотно винт упаковка Funnэль на открытом конце колонны. С помощью 1/16 дюймовый адаптер и трубку для крепления пустой шприц (5 мл) к нижней выходе из колонки. Убедитесь в том, чтобы исправить это газонепроницаемая к защитной колонкой.
- Нанести несколько капель суспензии материала шарика в верхней части воронки. Потяните поршень шприца, чтобы сосать суспензии через воронку в защитной колонкой. Применение более суспензии материала шарик на воронку и продолжать процедуру до тех пор столбец охрана не будет полностью заполнен шарик материала. Удалить воронку и поместить фильтр и фильтр кольцо на открытом конце. Плотно завинтить крышку колонки на колонку охраны и удалите шприц с другой стороны. Закройте оба конца столбцов охраны пробками, чтобы избежать высыхания из столбца материала.
4. Настройка системы для обмена водорода масс-спектрометрии (HX-MS)
- Подключите колонки ловушки с системой ВЭЖХ (рис. 1). Равновесие, установив расход столбецнасос до 0,4 мл / мин с 0,1% муравьиной кислоты в качестве растворителя. Не подключайте колонки пепсина, ни аналитической колонки еще.
- Калибровка масс-спектрометра и соединить выпускное отверстие ВЭЖХ на источник масс-спектрометра.
5. Определение динамического диапазона бирже
- Подготовка сверхчистой растворитель А (0,1% муравьиная кислота в воде) и растворитель В ультрачистой (0,1% муравьиной кислоты в ацетонитриле); готовые смешанные растворители являются коммерчески доступными. Продуйте ВЭЖХ насосы. Настройка хроматографии и методов масс-спектрометрии в программном обеспечении управления путем выбора программы ступенчатым градиентом после короткого шага обессоливания. Для полной длины Hsp90 использовать шаг обессоливания 1-2 мин с последующим переключением 6-ходовой клапан с опреснения / НАГРУЗКИ для элюирования и ступенчатый градиент от 90% растворителя A/10% растворителя B до 5% растворителя A/95% растворителя Б. Перед инъекцией установить Форсунка для загрузки и 6-ходовой клапан для опреснения / положение загрузки.
Примечание: Не используйте пепсин или аналитическую колонку в течение этого эксперимента. - Подготовка 100-200 пмоль Hsp90 в 1-10 мкл H 2 O буфера и инкубировать в течение 10 мин при 30 ° С Добавить температуру доводили D 2 O буфер, чтобы довести объем выборки до 100 мкл и инкубировать точно в течение определенного периода времени (например, 10 сек, 100 сек, 1000 сек). Добавить 100 мкл закалки буфер и перемешать с помощью пипетки вверх и вниз. Введите образец 200 мкл в инъекционный порт впрыска клапана с помощью шприца Гамильтона. Запустите программу хроматографии и переключите Форсунка в инъекционные положении. После 2 мин переключения 6-ходовой клапан с опреснения / погрузки для элюирования. Повторите это по крайней мере три временных точках.
- Повторите шаг 5.2, но добавить 2-3x избыток Sti1 к образцу Hsp90 до инкубации в течение 10 мин при 30 ° С
- Определить белковые массы полнометражные по деконволюции спектров в средствах массовой программного обеспечения спектрометрии. Рассчитать количество incorporatред дейтроны путем сравнения молекулярную массу полной длины Hsp90 с наблюдаемой массы после каждого запуска (например, 10 сек, 100 сек, 1000 сек).
- Участок объединенной дейтронов для Hsp90 в отсутствие и в присутствии Sti1 (ось у) в зависимости от времени (ось х). Определить временной точке в динамическом диапазоне, где различие между двумя кривыми максимальна. Используйте это значение для времени инкубации в D 2 O при определении Hsp90-Sti1 интерфейс и динамику на уровне пептидов.
6. Определение язвенной пептидов Использование MS / MS Spectra
- Подключите колонки пепсина и аналитическую колонку в систему.
- Настройте параметры для хроматографии и масс-спектрометрии в программном обеспечении управления, выбирая градиент тип и метод масс-спектрометрии. Выберите длинное градиент (например, более чем 90 мин), чтобы обеспечить хорошее разрешение хроматографического. Включение MS / MS спектры на масс-спектрометре.
Примечание: Хорошо разрешениемТион на ВЭЖХ и высокой точностью массы имеют наивысший приоритет в этом шаге. - Подготовка 100-200 пмоль Hsp90 в 100 мкл H 2 O буфера. Добавить 100 мкл закалки буфер и перемешать с помощью пипетки вверх и вниз. Введите образец 200 мкл в инъекционный порт впрыска клапана с помощью шприца Гамильтона. Запустите программу хроматографии и переключите Форсунка в инъекционные положении. После 2 мин переключения 6-ходовой клапан с опреснения / погрузки элюироваться позицию.
Примечание: Если больше чем один белок должен быть проанализирован в HX-МС (т.е. интерфейсы белок-белкового взаимодействия) пептиды для каждого белка должны быть определены индивидуально. - Определить язвенной пептиды Hsp90 с помощью функции поиска базу данных (то есть талисман) для полученных пептидов.
Примечание: Можно использовать пользовательскую базу данных как целью является определить, как много язвенной пептиды, насколько это возможно и анализ делается с очищенным белком. Образец чистоты будет чпр. должны быть приняты во внимание. - Повторите этот шаг без MS / MS и с градиентом, который будет использоваться для фактического эксперимента HX. Для Hsp90 и Sti1 использовать 10 мин градиент от 90% растворителя A/10% растворителя B в 45% растворителя A/55% растворителя Б.
Примечание: градиенты, как правило, между 5-15 мин и очень зависят от сложности выборки и технические характеристики системы HX. - Определить времени удерживания язвенной пептидов, идентифицированных в 6,4 градиента, используемого в 6,5 и создать список, содержащий 1.) Пептидной последовательности 2.) Пептид государственное обвинение и 3.) Время удерживания. Это будет использоваться для идентификации каждого пептида после HX экспериментов.
Примечание: Помните, что пептиды с небольшой м / г различий, идентичный уровня заряда и одинаковое время удерживания может быть источником двусмысленности.
7. Идентификация взаимодействию Интерфейсы белок-белковых
- Настройте градиент и метод масс-спектрометрии в контрольной Softwarэ. Используйте линейный градиент на 10 минут с 90% растворителя A/10% растворителя B до 45% растворителя A/55% B. растворителя Загрузите массовый метод спектрометрии, который оптимизирован для обнаружения масс между 300-1500 т / г, хотя большинство из пептиды будет ниже 1000 м / г. Установите Форсунка в положение нагрузки, 6-ходовой клапан в НАГРУЗКА / обессоливания позиции. Установка скорости потока загрузочного насоса к 0,4 мл / мин.
Примечание: длина и тип градиента образец зависит и может должны быть оптимизированы. Более длинные градиенты улучшить разрешение хроматографии но уменьшить включение дейтронов в белки из-за бэк-обмен. Выбранные методы должны быть одинаковыми для всех экспериментах для сравнения. - Для справки unexchanged подготовить 20-100 пмоль Hsp90 в 100 мкл H 2 O буфера. Добавить 100 мкл ледяной закалки буфер, пипетки вверх и вниз в два раза и ввести образец в инъекции клапана ВЭЖХ. Сразу запустить программу хроматографии и программные средствазуд впрыска клапан в инъекционные положении. После 2 мин переключения 6-ходовой клапан с опреснения / погрузки элюироваться позицию. Делайте это в течение Hsp90 и Sti1 индивидуально и для смеси белков.
- Подготовка 20-100 пмоль Hsp90 в объеме 1-5 мкл. Добавить температуру доводили D 2 O буфер, чтобы довести объем выборки до 100 мкл и инкубировать точно в течение определенного периода времени (например, 30 с; для конформационные динамика см. примечание). Добавить 100 мкл ледяной закалки буфера, пипетки вверх и вниз дважды и быстро впрыснуть 200 мкл в инъекции клапана ВЭЖХ. Сразу запустить программу хроматографии и переключите Форсунка в инъекционные положении. После 2 мин переключения 6-ходовой клапан с опреснения / погрузки элюироваться позицию. Для этого для каждого белка по отдельности, чтобы определить дейтронов включение в каждого пептида в отсутствии взаимодействующего белка.
Примечание: При изучении динамики CONFORформационной изменения, повторить эксперимент с различным временем инкубации Hsp90 в D 2 O буфера. Попытка охватить широкий шкалу времени путем выбора времени инкубации логарифмически (например, 10 сек, 30 сек, 100 сек, 300 сек, 1000 сек и т.д.). D 2 O буфера и буфера выборок должны быть точно идентична для изотопа водорода исключением. - Подготовка равное количество 100% контрольного образца (20-100 пмоль) и добавить D 2 O буфера, чтобы довести объем выборки до 100 мкл. Добавить 100 мкл ледяной закалки буфера, пипетки вверх и вниз дважды и быстро впрыснуть 200 мкл в инъекции клапана ВЭЖХ. Сразу запустить программу хроматографии и переключите Форсунка в инъекционные положении. После 2 мин переключения 6-ходовой клапан с опреснения / погрузки элюироваться позицию.
- Для определения поверхности взаимодействия смешать Hsp90 с по крайней мере, 2-кратного избытка Sti1 чтобы сдвинуть равновесие в связанном состоянии (см. примечание) и инкубироватьпри желаемой температуре до образования комплекса не находится в равновесии. Добавить температуру доводили D 2 O буфер, чтобы довести объем выборки до 100 мкл и инкубировать точно в течение определенного периода времени (например, 30 сек). Добавить 100 мкл ледяной закалки буфера, пипетки вверх и вниз дважды и быстро вводить в инъекции клапана ВЭЖХ. После 2 мин переключения 6-ходовой клапан с опреснения / погрузки элюироваться позицию. Повторите этот эксперимент с 20-100 пмоль Sti1 и сверх Hsp90.
Примечание: абсолютные концентрации зависит от диссоциации константы равновесия взаимодействия. В идеале, после разбавления в D 2 O концентрация белка добавляют в избытке должно быть по крайней мере в 10 раз K D (соответствует> 85% нижнего концентрированной белок связан). - Анализ полученных данных с соответствующим программным обеспечением и использовать время удерживания определено на этапе 6,5 найти каждого пептида в анализе. РасчетыТе центр тяжести распределения изотопов для unexchanged белка (шаг 7.2) и для HX экспериментов (шаг 7.3).
Примечание: Несмотря на то, изотопные узоры обмениваемых пептидов отличаться от их unexchanged коллегами, как знание о зарядового состояния пептида и высокая масса точность масс-спектрометра позволяет легко определения центров тяжести. - Сравните включение дейтронов белка-мишени только и с превышением связывания партнера. Это может быть сделано автоматически с коммерческим программным обеспечением или вручную с электронными таблицами. Значения контрольного образца 100% обозначим максимальный обмен на каждого пептида и может быть использовано для определения количества D 2 O этикетка потерянного в ходе эксперимента из-за задней обмена.
Результаты
Hsp90 представляет собой молекулярный сопровождающий в дрожжах и член шаперонной семьи Hsp90. Идя через сложную АТФазы цикла она помогает поздние этапы складывания многих клиентов белка. Эффективное складывающиеся требуется передачу клиентов от Hsp70 и взаимодействие со-шаперонной Sti1/Hop. Sti1...
Обсуждение
Связывание партнера взаимодействия с белком неизбежно приводит к изменениям в доступности растворителя на сайте связывания. Кроме того, многие белки подвергаются динамические конформационные изменения при связывании, которые влияют на другие регионы, чем действующей связывающей ин...
Раскрытие информации
Нам нечего раскрывать.
Благодарности
Мы благодарим М. Boysen замечания по рукописи. Этот проект был профинансирован Deutsche Forschungsgemeinschaft (SFB638 и MA 1278/4-1 в MPM и кластера передового опыта: CellNetworks EXC 81/1). MPM является следователь кластера передового опыта: CellNetworks.
Материалы
| Name | Company | Catalog Number | Comments |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
Ссылки
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5'-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O'Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены