このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
高スループット/高含有スクリーニングに適応した細胞内マイコバクテリアの定量化のための顕微鏡表現型アッセイ
要約
ここでは、小干渉合成RNA(siRNA)、化学化合物、および 結核 菌変異体ライブラリーのハイスループット/高含有スクリーンに適用可能な表現型アッセイについて述べている。この方法は、自動共焦点顕微鏡を用いた蛍光標識された宿主細胞内の蛍光標識型 結核 の検出に依存している。
要約
治療とワクチンが利用できるにもかかわらず、結核(TB)は世界で最も致命的で広範囲にわたる細菌感染症の1つです。数十年以来、多剤および広範囲に薬剤耐性株の突然のバーストは、結核の制御のための深刻な脅威です。そのため、結核の原因 物質である結核 菌(Mtb)にとって重要な新しい標的や経路を特定し、結核薬となりうる新しい化学物質を探ることが不可欠です。1つのアプローチは、干し草の山で針の検索を可能にする大規模なライブラリの遺伝的および化学的スクリーンに適した方法を設定することです。そのために、自動共焦点顕微鏡を用いて蛍光標識された宿主細胞内で蛍光標識 Mtb を検出することに頼る表現型アッセイを開発しました。この インビトロ アッセイは 、Mtbのホストへのコロニー形成プロセスの画像ベースの定量化を可能にし、siRNA-、化学化合物、または Mtb 変異体ライブラリのスクリーンに適した384ウェルマイクロプレート形式に最適化されました。その後、画像はマルチパラメトリック分析のために処理され、宿主細胞内の Mtbの病因に関する読み取り結果を提供します。
概要
過去数年間に報告された新興および再興性感染病原体の中で、結核菌(Mtb)は、2011年に140万人の死亡と870万人の新しい感染症の原因となる著名な場所を保持しています(世界結核レポート2012、www.who.int/topics/tuberculosis/en/)。多剤療法の利用可能性にもかかわらず、感染者数は依然として増加しており、多剤耐性(MDR)ならびに広範囲に薬剤耐性(XDR)Mtbは急速に世界中に広がっている1。また、Mtb抗原の存在を考慮すると、全世界の人口の3分の1がMtbによって潜感染していると考えられることは明らかである。したがって、Mtbと戦う新しい手段が緊急に必要とされています。この文脈で、我々は、自動共焦点蛍光顕微鏡3によるMtbの浸潤および増殖を宿主細胞に監視することに頼るインビトロ視覚表現型アッセイを開発した。384ウェルマイクロタイタープレートでのアッセイの適応は、自動画像取得および分析と組み合わせて、化合物、siRNAおよび細菌変異体の中規模ライブラリーの高含有量/ハイスループットスクリーニング(HC/HTS)を可能にした。この表現型アッセイに関するゲノムワイドRNAiライブラリのスクリーニングにより、Mtb人身売買や細胞内複製に関与する主要な宿主因子の同定が可能になっただけでなく、結核菌によって悪用される宿主経路の解明も可能になった。この特定の表現形学アッセイのもう一つの適応は、Mtbの食前体内持続性に不可欠な細菌因子の同定であった。例えば、ファゴソーム成熟の逮捕は、マクロファージにおけるMtbの生存および複製を促進する主要なメカニズムの1つと考えられている。蛍光標識酸性コンパートメントにおけるMtbノックアウト変異体の細胞内局在のモニタリングにより、生存プロセス4に関与する細菌遺伝子の同定が可能となった。最後に、Mtbの高含有イメージングは、細胞内細菌増殖などの様々な現象を阻害するための薬剤効率を定量化する優れた方法も提供する3。全体として、このタイプの高スループット表現型アッセイは、結核に対する創薬を加速することを可能にし、これらの異なるアプローチによって収集されたデータは、Mtbによって行われるホスト操作のより良い理解に寄与する。
プロトコル
1. ハイスループットゲノムワイドsiRNAスクリーニング
緑色蛍光タンパク質(GFP)を発現するMtb H37Rvに感染したヒトII型気球モデルA549細胞系でスクリーニングを行う。この手順の概要は 、図 1Aに示します。
- 1x siRNAバッファーで保存されている乾燥したsiRNAライブラリ(96ウェルプレート)を4μMの濃度に達し、その混合物の10μlを384ウェルの娘プレート(ドータープレート1)に移します。
- 10 μlの1x siRNAバッファーを娘プレート1に加え、siRNAを2倍希釈します。siRNA再懸濁後、プレートは剥離可能なアルミニウムシールで密封され、少なくとも6ヶ月および2〜3年まで-20°Cで保存することができますが、貯蔵時間はsiRNAライブラリメーカーの推奨によって異なる場合があります。
- 娘プレート1のsiRNAを娘プレート2に希釈し、500nMの濃度に達した。siRNA再懸濁後、プレートは剥離可能なアルミニウムシールで密封され、少なくとも6ヶ月および2〜3年まで-20°Cで保存することができますが、貯蔵時間はsiRNAライブラリメーカーの推奨によって異なる場合があります。
- 使用前に、室温で娘プレート2を解凍する。
- 2.5 μlのsiRNAを娘プレート2から取り出し、384ウェルアッセイプレートに入れる。
- ステップ1.5の同じ384ウェルアッセイプレートで、2.5 μlの陰性および正のコントロールsiRNAをそれぞれのウェルに加えます。
- トランスフェクション試薬を1x D-PBSで希釈して、各ウェルに0.1μlトランスフェクション試薬を提供するのに十分な溶液を得て、希釈トランスフェクション溶液を室温で5分間前キューバ化します。
- トランスフェクション試薬/PBS溶液混合物をアッセイプレートの各ウェルに7.5 μl添加し、室温で30分間インキュベートします。
- 10%のウシ胎児血清(FBS)を添加したRPMI 1640培地に懸濁したA549細胞(1,500細胞/ウェル)の40 μlを加えます。5%CO2を含む雰囲気中で37°Cで3日間培養期間の細胞を維持する。これらの細胞は24時間ごとに分裂し、したがって約12,000個の細胞がトランスフェクションの3日後にウェル内にある。
- 2週間前のGFP発現 Mtb H37Rv培養物をD-PBS(MgCl2 およびCaCl2から無料)で4,000 x gで5分間遠心して洗浄を廃棄します。このステップを 3x. 繰り返します(GFP-Mtb H37Rv カルチャ条件の場合はプロトコル 3 を参照してください)。
- 10%FBSを添加したRPMI 1640培地の10mlで細菌ペレットを懸濁し、室温で1時間デカントして、細菌凝集体を堆積物にできるようにします。
- 細菌の上清を収集し、測定OD600(OD 600 は0.6-0.8の間でなければならない)とGFP-蛍光(RFU値)をマイクロプレートリーダーを使用して細菌の濃度を決定します。同じ条件で調製された別の培養物に対して実験前に生成されたRFU値=f(CFU値)を表示する参照回帰線を用いて懸濁液の力価を算出する。感染の多重度(MOI)5に対応する2.4 x 106 個の細菌/mlを含む細菌懸濁液を調製します。
- 384ウェルアッセイプレート内の培地を取り出し、25μlの新たに調製した細菌懸濁液を加えます。
- 384ウェルアッセイプレートを5%CO2を含む雰囲気で5時間37°Cでインキュベートする。
- 培地を取り出し、10%FBS 3xを添加したRPMI培地で細胞を静かに洗います。
- 残りの細胞外細菌を殺すために、5%CO2を含む大気中で、50μg/mlのアミカシンを含む50μlの新鮮なRPMI-FBS培地を37°Cで1時間処理する。
- 培地含有アミカシンを取り出し、10%FBSを添加した新鮮なRPMI培地を50μl加えます。384ウェルアッセイプレートを5%CO2を含む雰囲気で5日間、37°Cでインキュベートする。
- 画像取得前に、PBSで作りたての30μg/mlのDAPI(最終濃度5μg/ml)を10 μl加え、37°Cで10分間インキュベートします。
- プレートを自動共焦点顕微鏡に積み込みます。

ここをクリックすると、より大きな画像が表示されます。 - 露出パラメータを設定します。放出フィルター520 nmを用いた励起レーザー450nmおよびGFP蛍光を用いた励起レーザー405 nmを用いたDAPI蛍光を記録する。
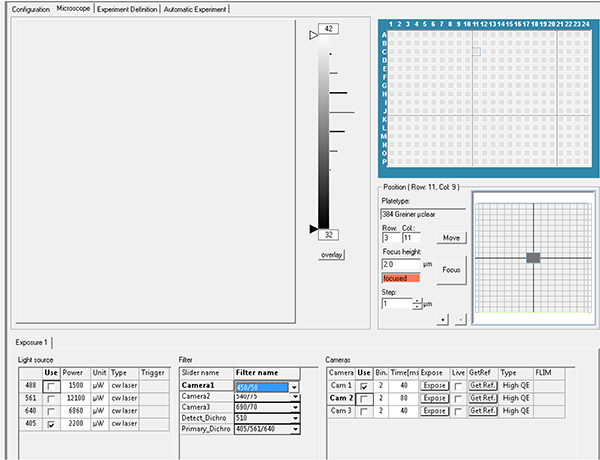
ここをクリックすると、より大きな画像が表示されます。
ここをクリックすると、より大きな画像が表示されます。 - 取得する各ウェルのウェルとフィールドを選択し、レイアウトパラメータとサブレイアウトパラメータと呼びます。
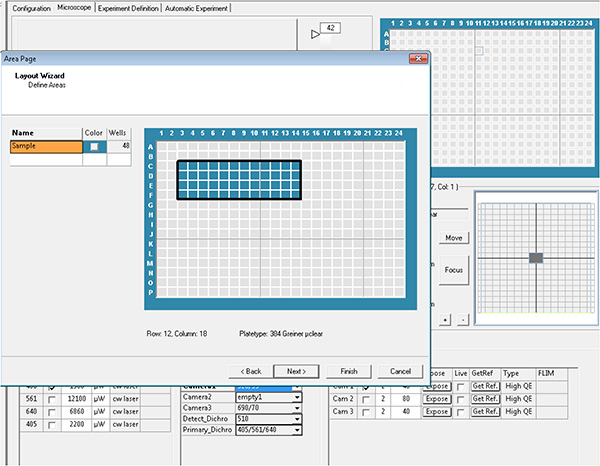
ここをクリックすると、より大きな画像が表示されます。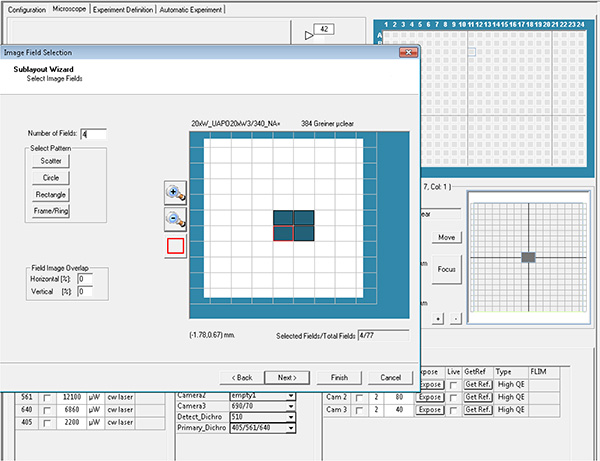
ここをクリックすると、より大きな画像が表示されます。 - 手順 1.20 および 1.21 のパラメータを使用して実験ファイルを生成し、自動取得を実行します。
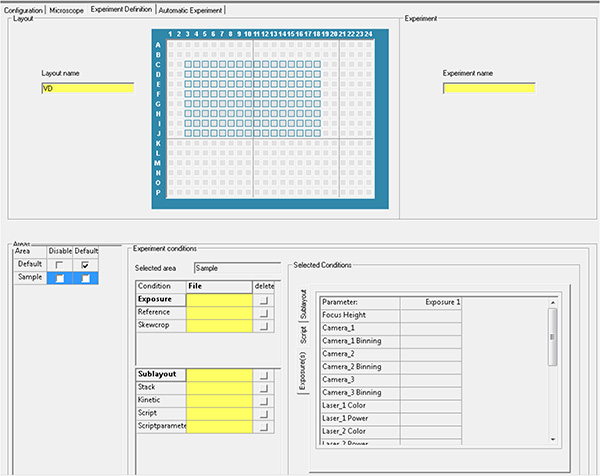
ここをクリックすると、より大きな画像が表示されます。
ここをクリックすると、より大きな画像が表示されます。 - リモート サーバーにイメージを転送します。
ここをクリックすると、より大きな画像が表示されます。 - 画像解析ソフトウェアを使用して画像を評価します。ピクセル強度特性アルゴリズムを用いて、核検出アルゴリズムとGFPチャンネルからの細菌領域を用いてDAPIチャネルから細胞核を検出する(図4A)。
注: このプロトコルは、細胞内 Mtb 成長に対する遺伝子サイレンシングの効果を調べるように最適化されています。 Mtb は、最適な条件で20時間ごとに分割する低成長細菌です。感染後5日後、細胞外 Mtb の量は、細胞内のリシスがない場合にはまだ少なく、分析の質に影響を与えなかった。このプロトコルは、広範囲に放出され、新しい細胞に感染することができる マイコバクテリアススメグマティスや大腸菌 のような急速成長細菌を使用してsiRNAスクリーンに適応する抗生物質治療およびインキュベーション時間の長さの点で最適化されなければならない。
2. ハイスループット複合スクリーニング
Mtb H37Rv感染宿主細胞に対してスクリーニングを行った。この手順の概要は 図 1Bで説明しています。
- DMSO 100%で可溶化した化合物ライブラリーを含む384ウェルのマザープレートを解凍する。10%FBSを添加したRPMI 1640培地10μlを含む384ウェルのドータープレートに0.5 μlの化合物を移します。
- 2週間前のGFP発現 Mtb H37Rv培養物をD-PBS(MgCl2 およびCaCl2から無料)で4,000 x gで5分間遠心して洗浄を廃棄します。このステップ 3x を繰り返します(GFP-Mtb H37Rv カルチャ条件の場合は、プロトコル 3 を参照してください)。
- 10%FBSを添加したRPMI 1640培地の10mlで細菌ペレットを懸濁し、室温で1時間デカントして、細菌凝集体を堆積物にできるようにします。
- 細菌の上清を収集し、マイクロプレートリーダーを使用してOD600(OD 600は0.6-0.8の間でなければならない)とGFP蛍光(RFU値)を測定します。実験前に生成されたRFU値=f(CFU値)を表示する参照回帰線を用いて懸濁液の力価を計算する。典型的な濃度は1 x 108の細菌/mlである。
- 収穫 6 日古い初代ヒトマクロファージRPMI 1640 培地で 10% ウシ胎児血清 (FBS) および 50 ng/ml 組み換えヒト M-CSF (ヒト末梢血単球細胞の精製およびマクロファージ分化についてはプロトコル 4 参照) を補充した。
- 希釈した一次細胞を異なるMOIで、1〜5の範囲で、37°Cで2時間90rpmで軽度の揺れを伴う懸濁液でインキュベートする。
- 感染した細胞を350xgで遠心分離して洗浄し、細胞外細菌を除去する。各遠心分離工程の後、10%FBSを添加したRPMI 1640培地中のペレットを再懸濁する。この手順を 2x 繰り返します。
- 10%FBSとアミカシンを50 μg/mlで補充したRPMI 1640培地中の感染細胞を一時停止し、37°Cで1時間の軽い揺れを伴う懸濁液をインキュベートする。
- アミカシンを含む培地を350xgで遠心分離して除去し、10%FBSおよび50ng/ml組み換えヒトM-CSFを添加した完全なRPMI 1640培地で感染細胞を洗浄する。1 回繰り返します。
- ステップ2.1の同じアッセイプレートに40μlの感染マクロファージ懸濁液を加え、すでに10μlの化合物希釈液を含んでいる。各ウェルのDMSOの最終濃度は1%に達しました。
- 5%CO2を含む雰囲気の中で37°Cで5日間アッセイプレートをインキュベートする。
- 細胞透過性の遠赤色蛍光色素で生細胞を染色する。
- プレートを自動共焦点顕微鏡に積み込みます。

ここをクリックすると、より大きな画像が表示されます。 - 露出パラメータを設定します。発光フィルター520 nmを用いた励起レーザー488nmを用いた発光フィルター690 nmおよびGFP蛍光を用いた励起レーザー640 nmを用いた極赤色蛍光を記録する。

ここをクリックすると、より大きな画像が表示されます。
ここをクリックすると、より大きな画像が表示されます。 - 取得する各ウェルのウェルとフィールドを選択し、レイアウトパラメータとサブレイアウトパラメータと呼びます。

ここをクリックすると、より大きな画像が表示されます。
ここをクリックすると、より大きな画像が表示されます。 - 手順 2.14 および 2.15 のパラメータを使用して実験ファイルを生成し、自動取得を実行します。

ここをクリックすると、より大きな画像が表示されます。
ここをクリックすると、より大きな画像が表示されます。 - リモート サーバーにイメージを転送します。

ここをクリックすると、より大きな画像が表示されます。 - 画像解析ソフトウェアを使用して画像を評価します。ピクセル強度特性アルゴリズムを使用して、GFPチャンネルから、ピクセル強度特性アルゴリズムと細菌領域を用いて遠赤色チャネルから細胞領域を検出する(図4B)。
注:このプロトコルは、蛍光タンパク質を発現する変異体(1つのウェル/ワン変異体)によって化合物を置換することによってMtb変異ライブラリースクリーニングに適応することができる(図1C、Brodinらも参照)。4)蛍光変異体は、まずウェル(ウェルあたり20μlの細菌懸濁液)に播種される。細菌は、その後、細胞懸濁液の30 μlによって回収されます。350xgで1分間遠心分離した後、プレートを5%CO2を含む雰囲気中で37°Cでインキュベートする。インキュベーション時間とMOIはアッセイに依存します。例として、ファゴソーム酸性化などの初期の細胞事象を可視化するために、細胞は1〜20の範囲のMOIで2時間感染することができる。リソソームは、5%CO2を含む雰囲気の中で37°Cで1.5時間2μMのリソトラッカー色素を使用して染色され、10%ホルマリンまたは4%パラホルムアルデヒド(PFA)で固定されます。共焦点画像は、リソソーム検出および細胞内局在化のための適切なアルゴリズムを特徴とする画像解析スクリプトを用いて取得し、最終的に解析される。
3. 緑色蛍光タンパク質結核菌H37Rv(GFP-H37Rv)の培養条件を発現
長期保存のために、GFP-H37RvはD-PBSで凍結した(バイアルあたり約1 x 108 マイコバクテリア)。
- ミドルブルックOADC濃縮10%、グリセロール0.5%、Tween-80 0.05%、ハイグロマイシンB(50μg/ml)を添加した7H9スープ培地50mlを含むエルレンマイヤーフラスコ中のGFP-H37Rvの冷凍在庫バイアルを再懸濁します。
- 37°Cで8日間インキュベートする。
- GFP-H37Rv培養のOD600 を測定する。
- GFP-H37Rv培養液を希釈して、ミドルブルックOADC濃縮10%、 グリセロール0.5%、Tween-80 0.05%、ハイグロマイシンB(50μg/ml)を添加した新鮮な7H9ブロス培地でOD 600=0.1を得る。
- GFP-H37Rvを37°Cで8日間インキュベートしてから、アッセイに使用してください。
4. 全血またはバフィーコート調製からのヒト末梢血単球細胞精製
- 1%FBSを含む1x D-PBS(MgCl 2およびCaCl2から解放)で血液ポーチ2xを希釈します。
- 400 x g で Ficoll 密度勾配遠心分離により 20 分間、単球を分離します。
- 単球を収集します。
- 1x D-PBS(MgCl2 およびCaCl2から自由)で1%FBSを含む1%FBSを室温で10分間400xgで遠心分離して、単球3xを洗浄します。
- 細胞を1 x 107の細胞 /mlまで濃縮します。
- メーカーのプロトコルに従ってCD14磁気ビーズを使用して単球を精製します(材料を参照)。
- CD14-単球精製後、細胞を1.5 x 106細胞/ml RPMI 1640で播種し、10%FBSおよび40ng/mlのヒトマクロファージコロニー刺激因子(hM-CSF)を補い、5%CO2を含む雰囲気で37°Cで4日間インキュベートした。
- 4日後、培地を新鮮なRPMI 1640で交換し、10%FBSと40ng/mlのhM-CSFで補い、5%CO2を含む雰囲気中で37°Cで2日間インキュベートした。
- 6日後、細胞はアッセイに使用することができる。
結果
ハイスループットゲノムワイドsiRNAスクリーニング
Mtb は、他のいくつかの肺上皮細胞と同様 に、インビトロで 免疫細胞を植民地化することができる。例えば 、Mtbは、ヒトII型肺球5-7のモデルとして一般的に使用されているA549上皮細胞に感染し、損傷を与えることができる。Dectin-1は 、Mtb 取り込み、A549細胞における細胞内抗菌増殖に対?...
ディスカッション
ここでは、蛍光標識された宿主細胞に感染するためにGFP発現 Mtb H37Rv株を用いた表現型アッセイに必要な方法を説明し、高含有量/ハイスループットスクリーンに適しています。このプロトコルは、広範囲の化合物、蛍光プローブおよび Mtb 変異体に適用することができる。上述した各プロトコルについて、画像取得前に固定および免疫標識のステップを行うことができた。20X(NA 0....
開示事項
利益相反は宣言されていません。
謝辞
この作業に対する財政的支援は、欧州共同体(ERC-STG INTRACELLTBグラントn°260901、MM4TBグラントn°260872)、アジェンス・ナショナル・ド・レシェルシュ、フェダー(12001407(D-AL)エクイペックス・イマジックス・バイオメッド)、および地域ノルド・パ・ド・カレーによって提供されました。私たちは、プラットフォームBICeLからガスパール・デロイソン、エリザベス・ヴェルクマイスター、アントニーノ・ボンジョヴァンニ、フランク・ラフォントの技術支援を感謝しています。
資料
| Name | Company | Catalog Number | Comments |
| µclear-plate black, 384-well | Greiner Bio-One | 781091 | 127.8/86/15 MM with Lid, TC treated |
| CellCarrier 384-well plate | PerkinElmer | 6007550 | Black, Clear Bottom, with Lid, TC treated |
| V-bottom white, 384-well plate | Greiner Bio-One | 781280 | |
| sealing tape, breathable, sterile | Corning | 3345 | |
| Lipofectamine RNAiMax | Life Technologies | 13778150 | Transfection reagent |
| Dimethyl sulfoxide | Sigma-Aldrich | 34943 | |
| RPMI 1640 + GlutaMAX-I | Life Technologies | 61870-010 | Cell culture medium |
| D-PBS 1x [-]MgCl2/[-]CaCl2 | Life Technologies | 14190-094 | Dulbecco's Phosphate Salin Buffer |
| D-PBS 1x [+]MgCl2/[+]CaCl2 | Life Technologies | 14190-091 | Dulbecco's Phosphate Salin Buffer |
| Fetal bovine serum | Life Technologies | 2610040-79 | |
| Ficoll Paque PLUS | Dutscher | 17-1440-03 | Ficoll for Peripherical Blood Monocyte Cells purification |
| CD14 MicroBeads, human | Miltenyi | 130-050-201 | Purification of CD14+ Monocytes |
| Human M-CSF, premium gr. (1000 μg) | Miltenyi | 130-096-493 | Macrophage Colony Stimulating Factor |
| LS Columns | Miltenyi | 130-042-401 | Columns for CD14+ Monocytes isolation |
| Tween 80 | Euromedex | 2002-A | Mycobacteria culture |
| Glycerol high purity | Euromedex | 50405-EX | Mycobacteria culture |
| Middlebrook OADC enrichment | Becton-Dickinson | 211886 | Mycobacteria culture |
| 7H9 | Becton-Dickinson | W1701P | Mycobacteria culture |
| Versene 1x | Life Technologies | 15040033 | Nonenzymatic cell dissociation solution |
| DAPI | Life Technologies | D1306 | Nuclei dye |
| Hoechst 33342 | Life Technologies | H3570 | Nuclei dye |
| Syto60 | Life Technologies | S11342 | Nuclei/cytoplasm dye |
| Formalin | Sigma-Aldrich | HT5014 | Cell fixation solution |
| siRNA targeting Dectin-1 | Santa-Cruz | sc-63276 | |
| *siGenome* Nontargeted siRNA pool | Dharmacon | D-001206-14 | |
| Rifampicin | Sigma-Aldrich | R3501 | antibiotic |
| Isoniazid (INH) | Sigma-Aldrich | I3377-50G | antibiotic |
| Hygromycin B | Life Technologies | 10687-010 | antibiotic |
| Amikacin | Sigma-Aldrich | A1774 | antibiotic |
| Automated Confocal Microscope OPERA | PerkinElmer | Image acquisition | |
| Columbus 2.3.1 Server Database | PerkinElmer | Data transfer, storage, and analysis | |
| Acapella 2.6 software | PerkinElmer | Image-based analysis | |
| GraphPad Prism5 software | GraphPad | Statistical analysis | |
| Excel 2010 | Microsoft | Statistical analysis |
参考文献
- Gandhi, N. R., et al. Multidrug-resistant and extensively drug-resistant tuberculosis: a threat to global control of tuberculosis. Lancet. 375, 1830-1843 (2010).
- Barry, C. E., et al. The spectrum of latent tuberculosis: rethinking the biology and intervention strategies. Nat. Rev. Microbiol. 7, 845-855 (2009).
- Christophe, T., Ewann, F., Jeon, H. K., Cechetto, J., Brodin, P. High-content imaging of Mycobacterium tuberculosis-infected macrophages: an in vitro model for tuberculosis drug discovery. Future Med. Chem. 2, 1283-1293 (2010).
- Brodin, P., et al. High content phenotypic cell-based visual screen identifies Mycobacterium tuberculosis acyltrehalose-containing glycolipids involved in phagosome remodeling. PLoS Pathog. 6, (2010).
- Bermudez, L. E., Goodman, J. Mycobacterium tuberculosis invades and replicates within type II alveolar cells. Infect. Immun. 64, 1400-1406 (1996).
- Dobos, K. M., Spotts, E. A., Quinn, F. D., King, C. H. Necrosis of lung epithelial cells during infection with Mycobacterium tuberculosis is preceded by cell permeation. Infect. Immun. 68, 6300-6310 (2000).
- McDonough, K. A., Kress, Y. Cytotoxicity for lung epithelial cells is a virulence-associated phenotype of Mycobacterium tuberculosis. Infect. Immun. 63, 4802-4811 (1995).
- Lee, H. M., Yuk, J. M., Shin, D. M., Jo, E. K. Dectin-1 is inducible and plays an essential role for mycobacteria-induced innate immune responses in airway epithelial cells. J. Clin. Immunol. 29, 795-805 (2009).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J. Biomol. Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat. Methods. 6, 569-575 (2009).
- Carralot, J. P., et al. Automated high-throughput siRNA transfection in raw 264.7 macrophages: a case study for optimization procedure. J. Biomol. Screen. 14, 151-160 (2009).
- Zhang, X. D. Illustration of SSMD, z score, SSMD*, z* score, and t statistic for hit selection in RNAi high-throughput screens. J. Biomol. Screen. 16, 775-785 (2011).
- Song, O. R., et al. Confocal-based method for quantification of diffusion kinetics in microwell plates and its application for identifying a rapid mixing method for high-content/throughput screening. J. Biomol. Screen. 15, 138-147 (2010).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved