Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un test phénotypique microscopique pour la quantification des mycobactéries intracellulaires adaptées au criblage à haut débit /haute teneur
Dans cet article
Résumé
Ici, nous décrivons une analyse phénotypique applicable aux écrans à haut débit/à haute teneur de l’ARN synthétique de petit-interférent (siRNA), du composé chimique, et des bibliothèques mutantes de tuberculose de mycobactérie. Cette méthode repose sur la détection de Mycobacterium tuberculosis marqué par fluorescence dans une cellule hôte marqué par fluorescence à l’aide d’une microscopie confocale automatisée.
Résumé
Malgré la disponibilité du traitement et du vaccin, la tuberculose (TB) reste l’une des infections bactériennes les plus mortelles et les plus répandues dans le monde. Depuis plusieurs décennies, l’explosion soudaine de souches multirésistantes et largement résistantes aux médicaments constitue une menace sérieuse pour le contrôle de la tuberculose. Par conséquent, il est essentiel d’identifier de nouvelles cibles et voies critiques pour l’agent causal de la tuberculose, Mycobacterium tuberculosis (Mtb)et de rechercher de nouveaux produits chimiques qui pourraient devenir des médicaments antituberculeux. Une approche consiste à mettre en place des méthodes adaptées aux écrans génétiques et chimiques des bibliothèques à grande échelle permettant la recherche d’une aiguille dans une botte de foin. À cette fin, nous avons développé un test phénotypique en nous appuyant sur la détection de Mtb marqué par fluorescence dans les cellules hôtes marqués par fluorescence à l’aide de la microscopie confocale automatisée. Ce test in vitro permet une quantification basée sur l’image du processus de colonisation de Mtb dans l’hôte et a été optimisé pour le format de microplaque de 384 puits, qui convient aux cribles de siRNA, de composé chimique ou de Mtb mutant-bibliothèques. Les images sont ensuite traitées pour l’analyse multiparamétrique, qui fournit la lecture inférer sur la pathogénie de Mtb dans les cellules hôtes.
Introduction
Parmi les agents pathogènes infectieux émergents et réémergents signalés au cours des dernières années, Mycobacterium tuberculosis (Mtb)occupe une place de choix en étant responsable de 1,4 million de décès et de 8,7 millions de nouvelles infections en 2011 (Global tuberculosis report 2012, www.who.int/topics/tuberculosis/en/). Malgré la disponibilité de thérapies multidrogues, le nombre de personnes infectées est toujours en hausse et multirésistante (MDR) ainsi que le Mtb très résistant aux médicaments (XDR) se propagent rapidement dans le monde entier1. De plus, si l’on tient compte de la présence d’antigènes Mtb, il est évident qu’un tiers de la population mondiale est considéré comme étant infecté de manière latente par Mtb. Statistiquement, dans un cas sur dix, il y a évolution vers la forme active de la maladie avec les symptômes cliniques suivants2. Par conséquent, de nouveaux moyens de lutter contre le VTT sont nécessaires de toute urgence. Dans ce contexte, nous avons développé un test phénotypique visuel in vitro s’appuyant sur la surveillance de l’invasion et de la multiplication du Mtb dans les cellules hôtes par microscopie confocale automatisée à fluorescence3. L’adaptation du test dans des plaques de microtitration de 384 puits en combinaison avec l’acquisition et l’analyse automatisées d’images, a permis un criblage à haute teneur / haut débit (HC / HTS) de bibliothèques à moyenne échelle de composés, de siARN et de mutants bactériens. Le criblage d’une bibliothèque d’ARNi à l’échelle du génome sur ce dosage phénotypique a ainsi permis d’identifier les principaux facteurs hôtes impliqués dans le trafic de Mtb et la réplication intracellulaire mais aussi l’élucidation des voies de l’hôte exploitées par le bacille tuberculeux. Une autre adaptation de ce test phénotypique particulier était pour l’identification des facteurs bactériens essentiels à la persistance intra-phosomique de Mtb. Par exemple, l’arrêt de la maturation du phagosome est considéré comme l’un des principaux mécanismes qui facilitent la survie et la réplication du Mtb dans les macrophages. La surveillance de la localisation subcellulaire des mutants knock-out Mtb dans les compartiments acides marqués par fluorescence a permis d’identifier les gènes bactériens impliqués dans le processus de survie4. Enfin, l’imagerie à haute teneur de Mtb offre également une excellente méthode pour quantifier l’efficacité des médicaments pour inhiber divers phénomènes tels que la croissance bactérienne intracellulaire3. Au total, ce type de dosage phénotypique à haut débit permet d’accélérer la découverte de médicaments contre la tuberculose et les données collectées par ces différentes approches contribuent à une meilleure compréhension de la manipulation de l’hôte exercée par Mtb.
Protocole
1. Criblage à haut débit de l’ARNsi à l’échelle du génome
Criblage effectué dans une lignée cellulaire humaine de pneumocytes de type II modèle A549 lors de l’infection par Mtb H37Rv exprimant la protéine fluorescente verte (GFP). Cette procédure est décrite à la figure 1A.
- Remettre en suspension la bibliothèque d’ARNsi séchés qui est stockée dans des plaques mères (plaques de 96 puits) avec 1x tampon d’ARNsi pour atteindre une concentration de 4 μM, puis transférer 10 μl du mélange dans une plaque fille de 384 puits (plaque fille 1).
- Ajouter 10 μl de tampon d’ARNsi 1x dans la plaque fille 1 pour diluer l’ARNsi par 2 fois. Après la remise en suspension de l’ARNsi, les plaques sont scellées avec un joint en aluminium pelable et peuvent être stockées à -20 ° C au moins 6 mois et jusqu’à 2-3 ans, mais le temps de stockage peut varier en fonction des recommandations du fabricant de la bibliothèque siRNA.
- Diluer les siARN de la plaque fille 1 en plaque fille 2 pour atteindre une concentration de 500 nM. Après la remise en suspension de l’ARNsi, les plaques sont scellées avec un joint en aluminium pelable et peuvent être stockées à -20 ° C au moins 6 mois et jusqu’à 2-3 ans, mais le temps de stockage peut varier en fonction des recommandations du fabricant de la bibliothèque siRNA.
- Avant utilisation, décongeler la plaque fille 2 à température ambiante.
- Prendre 2,5 μl d’ARNsi de la plaque fille 2 et placer dans une plaque d’analyse à 384 puits.
- Dans la même plaque d’analyse de 384 puits à l’étape 1.5, ajouter 2,5 μl d’ARNsi témoin négatif et positif à leurs puits respectifs.
- Diluer le réactif de transfection dans 1x D-PBS pour donner suffisamment de solution pour fournir 0,1 μl de réactif de transfection dans chaque puits et pré-incuber la solution de transfection diluée à température ambiante pendant 5 min.
- Ajouter 7,5 μl du mélange réactif de transfection/solution PBS à chaque puits de la plaque d’essai et incuber pendant 30 min à température ambiante.
- Ajouter 40 μl de cellules A549 (1 500 cellules/puits) en suspension dans un milieu RPMI 1640 complété par 10 % de sérum fœtal bovin (SBF). Maintenir les cellules pendant une période d’incubation de 3 jours à 37 °C dans une atmosphère contenant 5 % deCO2. Ces cellules se divisent toutes les 24 heures, soit environ 12 000 cellules se trouvent dans les puits trois jours après la transfection.
- Laver une culture Mtb H37Rv exprimant le GFP de deux semaines avec du D-PBS (exempt de MgCl2 et CaCl2)par centrifugation à 4 000 x g pendant 5 min et jeter les lavages. Répétez cette étape 3x. (Pour des conditions de culture deGFP-Mtb H37Rv voir le Protocole 3).
- Suspendre le culot bactérien dans 10 ml de milieu RPMI 1640 complété par 10% de FBS et décanter pendant 1 heure à température ambiante pour permettre aux agrégats bactériens de sédimenter.
- Recueillir le surnageant bactérien et mesurer la DO600 (LA600 doit être comprise entre 0,6 et 0,8) et la fluorescence GFP (valeur RFU) à l’aide d’un lecteur de microplaques pour déterminer la concentration bactérienne. Calculer le titre de la suspension à l’aide d’une droite de régression de référence affichant la valeur RFU = f (valeur CFU) qui avait été générée avant l’expérience sur une autre culture qui avait été préparée dans les mêmes conditions. Préparer une suspension bactérienne contenant 2,4 x 106 bactéries/ml, ce qui correspond à une multiplicité d’infection (MOI) de 5.
- Retirer le milieu dans la plaque d’analyse de 384 puits et ajouter 25 μl de suspension bactérienne fraîchement préparée.
- Incuber la plaque d’analyse à 384 puits à 37 °C pendant 5 heures dans une atmosphère contenant 5 % deCO2.
- Retirez le milieu et lavez doucement les cellules avec un milieu RPMI complété par 10% FBS 3x.
- Pour tuer les bactéries extracellulaires restantes, traiter les cellules avec 50 μl de milieu RPMI-FBS frais contenant 50 μg/ml d’amikacine à 37 °C pendant 1 h dans une atmosphère contenant 5 % deCO2.
- Retirer l’amikacine contenant du milieu et ajouter 50 μl de milieu RPMI frais complété par 10 % de FBS. Incuber la plaque d’analyse à 384 puits à 37 °C pendant 5 jours dans une atmosphère contenant 5 % deCO2.
- Avant l’acquisition de l’image, ajouter 10 μl de 30 μg/ml de DAPI fraîchement préparés dans du PBS (concentration finale de 5 μg/ml) et incuber pendant 10 min à 37 °C.
- Chargez la plaque dans un microscope confocal automatisé.

Cliquez ici pour agrandir l’image. - Définissez les paramètres d’exposition. Enregistrer la fluorescence DAPI à l’aide d’un laser d’excitation 405 nm avec filtre d’émission de 450 nm et la fluorescence GFP à l’aide d’un laser d’excitation de 488 nm avec filtre d’émission de 520 nm.
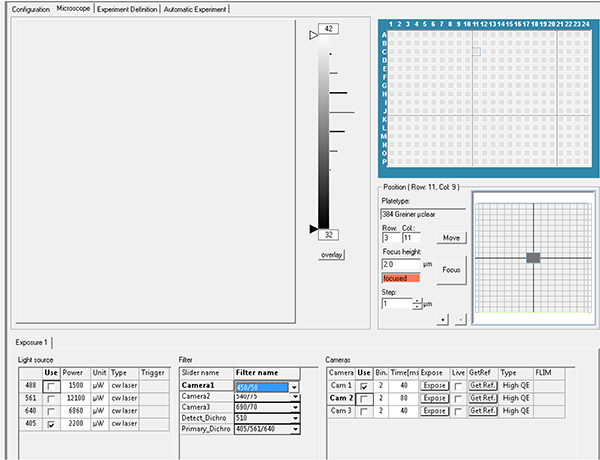
Cliquez ici pour agrandir l’image.
Cliquez ici pour agrandir l’image. - Sélectionnez les puits et les champs de chaque puits à acquérir, ce qui est ensuite appelé paramètres de mise en page et de sous-disposition.
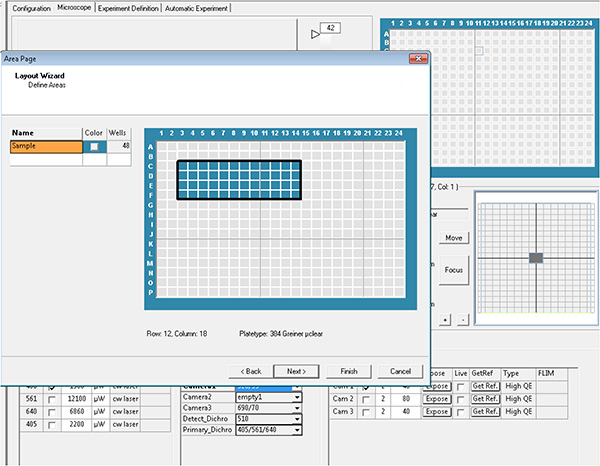
Cliquez ici pour agrandir l’image.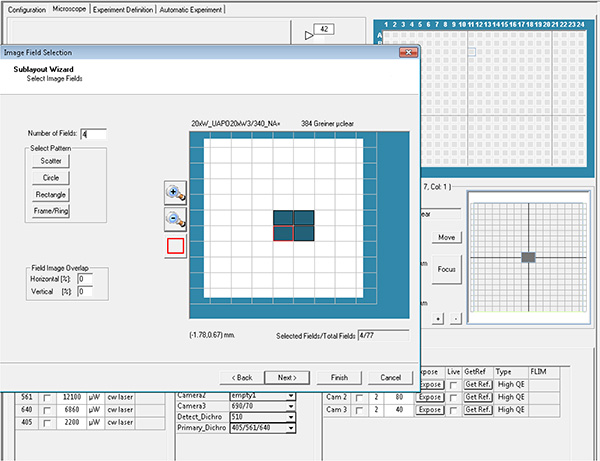
Cliquez ici pour agrandir l’image. - Générez le fichier d’expérience à l’aide des paramètres des étapes 1.20 et 1.21, puis exécutez l’acquisition automatique.
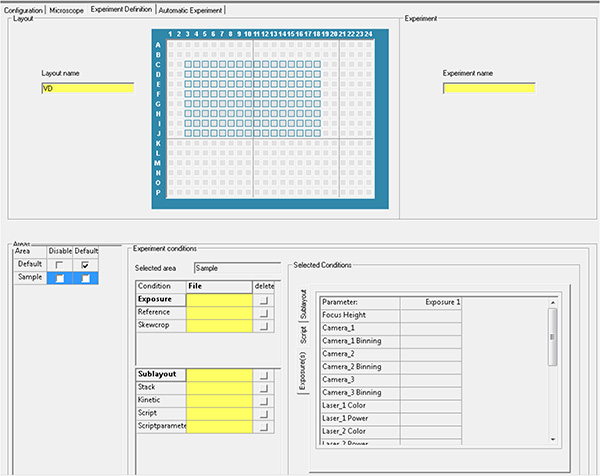
Cliquez ici pour agrandir l’image.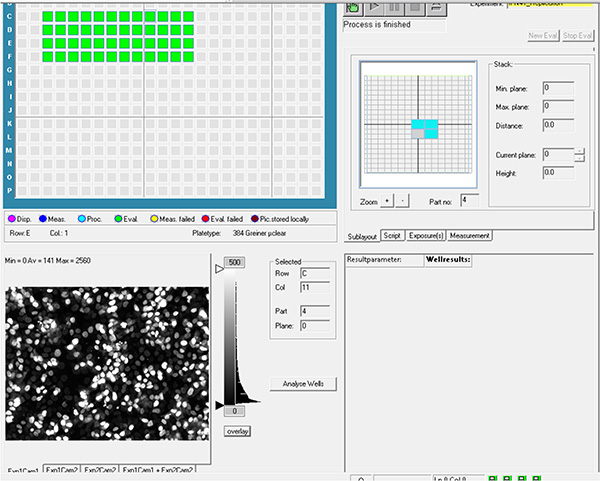
Cliquez ici pour agrandir l’image. - Transférer des images vers un serveur distant.
Cliquez ici pour agrandir l’image. - Évaluer les images à l’aide d’un logiciel d’analyse d’images. Détecter les noyaux cellulaires du canal DAPI à l’aide de l’algorithme de détection des noyaux et la zone bactérienne du canal GFP à l’aide de l’algorithme des propriétés d’intensité des pixels (Figure 4A).
Remarque: Ce protocole est optimisé pour étudier l’effet du silençage génique sur la croissance intracellulaire de Mtb. Le VTT est une bactérie à croissance lente qui se divise toutes les 20 heures dans des conditions optimales. Après 5 jours post-infection, la quantité de VTT extracellulaire est encore faible en l’absence de lyse cellulaire et n’a pas affecté la qualité de l’analyse. Ce protocole doit être optimisé en termes de durée de traitement antibiotique et de temps d’incubation pour être adapté aux criblages siRNA utilisant des bactéries à croissance rapide comme Mycobacteria smegmatis et Escherichia coli qui sont largement libérées et peuvent infecter de nouvelles cellules.
2. Criblage composé à haut débit
Dépistage effectué sur des cellules hôtes infectées par Mtb H37Rv. Cette procédure est décrite à la figure 1B.
- Décongeler les plaques mères de 384 puits contenant la bibliothèque composée solubilisée dans le DMSO à 100%. Transférer 0,5 μl des composés dans des plaques filles de 384 puits contenant 10 μl de milieu RPMI 1640 complété par 10 % de FBS.
- Laver une culture Mtb H37Rv exprimant le GFP de deux semaines avec du D-PBS (exempt de MgCl2 et CaCl2)par centrifugation à 4 000 x g pendant 5 min et jeter les lavages. Répétez cette étape 3x (pour les conditions de cultureGFP-Mtb H37Rv voir protocole 3).
- Suspendre le culot bactérien dans 10 ml de milieu RPMI 1640 complété par 10% de FBS et décanter pendant 1 heure à température ambiante pour permettre aux agrégats bactériens de sédimenter.
- Recueillir le surnageant bactérien et mesurer la DO600 (DO600 doit être comprise entre 0,6 et 0,8) et la fluorescence GFP (valeur RFU) à l’aide d’un lecteur de microplaque. Calculer le titre de la suspension à l’aide d’une droite de régression de référence affichant la valeur RFU = f (valeur CFU) qui avait été générée avant l’expérience. La concentration typique est de 1 x 108 bactéries/ml.
- Récoltez des macrophages humains primaires âgés de 6 jours à 4 x 105 cellules/ml dans le milieu RPMI 1640 complété par 10% de sérum fœtal bovin (FBS) et 50 ng/ml de M-CSF humain recombinant (pour la purification des cellules monocytaires du sang périphérique humain et la différenciation des macrophages, voir le protocole 4).
- Incuber les cellules primaires diluées avec des bacilles à différents MOI, allant de 1 à 5, en suspension avec des secousses légères à 90 rpm pendant 2 heures à 37 °C.
- Laver les cellules infectées par centrifugation à 350 x g pour éliminer les bactéries extracellulaires. Après chaque étape de centrifugation, ressusciter la pastille dans un milieu RPMI 1640 complété par 10% de FBS. Répétez cette étape 2x.
- Suspendre les cellules infectées dans le milieu RPMI 1640 complété par 10 % de FBS et d’amikacine à 50 μg/ml et incuber la suspension avec des secousses légères pendant 1 h à 37 °C.
- Retirer le milieu de culture cellulaire contenant de l’amikacine par centrifugation à 350 x g et laver les cellules infectées avec un milieu RPMI 1640 complet complété par 10 % de FBS et 50 ng/ml de M-CSF humain recombinant. Répétez une fois.
- Ajouter 40 μl de la suspension de macrophages infectés dans la même plaque d’essai à l’étape 2.1, qui contient déjà 10 μl des dilutions composées. La concentration finale de DMSO dans chaque puits a maintenant atteint 1%.
- Incuber les plaques d’essai pendant 5 jours à 37 °C dans une atmosphère contenant 5 % deCO2.
- Colorer les cellules vivantes avec un colorant fluorescent rouge lointain perméant.
- Chargez la plaque dans un microscope confocal automatisé.

Cliquez ici pour agrandir l’image. - Définissez les paramètres d’exposition. Enregistrer la fluorescence rouge lointain à l’aide d’un laser d’excitation 640 nm avec filtre d’émission de 690 nm et la fluorescence GFP à l’aide d’un laser d’excitation de 488 nm avec filtre d’émission de 520 nm.

Cliquez ici pour agrandir l’image.
Cliquez ici pour agrandir l’image. - Sélectionnez les puits et les champs de chaque puits à acquérir, ce qui est ensuite appelé paramètres de mise en page et de sous-disposition.

Cliquez ici pour agrandir l’image.
Cliquez ici pour agrandir l’image. - Générez le fichier d’expérience à l’aide des paramètres des étapes 2.14 et 2.15 et exécutez l’acquisition automatique.

Cliquez ici pour agrandir l’image.
Cliquez ici pour agrandir l’image. - Transférer des images vers un serveur distant.

Cliquez ici pour agrandir l’image. - Évaluer les images à l’aide d’un logiciel d’analyse d’images. Détecter la zone cellulaire du canal rouge lointain à l’aide de l’algorithme des propriétés d’intensité des pixels et la zone bactérienne du canal GFP à l’aide de l’algorithme des propriétés d’intensité des pixels(Figure 4B).
Remarque : Ce protocole peut être adapté pour le criblage de la bibliothèque mutante Mtb en remplaçant les composés par des mutants exprimant une protéine fluorescente (Un puits/Un mutant) (Figure 1C, voir aussi Brodin et al. 4). Les mutants fluorescents sont d’abord ensemencés dans des puits (20 μl de suspension bactérienne par puits). Les bactéries sont ensuite récupérées par 30 μl de suspension cellulaire. Après centrifugation à 350 x g pendant 1 min, la plaque est incubée à 37 °C dans une atmosphère contenant 5 % deCO2. Le temps d’incubation et le MOI dépendent du test. Par exemple, pour la visualisation des premiers événements cellulaires tels que l’acidification des phagosomes, les cellules peuvent être infectées pendant 2 heures avec moi allant de 1-20. Les lysosomes sont colorés à l’aide du colorant Lysotracker à 2 μM pendant 1,5 h à 37 °C dans une atmosphère contenant 5 % deCO2, puis fixés avec 10 % de forcine ou 4 % de paraformaldéhyde (PFA). Les images confocales sont acquises et finalement analysées à l’aide de scripts d’analyse d’images comportant des algorithmes appropriés pour la détection des lysosomes et la localisation subcellulaire4.
3. Protéine fluorescente verte exprimant mycobacterium tuberculosis H37Rv (GFP-H37Rv) Conditions de culture
Pour le stockage à long terme, les GFP-H37Rv ont été congelés dans du D-PBS (environ 1 x10 8 mycobactéries par flacon).
- Résurciez un flacon congelé de GFP-H37Rv dans une fiole d’Erlenmeyer contenant 50 ml de bouillon 7H9 additionnés d’enrichissement en OADC de Middlebrook à 10 %, de glycérol à 0,5 %, de Tween-80 à 0,05 % et d’hygromycine B (50 μg/ml).
- Incuber 8 jours à 37 °C.
- Mesurer laDO 600 de la culture GFP-H37Rv.
- Diluer la culture GFP-H37Rv pour obtenirla DO 600 = 0,1 dans un bouillon 7H9 frais complété par un enrichissement en OADC de Middlebrook à 10 %, du glycérol à 0,5 %, du tween-80 à 0,05 % et de l’hygromycine B (50 μg/ml).
- Incuber le GFP-H37Rv à 37 °C pendant 8 jours de plus avant de l’utiliser pour le dosage.
4. Purification des cellules monocytaires du sang périphérique humain à partir de la préparation de sang total ou de buffy-coat
- Diluer la poche sanguine 2x dans 1x D-PBS (exempt deMgCl2 et CaCl2)contenant 1% fbs.
- Isoler les monocytes par centrifugation de gradient de densité de Ficoll à 400 x g pendant 20 min.
- Recueillir les monocytes isolés.
- Laver les monocytes 3x avec 1x D-PBS (exempt deMgCl2 etCaCl2)contenant 1% de FBS par centrifugation à 400 x g pendant 10 min à température ambiante.
- Concentrer les cellules jusqu’à 1 x 107 cellules/ml.
- Purifier les monocytes à l’aide de billes magnétiques CD14 selon le protocole du fabricant (voir Matériaux).
- Après purification des cd14-monocytes, ensemencer les cellules à 1,5 x10 6 cellules/ml dans RPMI 1640 complété par 10% FBS et 40 ng/ml de macrophages humains Colony Stimulating Factor (hM-CSF) et incubés pendant 4 jours à 37 °C dans une atmosphère contenant 5% deCO2.
- Après 4 jours, remplacer le milieu par du RPMI 1640 frais complété par 10% de FBS et 40 ng/ml de hM-CSF et incubé pendant 2 jours à 37°C dans une atmosphère contenant 5% deCO2.
- Après 6 jours, les cellules peuvent être utilisées pour le test.
Résultats
Criblage à haut débit de l’ARNsi à l’échelle du génome
Mtb est capable de coloniser les cellules immunitaires in vitro ainsi que plusieurs autres cellules épithéliales pulmonaires. Par exemple, Mtbest capable d’infecter et d’endommager les cellules épithéliales A549 qui sont couramment utilisées comme modèle pour les pneumocytes humains de type II5-7. Dectin-1 a été rapporté comme un récepteur de la cellule hôte impliqué dans l’abso...
Discussion
Nous décrivons ici les méthodes requises pour un essai phénotypique utilisant une contrainte de Mtb H37Rv exprimant GFP pour infecter les cellules hôtes fluorescentement étiquetées, ce qui le rend approprié pour des écrans à haute teneur/à haut débit. Ce protocole pourrait être appliqué à un large éventail de composés, de sondes fluorescentes et de mutants Mtb. Pour chaque protocole décrit ci-dessus, des étapes de fixation et d’immunomarquage pourraient être effectuées avant l’ac...
Déclarations de divulgation
Aucun conflit d’intérêts déclaré.
Remerciements
Ce travail a été soutenu financièrement par la Communauté européenne (ERC-STG INTRACELLTB Grant n° 260901, MM4TB Grant n° 260872), l’Agence Nationale de Recherche, le Feder (12001407 (D-AL) Equipex Imaginex BioMed), et la Région Nord Pas de Calais. Nous remercions Gaspard Deloison, Elizabeth Werkmeister, Antonino Bongiovanni et Frank Lafont de la plateforme BICeL pour leur assistance technique.
matériels
| Name | Company | Catalog Number | Comments |
| µclear-plate black, 384-well | Greiner Bio-One | 781091 | 127.8/86/15 MM with Lid, TC treated |
| CellCarrier 384-well plate | PerkinElmer | 6007550 | Black, Clear Bottom, with Lid, TC treated |
| V-bottom white, 384-well plate | Greiner Bio-One | 781280 | |
| sealing tape, breathable, sterile | Corning | 3345 | |
| Lipofectamine RNAiMax | Life Technologies | 13778150 | Transfection reagent |
| Dimethyl sulfoxide | Sigma-Aldrich | 34943 | |
| RPMI 1640 + GlutaMAX-I | Life Technologies | 61870-010 | Cell culture medium |
| D-PBS 1x [-]MgCl2/[-]CaCl2 | Life Technologies | 14190-094 | Dulbecco's Phosphate Salin Buffer |
| D-PBS 1x [+]MgCl2/[+]CaCl2 | Life Technologies | 14190-091 | Dulbecco's Phosphate Salin Buffer |
| Fetal bovine serum | Life Technologies | 2610040-79 | |
| Ficoll Paque PLUS | Dutscher | 17-1440-03 | Ficoll for Peripherical Blood Monocyte Cells purification |
| CD14 MicroBeads, human | Miltenyi | 130-050-201 | Purification of CD14+ Monocytes |
| Human M-CSF, premium gr. (1000 μg) | Miltenyi | 130-096-493 | Macrophage Colony Stimulating Factor |
| LS Columns | Miltenyi | 130-042-401 | Columns for CD14+ Monocytes isolation |
| Tween 80 | Euromedex | 2002-A | Mycobacteria culture |
| Glycerol high purity | Euromedex | 50405-EX | Mycobacteria culture |
| Middlebrook OADC enrichment | Becton-Dickinson | 211886 | Mycobacteria culture |
| 7H9 | Becton-Dickinson | W1701P | Mycobacteria culture |
| Versene 1x | Life Technologies | 15040033 | Nonenzymatic cell dissociation solution |
| DAPI | Life Technologies | D1306 | Nuclei dye |
| Hoechst 33342 | Life Technologies | H3570 | Nuclei dye |
| Syto60 | Life Technologies | S11342 | Nuclei/cytoplasm dye |
| Formalin | Sigma-Aldrich | HT5014 | Cell fixation solution |
| siRNA targeting Dectin-1 | Santa-Cruz | sc-63276 | |
| *siGenome* Nontargeted siRNA pool | Dharmacon | D-001206-14 | |
| Rifampicin | Sigma-Aldrich | R3501 | antibiotic |
| Isoniazid (INH) | Sigma-Aldrich | I3377-50G | antibiotic |
| Hygromycin B | Life Technologies | 10687-010 | antibiotic |
| Amikacin | Sigma-Aldrich | A1774 | antibiotic |
| Automated Confocal Microscope OPERA | PerkinElmer | Image acquisition | |
| Columbus 2.3.1 Server Database | PerkinElmer | Data transfer, storage, and analysis | |
| Acapella 2.6 software | PerkinElmer | Image-based analysis | |
| GraphPad Prism5 software | GraphPad | Statistical analysis | |
| Excel 2010 | Microsoft | Statistical analysis |
Références
- Gandhi, N. R., et al. Multidrug-resistant and extensively drug-resistant tuberculosis: a threat to global control of tuberculosis. Lancet. 375, 1830-1843 (2010).
- Barry, C. E., et al. The spectrum of latent tuberculosis: rethinking the biology and intervention strategies. Nat. Rev. Microbiol. 7, 845-855 (2009).
- Christophe, T., Ewann, F., Jeon, H. K., Cechetto, J., Brodin, P. High-content imaging of Mycobacterium tuberculosis-infected macrophages: an in vitro model for tuberculosis drug discovery. Future Med. Chem. 2, 1283-1293 (2010).
- Brodin, P., et al. High content phenotypic cell-based visual screen identifies Mycobacterium tuberculosis acyltrehalose-containing glycolipids involved in phagosome remodeling. PLoS Pathog. 6, (2010).
- Bermudez, L. E., Goodman, J. Mycobacterium tuberculosis invades and replicates within type II alveolar cells. Infect. Immun. 64, 1400-1406 (1996).
- Dobos, K. M., Spotts, E. A., Quinn, F. D., King, C. H. Necrosis of lung epithelial cells during infection with Mycobacterium tuberculosis is preceded by cell permeation. Infect. Immun. 68, 6300-6310 (2000).
- McDonough, K. A., Kress, Y. Cytotoxicity for lung epithelial cells is a virulence-associated phenotype of Mycobacterium tuberculosis. Infect. Immun. 63, 4802-4811 (1995).
- Lee, H. M., Yuk, J. M., Shin, D. M., Jo, E. K. Dectin-1 is inducible and plays an essential role for mycobacteria-induced innate immune responses in airway epithelial cells. J. Clin. Immunol. 29, 795-805 (2009).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J. Biomol. Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat. Methods. 6, 569-575 (2009).
- Carralot, J. P., et al. Automated high-throughput siRNA transfection in raw 264.7 macrophages: a case study for optimization procedure. J. Biomol. Screen. 14, 151-160 (2009).
- Zhang, X. D. Illustration of SSMD, z score, SSMD*, z* score, and t statistic for hit selection in RNAi high-throughput screens. J. Biomol. Screen. 16, 775-785 (2011).
- Song, O. R., et al. Confocal-based method for quantification of diffusion kinetics in microwell plates and its application for identifying a rapid mixing method for high-content/throughput screening. J. Biomol. Screen. 15, 138-147 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon