È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un test fenotipico microscopico per la quantificazione dei micobatteri intracellulari adattati per lo screening ad alta produttività / alto contenuto
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui descriviamo un saggio fenotipico applicabile agli schermi ad alta produttività /alto contenuto di RNA sintetico (siRNA), composto chimico e librerie mutanti Mycobacterium tuberculosis. Questo metodo si basa sul rilevamento di Mycobacterium tuberculosis con etichetta fluorescente all'interno di una cellula ospite etichettata fluorescentmente utilizzando la microscopia confocale automatizzata.
Abstract
Nonostante la disponibilità di terapia e vaccino, la tubercolosi (TB) rimane una delle infezioni batteriche più mortali e diffuse al mondo. Da diversi decenni, l'improvvisa esplosione di ceppi multi- ed estesamente resistenti ai farmaci è una seria minaccia per il controllo della tubercolosi. Pertanto, è essenziale identificare nuovi obiettivi e percorsi critici per l'agente causale della tubercolosi, Mycobacterium tuberculosis (Mtb)e cercare nuove sostanze chimiche che potrebbero diventare farmaci per la tubercolosi. Un approccio è quello di mettere a punto metodi adatti agli schermi genetici e chimici delle biblioteche su larga scala che consentano la ricerca di un ago in un pagliaio. A tal fine, abbiamo sviluppato un saggio fenotipico basato sul rilevamento di Mtb etichettate fluorescentmente all'interno di cellule ospiti etichettate fluorescentmente utilizzando la microscopia confocale automatizzata. Questo saggio in vitro consente una quantificazione basata sull'immagine del processo di colonizzazione della Mtb nell'ospite ed è stato ottimizzato per il formato micropiatta 384-well, che è corretto per schermi di siRNA-, composto chimico- o mtb mutante-librerie. Le immagini vengono quindi elaborate per l'analisi multiparametrica, che fornisce l'inferenza di lettura sulla patogenesi della Mtb all'interno delle cellule ospiti.
Introduzione
Tra gli agenti patogeni infettivi emergenti e riemergendo segnalati negli ultimi anni, Mycobacterium tuberculosis (Mtb) occupa un posto di primo piano essendo responsabile di 1,4 milioni di morti e 8,7 milioni di nuove infezioni nel 2011 (Global tuberculosis report 2012, www.who.int/topics/tuberculosis/en/). Nonostante la disponibilità di terapie multifarmaco, il numero di persone infette è ancora in aumento e la mtb multifarmaco resistente (MDR) e ampiamente resistente ai farmaci (XDR) si sta rapidamente diffondendo in tutto ilmondo 1. Inoltre, quando si prende in considerazione la presenza di antigeni mtb, è evidente che un terzo della popolazione globale è considerato latentemente infettato dalla Mtb. Statisticamente, in un caso su dieci, vi è evoluzione verso la forma attiva della malattia con successivi sintomi clinici2. Pertanto, sono urgentemente necessari nuovi mezzi per combattere la Mtb. In questo contesto, abbiamo sviluppato un saggio fenotipico visivo in vitro basato sul monitoraggio dell'invasione e della moltiplicazione delle Mtb nelle cellule ospiti mediante microscopia a fluorescenza confocale automatizzata3. L'adattamento del saggio in piastre microtiter a 384 pozzi in combinazione con l'acquisizione e l'analisi automatizzate delle immagini, ha permesso lo screening ad alto contenuto / alta produttività (HC / HTS) di librerie su scala media di composti, siRNA e mutanti batterici. Lo screening di una libreria RNAi a livello genomico su questo saggio fenotipico ha quindi permesso l'identificazione dei principali fattori ospiti coinvolti nel traffico di Mtb e nella replicazione intracellulare, ma anche la spiegazione delle vie ospiti sfruttate dal bacillo tubercolare. Un altro adattamento di questo particolare saggio fenotipico è stato per l'identificazione di fattori batterici essenziali per la persistenza intrafagosomale della Mtb. Ad esempio, l'arresto della maturazione fagoma è considerato uno dei principali meccanismi che facilita la sopravvivenza e la replicazione della Mtb nel macrofago. Il monitoraggio della localizzazione subcellulare dei mutanti knock-out mtb in compartimenti acido-etichettati fluorescentmente ha permesso l'identificazione di geni batterici coinvolti nel processo di sopravvivenza4. Infine, l'imaging ad alto contenuto di Mtb offre anche un metodo eccellente per quantificare l'efficienza del farmaco per inibire vari fenomeni come la crescita batterica intracellulare3. Complessivamente, questo tipo di saggio fenotipico ad alta produttività consente di accelerare la scoperta di farmaci contro la TBC e i dati raccolti da questi diversi approcci contribuiscono a una migliore comprensione della manipolazione dell'ospite esercitata da Mtb.
Protocollo
1. Screening del siRNA a livello genomico ad alta produttività
Screening eseguito in una linea cellulare umana di pneumociti di tipo II modello A549 in caso di infezione da Mtb H37Rv che esprime proteine fluorescenti verdi (GFP). Questa procedura è descritta nella figura 1A.
- Rimostrare la libreria di siRNA essiccata che viene conservata in piastre madre (piastre a 96 pozzoli) con tampone di siRNA 1x per raggiungere una concentrazione di 4 μM, quindi trasferire 10 μl della miscela in una piastra figlia di 384 po '(piastra figlia 1).
- Aggiungere 10 μl di tampone di siRNA 1x nella piastra figlia 1 per diluire il siRNA di 2 volte. Dopo la resopensione del siRNA, le piastre sono sigillate con guarnizione in alluminio sbucciabile e possono essere conservate a -20 °C almeno 6 mesi e fino a 2-3 anni, ma il tempo di conservazione può variare a seconda delle raccomandazioni del produttore della libreria siRNA.
- Diluire i siRNA nella piastra figlia 1 nella piastra figlia 2 per raggiungere una concentrazione di 500 nM. Dopo la resopensione del siRNA, le piastre sono sigillate con guarnizione in alluminio sbucciabile e possono essere conservate a -20 °C almeno 6 mesi e fino a 2-3 anni, ma il tempo di conservazione può variare a seconda delle raccomandazioni del produttore della libreria siRNA.
- Prima dell'uso, scongelare la piastra figlia 2 a temperatura ambiente.
- Prendere 2,5 μl di siRNA dalla piastra figlia 2 e posizionare in una piastra di dosaggio di 384.
- Nella stessa piastra di dosaggio di 384 pozzi nel passaggio 1.5, aggiungere 2,5 μl di siRNA di controllo negativo e positivo ai rispettivi pozzi.
- Diluire il reagente di trasfezione in 1x D-PBS per produrre una soluzione sufficiente a fornire un reagente di trasfezione da 0,1 μl in ogni pozzo e preincubare la soluzione di trasfezione diluita a temperatura ambiente per 5 minuti.
- Aggiungere 7,5 μl della miscela di soluzione reagente di trasfezione/PBS ad ogni pozzo nella piastra di dosaggio e incubare per 30 minuti a temperatura ambiente.
- Aggiungere 40 μl di cellule A549 (1.500 cellule/pozzo) sospese in mezzo RPMI 1640 integrato con siero bovino fetale al 10% (FBS). Mantenere le cellule per un periodo di incubazione di 3 giorni a 37 °C in un'atmosfera contenente il 5% di CO2. Queste cellule si dividono ogni 24 ore, quindi circa 12.000 cellule si trovano nei pozzi tre giorni dopo la trasfezione.
- Lavare una coltura mtb H37Rv di due settimane con D-PBS (privo di MgCl2 e CaCl2)con centrifugazione a 4.000 x g per 5 minuti e scartare i lavaggi. Ripetere questo passaggio 3x. (Per le condizioni di coltura GFP-Mtb H37Rv vedere protocollo 3).
- Sospendere il pellet batterico in 10 ml di mezzo RPMI 1640 integrato con FBS al 10% e decantare per 1 ora a temperatura ambiente per consentire aggregati batterici ai sedimenti.
- Raccogliere il supernatante batterico e misurare OD600 (OD600 deve essere compreso tra 0,6-0,8) e GFP-fluorescenza (valore RFU) utilizzando un lettore di micropiacche per determinare la concentrazione batterica. Calcolare il carattere della sospensione utilizzando una linea di regressione di riferimento che mostra il valore RFU = f (valore CFU) generato prima dell'esperimento su un'altra coltura preparata nelle stesse condizioni. Preparare sospensioni batteriche contenenti 2,4 x 106 batteri/ml, che corrisponde a una molteplicità di infezione (MOI) di 5.
- Rimuovere il mezzo nella piastra di dosaggio da 384 e aggiungere 25 μl di sospensione batterica preparata al momento.
- Incubare la piastra di dosaggio a 384°C a 37 °C per 5 ore in un'atmosfera contenente il 5% di CO2.
- Rimuovere il mezzo e lavare delicatamente le celle con mezzo RPMI integrato con 10% FBS 3x.
- Per uccidere i restanti batteri extracellulari, trattare le cellule con 50 μl di mezzo RPMI-FBS fresco contenente 50 μg/ml di aikacina a 37 °C per 1 ora in un'atmosfera contenente il 5% di CO2.
- Rimuovere l'amikacina contenente mezzo e aggiungere 50 μl di mezzo RPMI fresco integrato con FBS al 10%. Incubare la piastra di dosaggio a 384°C a 37 °C per 5 giorni in un'atmosfera contenente il 5% di CO2.
- Prima dell'acquisizione dell'immagine, aggiungere 10 μl di 30 μg/ml di DAPI appena preparati in PBS (concentrazione finale 5 μg/ml) e incubare per 10 min a 37 °C.
- Caricare la piastra nel microscopio confocale automatizzato.

Clicca qui per visualizzare l'immagine più grande. - Impostare i parametri di esposizione. Registrare la fluorescenza DAPI utilizzando il laser di eccitazione 405 nm con filtro di emissione 450 nm e fluorescenza GFP utilizzando laser di eccitazione 488 nm con filtro di emissione 520 nm.
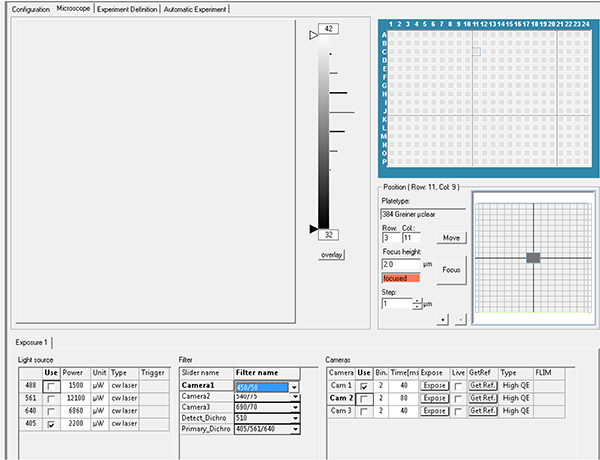
Clicca qui per visualizzare l'immagine più grande.
Clicca qui per visualizzare l'immagine più grande. - Selezionare i pozzi e i campi in ogni pozzo da acquisire, che viene quindi definito parametri di layout e sottolayout.
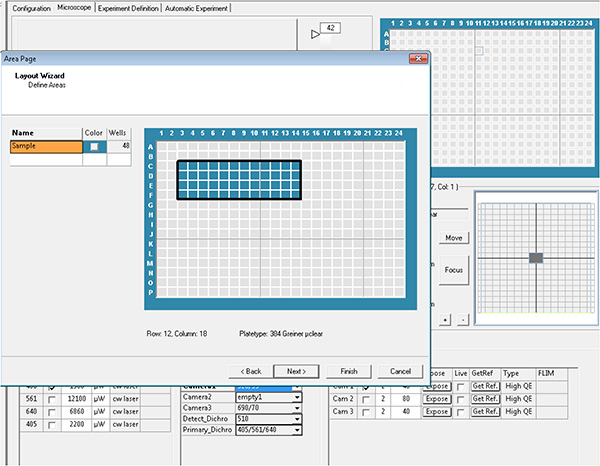
Clicca qui per visualizzare l'immagine più grande.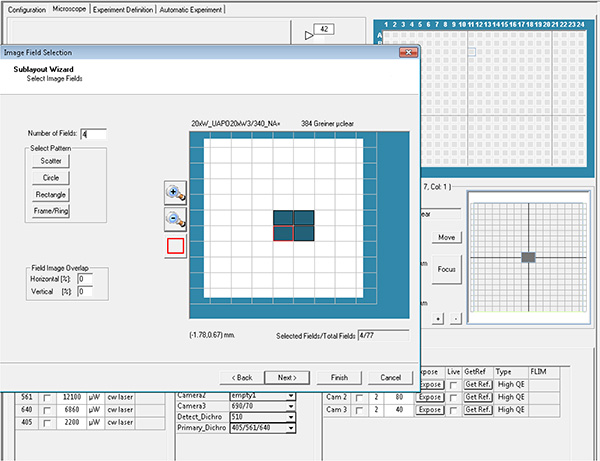
Clicca qui per visualizzare l'immagine più grande. - Generare il file di esperimento utilizzando i parametri dei passaggi 1.20 e 1.21 ed eseguire l'acquisizione automatica.
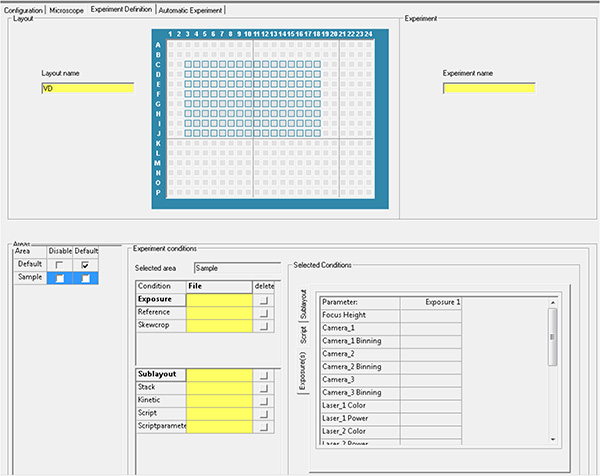
Clicca qui per visualizzare l'immagine più grande.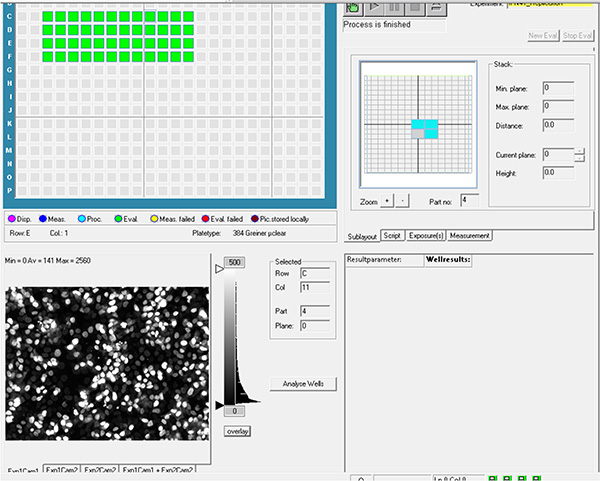
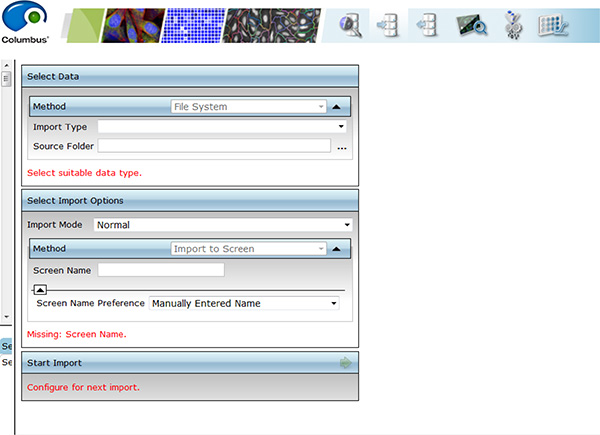
Clicca qui per visualizzare l'immagine più grande. - Trasferire immagini su un server remoto.
Clicca qui per visualizzare l'immagine più grande. - Valutare le immagini utilizzando il software di analisi delle immagini. Rilevare i nuclei cellulari dal canale DAPI utilizzando l'algoritmo di rilevamento dei nuclei e l'area batterica dal canale GFP utilizzando l'algoritmo delle proprietà di intensità dei pixel (Figura 4A).
Nota: Questo protocollo è ottimizzato per studiare l'effetto del silenziamento genico sulla crescita intracellulare della Mtb. Mtb è un batterio a crescita lenta che si divide ogni 20 ore in condizioni ottimali. Dopo 5 giorni dopo l'infezione la quantità di Mtb extracellulare è ancora bassa in assenza di lisi cellulare e non ha influito sulla qualità dell'analisi. Questo protocollo deve essere ottimizzato in termini di durata del trattamento antibiotico e tempo di incubazione da adattare per gli schermi siRNA utilizzando batteri a crescita rapida come Mycobacteria smegmatis ed Escherichia coli che vengono ampiamente rilasciati e possono infettare nuove cellule.
2. Screening composto ad alta produttività
Screening eseguito su cellule ospiti infette mtb H37Rv. Questa procedura è descritta nella figura 1B.
- Scongelare le piastre materne da 384 pozzi contenenti la libreria composta solubilizzata in DMSO al 100%. Trasferire 0,5 μl dei composti in piastre figlie da 384 pozzi contenenti 10 μl di mezzo RPMI 1640 integrato con FBS al 10%.
- Lavare una coltura mtb H37Rv di due settimane con D-PBS (privo di MgCl2 e CaCl2)con centrifugazione a 4.000 x g per 5 minuti e scartare i lavaggi. Ripetere questo passaggio 3x (per le condizioni di colturaGFP- Mtb H37Rv vedere protocollo 3).
- Sospendere il pellet batterico in 10 ml di mezzo RPMI 1640 integrato con FBS al 10% e decantare per 1 ora a temperatura ambiente per consentire aggregati batterici ai sedimenti.
- Raccogliere il supernatante batterico e misurare OD600 (OD600 deve essere compreso tra 0,6-0,8) e GFP-fluorescenza (valore RFU) utilizzando un lettore di micropiatta. Calcolare il carattere della sospensione utilizzando una linea di regressione di riferimento che visualizza il valore RFU = f (valore CFU) generato prima dell'esperimento. La concentrazione tipica è di 1 x 108 batteri/ml.
- Raccogliere macrofagi umani primari di 6 giorni a 4 x 105 cellule/ml in mezzo RPMI 1640 integrato con siero bovino fetale al 10% (FBS) e M-CSF umano ricombinante da 50 ng/ml (per la purificazione delle cellule monocite del sangue periferico umano e differenziazione dei macrofagi vedi protocollo 4).
- Incubare le cellule primarie diluite con bacilli a diversi MOI, che vanno da 1 a 5, in sospensione con lievi scosse a 90 giri/min per 2 ore a 37 °C.
- Lavare le cellule infette per centrifugazione a 350 x g per rimuovere i batteri extracellulari. Dopo ogni fase di centrifugazione, rimorsi il pellet in mezzo RPMI 1640 integrato con FBS al 10%. Ripetere questo passaggio 2x.
- Sospendere le cellule infette in mezzo RPMI 1640 integrato con FBS al 10% e amikacina a 50 μg/ml e incubare la sospensione con lievi scosse per 1 ora a 37 °C.
- Rimuovere il mezzo di coltura cellulare contenente amikacina mediante centrifugazione a 350 x g e lavare le cellule infette con un mezzo RPMI 1640 completo integrato con FBS al 10% e M-CSF umano ricombinante da 50 ng/ml. Ripeti una volta.
- Aggiungere 40 μl della sospensione dei macrofagi infetti nella stessa piastra di dosaggio nel passaggio 2.1, che contiene già 10 μl delle diluizioni composte. La concentrazione finale di DMSO in ogni pozzo ha ora raggiunto l'1%.
- Incubare le piastre di dosaggio per 5 giorni a 37 °C in un'atmosfera contenente il 5% di CO2.
- Macchiare le cellule vive con colorante fluorescente far-red permeante alle cellule.
- Caricare la piastra nel microscopio confocale automatizzato.

Clicca qui per visualizzare l'immagine più grande. - Impostare i parametri di esposizione. Registrare la fluorescenza far-red utilizzando il laser di eccitazione 640 nm con filtro di emissione 690 nm e fluorescenza GFP utilizzando laser di eccitazione 488 nm con filtro di emissione 520 nm.

Clicca qui per visualizzare l'immagine più grande.
Clicca qui per visualizzare l'immagine più grande. - Selezionare i pozzi e i campi in ogni pozzo da acquisire, che viene quindi definito parametri di layout e sottolayout.

Clicca qui per visualizzare l'immagine più grande.
Clicca qui per visualizzare l'immagine più grande. - Generare il file di esperimento utilizzando i parametri dei passaggi 2.14 e 2.15 ed eseguire l'acquisizione automatica.

Clicca qui per visualizzare l'immagine più grande.
Clicca qui per visualizzare l'immagine più grande. - Trasferire immagini su un server remoto.

Clicca qui per visualizzare l'immagine più grande. - Valutare le immagini utilizzando il software di analisi delle immagini. Rilevare l'area cellulare dal canale rosso lontano utilizzando l'algoritmo delle proprietà dell'intensità dei pixel e l'area batterica dal canale GFP utilizzando l'algoritmo delle proprietà di intensità dei pixel (Figura 4B).
Nota: Questo protocollo può essere adattato per lo screening della libreria mutante mtb sostituendo i composti con mutanti che esprimono una proteina fluorescente (un mutante bene/uno) (Figura 1C, vedi anche Brodin et al. 4). I mutanti fluorescenti vengono seminati per la prima volta in pozzi (20 μl di sospensione batterica per pozzo). I batteri vengono quindi recuperati di 30 μl di sospensione cellulare. Dopo la centrifugazione a 350 x g per 1 min, la piastra viene incubata a 37 °C in un'atmosfera contenente il 5% di CO2. Il tempo di incubazione e il MOI dipendono dal saggio. Ad esempio, per la visualizzazione dei primi eventi cellulari come l'acidificazione fagosoma, le cellule possono essere infettate per 2 ore con MOI che vanno da 1 a 20. I lisosomi sono macchiati usando colorante Lysotracker a 2 μM per 1,5 ore a 37 °C in un'atmosfera contenente il 5% di CO2 e quindi fissati con formalina al 10% o paraformaldeide al 4% (PFA). Le immagini confocali vengono acquisite e infine analizzate utilizzando script di analisi delle immagini con algoritmi appropriati per il rilevamento dei lisosomi e la localizzazione subcellulare4.
3. Proteina fluorescente verde che esprime mycobacterium tuberculosis H37Rv (GFP-H37Rv) Condizioni di coltura
Per la conservazione a lungo termine, GFP-H37Rv è stato congelato in D-PBS (circa 1 x 108 micobatteri per flaconcino).
- Resuspend una fiala congelata di GFP-H37Rv in un matraccio Erlenmeyer contenente 50 ml di mezzo brodo 7H9 integrato con arricchimento Middlebrook OADC 10%, Glicerolo 0,5%, Tween-80 0,05% e Igromicina B (50 μg/ml).
- Incubare 8 giorni a 37 °C.
- Misurare l'OD600 della cultura GFP-H37Rv.
- Diluire la coltura GFP-H37Rv per ottenere OD600 = 0,1 in mezzo brodo fresco 7H9 integrato con arricchimento Middlebrook OADC 10%, glicerolo 0,5%, Tween-80 0,05% e igromicina B (50 μg/ml).
- Incubare il GFP-H37Rv a 37 °C per altri 8 giorni prima dell'uso per il saggio.
4. Purificazione umana delle cellule monocite del sangue periferico dall'intero sangue o dalla preparazione del mantello di buffy
- Diluire la busta di sangue 2x in 1x D-PBS (privo di MgCl2 e CaCl2) contenente 1% FBS.
- Isolare i monociti per centrifugazione gradiente densità Ficoll a 400 x g per 20 min.
- Raccogli i monociti isolati.
- Lavare i monociti 3x con 1x D-PBS (privo di MgCl2 e CaCl2)contenente 1% FBS per centrifugazione a 400 x g per 10 min a temperatura ambiente.
- Concentrare le cellule fino a 1 x 107 cellule/ml.
- Purificare i monociti utilizzando perline magnetiche CD14 secondo il protocollo del produttore (vedi Materiali).
- Dopo la purificazione dei monociti CD14, seminare le cellule a 1,5 x10 6 cellule/ml in RPMI 1640 integrate con FBS al 10% e 40 ng/ml di fattore stimolante della colonia di macrofagi umani (hM-CSF) e incubate per 4 giorni a 37 °C in un'atmosfera contenente il 5% di CO2.
- Dopo 4 giorni sostituire il mezzo con RPMI fresco 1640 completato con FBS al 10% e 40 ng/ml di hM-CSF e incubato per 2 giorni a 37 °C in un'atmosfera contenente il 5% di CO2.
- Dopo 6 giorni, le cellule possono essere utilizzate per il saggio.
Risultati
Screening del siRNA a livello genomico ad alta produttività
Mtb è in grado di colonizzare le cellule immunitarie in vitro così come molte altre cellule epiteliali polmonari. Ad esempio, Mtb èin grado di infettare e danneggiare le cellule epiteliali A549 che sono comunemente utilizzate come modello per gli pneumociti di tipo IIumano 5-7. La dectina-1 è stata segnalata come recettore delle cellule ospiti coinvolto nell'assorbimento della mtb, nell...
Discussione
Descriviamo qui i metodi necessari per un saggio fenotipico utilizzando un ceppo Mtb H37Rv che esprime GFP per infettare le cellule ospiti etichettate fluorescentmente, il che lo rende appropriato per schermi ad alto contenuto / ad alta produttività. Questo protocollo potrebbe essere applicato a una vasta gamma di composti, sonde fluorescenti e mutanti Mtb. Per ogni protocollo sopra descritto, è possibile eseguire passaggi di fissazione e immunoetichettatura prima dell'acquisizione dell'immagine. Util...
Divulgazioni
Nessun conflitto di interessi dichiarato.
Riconoscimenti
Il sostegno finanziario a questo lavoro è stato fornito dalla Comunità europea (sovvenzione ERC-STG INTRACELLTB n° 260901, sovvenzione MM4TB n° 260872), dall'Agence Nationale de Recherche, dalla Feder (12001407 (D-AL) Equipex Imaginex BioMed) e dalla regione Nord Pas de Calais. Riconosciamo con gratitudine l'assistenza tecnica di Gaspard Deloison, Elizabeth Werkmeister, Antonino Bongiovanni e Frank Lafont dalla piattaforma BICeL.
Materiali
| Name | Company | Catalog Number | Comments |
| µclear-plate black, 384-well | Greiner Bio-One | 781091 | 127.8/86/15 MM with Lid, TC treated |
| CellCarrier 384-well plate | PerkinElmer | 6007550 | Black, Clear Bottom, with Lid, TC treated |
| V-bottom white, 384-well plate | Greiner Bio-One | 781280 | |
| sealing tape, breathable, sterile | Corning | 3345 | |
| Lipofectamine RNAiMax | Life Technologies | 13778150 | Transfection reagent |
| Dimethyl sulfoxide | Sigma-Aldrich | 34943 | |
| RPMI 1640 + GlutaMAX-I | Life Technologies | 61870-010 | Cell culture medium |
| D-PBS 1x [-]MgCl2/[-]CaCl2 | Life Technologies | 14190-094 | Dulbecco's Phosphate Salin Buffer |
| D-PBS 1x [+]MgCl2/[+]CaCl2 | Life Technologies | 14190-091 | Dulbecco's Phosphate Salin Buffer |
| Fetal bovine serum | Life Technologies | 2610040-79 | |
| Ficoll Paque PLUS | Dutscher | 17-1440-03 | Ficoll for Peripherical Blood Monocyte Cells purification |
| CD14 MicroBeads, human | Miltenyi | 130-050-201 | Purification of CD14+ Monocytes |
| Human M-CSF, premium gr. (1000 μg) | Miltenyi | 130-096-493 | Macrophage Colony Stimulating Factor |
| LS Columns | Miltenyi | 130-042-401 | Columns for CD14+ Monocytes isolation |
| Tween 80 | Euromedex | 2002-A | Mycobacteria culture |
| Glycerol high purity | Euromedex | 50405-EX | Mycobacteria culture |
| Middlebrook OADC enrichment | Becton-Dickinson | 211886 | Mycobacteria culture |
| 7H9 | Becton-Dickinson | W1701P | Mycobacteria culture |
| Versene 1x | Life Technologies | 15040033 | Nonenzymatic cell dissociation solution |
| DAPI | Life Technologies | D1306 | Nuclei dye |
| Hoechst 33342 | Life Technologies | H3570 | Nuclei dye |
| Syto60 | Life Technologies | S11342 | Nuclei/cytoplasm dye |
| Formalin | Sigma-Aldrich | HT5014 | Cell fixation solution |
| siRNA targeting Dectin-1 | Santa-Cruz | sc-63276 | |
| *siGenome* Nontargeted siRNA pool | Dharmacon | D-001206-14 | |
| Rifampicin | Sigma-Aldrich | R3501 | antibiotic |
| Isoniazid (INH) | Sigma-Aldrich | I3377-50G | antibiotic |
| Hygromycin B | Life Technologies | 10687-010 | antibiotic |
| Amikacin | Sigma-Aldrich | A1774 | antibiotic |
| Automated Confocal Microscope OPERA | PerkinElmer | Image acquisition | |
| Columbus 2.3.1 Server Database | PerkinElmer | Data transfer, storage, and analysis | |
| Acapella 2.6 software | PerkinElmer | Image-based analysis | |
| GraphPad Prism5 software | GraphPad | Statistical analysis | |
| Excel 2010 | Microsoft | Statistical analysis |
Riferimenti
- Gandhi, N. R., et al. Multidrug-resistant and extensively drug-resistant tuberculosis: a threat to global control of tuberculosis. Lancet. 375, 1830-1843 (2010).
- Barry, C. E., et al. The spectrum of latent tuberculosis: rethinking the biology and intervention strategies. Nat. Rev. Microbiol. 7, 845-855 (2009).
- Christophe, T., Ewann, F., Jeon, H. K., Cechetto, J., Brodin, P. High-content imaging of Mycobacterium tuberculosis-infected macrophages: an in vitro model for tuberculosis drug discovery. Future Med. Chem. 2, 1283-1293 (2010).
- Brodin, P., et al. High content phenotypic cell-based visual screen identifies Mycobacterium tuberculosis acyltrehalose-containing glycolipids involved in phagosome remodeling. PLoS Pathog. 6, (2010).
- Bermudez, L. E., Goodman, J. Mycobacterium tuberculosis invades and replicates within type II alveolar cells. Infect. Immun. 64, 1400-1406 (1996).
- Dobos, K. M., Spotts, E. A., Quinn, F. D., King, C. H. Necrosis of lung epithelial cells during infection with Mycobacterium tuberculosis is preceded by cell permeation. Infect. Immun. 68, 6300-6310 (2000).
- McDonough, K. A., Kress, Y. Cytotoxicity for lung epithelial cells is a virulence-associated phenotype of Mycobacterium tuberculosis. Infect. Immun. 63, 4802-4811 (1995).
- Lee, H. M., Yuk, J. M., Shin, D. M., Jo, E. K. Dectin-1 is inducible and plays an essential role for mycobacteria-induced innate immune responses in airway epithelial cells. J. Clin. Immunol. 29, 795-805 (2009).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J. Biomol. Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat. Methods. 6, 569-575 (2009).
- Carralot, J. P., et al. Automated high-throughput siRNA transfection in raw 264.7 macrophages: a case study for optimization procedure. J. Biomol. Screen. 14, 151-160 (2009).
- Zhang, X. D. Illustration of SSMD, z score, SSMD*, z* score, and t statistic for hit selection in RNAi high-throughput screens. J. Biomol. Screen. 16, 775-785 (2011).
- Song, O. R., et al. Confocal-based method for quantification of diffusion kinetics in microwell plates and its application for identifying a rapid mixing method for high-content/throughput screening. J. Biomol. Screen. 15, 138-147 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon