Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein mikroskopischer phänotypischer Assay zur Quantifizierung intrazellulärer Mykobakterien angepasst für High-Throughput/High-Content Screening
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier beschreiben wir einen phänotypischen Assay, der auf die High-Throughput/High-Content-Screens von klein-interferierender synthetischer RNA (siRNA), chemischer Verbindung und Mycobacterium tuberculosis mutierte Bibliotheken anwendbar ist. Diese Methode beruht auf dem Nachweis von fluoreszierend gekennzeichneter Mycobacterium tuberculosis in fluoreszierend markierten Wirtszellen mittels automatisierter konfokaler Mikroskopie.
Zusammenfassung
Trotz der Verfügbarkeit von Therapie und Impfstoff ist Tuberkulose (TB) nach wie vor eine der tödlichsten und am weitesten verbreiteten bakteriellen Infektionen der Welt. Seit mehreren Jahrzehnten stellt der plötzliche Ausbruch multi- und extensiv-resistenter Stämme eine ernsthafte Bedrohung für die Bekämpfung der Tuberkulose dar. Daher ist es wichtig, neue Ziele und Wege zu identifizieren, die für den Erreger der Tuberkulose, Mycobacterium tuberculosis (Mtb) entscheidend sind, und nach neuartigen Chemikalien zu suchen, die zu TB-Medikamenten werden könnten. Ein Ansatz besteht darin, Methoden einzurichten, die für die genetischen und chemischen Siebs von Großbibliotheken geeignet sind und die Suche nach einer Nadel in einem Heuhaufen ermöglichen. Zu diesem Zweck entwickelten wir einen phänotypchen assay, der sich auf den Nachweis von fluoreszierend beschriftetem Mtb in fluoreszierend gekennzeichneten Wirtszellen mittels automatisierter konfokaler Mikroskopie stützt. Dieser In-vitro-Assay ermöglicht eine bildbasierte Quantifizierung des Kolonisationsprozesses von Mtb in den Wirt und wurde für das 384-Well-Mikroplattenformat optimiert, das für Siebe von siRNA-, chemischen Compound- oder Mtb-Mutationsbibliotheken passt. Die Bilder werden dann für die multiparametrische Analyse verarbeitet, die eine Auslesung der Pathogenese von Mtb in Wirtszellen liefert.
Einleitung
Unter den in den letzten Jahren gemeldeten neu auftretenden und wieder auftauchenden infektionserösen Krankheitserregern ist Mycobacterium tuberculosis (Mtb) für 1,4 Millionen Todesfälle und 8,7 Millionen Neuinfektionen im Jahr 2011 verantwortlich (Global tuberculosis report 2012, www.who.int/topics/tuberculosis/en/). Trotz der Verfügbarkeit von Multidrug-Therapien ist die Zahl der Infizierten immer noch auf dem Vormarsch und multiresistent (MDR) sowie extensiv medikamentenresistent (XDR) Mtb breiten sich schnell auf der ganzen Welt aus1. Darüber hinaus ist es unter Berücksichtigung des Vorhandenseins von Mtb-Antigenen offensichtlich, dass ein Drittel der Weltbevölkerung als latent von Mtb infiziert angesehen wird. Statistisch gesehen gibt es in einem von zehn Fällen eine Entwicklung hin zur aktiven Form der Krankheit mit nachfolgenden klinischen Symptomen2. Daher sind neue Mittel zur Bekämpfung von Mtb dringend erforderlich. In diesem Zusammenhang entwickelten wir einen in vitro visuellen phänotypchen Assay, der sich auf die Überwachung der Mtb-Invasion und -Multiplikation in Wirtszellen durch automatisierte konfokale Fluoreszenzmikroskopie3stützt. Die Anpassung des Assays in 384-Well-Mikrotiterplatten in Kombination mit automatisierter Bildaufnahme und -analyse ermöglichte High-Content/High-Throughput Screening (HC/HTS) von mittelgroßen Bibliotheken von Verbindungen, siRNAs und bakteriellen Mutanten. Das Screening einer genomweiten RNAi-Bibliothek auf diesem phänotypischen Assay ermöglichte somit die Identifizierung der wichtigsten Wirtsfaktoren, die am Mtb-Handel und der intrazellulären Replikation beteiligt sind, aber auch die Aufklärung von Wirtspfaden, die vom Tuberkel-Bacillus ausgenutzt werden. Eine weitere Anpassung dieses speziellen phänotypischen Assays war die Identifizierung von bakteriellen Faktoren, die für die intraphagosomale Persistenz von Mtb unerlässlich sind. Zum Beispiel wird die Verhaftung der Phagosomenreifung als einer der wichtigsten Mechanismen betrachtet, die das Überleben und die Replikation von Mtb in Makrophagen erleichtern. Die Überwachung der subzellulären Lokalisation vonMtb-Knock-out-Mutanten in fluoreszierend-säuerlichen Kompartimenten ermöglichte die Identifizierung von bakteriellen Genen, die am Überlebensprozess beteiligt sind 4 . Schließlich bietet die hochklassige Bildgebung von Mtb auch eine ausgezeichnete Methode zur Quantifizierung der Arzneimitteleffizienz zur Hemmung verschiedener Phänomene wie intrazelluläres Bakterienwachstum3. Insgesamt ermöglicht diese Art von hochdurchsatz-phänotypischem Assay eine beschleunigte Arzneimittelentdeckung gegen TB und die von diesen verschiedenen Ansätzen gesammelten Daten tragen zu einem besseren Verständnis der von Mtbausgeübten Host-Manipulation bei.
Protokoll
1. High-Throughput Genom-wide siRNA Screening
Screening durchgeführt in einem menschlichen Typ-II-Pneumozyten Modell A549 Zelllinie nach einer Infektion mit Mtb H37Rv, die grünes fluoreszierendes Protein (GFP) exzessiert. Dieses Verfahren ist in Abbildung 1Abeschrieben.
- Setzen Sie die getrocknete siRNA-Bibliothek, die in Mutterplatten (96-Well-Platten) mit 1x siRNA-Puffer gelagert wird, auf eine Konzentration von 4 m auf, und übertragen Sie dann 10 l des Gemischs in eine 384-Well-Tochterplatte (Tochterplatte 1).
- Fügen Sie 10 l 1x 1x siRNA Puffer in Tochterplatte 1 hinzu, um siRNA um das 2-fache zu verdünnen. Nach der siRNA-Resuspension werden platten mit abziehbarer Aluminiumdichtung versiegelt und können bei -20 °C mindestens 6 Monate und bis zu 2-3 Jahren gelagert werden, aber die Lagerzeit kann je nach den Empfehlungen des Herstellers der siRNA-Bibliothek variieren.
- SiRNAs in Tochterplatte 1 in Tochterplatte 2 verdünnen, um eine Konzentration von 500 nM zu erreichen. Nach der siRNA-Resuspension werden platten mit abziehbarer Aluminiumdichtung versiegelt und können bei -20 °C mindestens 6 Monate und bis zu 2-3 Jahren gelagert werden, aber die Lagerzeit kann je nach den Empfehlungen des Herstellers der siRNA-Bibliothek variieren.
- Vor Gebrauch auftauen Tochterplatte 2 bei Raumtemperatur.
- Nehmen Sie 2,5 l siRNA von Tochterplatte 2 und legen Sie sie in eine 384-Well-Assay-Platte.
- In der gleichen 384-Well-Assay-Platte in Schritt 1.5 fügen Sie ihren jeweiligen Brunnen 2,5 l negative und positive KontrollsiRNA hinzu.
- Verdünnen Sie das Transfektionsreagenz in 1x D-PBS, um genügend Lösung zu liefern, um 0,1 l Transfektionsreagenz in jedem Brunnen bereitzustellen, und die verdünnte Transfektionslösung bei Raumtemperatur für 5 min vorinkubieren.
- Fügen Sie jedem Brunnen in der Assayplatte 7,5 l des Transfektionsreagenz/PBS-Lösungsgemisches hinzu und brüten Sie 30 min bei Raumtemperatur.
- Fügen Sie 40 l A549-Zellen (1.500 Zellen/well) hinzu, die in RPMI 1640 Medium suspendiert sind, ergänzt mit 10% fetalem Rinderserum (FBS). Halten Sie Zellen für eine 3-tägige Inkubationszeit bei 37 °C in einer Atmosphäre, die 5%CO2enthält. Diese Zellen teilen sich alle 24 Stunden, so dass sich etwa 12.000 Zellen drei Tage nach der Transfektion in den Brunnen befinden.
- Waschen Sie eine zwei Wochen alte GFP-ausdruckende Mtb H37Rv Kultur mit D-PBS (frei von MgCl2 und CaCl2) durch Zentrifugation bei 4.000 x g für 5 min und entsorgen Sie die Wäsche. Wiederholen Sie diesen Schritt 3x. (Für GFP-Mtb H37Rv Kulturbedingungen siehe Protokoll 3).
- Setzen Sie das bakterielle Pellet in 10 ml RPMI 1640 Medium, ergänzt mit 10% FBS und Dekanat für 1 Stunde bei Raumtemperatur, um bakterielle Aggregate sedimentieren zu lassen.
- Sammeln Sie den bakteriellen Überstand und messen Sie OD600 (OD600 sollte zwischen 0,6-0,8 liegen) und GFP-Fluoreszenz (RFU-Wert) mit einem Mikroplattenleser, um die bakterielle Konzentration zu bestimmen. Berechnen Sie den Titer der Suspension mit einer Referenzregressionslinie, die den RFU-Wert = f (CFU-Wert) anzeigt, der vor dem Experiment an einer anderen Kultur generiert wurde, die unter den gleichen Bedingungen vorbereitet worden war. Bereiten Sie bakterielle Suspension mit 2,4 x 106 Bakterien/ml vor, was einer Infektionsvielzahl (MOI) von 5 entspricht.
- Entfernen Sie das Medium in der 384-Well-Assay-Platte und fügen Sie 25 l frisch zubereitete bakterielle Suspension hinzu.
- Inkubieren Sie die 384-Well-Assayplatte bei 37 °C für 5 Stunden in einer Atmosphäre mit 5%CO2.
- Entfernen Sie das Medium und waschen Sie die Zellen vorsichtig mit RPMI Medium ergänzt mit 10% FBS 3x.
- Um die verbleibenden extrazellulären Bakterien abzutöten, behandeln Sie die Zellen mit 50 l frischem RPMI-FBS-Medium, das 50 g/ml Amikacin bei 37 °C für 1 Stunde in einer Atmosphäre mit 5%CO2enthält.
- Entfernen Sie das mittelhaltige Amikacin und fügen Sie 50 l frisches RPMI-Medium hinzu, das mit 10% FBS ergänzt wird. Inkubieren Sie die 384-Well-Assayplatte bei 37 °C 5 Tage lang in einer Atmosphäre, die 5%CO2enthält.
- Vor der Bildaufnahme 10 l frisch zubereitete 30 g/ml DAPI in PBS (Endkonzentration 5 g/ml) hinzufügen und 10 min bei 37 °C inkubieren.
- Laden Sie die Platte in das automatische konfokale Mikroskop.

Klicken Sie hier, um ein größeres Bild anzuzeigen. - Legen Sie die Belichtungsparameter fest. Aufnahme der DAPI-Fluoreszenz mit Anregungslaser 405 nm mit Emissionsfilter 450 nm und GFP-Fluoreszenz mit Anregungslaser 488 nm mit Emissionsfilter 520 nm.
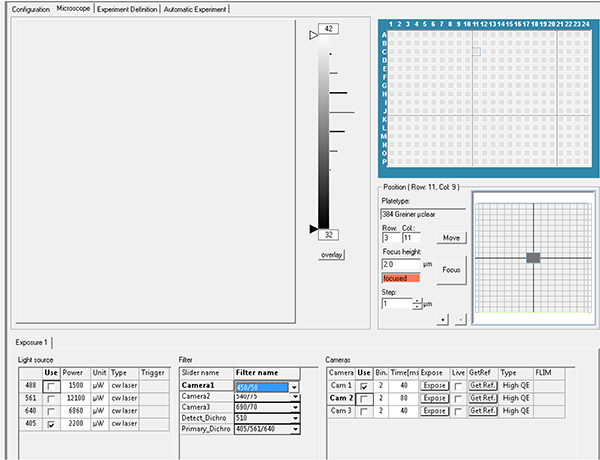
Klicken Sie hier, um ein größeres Bild anzuzeigen.
Klicken Sie hier, um ein größeres Bild anzuzeigen. - Wählen Sie die Brunnen und die Felder in jedem zu erwerbenden Brunnen aus, der dann als Layout- und Unterlayoutparameter bezeichnet wird.
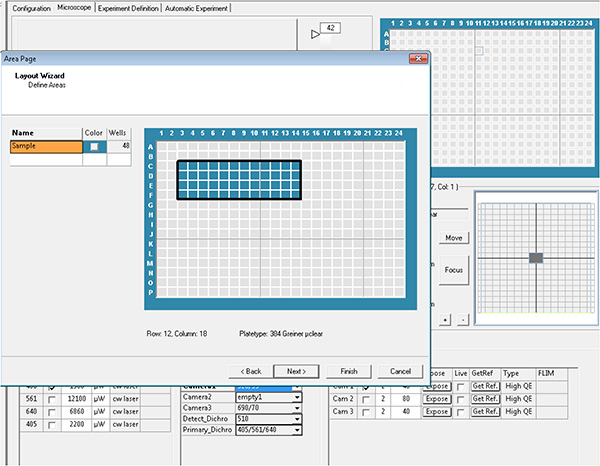
Klicken Sie hier, um ein größeres Bild anzuzeigen.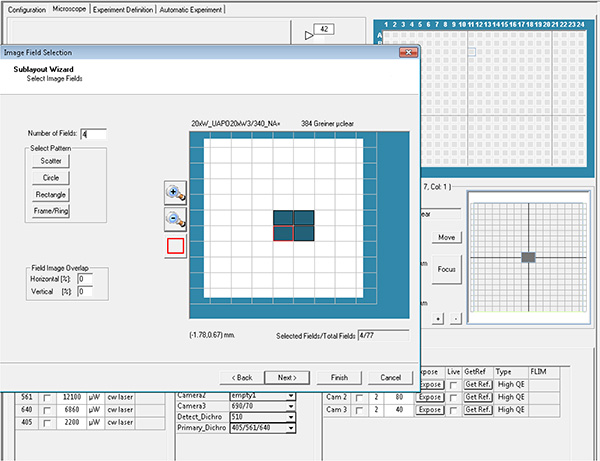
Klicken Sie hier, um ein größeres Bild anzuzeigen. - Generieren Sie die Experimentierdatei mit den Parametern aus den Schritten 1.20 und 1.21, und führen Sie die automatische Erfassung aus.
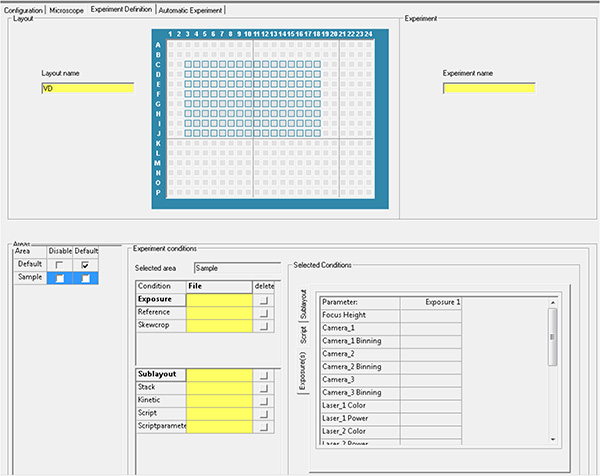
Klicken Sie hier, um ein größeres Bild anzuzeigen.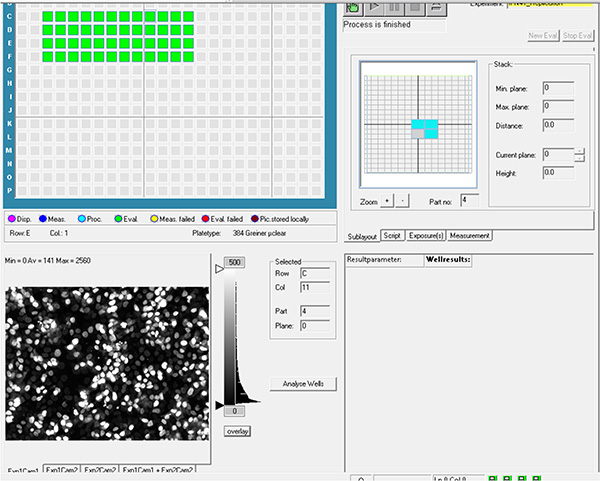
Klicken Sie hier, um ein größeres Bild anzuzeigen. - Übertragen Sie Bilder auf den Remote-Server.
Klicken Sie hier, um ein größeres Bild anzuzeigen. - Bewerten Sie Bilder mit Bildanalysesoftware. Erkennen Sie Zellkerne aus dem DAPI-Kanal mithilfe des Kernerkennungsalgorithmus und des Bakterienbereichs aus dem GFP-Kanal mithilfe des Algorithmus für die Eigenschaften der Pixelintensität (Abbildung 4A).
Hinweis: Dieses Protokoll wurde optimiert, um die Wirkung von Gen-Silencing auf das intrazelluläre Mtb-Wachstum zu untersuchen. Mtb ist ein langsam wachsendes Bakterium, das sich alle 20 Stunden unter optimalen Bedingungen teilt. Nach 5 Tagen nach der Infektion ist die Menge an extrazellulärem Mtb ohne Zelllyse immer noch gering und hatte keinen Einfluss auf die Qualität der Analyse. Dieses Protokoll muss hinsichtlich der Länge der Antibiotikabehandlung und der Inkubationszeit optimiert werden, um für siRNA-Bildschirme mit schnell wachsende Bakterien wie Mykobakterien smegmatis und Escherichia coli angepasst zu werden, die extensiv freigesetzt werden und neue Zellen infizieren können.
2. Hochdurchsatz Compound Screening
Screening an Mtb H37Rv infizierten Wirtszellen durchgeführt. Dieses Verfahren ist in Abbildung 1Bbeschrieben.
- Die 384-Well-Mutterplatten, die die in DMSO zu 100% lösliche zusammengesetzte Bibliothek enthalten, auftauen. Übertragen Sie 0,5 l der Verbindungen in 384-Well-Tochterplatten, die 10 l RPMI 1640 Medium enthalten, ergänzt mit 10% FBS.
- Waschen Sie eine zwei Wochen alte GFP-ausdruckende Mtb H37Rv Kultur mit D-PBS (frei von MgCl2 und CaCl2) durch Zentrifugation bei 4.000 x g für 5 min und entsorgen Sie die Wäsche. Wiederholen Sie diesen Schritt 3x (Für GFP-Mtb H37Rv Kulturbedingungen siehe Protokoll 3).
- Setzen Sie das bakterielle Pellet in 10 ml RPMI 1640 Medium, ergänzt mit 10% FBS und Dekanat für 1 Stunde bei Raumtemperatur, um bakterielle Aggregate sedimentieren zu lassen.
- Sammeln Sie den bakteriellen Überstand und messen Sie OD600 (OD600 sollte zwischen 0,6-0,8 liegen) und GFP-Fluoreszenz (RFU-Wert) mit einem Mikroplattenleser. Berechnen Sie den Titer der Suspension mit einer Referenzregressionslinie, die den RFU-Wert = f (CFU-Wert) anzeigt, der vor dem Experiment generiert wurde. Typische Konzentration beträgt 1 x 108 Bakterien/ml.
- Ernte 6 Tage alte primäre menschliche Makrophagen bei 4 x 105 Zellen/ml in RPMI 1640 Medium ergänzt mit 10% fetalem Rinderserum (FBS) und 50 ng/ml rekombinantem menschlichen M-CSF (Für menschliche periphere Blutmonozytenzellen Reinigung und Makrophagen Differenzierung siehe Protokoll 4).
- Inkubieren Sie die verdünnten Primärzellen mit Bazillen bei verschiedenen MOI, von 1-5, in Suspension mit leichtem Schütteln bei 90 Rprossen für 2 Stunden bei 37 °C.
- Waschen Sie die infizierten Zellen durch Zentrifugation bei 350 x g, um die extrazellulären Bakterien zu entfernen. Nach jedem Zentrifugationsschritt das Pellet in RPMI 1640 medium, ergänzt mit 10% FBS, wieder aufsetzen. Wiederholen Sie diesen Schritt 2x.
- Setzen Sie die infizierten Zellen in RPMI 1640 Medium, ergänzt mit 10% FBS und Amikacin, bei 50 g/ml aus und inkubieren Sie die Suspension mit leichtem Schütteln für 1 Stunde bei 37 °C.
- Entfernen Sie das amikacinhaltige Zellkulturmedium durch Zentrifugation bei 350 x g und waschen Sie die infizierten Zellen mit dem vollständigen RPMI 1640 Medium, ergänzt mit 10% FBS und 50 ng/ml rekombinantem humanem M-CSF. Wiederholen Sie dies einmal.
- Fügen Sie in Schritt 2.1 40 l der infizierten Makrophagensuspension in die gleiche Assayplatte ein, die bereits 10 l der zusammengesetzten Verdünnungen enthält. Die endgültige Konzentration von DMSO in jedem Brunnen hat nun 1% erreicht.
- Inkubieren Sie die Assayplatten 5 Tage lang bei 37 °C in einer Atmosphäre, die 5%CO2enthält.
- Färben Sie die lebenden Zellen mit zelldurchlässigen, weitroten Fluoreszenzfarbstoffen.
- Laden Sie die Platte in das automatische konfokale Mikroskop.

Klicken Sie hier, um ein größeres Bild anzuzeigen. - Legen Sie die Belichtungsparameter fest. Zeichnen Sie die weitrote Fluoreszenz mit Anregungslaser 640 nm mit Emissionsfilter 690 nm und GFP-Fluoreszenz mit Anregungslaser 488 nm mit Emissionsfilter 520 nm auf.

Klicken Sie hier, um ein größeres Bild anzuzeigen.
Klicken Sie hier, um ein größeres Bild anzuzeigen. - Wählen Sie die Brunnen und die Felder in jedem zu erwerbenden Brunnen aus, der dann als Layout- und Unterlayoutparameter bezeichnet wird.

Klicken Sie hier, um ein größeres Bild anzuzeigen.
Klicken Sie hier, um ein größeres Bild anzuzeigen. - Generieren Sie die Experimentierdatei mit den Parametern aus den Schritten 2.14 und 2.15, und führen Sie die automatische Erfassung aus.

Klicken Sie hier, um ein größeres Bild anzuzeigen.
Klicken Sie hier, um ein größeres Bild anzuzeigen. - Übertragen Sie Bilder auf den Remote-Server.

Klicken Sie hier, um ein größeres Bild anzuzeigen. - Bewerten Sie Bilder mit Bildanalysesoftware. Erkennen Sie den Zellbereich aus dem rot-roten Kanal mithilfe des Algorithmus für die Pixelintensitätseigenschaften und des Bakterienbereichs aus dem GFP-Kanal mithilfe des Algorithmus für die Pixelintensitätseigenschaften (Abbildung 4B).
Hinweis: Dieses Protokoll kann für mtb mutierte Bibliothek Screening angepasst werden, indem Verbindungen durch Mutanten, die ein fluoreszierendes Protein (One well/One mutant)(Abbildung 1C, siehe auch Brodin et al. 4). Fluoreszierende Mutanten werden zuerst in Brunnen ausgesät (20 l bakterielle Suspension pro Brunnen). Die Bakterien werden dann durch 30 l Zellsuspension zurückgewonnen. Nach zentrifugieren bei 350 x g für 1 min wird die Platte bei 37 °C in einer Atmosphäre mit 5%CO2inkubiert. Inkubationszeit und MOI hängen vom Test ab. Zum Beispiel für die Visualisierung von frühen zellulären Ereignissen wie Phagosomenversauerung, können die Zellen für 2 Stunden mit MOI im Bereich von 1-20 infiziert werden. Lysosomen werden in einer Atmosphäre mit 5 %CO2-Gehalt mit Lysotracker-Farbstoff mit 2 m bei 1,5 stunden bei 37 °C gefärbt und anschließend entweder mit 10 % Formalin oder 4 % Paraformaldehyd (PFA) fixiert. Konfokale Bilder werden erfasst und schließlich mit Bildanalyseskripten mit geeigneten Algorithmen für die Lysosomenerkennung und subzelluläre Lokalisierung analysiert4.
3. Grünes fluoreszierendes Protein, das Mycobacterium tuberculosis H37Rv (GFP-H37Rv) Kulturbedingungen ausdrückt
Zur Langzeitlagerung wurden GFP-H37Rv in D-PBS eingefroren (ca. 1 x 108 Mykobakterien pro Durchstechflasche).
- Eine gefrorene Durchstechflasche GFP-H37Rv in einem Erlenmeyerkolben mit 50 ml 7H9-Brühenmedium, ergänzt mit Middlebrook OADC-Anreicherung 10%, Glycerin 0,5%, Tween-80 0,05% und Hygromycin B (50 g/ml) wieder aufsetzen.
- 8 Tage bei 37 °C inkubieren.
- Messen Sie OD600 der GFP-H37Rv-Kultur.
- Verdünnen Sie die GFP-H37Rv-Kultur, um OD600 = 0,1 in frischem 7H9-Brühenmedium zu erhalten, ergänzt mit Middlebrook OADC-Anreicherung 10%, Glycerin 0,5%, Tween-80 0,05% und Hygromycin B (50 g/ml).
- Inkubieren Sie den GFP-H37Rv bei 37 °C für 8 Tage mehr vor der Anwendung für den Test.
4. Humane periphere Blutmonozytenzellen Reinigung aus Vollblut oder Buffy-Coat-Präparation
- Verdünnen Sie den Blutbeutel 2x in 1x D-PBS (frei von MgCl2 und CaCl2) mit 1% FBS.
- Isolieren Sie die Monozyten durch Ficoll DichteGradientzentrifugation bei 400 x g für 20 min.
- Sammeln Sie die isolierten Monozyten.
- Waschen Sie die Monozyten 3x mit 1x D-PBS (frei von MgCl2 und CaCl2) mit 1% FBS durch Zentrifugation bei 400 x g für 10 min bei Raumtemperatur.
- Konzentrieren Sie die Zellen auf 1 x 107 Zellen/ml.
- Reinigen Sie Monozyten mit CD14-Magnetperlen gemäß dem Herstellerprotokoll (siehe Materialien).
- Nach der Reinigung von CD14-Monozyten werden die Zellen bei 1,5 x 106 Zellen/ml in RPMI 1640 mit 10% FBS und 40 ng/ml humanem Makrophagen-Koloniestimulierenden Faktor (hM-CSF) ergänzt und 4 Tage lang bei 37 °C in einer Atmosphäre mit 5%CO2inkubiert.
- Nach 4 Tagen das Medium durch frisches RPMI 1640 ersetzen, ergänzt durch 10% FBS und 40 ng/ml hM-CSF und 2 Tage bei 37 °C in einer Atmosphäre mit 5%CO2inkubiert.
- Nach 6 Tagen können die Zellen für den Test verwendet werden.
Ergebnisse
Genom-weites SiRNA-Screening mit hohem Durchsatz
Mtb ist in der Lage, Immunzellen in vitro sowie mehrere andere Lungenepithelzellen zu kolonisieren. Zum Beispiel ist Mtbin der Lage, A549 Epithelzellen zu infizieren und zu schädigen, die häufig als Modell für menschliche Typ-II-Pneumozyten5-7verwendet werden. Dectin-1 wurde als Wirtzellrezeptor berichtet, der an der Mtb-Aufnahme, proflammatorischen Reaktion und antibakteriellen Wirkung auf intrazel...
Diskussion
Wir beschreiben hier die Methoden, die für einen phänotypischen Test mit einem GFP-exezierenden Mtb H37Rv-Stamm erforderlich sind, um fluoreszierend markierte Wirtszellen zu infizieren, was ihn für Bildschirme mit hohem Gehalt/Hohem Durchsatz geeignet macht. Dieses Protokoll könnte auf eine breite Palette von Verbindungen, Fluoreszenzsonden und Mtb-Mutationen angewendet werden. Für jedes oben beschriebene Protokoll können fixierende und immunkende Schritte vor der Bildaufnahme durchgeführt werden...
Offenlegungen
Es wurden keine Interessenkonflikte angemeldet.
Danksagungen
Finanzielle Unterstützung für diese Arbeiten wurden von der Europäischen Gemeinschaft (ERC-STG INTRACELLTB Grant n° 260901, MM4TB Grant n° 260872), der Agence Nationale de Recherche, der Feder (12001407 (D-AL) Equipex Imaginex BioMed BioMed) und der Region Nord Pas de Calais gewährt. Wir danken der technischen Unterstützung von Gaspard Deloison, Elizabeth Werkmeister, Antonino Bongiovanni und Frank Lafont von der Plattform BICeL.
Materialien
| Name | Company | Catalog Number | Comments |
| µclear-plate black, 384-well | Greiner Bio-One | 781091 | 127.8/86/15 MM with Lid, TC treated |
| CellCarrier 384-well plate | PerkinElmer | 6007550 | Black, Clear Bottom, with Lid, TC treated |
| V-bottom white, 384-well plate | Greiner Bio-One | 781280 | |
| sealing tape, breathable, sterile | Corning | 3345 | |
| Lipofectamine RNAiMax | Life Technologies | 13778150 | Transfection reagent |
| Dimethyl sulfoxide | Sigma-Aldrich | 34943 | |
| RPMI 1640 + GlutaMAX-I | Life Technologies | 61870-010 | Cell culture medium |
| D-PBS 1x [-]MgCl2/[-]CaCl2 | Life Technologies | 14190-094 | Dulbecco's Phosphate Salin Buffer |
| D-PBS 1x [+]MgCl2/[+]CaCl2 | Life Technologies | 14190-091 | Dulbecco's Phosphate Salin Buffer |
| Fetal bovine serum | Life Technologies | 2610040-79 | |
| Ficoll Paque PLUS | Dutscher | 17-1440-03 | Ficoll for Peripherical Blood Monocyte Cells purification |
| CD14 MicroBeads, human | Miltenyi | 130-050-201 | Purification of CD14+ Monocytes |
| Human M-CSF, premium gr. (1000 μg) | Miltenyi | 130-096-493 | Macrophage Colony Stimulating Factor |
| LS Columns | Miltenyi | 130-042-401 | Columns for CD14+ Monocytes isolation |
| Tween 80 | Euromedex | 2002-A | Mycobacteria culture |
| Glycerol high purity | Euromedex | 50405-EX | Mycobacteria culture |
| Middlebrook OADC enrichment | Becton-Dickinson | 211886 | Mycobacteria culture |
| 7H9 | Becton-Dickinson | W1701P | Mycobacteria culture |
| Versene 1x | Life Technologies | 15040033 | Nonenzymatic cell dissociation solution |
| DAPI | Life Technologies | D1306 | Nuclei dye |
| Hoechst 33342 | Life Technologies | H3570 | Nuclei dye |
| Syto60 | Life Technologies | S11342 | Nuclei/cytoplasm dye |
| Formalin | Sigma-Aldrich | HT5014 | Cell fixation solution |
| siRNA targeting Dectin-1 | Santa-Cruz | sc-63276 | |
| *siGenome* Nontargeted siRNA pool | Dharmacon | D-001206-14 | |
| Rifampicin | Sigma-Aldrich | R3501 | antibiotic |
| Isoniazid (INH) | Sigma-Aldrich | I3377-50G | antibiotic |
| Hygromycin B | Life Technologies | 10687-010 | antibiotic |
| Amikacin | Sigma-Aldrich | A1774 | antibiotic |
| Automated Confocal Microscope OPERA | PerkinElmer | Image acquisition | |
| Columbus 2.3.1 Server Database | PerkinElmer | Data transfer, storage, and analysis | |
| Acapella 2.6 software | PerkinElmer | Image-based analysis | |
| GraphPad Prism5 software | GraphPad | Statistical analysis | |
| Excel 2010 | Microsoft | Statistical analysis |
Referenzen
- Gandhi, N. R., et al. Multidrug-resistant and extensively drug-resistant tuberculosis: a threat to global control of tuberculosis. Lancet. 375, 1830-1843 (2010).
- Barry, C. E., et al. The spectrum of latent tuberculosis: rethinking the biology and intervention strategies. Nat. Rev. Microbiol. 7, 845-855 (2009).
- Christophe, T., Ewann, F., Jeon, H. K., Cechetto, J., Brodin, P. High-content imaging of Mycobacterium tuberculosis-infected macrophages: an in vitro model for tuberculosis drug discovery. Future Med. Chem. 2, 1283-1293 (2010).
- Brodin, P., et al. High content phenotypic cell-based visual screen identifies Mycobacterium tuberculosis acyltrehalose-containing glycolipids involved in phagosome remodeling. PLoS Pathog. 6, (2010).
- Bermudez, L. E., Goodman, J. Mycobacterium tuberculosis invades and replicates within type II alveolar cells. Infect. Immun. 64, 1400-1406 (1996).
- Dobos, K. M., Spotts, E. A., Quinn, F. D., King, C. H. Necrosis of lung epithelial cells during infection with Mycobacterium tuberculosis is preceded by cell permeation. Infect. Immun. 68, 6300-6310 (2000).
- McDonough, K. A., Kress, Y. Cytotoxicity for lung epithelial cells is a virulence-associated phenotype of Mycobacterium tuberculosis. Infect. Immun. 63, 4802-4811 (1995).
- Lee, H. M., Yuk, J. M., Shin, D. M., Jo, E. K. Dectin-1 is inducible and plays an essential role for mycobacteria-induced innate immune responses in airway epithelial cells. J. Clin. Immunol. 29, 795-805 (2009).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J. Biomol. Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat. Methods. 6, 569-575 (2009).
- Carralot, J. P., et al. Automated high-throughput siRNA transfection in raw 264.7 macrophages: a case study for optimization procedure. J. Biomol. Screen. 14, 151-160 (2009).
- Zhang, X. D. Illustration of SSMD, z score, SSMD*, z* score, and t statistic for hit selection in RNAi high-throughput screens. J. Biomol. Screen. 16, 775-785 (2011).
- Song, O. R., et al. Confocal-based method for quantification of diffusion kinetics in microwell plates and its application for identifying a rapid mixing method for high-content/throughput screening. J. Biomol. Screen. 15, 138-147 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten