É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um ensaio fenotípico microscópico para quantificação de micobactérias intracelulares adaptadas para triagem de alto rendimento/alto conteúdo
Neste Artigo
Resumo
Aqui, descrevemos um ensaio fenotípico aplicável às telas de alto rendimento/alto conteúdo de pequenas interfissões de RNA sintético (siRNA), composto químico e bibliotecas mutantes mycobacterium tuberculosis. Este método baseia-se na detecção de Mycobacterium tuberculosis rotulado fluorescentemente dentro de célula hospedeira fluorescente rotulada usando microscopia confocal automatizada.
Resumo
Apesar da disponibilidade de terapia e vacina, a tuberculose (TB) continua sendo uma das infecções bacterianas mais letais e generalizadas do mundo. Desde várias décadas, a súbita explosão de cepas multi e extensivamente resistentes a medicamentos é uma séria ameaça para o controle da tuberculose. Por isso, é essencial identificar novos alvos e caminhos críticos para o agente causador da tuberculose, Mycobacterium tuberculosis (Mtb)e buscar novos produtos químicos que possam se tornar medicamentos para TB. Uma abordagem é criar métodos adequados para as telas genéticas e químicas de bibliotecas de grande escala que permitem a busca de uma agulha em um palheiro. Para isso, desenvolvemos um ensaio fenotípico que conta com a detecção de Mtb rotulado fluorescentemente dentro de células host fluorescentes rotuladas usando microscopia confocal automatizada. Este ensaio in vitro permite uma quantificação baseada em imagem do processo de colonização de Mtb para o hospedeiro e foi otimizado para o formato de microplacão de 384 poços, que é adequado para telas de siRNA-, composto químico ou bibliotecas mutantes Mtb. As imagens são então processadas para análise multiparamétrica, que fornece leitura sobre a patogênese de Mtb dentro das células hospedeiras.
Introdução
Entre os patógenos infecciosos emergentes e ressurgindo relatados nos últimos anos, o Mycobacterium tuberculosis (Mtb)ocupa um lugar de destaque sendo responsável por 1,4 milhão de mortes e 8,7 milhões de novas infecções em 2011 (Relatório Global de Tuberculose 2012, www.who.int/topics/tuberculosis/en/). Apesar da disponibilidade de terapias multidroga, o número de pessoas infectadas ainda está em ascensão e a resistência multidroga (MDR), bem como mtb extensivamente resistente a medicamentos (XDR) estão se espalhando rapidamente por todo o mundo1. Além disso, ao levar em consideração a presença de antígenos de Mtb, é evidente que um terço da população global é considerada latentemente infectada pelo Mtb. Estatisticamente, em um caso em cada dez, há evolução para a forma ativa da doença com sintomas clínicos subsequentes2. Portanto, novos meios para combater o Mtb são urgentemente necessários. Nesse contexto, desenvolvemos um ensaio fenotípico visual in vitro que conta com o monitoramento da invasão e multiplicação de Mtb em células hospedeiras por microscopia automatizada de fluorescência confocal3. A adaptação do ensaio em placas de microtização de 384 poços em combinação com aquisição e análise automatizada de imagens permitiu a triagem de alto conteúdo/high-throughput (HC/HTS) de bibliotecas de média escala de compostos, siRNAs e mutantes bacterianos. A triagem de uma biblioteca RNAi de genoma em larga neste ensaio fenotípico permitiu assim a identificação dos principais fatores hospedeiros envolvidos no tráfico de Mtb e na replicação intracelular, mas também a elucidação de vias hospedeiras exploradas pelo bacilo tubérculo. Outra adaptação deste ensaio fenotípico em particular foi para a identificação de fatores bacterianos essenciais à persistência intra-fagossômica de Mtb. Por exemplo, a prisão do amadurecimento fágora é considerada como um dos principais mecanismos que facilita a sobrevivência e a replicação do Mtb no macrófago. O monitoramento da localização subcelular de mutantes eliminados de Mtb em compartimentos fluorescentes rotulados-ácidos permitiu a identificação de genes bacterianos envolvidos no processo de sobrevivência4. Finalmente, a imagem de alto teor de Mtb também oferece um excelente método para quantificar a eficiência das drogas para inibir vários fenômenos como o crescimento bacteriano intracelular3. Ao todo, esse tipo de ensaio fenotípico de alto rendimento permite acelerar a descoberta de drogas contra a TB e os dados coletados por essas diferentes abordagens contribuem para uma melhor compreensão da manipulação do hospedeiro exercida pelo Mtb.
Protocolo
1. Triagem de siRNA de genoma de alto rendimento
Triagem realizada em uma linha celular tipo-II humana modelo A549 após infecção com Mtb H37Rv expressando proteína fluorescente verde (GFP). Este procedimento está descrito na Figura 1A.
- Resuspend a biblioteca de siRNA seca que é armazenada em placas maternas (placas de 96 poços) com 1x tampão siRNA para atingir uma concentração de 4 μM, em seguida, transferir 10 μl da mistura para uma placa filha de 384 poços (placa filha 1).
- Adicione 10 μl de tampão de siRNA de 1x na placa da filha 1 para diluir o siRNA em 2 vezes. Após a ressuspensão do siRNA, as placas são seladas com vedação de alumínio descascado e podem ser armazenadas a -20 °C pelo menos 6 meses e até 2-3 anos, mas o tempo de armazenamento pode variar dependendo das recomendações do fabricante da biblioteca siRNA.
- Diluir siRNAs na placa da filha 1 no prato da filha 2 para atingir uma concentração de 500 nM. Após a ressuspensão do siRNA, as placas são seladas com vedação de alumínio descascado e podem ser armazenadas a -20 °C pelo menos 6 meses e até 2-3 anos, mas o tempo de armazenamento pode variar dependendo das recomendações do fabricante da biblioteca siRNA.
- Antes de usar, descongele o prato da filha 2 em temperatura ambiente.
- Pegue 2,5 μl de siRNA da placa da filha 2 e coloque em uma placa de ensaio de 384 poços.
- Na mesma placa de ensaio de 384 poços na etapa 1.5, adicione 2,5 μl de controle negativo e positivo siRNA aos seus respectivos poços.
- Diluir o reagente de transfecção em 1x D-PBS para produzir solução suficiente para fornecer reagente de transfecção de 0,1 μl em cada poço e preinubar a solução de transfecção diluída à temperatura ambiente por 5 minutos.
- Adicione 7,5 μl da mistura de solução de reagente/PBS de transfecção a cada poço na placa de ensaio e incubar por 30 minutos à temperatura ambiente.
- Adicione 40 μl de células A549 (1.500 células/poço) suspensas em RPMI 1640 médio suplementado com 10% de soro bovino fetal (FBS). Manter as células por um período de incubação de 3 dias a 37 °C em uma atmosfera contendo 5% de CO2. Essas células se dividem a cada 24 horas, portanto cerca de 12.000 células estão nos poços três dias após a transfecção.
- Lave uma cultura MTB H37Rv de duas semanas de idade expressando com D-PBS (livre de MgCl2 e CaCl2) por centrifugação a 4.000 x g por 5 min e descarte as lavagens. Repita esta etapa 3x. (Para condições de culturaGFP-Mtb H37Rv ver Protocolo 3).
- Suspenda a pelota bacteriana em 10 ml de RPMI 1640 médio suplementado com 10% de FBS e decante por 1 hora à temperatura ambiente para permitir agregados bacterianos aos sedimentos.
- Recolher o supernascer bacteriano e medir OD600 (OD600 deve estar entre 0,6-0,8) e GFP-fluorescência (valor RFU) usando um leitor de microplaca para determinar a concentração bacteriana. Calcule o título da suspensão utilizando uma linha de regressão de referência exibindo valor RFU = f (valor da UFC) que havia sido gerado antes do experimento em outra cultura que havia sido preparada nas mesmas condições. Prepare a suspensão bacteriana contendo 2,4 x 106 bactérias/ml, o que corresponde a uma multiplicidade de infecção (MOI) de 5.
- Retire o meio na placa de ensaio de 384 poços e adicione 25 μl de suspensão bacteriana recém-preparada.
- Incubar a placa de ensaio de 384 poços a 37 °C por 5 horas em uma atmosfera contendo 5% de CO2.
- Remova o meio e lave suavemente as células com meio RPMI suplementado com 10% de FBS 3x.
- Para matar as bactérias extracelulares restantes, trate as células com 50 μl de meio RPMI-FBS fresco contendo 50 μg/ml de amikacina a 37 °C por 1 hora em uma atmosfera contendo 5% de CO2.
- Remova a amikacina de média contenção e adicione 50 μl de meio RPMI fresco complementado com 10% de FBS. Incubar a placa de ensaio de 384 poços a 37 °C durante 5 dias em uma atmosfera contendo 5% de CO2.
- Antes da aquisição de imagem, adicione 10 μl de 30 μg/ml recém-preparados de DAPI em PBS (concentração final 5 μg/ml) e incubar por 10 min a 37 °C.
- Carregue a placa em microscópio confocal automatizado.

Clique aqui para ver imagem maior. - Defina os parâmetros de exposição. Fluorescência de REGISTRO DAPI usando laser de excitação 405 nm com filtro de emissão 450 nm e fluorescência GFP usando laser de excitação 488 nm com filtro de emissão 520 nm.
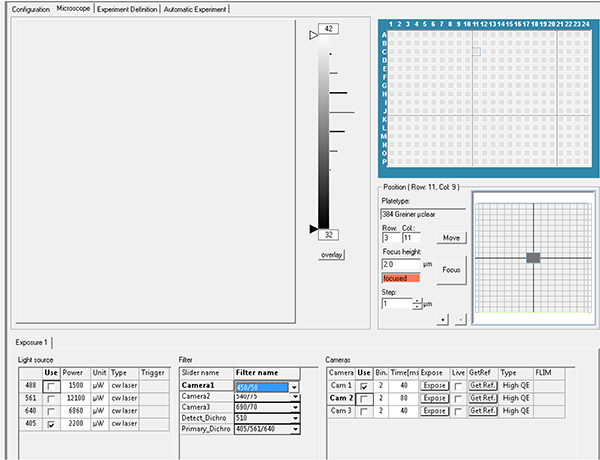
Clique aqui para ver imagem maior.
Clique aqui para ver imagem maior. - Selecione os poços e os campos em cada poço a ser adquirido, que é então chamado de layout e parâmetros de subcamada.
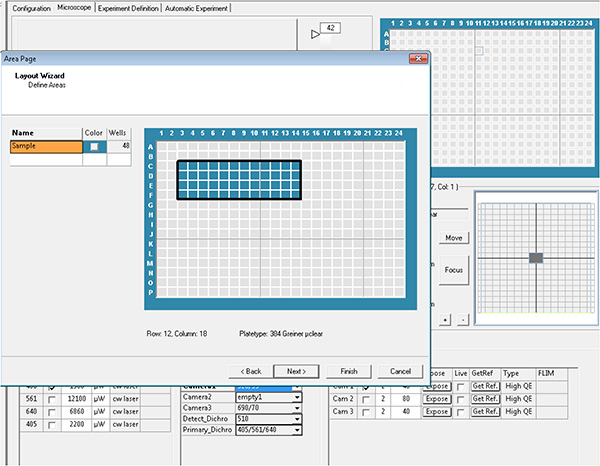
Clique aqui para ver imagem maior.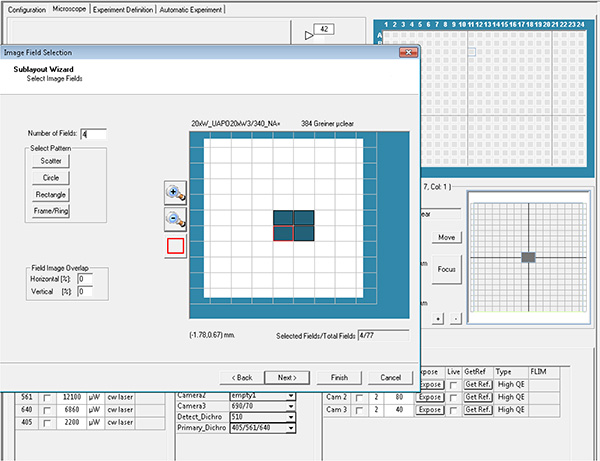
Clique aqui para ver imagem maior. - Gere o arquivo de experimento usando os parâmetros das etapas 1.20 e 1.21, e execute a aquisição automática.
Clique aqui para ver imagem maior.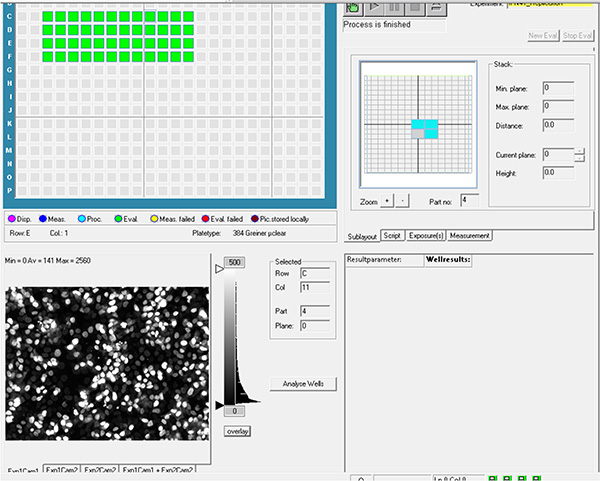
Clique aqui para ver imagem maior. - Transfira imagens para o servidor remoto.
Clique aqui para ver imagem maior. - Avalie imagens usando software de análise de imagens. Detecte núcleos celulares do canal DAPI usando algoritmo de detecção de núcleos e a área bacteriana do canal GFP usando o algoritmo de propriedades de intensidade de pixel(Figura 4A).
Nota: Este protocolo é otimizado para estudar o efeito do silenciamento genético no crescimento de Mtb intracelular. Mtb é uma bactéria de crescimento lento que divide a cada 20 horas em condições ideais. Após 5 dias após a infecção, a quantidade de Mtb extracelular ainda é baixa na ausência de lise celular e não afetou a qualidade da análise. Este protocolo deve ser otimizado em termos de tempo de tratamento com antibióticos e tempo de incubação para ser adaptado para telas de siRNA usando bactérias de crescimento rápido como Mycobacteria smegmatis e Escherichia coli que são amplamente liberadas e podem infectar novas células.
2. Triagem composta de alto rendimento
Triagem realizada em células hospedeiras infectadas por Mtb H37Rv. Este procedimento está descrito na Figura 1B.
- Descongele as placas-mãe de 384 poços contendo a biblioteca composta solubilizada em DMSO 100%. Transfira 0,5 μl dos compostos em placas filhas de 384 poços contendo 10 μl de RPMI 1640 médio suplementado com 10% de FBS.
- Lave uma cultura MTB H37Rv de duas semanas de idade expressando com D-PBS (livre de MgCl2 e CaCl2) por centrifugação a 4.000 x g por 5 min e descarte as lavagens. Repita esta etapa 3x (Para condições de culturaGFP-Mtb H37Rv ver Protocolo 3).
- Suspenda a pelota bacteriana em 10 ml de RPMI 1640 médio suplementado com 10% de FBS e decante por 1 hora à temperatura ambiente para permitir agregados bacterianos aos sedimentos.
- Recolher o supernascer bacteriano e medir OD600 (OD600 deve estar entre 0,6-0,8) e GFP-fluorescência (valor RFU) usando um leitor de microplaca. Calcule o título da suspensão usando uma linha de regressão de referência exibindo valor RFU = f (valor da UFC) que havia sido gerado antes do experimento. A concentração típica é de 1 x 108 bactérias/ml.
- Colher macrófagos humanos primários de 6 dias de idade a 4 x 105 células/ml em RPMI 1640 médio suplementado com 10% de soro bovino fetal (FBS) e 50 ng/ml recombinando humano M-CSF (Para purificação de células de monócito de sangue periférico humano e diferenciação de macrófagos ver Protocolo 4).
- Incubar as células primárias diluídas com bacilos em diferentes MOI, variando de 1 a 5, em suspensão com leve agitação a 90 rpm por 2 horas a 37 °C.
- Lave as células infectadas por centrifugação a 350 x g para remover as bactérias extracelulares. Após cada etapa de centrifugação, resuspense a pelota em RPMI 1640 médio suplementado com 10% de FBS. Repita esta etapa 2x.
- Suspenda as células infectadas em RPMI 1640 médio suplementado com 10% de FBS e amikacina a 50 μg/ml e incubar a suspensão com leve agitação por 1 hora a 37 °C.
- Remova o meio de cultura celular contendo amikacina por centrifugação a 350 x g e lave as células infectadas com meio RPMI completo 1640 suplementado com 10% de FBS e 50 ng/ml humano recombinante M-CSF. Repita uma vez.
- Adicione 40 μl da suspensão dos macrófagos infectados na mesma placa de ensaio na etapa 2.1, que já contém 10 μl das diluições compostas. A concentração final de DMSO em cada poço chegou agora a 1%.
- Incubar as placas de ensaio por 5 dias a 37 °C em uma atmosfera contendo 5% de CO2.
- Manche as células vivas com corante fluorescente vermelho-permeante de células.
- Carregue a placa em microscópio confocal automatizado.

Clique aqui para ver imagem maior. - Defina os parâmetros de exposição. Registre fluorescência distante-vermelha usando laser de excitação de 640 nm com filtro de emissão 690 nm e fluorescência GFP usando laser de excitação 488 nm com filtro de emissão de 520 nm.

Clique aqui para ver imagem maior.
Clique aqui para ver imagem maior. - Selecione os poços e os campos em cada poço a ser adquirido, que é então chamado de layout e parâmetros de subcamada.

Clique aqui para ver imagem maior.
Clique aqui para ver imagem maior. - Gere o arquivo de experimento usando os parâmetros das etapas 2.14 e 2.15 e execute a aquisição automática.

Clique aqui para ver imagem maior.
Clique aqui para ver imagem maior. - Transfira imagens para o servidor remoto.

Clique aqui para ver imagem maior. - Avalie imagens usando software de análise de imagens. Detecte a área celular do canal vermelho usando o algoritmo de propriedades de intensidade de pixel e área bacteriana do canal GFP usando o algoritmo de propriedades de intensidade de pixel(Figura 4B).
Nota: Este protocolo pode ser adaptado para a triagem da biblioteca mutante Mtb substituindo compostos por mutantes que expressam uma proteína fluorescente (Um mutante bem/Um) (Figura 1C, veja também Brodin et al. 4). Mutantes fluorescentes são primeiro semeados em poços (20 μl de suspensão bacteriana por poço). As bactérias são então recuperadas por 30 μl de suspensão celular. Após centrifugação a 350 x g por 1 min, a placa é incubada a 37 °C em uma atmosfera contendo 5% de CO2. O tempo de incubação e o MOI dependem do ensaio. Como exemplo, para visualização de eventos celulares precoces, como acidificação de fagosome, as células podem ser infectadas por 2 horas com MOI variando de 1 a 20. Os lysosomos são manchados usando corante lysotracker a 2 μM por 1,5 h a 37 °C em uma atmosfera contendo 5% de CO 2 edepois fixado com 10% de formalina ou 4% de paraformaldeído (PFA). As imagens confocal são adquiridas e finalmente analisadas usando scripts de análise de imagem com algoritmos apropriados para detecção de lysososomes e localização subcelular4.
3. Proteína Fluorescente Verde Expressando Mycobacterium tuberculosis H37Rv (GFP-H37Rv) Condições de Cultura
Para armazenamento a longo prazo, o GFP-H37Rv foi congelado em D-PBS (cerca de 1 x 108 micobactérias por frasco).
- Resuspend um frasco de caldo congelado de GFP-H37Rv em um frasco Erlenmeyer contendo 50 ml de caldo 7H9 médio suplementado com enriquecimento Middlebrook OADC 10%, Glicerol 0,5%, Tween-80 0,05% e Hygromycin B (50 μg/ml).
- Incubar 8 dias a 37 °C.
- Medir OD600 da cultura GFP-H37Rv.
- Diluir a cultura GFP-H37Rv para obter OD600 = 0,1 em caldo fresco 7H9 complementado com enriquecimento Middlebrook OADC 10%, Glicerol 0,5%, Tween-80 0,05% e Hygromycin B (50 μg/ml).
- Incubar o GFP-H37Rv a 37 °C por mais 8 dias antes de usar para o ensaio.
4. Purificação de células de monócitos de sangue periférico humano a partir de preparação de sangue inteiro ou casaco de buffy
- Diluir a bolsa de sangue 2x em 1x D-PBS (livre de MgCl2 e CaCl2) contendo 1% de FBS.
- Isole os monócitos por Centrifugação gradiente de densidade ficoll a 400 x g por 20 min.
- Recolher os monócitos isolados.
- Lave os monócitos 3x com 1x D-PBS (livre de MgCl2 e CaCl2) contendo 1% de FBS por centrifugação a 400 x g por 10 minutos à temperatura ambiente.
- Concentre as células até 1 x 107 células/ml.
- Purificar monócitos usando contas magnéticas CD14 de acordo com o protocolo do fabricante (ver Materiais).
- Após a purificação de cd14-monócitos, semeou as células em 1,5 x 106 células/ml em RPMI 1640 complementada com 10% FBS e 40 ng/ml de Fator Estimulante da Colônia de Macrófagos Humanos (hM-CSF) e incubada por 4 dias a 37 °C em uma atmosfera contendo 5% CO2.
- Após 4 dias substitua o meio por RPMI fresco 1640 complementado com 10% de FBS e 40 ng/ml de hM-CSF e incubado por 2 dias a 37 °C em uma atmosfera contendo 5% de CO2.
- Após 6 dias, as células podem ser usadas para o ensaio.
Resultados
Triagem de siRNA de genoma de alto rendimento
Mtb é capaz de colonizar células imunes in vitro, bem como várias outras células epiteliais pulmonares. Por exemplo, o Mtbé capaz de infectar e danificar células epiteliais A549 que são comumente usadas como modelo para pneumócitos tipo II humano5-7. O Dectin-1 foi relatado como um receptor de células hospedeiras envolvido na absorção de Mtb, resposta proinflamatória e efeito antibacteriano no...
Discussão
Descrevemos aqui os métodos necessários para um ensaio fenotípico usando uma cepa Mtb H37Rv expressante gfp para infectar células host fluorescentes rotuladas, o que o torna apropriado para telas de alto conteúdo/alto rendimento. Este protocolo poderia ser aplicado a uma ampla gama de compostos, sondas fluorescentes e mutantes Mtb. Para cada protocolo descrito acima, as etapas de fixação e imunolabelamento poderiam ser realizadas antes da aquisição da imagem. Usamos um microscópio confocal flu...
Divulgações
Nenhum conflito de interesses declarado.
Agradecimentos
O apoio financeiro para este trabalho foi fornecido pela Comunidade Europeia (ERC-STG INTRACELLTB Grant n° 260901, MM4TB Grant n° 260872), pela Agence Nationale de Recherche, pela Feder (12001407 (D-AL) Equipex Imaginex BioMed) e pela Região Nord Pas de Calais. Agradecemos a assistência técnica de Gaspard Deloison, Elizabeth Werkmeister, Antonino Bongiovanni e Frank Lafont da plataforma BICeL.
Materiais
| Name | Company | Catalog Number | Comments |
| µclear-plate black, 384-well | Greiner Bio-One | 781091 | 127.8/86/15 MM with Lid, TC treated |
| CellCarrier 384-well plate | PerkinElmer | 6007550 | Black, Clear Bottom, with Lid, TC treated |
| V-bottom white, 384-well plate | Greiner Bio-One | 781280 | |
| sealing tape, breathable, sterile | Corning | 3345 | |
| Lipofectamine RNAiMax | Life Technologies | 13778150 | Transfection reagent |
| Dimethyl sulfoxide | Sigma-Aldrich | 34943 | |
| RPMI 1640 + GlutaMAX-I | Life Technologies | 61870-010 | Cell culture medium |
| D-PBS 1x [-]MgCl2/[-]CaCl2 | Life Technologies | 14190-094 | Dulbecco's Phosphate Salin Buffer |
| D-PBS 1x [+]MgCl2/[+]CaCl2 | Life Technologies | 14190-091 | Dulbecco's Phosphate Salin Buffer |
| Fetal bovine serum | Life Technologies | 2610040-79 | |
| Ficoll Paque PLUS | Dutscher | 17-1440-03 | Ficoll for Peripherical Blood Monocyte Cells purification |
| CD14 MicroBeads, human | Miltenyi | 130-050-201 | Purification of CD14+ Monocytes |
| Human M-CSF, premium gr. (1000 μg) | Miltenyi | 130-096-493 | Macrophage Colony Stimulating Factor |
| LS Columns | Miltenyi | 130-042-401 | Columns for CD14+ Monocytes isolation |
| Tween 80 | Euromedex | 2002-A | Mycobacteria culture |
| Glycerol high purity | Euromedex | 50405-EX | Mycobacteria culture |
| Middlebrook OADC enrichment | Becton-Dickinson | 211886 | Mycobacteria culture |
| 7H9 | Becton-Dickinson | W1701P | Mycobacteria culture |
| Versene 1x | Life Technologies | 15040033 | Nonenzymatic cell dissociation solution |
| DAPI | Life Technologies | D1306 | Nuclei dye |
| Hoechst 33342 | Life Technologies | H3570 | Nuclei dye |
| Syto60 | Life Technologies | S11342 | Nuclei/cytoplasm dye |
| Formalin | Sigma-Aldrich | HT5014 | Cell fixation solution |
| siRNA targeting Dectin-1 | Santa-Cruz | sc-63276 | |
| *siGenome* Nontargeted siRNA pool | Dharmacon | D-001206-14 | |
| Rifampicin | Sigma-Aldrich | R3501 | antibiotic |
| Isoniazid (INH) | Sigma-Aldrich | I3377-50G | antibiotic |
| Hygromycin B | Life Technologies | 10687-010 | antibiotic |
| Amikacin | Sigma-Aldrich | A1774 | antibiotic |
| Automated Confocal Microscope OPERA | PerkinElmer | Image acquisition | |
| Columbus 2.3.1 Server Database | PerkinElmer | Data transfer, storage, and analysis | |
| Acapella 2.6 software | PerkinElmer | Image-based analysis | |
| GraphPad Prism5 software | GraphPad | Statistical analysis | |
| Excel 2010 | Microsoft | Statistical analysis |
Referências
- Gandhi, N. R., et al. Multidrug-resistant and extensively drug-resistant tuberculosis: a threat to global control of tuberculosis. Lancet. 375, 1830-1843 (2010).
- Barry, C. E., et al. The spectrum of latent tuberculosis: rethinking the biology and intervention strategies. Nat. Rev. Microbiol. 7, 845-855 (2009).
- Christophe, T., Ewann, F., Jeon, H. K., Cechetto, J., Brodin, P. High-content imaging of Mycobacterium tuberculosis-infected macrophages: an in vitro model for tuberculosis drug discovery. Future Med. Chem. 2, 1283-1293 (2010).
- Brodin, P., et al. High content phenotypic cell-based visual screen identifies Mycobacterium tuberculosis acyltrehalose-containing glycolipids involved in phagosome remodeling. PLoS Pathog. 6, (2010).
- Bermudez, L. E., Goodman, J. Mycobacterium tuberculosis invades and replicates within type II alveolar cells. Infect. Immun. 64, 1400-1406 (1996).
- Dobos, K. M., Spotts, E. A., Quinn, F. D., King, C. H. Necrosis of lung epithelial cells during infection with Mycobacterium tuberculosis is preceded by cell permeation. Infect. Immun. 68, 6300-6310 (2000).
- McDonough, K. A., Kress, Y. Cytotoxicity for lung epithelial cells is a virulence-associated phenotype of Mycobacterium tuberculosis. Infect. Immun. 63, 4802-4811 (1995).
- Lee, H. M., Yuk, J. M., Shin, D. M., Jo, E. K. Dectin-1 is inducible and plays an essential role for mycobacteria-induced innate immune responses in airway epithelial cells. J. Clin. Immunol. 29, 795-805 (2009).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J. Biomol. Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat. Methods. 6, 569-575 (2009).
- Carralot, J. P., et al. Automated high-throughput siRNA transfection in raw 264.7 macrophages: a case study for optimization procedure. J. Biomol. Screen. 14, 151-160 (2009).
- Zhang, X. D. Illustration of SSMD, z score, SSMD*, z* score, and t statistic for hit selection in RNAi high-throughput screens. J. Biomol. Screen. 16, 775-785 (2011).
- Song, O. R., et al. Confocal-based method for quantification of diffusion kinetics in microwell plates and its application for identifying a rapid mixing method for high-content/throughput screening. J. Biomol. Screen. 15, 138-147 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados