Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Yüksek Verimli/Yüksek İçerikli Taramaya Uyarlanmış Hücre İçi Mikobakterilerin Nicelleştirilmesi için Mikroskobik Fenotipik Bir Tahlil
Bu Makalede
Özet
Burada, küçük müdahale eden sentetik RNA (siRNA), kimyasal bileşik ve Mycobacterium tuberculosis mutant kütüphanelerinin Yüksek verimli / Yüksek içerikli ekranları için geçerli olan fenotipik bir test açıklıyoruz. Bu yöntem, otomatik konfokal mikroskopi kullanılarak floresan etiketli konak hücre içinde floresan etiketli Mycobacterium tuberculosis'in tespitine dayanır.
Özet
Tedavi ve aşı mevcudiyetine rağmen, tüberküloz (TB) dünyadaki en ölümcül ve yaygın bakteriyel enfeksiyonlardan biri olmaya devam etmektedir. Birkaç on yıldan bu yana, çok ve yaygın olarak ilaca dirençli suşların ani patlaması tüberkülozun kontrolü için ciddi bir tehdittir. Bu nedenle, tüberkülozun etken maddesi olan Mycobacterium tuberculosis(Mtb)için kritik olan yeni hedeflerin ve yolların belirlenmesi ve TB ilaçları olabilecek yeni kimyasalların araştırılması esastır. Bir yaklaşım, samanlıkta iğne aranmasını sağlayan büyük ölçekli kütüphanelerin genetik ve kimyasal ekranlarına uygun yöntemler kurmaktır. Bu amaçla, otomatik konfokal mikroskopi kullanarak floresan etiketli konak hücreler içinde floresan etiketli Mtb'nin tespitine dayanan fenotipik bir test geliştirdik. Bu in vitro test, Mtb'nin kolonileşme sürecinin konakçıya görüntü bazlı nicelleştirilmesine izin verir ve siRNA, kimyasal bileşik veya Mtb mutant kütüphanelerinin ekranları için uygun olan 384 kuyu mikro plaka formatı için optimize edilmiştir. Görüntüler daha sonra konak hücreler içindeki Mtb patogenezinde okuma çıkarımını sağlayan çokparametrik analiz için işlenir.
Giriş
Son yıllarda bildirilen ortaya çıkan ve yeniden ortaya çıkan bulaşıcı patojenler arasında, Mycobacterium tuberculosis (Mtb),2011 yılında 1,4 milyon ölümden ve 8,7 milyon yeni enfeksiyondan sorumlu olarak önemli bir yer tutmaktadır (Küresel tüberküloz raporu 2012, www.who.int/topics/tuberculosis/en/). Multidrug tedavilerinin mevcudiyetine rağmen, enfekte insan sayısı hala artmaktadır ve çok halıya dayanıklı (MDR) ve yaygın olarak ilaca dirençli (XDR) Mtb hızla tüm dünyaya yayılmaktadır1. Ayrıca, Mtb antijenlerinin varlığı dikkate alındığında, küresel nüfusun üçte birinin Mtb tarafından gizlice enfekte olduğu düşünülmektedir. İstatistiksel olarak, on vakadan birinde, sonraki klinik semptomlarla hastalığın aktif formuna doğru evrim vardır2. Bu nedenle, Mtb ile savaşmak için acilen yeni araçlara ihtiyaç vardır. Bu kapsamda, otomatik konfokal floresan mikroskopisi3 . Tahlilin 384 kuyulu mikrotiter plakalarda otomatik görüntü alma ve analiz ile birlikte uyarlanması, orta ölçekli bileşik, siRNA ve bakteriyel mutant kütüphanelerinin Yüksek içerikli / Yüksek verimli Taramaya (HC / HTS) izin verdi. Bu fenotipik test üzerinde genom geniş bir RNAi kütüphanesinin taranması, böylece Mtb kaçakçılığı ve hücre içi replikasyonda yer alan temel konak faktörlerinin tanımlanmasını ve aynı zamanda tüberkül basil basili tarafından sömürülen konak yolların aydınlatılmasını sağladı. Bu özel fenotipik tahlilin bir diğer adaptasyonu da Mtb intra-fagozomal kalıcılık için gerekli bakteriyel faktörlerin tanımlanmasıydı. Örneğin, fagozom olgunlaşmasının tutuklanması, Mtb'nin makrofajda hayatta kalmasını ve replikasyonunu kolaylaştıran önemli mekanizmalardan biri olarak kabul edilir. Mtb nakavt mutantlarının floresan etiketli-asidik bölmelerde hücre altı lokalizasyonunun izlenmesi, sağkalım sürecinde yer alan bakteri genlerinin tanımlanmasına izin verdi4. Son olarak, Mtb'nin yüksek içerikli görüntülemesi, hücre içi bakteri üremesi gibi çeşitli fenomenleri inhibe etmek için ilaç verimliliğini ölçmek için mükemmel bir yöntem sunar3. Tamamen, bu tür yüksek verimli fenotipik test, TB'ye karşı ilaç keşfini hızlandırmaya izin verir ve bu farklı yaklaşımlarla toplanan veriler Mtbtarafından uygulanan konak manipülasyonunun daha iyi anlaşılmasına katkıda bulunur.
Protokol
1. Yüksek verimli Genom çapında siRNA Taraması
Yeşil Floresan Proteini (GFP) ifade eden Mtb H37Rv ile enfeksiyon üzerine insan Tip-II pnömosit modeli A549 hücre hattında tarama yapıldı. Bu yordam Şekil 1A'daözetlenmiştir.
- 4 μM konsantrasyona ulaşmak için 1x siRNA tamponu ile ana plakalarda (96 kuyu plakaları) depolanan kurutulmuş siRNA kütüphanesini yeniden kullanın, ardından karışımın 10 μl'lik kısmını 384 kuyulu bir kız plakasına (kız plakası 1) aktarın.
- SiRNA'yı 2 kat seyreltmek için 16 kız plakası 1'e 10 μl siRNA tamponu ekleyin. siRNA resüspensiyondan sonra, plakalar soyulabilir alüminyum conta ile kapatılır ve -20 °C'de en az 6 ay ve 2-3 yıla kadar saklanabilir, ancak depolama süresi siRNA kütüphane üreticisinin önerilerine bağlı olarak değişebilir.
- 500 nM konsantrasyona ulaşmak için kız plakası 1'deki siRNA'ları kız plakası 2'ye seyreltin. siRNA resüspensiyondan sonra, plakalar soyulabilir alüminyum conta ile kapatılır ve -20 °C'de en az 6 ay ve 2-3 yıla kadar saklanabilir, ancak depolama süresi siRNA kütüphane üreticisinin önerilerine bağlı olarak değişebilir.
- Kullanmadan önce, kız plakası 2'yi oda sıcaklığında çözün.
- Kız plakası 2'den 2,5 μl siRNA alın ve 384 kuyulu bir test plakasına yerleştirin.
- Adım 1.5'te aynı 384 kuyu test plakasında, ilgili kuyularına 2.5 μl negatif ve pozitif kontrol siRNA ekleyin.
- Transfeksiyon reaktifini 1x D-PBS'de seyrelterek her kuyuda 0,1 μl transfeksiyon reaktifi sağlayacak kadar çözelti elde edin ve seyreltilmiş transfeksiyon çözeltisini oda sıcaklığında 5 dakika önceden kesin.
- Test plakasındaki her kuyuya 7,5 μl transfeksiyon reaktifi/PBS çözelti karışımı ekleyin ve oda sıcaklığında 30 dakika kuluçkaya yatırın.
- %10 fetal sığır serumu (FBS) ile desteklenmiş RPMI 1640 ortamında askıya alınan 40 μl A549 hücresi (1.500 hücre/kuyu) ekleyin. %5 CO2içeren bir atmosferde 37 °C'de 3 günlük kuluçka süresi boyunca hücreleri koruyun. Bu hücreler her 24 saatte bir bölünür, böylece transfeksiyondan üç gün sonra yaklaşık 12.000 hücre kuyulardadır.
- İki haftalık GFP ifade eden Mtb H37Rv kültürünü D-PBS (MgCl 2 ve CaCl2'den ücretsiz) ile5dakika boyunca 4.000 x g'da santrifüjleme ile yıkayın ve yıkamaları atın. Bu adımı 3x tekrarlayın. (GFP-Mtb H37Rv kültür koşulları için bkz. protokol 3).
- Bakteriyel peleti% 10 FBS ile desteklenmiş 10 ml RPMI 1640 ortamında askıya alın ve bakteriyel agregaların tortulanmasına izin vermek için oda sıcaklığında 1 saat dekant.
- Bakteriyel süpernatantı toplayın ve bakteri konsantrasyonu belirlemek için bir mikro plaka okuyucu kullanarakOD 600 (OD 600 0.6-0.8 arasında olmalıdır) ve GFP-floresan (RFU değeri) ölçün. Aynı koşullarda hazırlanmış başka bir kültür üzerinde denemeden önce oluşturulan RFU değeri = f (CFU değeri) gösteren bir referans regresyon çizgisi kullanarak süspansiyonun titresini hesaplayın. 5'lik çok miktarda enfeksiyona (MOI) karşılık gelen2,4 x 10 6 bakteri/ml içeren bakteri süspansiyonu hazırlayın.
- 384 kuyulu test plakasındaki ortamı çıkarın ve 25 μl taze hazırlanmış bakteri süspansiyonu ekleyin.
- 384 kuyulu test plakasını% 5 CO2içeren bir atmosferde 5 saat boyunca 37 ° C'de kuluçkaya yatırın.
- Ortamı çıkarın ve hücreleri% 10 FBS 3x ile desteklenmiş RPMI ortamı ile hafifçe yıkayın.
- Kalan hücre dışı bakterileri öldürmek için, %5 CO2içeren bir atmosferde 1 saat boyunca 37 °C'de 50 μg/ml amikasin içeren 50 μl taze RPMI-FBS ortamı ile hücreleri tedavi edin.
- Orta içeren amikacin'i çıkarın ve% 10 FBS ile desteklenmiş 50 μl taze RPMI ortamı ekleyin. 384 kuyulu test plakasını% 5 CO2içeren bir atmosferde 5 gün boyunca 37 ° C'de kuluçkaya yatırın.
- Görüntü alımından önce, PBS'de 10 μl taze hazırlanmış 30 μg/ml DAPI ekleyin (son konsantrasyon 5 μg/ml) ve 37 °C'de 10 dakika kuluçkaya yatırın.
- Plakayı otomatik konfokal mikroskopa yükleyin.

Daha büyük resmi görüntülemek için burayı tıklatın. - Pozlama parametrelerini ayarlayın. Emisyon filtresi 450 nm ile excitation lazer 405 nm ve emisyon filtresi 520 nm ile excitation lazer 488 nm kullanarak GFP floresan kullanarak DAPI floresan kaydedin.
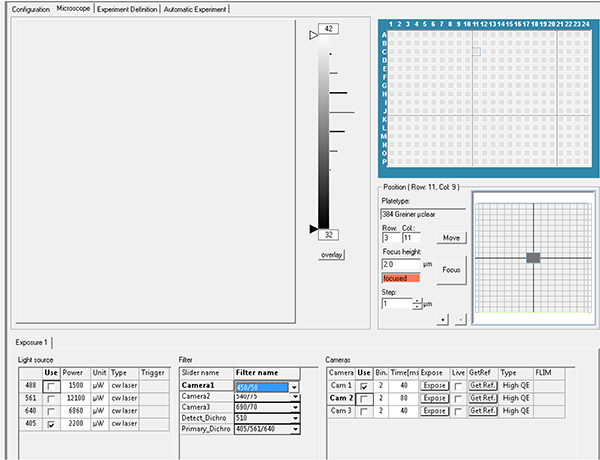
Daha büyük resmi görüntülemek için burayı tıklatın.
Daha büyük resmi görüntülemek için burayı tıklatın. - Elde edilecek her kuyudaki kuyuları ve alanları seçin, daha sonra düzen ve alt katman parametreleri olarak adlandırılır.
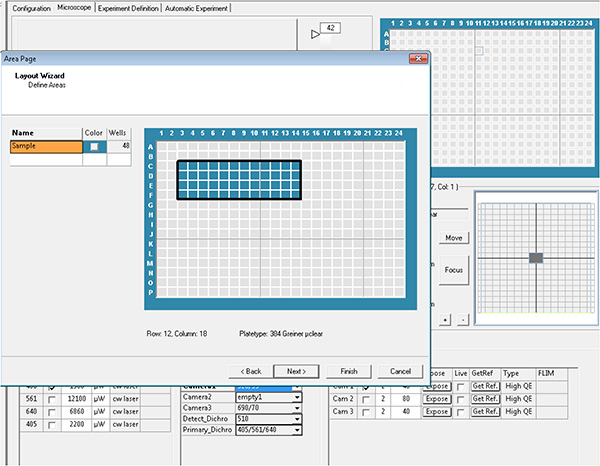
Daha büyük resmi görüntülemek için burayı tıklatın.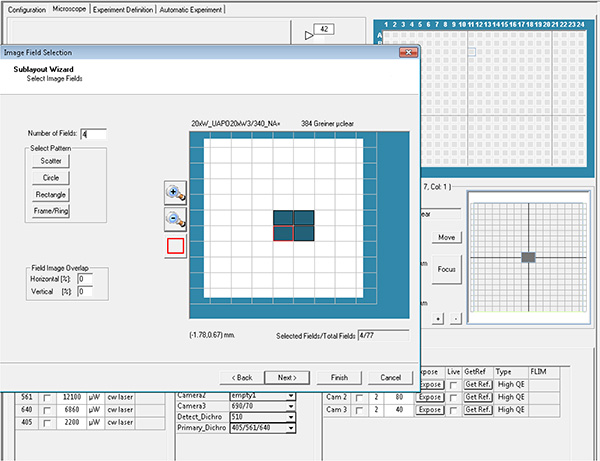
Daha büyük resmi görüntülemek için burayı tıklatın. - 1.20 ve 1.21 adımlarındaki parametreleri kullanarak deneme dosyasını oluşturun ve otomatik alma işlemini çalıştırın.
Daha büyük resmi görüntülemek için burayı tıklatın.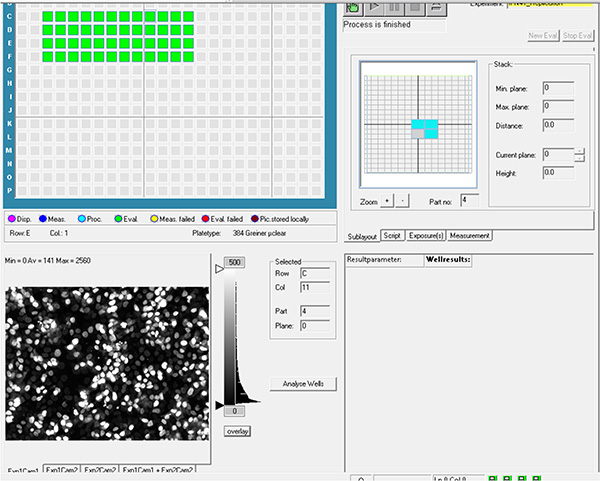
Daha büyük resmi görüntülemek için burayı tıklatın. - Görüntüleri uzak sunucuya aktarın.
Daha büyük resmi görüntülemek için burayı tıklatın. - Görüntü analiz yazılımını kullanarak görüntüleri değerlendirin. Çekirdek algılama algoritmasını kullanarak DAPI kanalından hücre çekirdeklerini ve piksel yoğunluğu özellikleri algoritmasını kullanarak GFP kanalından bakteri alanını tespit edin (Şekil 4A).
Not: Bu protokol, gen susturmasının hücre içi Mtb büyümesi üzerindeki etkisini incelemek için optimize edilmiştir. Mtb, her 20 saatte bir optimum koşullarda bölünen yavaş büyümeli bir bakteridir. Enfeksiyondan 5 gün sonra hücre dışı Mtb miktarı hücre lizizinin yokluğunda hala düşüktür ve analizin kalitesini etkilememiştir. Bu protokol, mycobacteria smegmatis ve Escherichia coli gibi yoğun olarak salınan ve yeni hücreleri enfekte edebilen hızlı büyüme bakterileri kullanılarak siRNA ekranları için uyarlanacak antibiyotik tedavisinin uzunluğu ve kuluçka süresi açısından optimize edilmelidir.
2. Yüksek verimli Bileşik Tarama
Tarama Mtb H37Rv enfekte konak hücrelerinde gerçekleştirildi. Bu yordam Şekil 1B'de özetlenmiştir.
- DMSO'da çözünen bileşik kütüphaneyi içeren 384 kuyulu ana plakaları %100 çözün. %10 FBS ile desteklenmiş 10 μl RPMI 1640 ortamı içeren 384 kuyulu kız plakalarındaki bileşiklerin 0,5 μl'sini aktarın.
- İki haftalık GFP ifade eden Mtb H37Rv kültürünü D-PBS (MgCl 2 ve CaCl2'den ücretsiz) ile5dakika boyunca 4.000 x g'da santrifüjleme ile yıkayın ve yıkamaları atın. Bu adımı 3x yineleyin (GFP-Mtb H37Rv kültür koşulları için bkz. protokol 3).
- Bakteriyel peleti% 10 FBS ile desteklenmiş 10 ml RPMI 1640 ortamında askıya alın ve bakteriyel agregaların tortulanmasına izin vermek için oda sıcaklığında 1 saat dekant.
- Bakteriyel süpernatantı toplayın ve mikro plaka okuyucu kullanarak OD600 (OD600 0.6-0.8 arasında olmalıdır) ve GFP floresanını (RFU değeri) ölçün. Denemeden önce oluşturulan RFU değeri = f (CFU değeri) görüntüleyen bir referans regresyon çizgisi kullanarak süspansiyonun titresini hesaplayın. Tipik konsantrasyon 1 x 108 bakteri/ml'dir.
- %10 fetal sığır serumu (FBS) ve50 ng/ml rekombinant insan M-CSF ile desteklenmiş RPMI 1640 ortamında 4 x 10 5 hücre/ml'de 6 günlük birincil insan makrofajlarını hasat edin (İnsan Periferik Kan Monosit Hücreleri saflaştırma ve makrofajların farklılaşması için bkz. Protokol 4).
- Seyreltilmiş birincil hücreleri, 1-5 arasında değişen farklı MOI'de basil ile kuluçkaya yaslanın, süspansiyonda 90 rpm'de 37 °C'de 2 saat hafif sallanarak.
- Hücre dışı bakterileri temizlemek için enfekte hücreleri 350 x g'da santrifüjleme ile yıkayın. Her santrifüj adımından sonra peletin %10 FBS ile desteklenmiş RPMI 1640 ortamında yeniden kullanılabilir. Bu adımı 2x tekrarlayın.
- RPMI 1640 ortamındaki enfekte hücreleri % 10 FBS ve amikacin ile 50 μg / ml'de askıya alın ve süspansiyonu 37 ° C'de 1 saat hafif sallama ile kuluçkaya yatırın.
- Amikacin içeren hücre kültürü ortamını 350 x g'da santrifüjleme ile çıkarın ve enfekte olmuş hücreleri% 10 FBS ve 50 ng / ml rekombinant insan M-CSF ile desteklenmiş eksiksiz RPMI 1640 ortamı ile yıkayın. Bir kez tekrarlayın.
- Zaten bileşik seyreltmelerin 10 μl'sini içeren 2.1 adımında aynı test plakasına 40 μl enfekte makrofaj süspansiyonu ekleyin. DMSO'nun her kuyudaki son konsantrasyonu artık % 1'e ulaştı.
- %5 CO2içeren bir atmosferde 37 °C'de 5 gün boyunca test plakalarını kuluçkaya yatırın.
- Canlı hücreleri hücre geçirgen uzak kırmızı floresan boya ile lekelenin.
- Plakayı otomatik konfokal mikroskopa yükleyin.

Daha büyük resmi görüntülemek için burayı tıklatın. - Pozlama parametrelerini ayarlayın. Emisyon filtresi 690 nm ile excitation lazer 640 nm ve emisyon filtresi 520 nm ile excitation lazer 488 nm kullanarak GFP floresan kullanarak uzak kırmızı floresan kaydedin.

Daha büyük resmi görüntülemek için burayı tıklatın.
Daha büyük resmi görüntülemek için burayı tıklatın. - Elde edilecek her kuyudaki kuyuları ve alanları seçin, daha sonra düzen ve alt katman parametreleri olarak adlandırılır.

Daha büyük resmi görüntülemek için burayı tıklatın.
Daha büyük resmi görüntülemek için burayı tıklatın. - 2.14 ve 2.15 adımlarındaki parametreleri kullanarak deneme dosyasını oluşturun ve otomatik alma işlemini çalıştırın.

Daha büyük resmi görüntülemek için burayı tıklatın.
Daha büyük resmi görüntülemek için burayı tıklatın. - Görüntüleri uzak sunucuya aktarın.

Daha büyük resmi görüntülemek için burayı tıklatın. - Görüntü analiz yazılımını kullanarak görüntüleri değerlendirin. Piksel yoğunluğu özellikleri algoritması kullanarak uzak kırmızı kanaldan hücre alanını ve piksel yoğunluğu özellikleri algoritması(Şekil 4B)kullanarak GFP kanalından bakteri alanını algılayın.
Not: Bu protokol, floresan proteini (Bir kuyu/Bir mutant) ifade eden mutantlar tarafından bileşiklerin değiştirilmesiyle Mtb mutant kütüphanesi taraması için uyarlanabilir (Şekil 1C, ayrıca bkz. 4). Floresan mutantlar ilk olarak kuyularda tohumlanır (kuyu başına 20 μl bakteri süspansiyonu). Bakteriler daha sonra 30 μl hücre süspansiyonu ile geri kazanilir. 1 dakika boyunca 350 x g'da santrifüjlemeden sonra, plaka% 5 CO2içeren bir atmosferde 37 ° C'de inkübe edilir. Kuluçka süresi ve MOI teste bağlıdır. Örnek olarak, fagozom asitleştirme gibi erken hücresel olayların görselleştirilmesi için, hücreler 1-20 arasında değişen MOI ile 2 saat boyunca enfekte edilebilir. Lizozomlar% 5 CO 2 içeren bir atmosferde 37 ° C'de 1.5 saat boyunca2 μM'de Lysotracker boyası kullanılarak lekelenir ve daha sonra% 10 formalin veya% 4 paraformaldehit (PFA) ile sabitlenir. Konfokal görüntüler, lizozom tespiti ve hücre altı yerelleştirme için uygun algoritmalara sahip görüntü analizi komut dosyaları kullanılarak elde edilir ve son olarak analiz edilir4.
3. Mikobakteri tüberkülozu ifade eden Yeşil Floresan Protein H37Rv (GFP-H37Rv) Kültür Koşulları
Uzun süreli depolama için, GFP-H37Rv D-PBS'de donduruldu (şişe başına yaklaşık 1 x10 8 mikobakteri).
- Middlebrook OADC zenginleştirmesi %10, Gliserol %0,5, Ara-80%0,05 ve Hygromycin B (50 μg/ml) ile desteklenmiş 50 ml 7H9 et suyu ortamı içeren bir Erlenmeyer şişesinde bir adet donmuş stok GFP-H37Rv şişesini yeniden depola.
- 37 °C'de 8 gün kuluçkaya yaslanın.
- GFP-H37Rv kültürünün OD600'lerini ölçün.
- GFP-H37Rv kültürünü seyrelterek OD600 = 0.1'i Middlebrook OADC zenginleştirmesi ile desteklenmiş taze 7H9 et suyu ortamında %10, Gliserol%0.5, Ara-80%0.05 ve Hygromycin B (50 μg/ml) ile destekleyin.
- Test için kullanmadan önce GFP-H37Rv'u 8 gün daha 37 °C'de kuluçkaya yatırın.
4. İnsan Periferik Kan Monosit Hücreleri Tam Kan veya Buffy-coat Preparatından Arındırma
- Kan kesesini % 1 FBS içeren 1x D-PBS'de (MgCl2 ve CaCl 2'den ücretsiz)2xseyreltin.
- Monositleri 20 dakika boyunca 400 x g'da Ficoll yoğunluk gradyan santrifüjleme ile izole edin.
- Yalıtılmış monositleri toplayın.
- Monositleri 3x'i oda sıcaklığında 10 dakika boyunca 400 x g'da santrifüjleme ile%1 FBS içeren 1x D-PBS (MgCl 2 ve CaCl2içermez) ile yıkayın.
- Hücreleri 1 x 107 hücre/ml'ye kadar konsantre edin.
- Cd14 manyetik boncukları kullanarak monositleri üreticinin protokolüne göre arındırın (bkz. Malzemeler).
- CD14-monositlerin saflaştırılmasından sonra, RPMI 1640'ta 1,5 x 106 hücre/ml'deki hücrelerin tohumlamaları% 10 FBS ve 40 ng / ml insan Makrofaj koloni uyarıcı faktörü (hM-CSF) ile tamamlanmış ve% 5 CO2içeren bir atmosferde 37 ° C'de 4 gün boyunca inkübe edilir.
- 4 gün sonra ortamı% 10 FBS ve 40 ng / ml hM-CSF ile tamamlanan ve% 5 CO 2 içeren bir atmosferde 37 ° C'de 2 gün boyunca inkübe edilen taze RPMI1640ile değiştirin.
- 6 gün sonra, hücreler test için kullanılabilir.
Sonuçlar
Yüksek verimli genom çapında siRNA taraması
Mtb, immün hücreleri in vitro ve diğer birkaç akciğer epitel hücresini kolonize edebilir. Örneğin, Mtb,insan tipi II pnömosilatlar için model olarak yaygın olarak kullanılan A549 epitel hücrelerini enfekte edebilir ve zarar verebilir5-7. Dectin-1, A549 hücrelerinde hücre içi mikobakteriyel büyüme üzerinde Mtb alımı, proinflamatuar yanıt ve antibakteriyel etkide yer alan bir konak h...
Tartışmalar
Burada, yüksek içerikli / Yüksek verimli ekranlar için uygun hale getiren floresan etiketli konak hücreleri enfekte etmek için GFP ifade eden bir Mtb H37Rv suşu kullanarak fenotipik bir test için gerekli yöntemleri açıklıyoruz. Bu protokol çok çeşitli bileşiklere, floresan problara ve Mtb mutantlarına uygulanabilir. Yukarıda açıklanan her protokol için, görüntü alımından önce fiksasyon ve immünolabeling adımları gerçekleştirilebilir. Görüntü elde etmek için 20X (NA 0....
Açıklamalar
Çıkar çatışması bildirilmedi.
Teşekkürler
Bu çalışma için finansal destek Avrupa Topluluğu (ERC-STG INTRACELLTB Grant n° 260901, MM4TB Grant n° 260872), Agence Nationale de Recherche, Feder (12001407 (D-AL) Equipex Imaginex BioMed) ve Nord Pas de Calais Bölgesi tarafından sağlanmıştır. BICeL platformundan Gaspard Deloison, Elizabeth Werkmeister, Antonino Bongiovanni ve Frank Lafont'un teknik yardımını minnetle kabul ediyoruz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| µclear-plate black, 384-well | Greiner Bio-One | 781091 | 127.8/86/15 MM with Lid, TC treated |
| CellCarrier 384-well plate | PerkinElmer | 6007550 | Black, Clear Bottom, with Lid, TC treated |
| V-bottom white, 384-well plate | Greiner Bio-One | 781280 | |
| sealing tape, breathable, sterile | Corning | 3345 | |
| Lipofectamine RNAiMax | Life Technologies | 13778150 | Transfection reagent |
| Dimethyl sulfoxide | Sigma-Aldrich | 34943 | |
| RPMI 1640 + GlutaMAX-I | Life Technologies | 61870-010 | Cell culture medium |
| D-PBS 1x [-]MgCl2/[-]CaCl2 | Life Technologies | 14190-094 | Dulbecco's Phosphate Salin Buffer |
| D-PBS 1x [+]MgCl2/[+]CaCl2 | Life Technologies | 14190-091 | Dulbecco's Phosphate Salin Buffer |
| Fetal bovine serum | Life Technologies | 2610040-79 | |
| Ficoll Paque PLUS | Dutscher | 17-1440-03 | Ficoll for Peripherical Blood Monocyte Cells purification |
| CD14 MicroBeads, human | Miltenyi | 130-050-201 | Purification of CD14+ Monocytes |
| Human M-CSF, premium gr. (1000 μg) | Miltenyi | 130-096-493 | Macrophage Colony Stimulating Factor |
| LS Columns | Miltenyi | 130-042-401 | Columns for CD14+ Monocytes isolation |
| Tween 80 | Euromedex | 2002-A | Mycobacteria culture |
| Glycerol high purity | Euromedex | 50405-EX | Mycobacteria culture |
| Middlebrook OADC enrichment | Becton-Dickinson | 211886 | Mycobacteria culture |
| 7H9 | Becton-Dickinson | W1701P | Mycobacteria culture |
| Versene 1x | Life Technologies | 15040033 | Nonenzymatic cell dissociation solution |
| DAPI | Life Technologies | D1306 | Nuclei dye |
| Hoechst 33342 | Life Technologies | H3570 | Nuclei dye |
| Syto60 | Life Technologies | S11342 | Nuclei/cytoplasm dye |
| Formalin | Sigma-Aldrich | HT5014 | Cell fixation solution |
| siRNA targeting Dectin-1 | Santa-Cruz | sc-63276 | |
| *siGenome* Nontargeted siRNA pool | Dharmacon | D-001206-14 | |
| Rifampicin | Sigma-Aldrich | R3501 | antibiotic |
| Isoniazid (INH) | Sigma-Aldrich | I3377-50G | antibiotic |
| Hygromycin B | Life Technologies | 10687-010 | antibiotic |
| Amikacin | Sigma-Aldrich | A1774 | antibiotic |
| Automated Confocal Microscope OPERA | PerkinElmer | Image acquisition | |
| Columbus 2.3.1 Server Database | PerkinElmer | Data transfer, storage, and analysis | |
| Acapella 2.6 software | PerkinElmer | Image-based analysis | |
| GraphPad Prism5 software | GraphPad | Statistical analysis | |
| Excel 2010 | Microsoft | Statistical analysis |
Referanslar
- Gandhi, N. R., et al. Multidrug-resistant and extensively drug-resistant tuberculosis: a threat to global control of tuberculosis. Lancet. 375, 1830-1843 (2010).
- Barry, C. E., et al. The spectrum of latent tuberculosis: rethinking the biology and intervention strategies. Nat. Rev. Microbiol. 7, 845-855 (2009).
- Christophe, T., Ewann, F., Jeon, H. K., Cechetto, J., Brodin, P. High-content imaging of Mycobacterium tuberculosis-infected macrophages: an in vitro model for tuberculosis drug discovery. Future Med. Chem. 2, 1283-1293 (2010).
- Brodin, P., et al. High content phenotypic cell-based visual screen identifies Mycobacterium tuberculosis acyltrehalose-containing glycolipids involved in phagosome remodeling. PLoS Pathog. 6, (2010).
- Bermudez, L. E., Goodman, J. Mycobacterium tuberculosis invades and replicates within type II alveolar cells. Infect. Immun. 64, 1400-1406 (1996).
- Dobos, K. M., Spotts, E. A., Quinn, F. D., King, C. H. Necrosis of lung epithelial cells during infection with Mycobacterium tuberculosis is preceded by cell permeation. Infect. Immun. 68, 6300-6310 (2000).
- McDonough, K. A., Kress, Y. Cytotoxicity for lung epithelial cells is a virulence-associated phenotype of Mycobacterium tuberculosis. Infect. Immun. 63, 4802-4811 (1995).
- Lee, H. M., Yuk, J. M., Shin, D. M., Jo, E. K. Dectin-1 is inducible and plays an essential role for mycobacteria-induced innate immune responses in airway epithelial cells. J. Clin. Immunol. 29, 795-805 (2009).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J. Biomol. Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat. Methods. 6, 569-575 (2009).
- Carralot, J. P., et al. Automated high-throughput siRNA transfection in raw 264.7 macrophages: a case study for optimization procedure. J. Biomol. Screen. 14, 151-160 (2009).
- Zhang, X. D. Illustration of SSMD, z score, SSMD*, z* score, and t statistic for hit selection in RNAi high-throughput screens. J. Biomol. Screen. 16, 775-785 (2011).
- Song, O. R., et al. Confocal-based method for quantification of diffusion kinetics in microwell plates and its application for identifying a rapid mixing method for high-content/throughput screening. J. Biomol. Screen. 15, 138-147 (2010).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır