このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
電圧クランプ
要約
この記事では、マレイミド色素の代わりに蛍光非天然アミノ酸(fUAA)を用いて、イオンチャネルの構造的再配列を調べる従来の電圧クランプ蛍光光度法(VCF)の強化について説明します。この手順には、 アフリカツメガエル卵母細胞DNA注入、RNA / fUAA共注入および同時電流および蛍光測定が含まれる。
要約
電圧クランプ蛍光測定法(VCF)は、蛍光および電流のリアルタイム測定が局所再編成および全体的機能について同時に報告する、エレクトロジェニック膜タンパク質の構造および機能を調査するために選択された技術であった1 。低温電子顕微鏡法やX線結晶解析などの高分解能構造技術は、目的のタンパク質の静的画像を提供しますが、VCFは動的再構成(蛍光)を動的機能データ(電気生理学)にリンクさせる動的構造データを提供します。最近まで、タンパク質の部位特異的蛍光標識に使用されたチオール反応性化学は、内因性のシステインを含むすべての利用可能なシステインが標識されるため、アプローチの範囲を制限した。従って、内在性システインを含まないタンパク質を構築することが必要であった。標識はまた、細胞外からアクセス可能な部位に限定された側。これは、直交tRNAおよびtRNAシンテターゼ対2を用いた終止コドン抑制に応答して、小さな蛍光プローブを特異的に組み込むための蛍光不自然なアミノ酸 (fUAA)の使用により変化した。 VCFの改善は、DNA注入(tRNA /シンテターゼ対)の2段階注入手順とそれに続くRNA / fUAA同時注入を必要とするのみである。今や、細胞内および埋設サイトの両方の標識が可能であり、VCFの使用が大幅に拡大している。 VCF技術は広範囲のタンパク質を研究する上で魅力的になり、より重要なことには多数の細胞質ゾル調節機構を調べることが可能になる。
概要
大腸菌 、酵母および哺乳動物細胞において、様々な化学的および物理的性質の200種以上の非天然アミノ酸がタンパク質に遺伝子組み換えされている3 。非天然アミノ酸は、直交操作されたtRNA /シンテターゼ対を介して特異的終止コドンに応答して組み込まれる。タンパク質を改変するための遺伝的アプローチは、タンパク質の構造および機能についての貴重な洞察を提供している。ここでは、蛍光UAAと組み合わせてVoltage-Clamp Fluorometry(VCF)を使用するためのプロトコルを紹介します。
VCFでは、蛍光プローブの周囲に局在する機能的データと構造的再構成の同時観察(約5Å)により、ミリ秒の分解能で動的情報を得ることができます1 。蛍光プローブは、タンパク質の局在化した移動の際に消光状態を変化させる。唯一の1〜2Åの移動は、蛍光の顕著な変化をもたらすのに十分である強度4 。標的タンパク質における目的部位の同定の後、部位は点突然変異によって突然変異される。古典的には、残渣はシステインに変異していたが、今では、遺伝的fUAA取り込みのためにアンバーストップコドン(TAG)が導入されている。次いで、タンパク質をin vitroで転写する。
ツメガエル卵母細胞は、他の発現系( 例えば、哺乳動物細胞)を使用することができるが、サイズが大きいため、操作が簡単で蛍光強度が高い(フルオロホアが多い)ため、ノイズ比に影響します。さらに、 アフリカツメガエルの卵母細胞は、内因性タンパク質2,8に由来するバックグラウンドが低く、tからのバックグラウンド蛍光に対する動物の極の遮蔽に対する暗色色素沈着彼は細胞質ゾルです。 アフリカツメガエル卵母細胞を外科的に除去し、fUAAに特異的な直交tRNA / tRNAシンテターゼ対をコードするDNAを卵母細胞の核に注入する。 6〜24時間のインキュベーション時間後、タンパク質RNAをfUAAと共に卵母細胞の細胞質ゾルに同時注入し、その後2〜3日間のインキュベーション時間をおこなう。 fUAA(光漂白)の損傷を防ぐために、蛍光色素を避けるために赤色光下でAnapを含む手順を実施しなければならない。
卵母細胞は、直立蛍光顕微鏡に取り付けられた切開卵母細胞クランプセットアップで研究され、電流および蛍光の変化が同時に記録される( 9,10 ) 。あるいは、2電極電圧クランプ1またはパッチクランプ構成11を使用することができる。蛍光は、低いRMSノイズを有する適切な波長によって励起され、高い増幅度を有する増幅器にリンクされたフォトダイオードを使用して記録される。
電圧クランプ蛍光測定に蛍光非天然アミノ酸(fUAA)を使用することにはいくつかの利点があります。 1つは、膜タンパク質の細胞質ゾル側へのアクセスである。 ( 例えば、 Ca 2+ - またはヌクレオチド結合部位、電位依存性イオンチャネルの高速および閉鎖状態の不活性化、細孔開口、モジュール結合)には、多くの調節プロセスが存在する。これらのプロセスはすべて、蛍光標識のためにアクセス可能になりました。
別の利点は、プローブのサイズが小さくなり、タンパク質の妨害が少なくなることである。これまで、3-(6-アセチルナフタレン-2-イルアミノ)-2-アミノプロパン酸(Anap)がアフリカツメガエル卵母細胞に使用されている唯一のfUAAであるfUAAの2つの直交tRNA / tRNAシンテターゼ対が設計されている図2に示すように 、"xref"> 8。 Anapは、272.3g / molの分子量を有する環境に敏感なフルオロフォアであり、トリプトファン12 ( 図1A、1B )よりわずかに大きいだけである。そのサイズが小さいため、リンカーを介して結合した従来のフルオロフォア(典型的には500g / mol超)と比較して、フルオロフォアによって導入される可能性のある立体効果はより少ない。さらに、Anapの場合、フルオロフォアはシステインに連結されたものよりもタンパク質骨格の近くに位置し、その結果、Anapはより局在化した再配列をプロービングしている。最後に、従来のVCFにおける内在性システインの除去は、UAA-VCFではもはや必須ではなく、したがって(i)タンパク質を(ほとんど)それらの天然状態にし、(ii)VCFを適用させる機能がシステイン置換によって改変され得るより広い範囲のタンパク質を研究する。

図1 :アナプスおよび蛍光スペクトル。 ( A )Anapの化学構造。 ( B )1nMのAnapに対する標準化された吸収スペクトルおよび発光スペクトル。これは、Anap蛍光の溶媒疎水性に対する感受性を実証する。発光スペクトルは、350nmで励起することによって得られた。 この図の拡大版を見るには、ここをクリックしてください。
蛍光UAAを使用することの欠点は、アミノアシル化tRNAの量が不足している場合、タンパク質の異種集団が停止コドンのリードスルー、翻訳再開、C末端トランケートタンパク質または内因性アミノアシル化とのクロストークに起因し得ることである。このようなリーク表現は、fUAAとtRNA / tRNAシンテターゼの対が存在しない場合には常にチェックする必要があります。トランスの問題に取り組んだ以前にN末端挿入部位のためにそれを回避する方法を提供する。しかしながら、fUAA、tRNAおよびtRNAシンテターゼが飽和量で存在する場合、漏出発現の可能性は低いままである。
fUAA-VCFと従来のVCFとの主要な手順の違いは、卵母細胞の注入および取り扱いである。 tRNAおよびtRNAシンテターゼ(pAnap)をコードするDNAの注入に続いて、タンパク質mRNAと同時注入されるか、またはインキュベーション溶液にアセトキシメチル(AM)エステルとして添加される、Anapの導入が続く。
Access restricted. Please log in or start a trial to view this content.
プロトコル
カエルの操作はカナダのガイドラインに従って行われ、モントリオール大学の倫理委員会(CDEA、プロトコル#15-042)によって承認された。
1.fUAA組込みのためのmRNA調製
- 立体配座の変化が起こると予想されるタンパク質の関心のある部位を選択する。この領域のアミノ酸を選択して、fUAAを置換する。
注:ポジションの選択は、予想される構造上の並び替えに基づいています。高分解能構造が存在し、予想される動きの仮説がある場合には、化学環境が変化するように配置しなければならない。これは、誘電率の変化(疎水性対親水性環境)、またはおそらく別のアミノ酸によるクエンチングのいずれかである可能性がある。最良のクエンチャーはトリプトファンです。 Anapはある状態(ファンデルワールス半径の重なり)でクエンチャーと接触していて、そこから解放されていなければなりませんもう一方。高解像度の構造やモデルが存在しない場合は、関心領域をスキャンする必要があります。いずれの場合も、発現および蛍光シグナルを得る確率を高めるために、いくつかの近くの場所を選択することが推奨される。タンパク質成熟および/または機能の間の立体効果を最小限に抑えるために、大きな芳香族アミノ酸(Phe、Trp、Tyr)を置換することを選択することができる。しかしながら、著者らは、置換アミノ酸にかかわらず、fUAA挿入の関心領域を走査することがより生産的であることを経験している。 - 部位特異的突然変異誘発を用いて、選択された部位にアンバー停止コドン(TAG)を挿入する。目的のタンパク質がアンバーストップコドン(TAG)で終わらないことを確認する。そうであれば、別のもの(オーカーまたはオパール停止コドン)に変異させる。 DNAを増幅し、単離し、配列決定する。 インビトロ転写16でタンパク質mRNAを得、20℃または80℃でmRNAを保存する。
- アフリカツメガエルのカエルのステージVまたはVIの卵母細胞を外科的に入手し、コラゲナーゼで脱顆粒する。
- 承認された動物プロトコール(ここでは、3-アミノ安息香酸エチルエステル)に従って適切な麻酔薬でカエルを麻酔する。彼らがつま先先端に優しいピンチ(離脱反射の喪失)に反応しなかった場合、それらは手術のために適切に麻酔される。
- 直ちに麻酔液からカエルを取り除き、澄んだ水で肌をすすいでください。このすすぎは、吸収されていない化学物質を皮膚表面から除去することによって動物がより深いレベルの麻酔に落ちるのを防ぎます。
- 一方の側から卵巣節を外科的に取り除き、2つの鉗子を用いて慎重に節を開ける。 1%(w / v)コラゲナーゼを含む「標準卵母細胞溶液」(SOS)中で卵母細胞を20〜30分間インキュベートし、攪拌する。脱胞化する。 SOS溶液で3回洗浄する。
- 大型の健康な卵子を個別に選択し、抗生物質(100U / mLペニシリン、100μg/ mLストレプトマイシン、10mg / 100mLカナマイシン)および5%ウマ血清を18℃で少なくとも4時間注入したバース溶液。
注:2〜4回の手術の後、4ヶ月間遅延させると、 アフリカツメガエルは、3-アミノ安息香酸エチルエステルとの長期インキュベーション(> 1時間)によって安楽死させる。
- DNAの核注入のためには、核に達し、卵母細胞の損傷を避けるために、細長い注射チップを準備する。注入チップにオイルを満たし、ナノインジェクタデバイスにマウントします。
- ステレオ顕微鏡下にナノインジェクタを取り付け、鉗子を使って先端の端を壊します。先端の内側に気泡が閉じ込められなくなるまでオイルを排出します。
- 1μLの0.1μg/#181; L立体顕微鏡下のパラフィルム上にNaOH(1N NaOHの1%)を含むヌクレアーゼフリーの水に入れ、DNAを注射チップに充填する。
- 40の卵母細胞を、抗生物質が補充されたバース溶液を含むメッシュコート注入皿に移す。
注:メッシュでコーティングされた注入ディッシュを作成するには、ポリスチレンペトリ皿に充填するために、800μmナイロンメッシュの適切なサイズの断片を切り取ってください。中央にクロロホルムを加え、メッシュを上に置きます。プラスチックがセットされるまでメッシュを平らにします。 - 卵母細胞核は動物の(暗い)極に位置するので、注射極を動物極の中心に向け、先端が動物の半球の中心近くに達するように倒す(またはRNA注入と比較して深さを2〜3倍にする) )。 9.2nLのpAnapを各卵母細胞の核に注入する。先端が細く、注入量が少ないと、注入が不規則になったり、先端が詰まったりすることがあります。空気中に注入して注射が機能するかどうかを確認することがあります。
注:DNAが核に正しく注入されるかどうかは不明です。したがって、卵母細胞の10〜40%がtRNA /シンテターゼの対を発現しないことを期待する。詳細については、ディスカッションを参照してください。 - 抗生物質と5%ウマ血清(HS)を添加した2 mLのBarth溶液に卵母細胞を18℃で6〜24時間インキュベートして、Anap特異的tRNAとtRNA合成酵素の強力な発現を可能にする。
注:DNAインキュベーション時間はRNA注入の数日前に持続することがありますが、発現は増加しません。 - RNA注入用にナノインジェクタを準備します(ステップ2.2と同じですが、注入チップはDNA注入ほど薄くする必要はありません)。 Anapの光退色を防ぐために、この時点から赤色光の下でのみ作業してください。
- 1μLの1mMのAnapと1μLの1μLの1-2μg/μLのmRNAを直接1枚のパラフィルムに混合し、注入チップに混合溶液を充填する。植物のすぐ下の膜(明るい)極に注入し、各pAnap注入卵母細胞に46nLを注入する。
注:必要なmRNA濃度は目的のタンパク質に依存します。 - ボックスの中で光から保護された卵母細胞をインキュベートするか、またはアルミホイルで包まれたBarthの溶液で抗生物質と5%ウマ血清を18℃で2〜3日間インキュベートする。新鮮なバースの溶液で毎日交換し、汚染を避けるために死んだ卵子を除去してください。
- 前述の18のように切開した卵母細胞電圧クランプ装置を設置する。
- 卵母細胞を置くための標準立体顕微鏡と蛍光測定を行うための顕微鏡との間でそれを動かすことができるスライダに記録チャンバを設置することによって、電気生理学的記録システムを直立蛍光顕微鏡に取り付ける( 図2c )。
注:切開卵母細胞のチャンバーの形状は、通常のトラを使用するのには適していません操作中の照明のための光を照らす。従って、赤フィルタを有する「グースネック」ハロゲンランプは、上から横に照明するために使用される。顕微鏡のコンデンサーを取り外して、電気生理学チャンバーのステージを下げる空間を作ることができます。 - フォトダイオード検出システムを蛍光顕微鏡のCマウント出口ポートに接続します( 図2a )。デジタル信号プロセッサ(DSP、アナログ/デジタル - デジタル/アナログ変換器)の第2の入力チャネルに光電流読み出し値を接続します。
- 蛍光励起のための光源として100 W、12 Vハロゲンランプを使用してください。
注記:あるいは、Hgバーナーを使用することもできますが、録画中に急速に光退色するのを防ぐために、強度を下げなければなりません。 LED照明は、それぞれのLEDが励起範囲内で有意な強度( 例えば、 Anapの場合〜350nm)を示す場合にのみ推奨されます。ほとんどの白色LEDは、UVスペクトルの遠くまで到達しません。 - Ins励起光源と顕微鏡の間に電気的に制御されたシャッターを設置し、そのコントロール(通常はTTLパルス)をDSPのデジタル出力に接続します。録音ソフトウェアのTTLパルスを計時してください(メーカーのドキュメントを参照)。録音の開始前にシャッターが約100ms開くようにしてください。このようにして、開口プロセス中の振動は記録を妨害しない。時間はシャッターの速度と振動に依存します。 図4に示すように、録音終了の5ms前にパルスを終了します。このようにして、全蛍光の値も記録される。
- フィルタキューブタレットに適切なフィルタキューブ(励起フィルタ、ダイクロイックミラー、エミッションフィルタ)を挿入します。 Anapでは、Ex:377 / 50nmバンドパス、ダイクロイック409nmロングパス、Em:470 / 40nmバンドパスを使用します。
- 以前に記載されているようにカットオープン卵母細胞の電圧クランプのための準備ステップに従ってください18 (寒天ブリッジ調製、卵母細胞をマウントする、サポニン透過性)。しかし、記録の前に蛍光体の漂白を避けるために、赤色光の下では常に作業してください。卵母細胞を置くときは、動物の極が上を向いていることを確認してください。動物極膜の下での色素沈着は、細胞質ゾル由来の自己蛍光に対して遮蔽し、したがってバックグラウンド蛍光を減少させる。
- チャンバーを顕微鏡にスライドさせ、4Xの対物レンズを使用して焦点を合わせます。
- 電圧感知V1電極(3M KCl)で卵母細胞を倒し、40倍の水浸漬(NA 0.8 - 0.9)の対物レンズに切り替える。上方を向いている動物の極に焦点を当てる。
- 赤いライトを消す。フィルタキューブタレットとフォトダイオードに接続された光出口ポートを回して、右のフィルタキューブを選択します。ハローグをオンにするそして2〜5秒間、シャッターを短く切り換えて、卵母細胞に由来するバックグラウンド蛍光強度を読み取る。記載された設定では、値はAnapのために約50-200pAでなければならない。
- クランプをオンにし、浴/ガードスイッチをアクティブにして、Iヘッドステージのつまみを回して膜電位(V1 - V2)を指示電位に調整します。
- 録音ソフトウェアで保持ポテンシャル、ステッププロトコル、パルス数や長さなどを選択します。電圧に依存する電流およびAnap蛍光強度を記録する。
- 同じタンパク質中の2つの位置を同時にモニターするために、細胞外および接近可能なアミノ酸をシステインに突然変異させ、他のシステインを除去してチオール化学による特異的標識を保証する。
- ステップ2.1-2.5を実行します。
- VCF記録の前に、標識溶液中の5μMTMR-マレイミド中の卵母細胞を15分間インキュベートする(または他の色素Anapと比較して非重複スペクトルで)。
- 余分な色素を除去するために標識溶液で卵母細胞を3回洗浄する。
- ステップ4.1-4.6を実行します。
- フィルタキューブタレットにTMR(励起フィルタ、ダイクロイックミラー、エミッションフィルタ)用の適切なフィルタキューブを挿入します。フィルタータレットを回してTMRフィルターキューブに切り替えます。
- ステップ4.4で説明したように、TMRのバックグラウンド蛍光を読み取る。
注:チオールケミストリで標識すると、膜内の非特異的な標識により高いバックグラウンド蛍光が生じます。したがって、TMRバックグラウンド蛍光はアンプを飽和させることがあります(> 2,000 pA)。その場合、光強度を減少させないでください。ただし、オフセット電流をフォトダイオードに加えることによって、バックグラウンド蛍光を減算するだけです。市販されているシステムでは、検出器システムの「サンプル&ホールド」機能を使用します。実験室でのバックグラウンド蛍光値(10倍濃度フィルターを使用)に注意してくださいこの値は記録されません(彩度)。 - ステップ4.6のように、電圧依存性電流およびTMR蛍光強度を同時に記録する。
2.卵母細胞の調製および注入
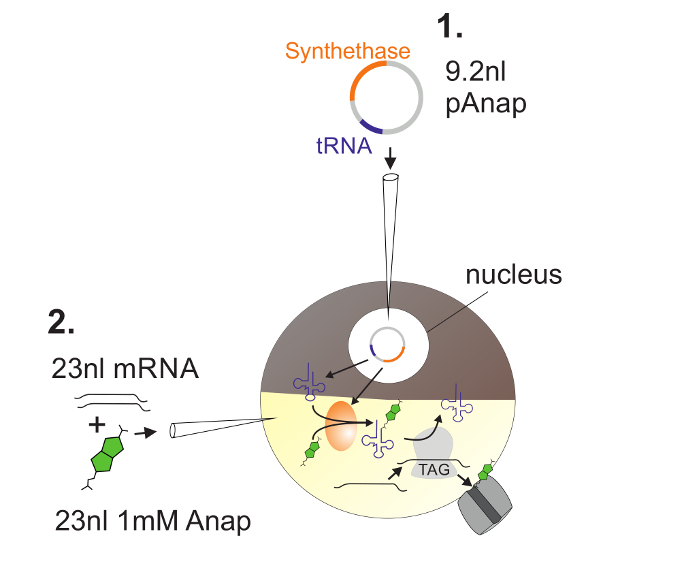
図 2 :Anap Incorporationの ための Xenopus 卵母細胞 へのDNAおよびRNA注入の 図 。
まず、pAnapをアフリカツメガエル卵母細胞の核に注入する( 1 )。 6〜24時間後、AnapおよびチャネルRNAを植物の極に同時注入する( 2 )。 Anapは、pAnapによってコードされるアミノアシル-tRNAシンテターゼによって、アンバーストップ抗コドンを有するtRNAと直角にアミノアシル化される。このようにして、アミノアシル化されたAnap-tRNAは、挿入されたアンバー停止コドンでリボソームによって認識されるl RNAを切断し、停止コドンの抑制およびAnapの挿入をもたらす。 この図の拡大版を見るには、ここをクリックしてください。
3. VCFセットアップ

図3 < / strong> :VCF設定。 ( A )顕微鏡内部の光路を示すVCFセットアップの側面図。フィルタキューブは、励起フィルタ、ダイクロイックミラー、および放出フィルタを含む。 ( B )選択された卵母細胞チャンバーの寸法は、上部チャンバー半径(1)に対して3.4cm、下部チャンバー長さ(2)については5.5cm、下部チャンバー幅(3)については1.4cm、そして中央チャンバー幅(4)については1.7cmである。 ( C )VCFセットアップの正面図。左の第1の接眼レンズは、卵母細胞を切開型電圧クランプチャンバーに取り付け、透過性化するためのものである。次に、チャンバーを第2接眼レンズの右側の顕微鏡下でスライドさせる。ここでは、V1電極を4X対物レンズを用いて卵母細胞に挿入し、水浸40X対物レンズを用いて蛍光を記録する。 この図の拡大版を見るには、ここをクリックしてください。
5. 2色VCF
Access restricted. Please log in or start a trial to view this content.
結果
図4は、pAnapおよびAnapの存在下で高速不活性除去(IR)、L382stop-W434Fを有するシェーカーチャネルを発現する卵母細胞から得られたVCF記録の例を示す。 W434F突然変異はイオン性カリウム電流を遮断し、過渡ゲーティング電荷の移動(ゲーティング電流)を測定することを可能にする。脱分極時のゲーティング電流(上部トレース)とAnap蛍光強度の変化(下?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
tRNA-シンテターゼと共に連続的に転写されるtRNAのin vivoアミノアシル化は、蛍光測定のための高発現レベルを得ることを可能にする。効率的なfUAA取り込みのためには、pAnapが核内に正確に注入されることが重要である。核の正確な位置の不確定性のために、DNA注射の10〜40%が失敗すると予想され、非発現(または漏出発現)卵母細胞が生じる。したがって、AnapとpAnapの非存在下で発現?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者は何も開示することはない。
謝辞
pAnapはPeter Schultz博士(Scripps Research Institute)からの贈り物でした。この研究は、カナダ保健研究助成金MOP-102689とMOP-136894(RBへ)とカナダ財団法人イノベーショングラント950-225005によって資金提供されました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Solutions | |||
| Barth's solution | |||
| NaCl | Sigma-Aldrich | S7653 | 90 mM |
| KCl | Fisher Scientific | BP366-500 | 3 mM |
| MgSO4 | Sigma-Aldrich | M-9397 | 0.82 mM |
| CaCl2 | Sigma-Aldrich | C-7902 | 0.41 mM |
| Ca(NO3)2 | Sigma-Aldrich | C-1396 | 0.33 mM |
| HEPES | Sigma-Aldrich | H4034 | 5 mM |
| NaOH hydrate | BDH | BDH7225-4 | pH 7.6 |
| Penicilin | Invitrogen | 15140122 | 100 U/mL |
| Streptomycin | Invitrogen | 15140122 | 100 µg/mL |
| Kanamycin | Invitrogen | 15160054 | 10 mg/100mL |
| Horse Serum (HS) | Invitrogen | 16050122 | 5% |
| SOS Standard Oocyte Solution | |||
| NaCl | Sigma-Aldrich | 746398 | 102 mM |
| KCl | Sigma-Aldrich | 746436 | 3 mM |
| MgCl2 | Sigma-Aldrich | M9272 | 1 mM |
| HEPES | Sigma-Aldrich | H4034 | 5 mM |
| External recording solution | |||
| N-methyl-D-glucamine (NMDG) | Alfa Aesar | L14282 | 115 mM |
| HEPES | Sigma-Aldrich | H4034 | 10 mM |
| Calcium hydroxide | Sigma-Aldrich | 239232 | 2 mM |
| MES hydrate | Sigma-Aldrich | 258105 | pH 7.2 |
| Internal recording solution | |||
| N-methyl-D-glucamine (NMDG) | Alfa Aesar | L14282 | 115 mM |
| HEPES | Sigma-Aldrich | H4034 | 10 mM |
| Ethylenediamine Tetraacetic Acid (EDTA) | Fisher Scientific | E478-500 | 2 mM |
| MES hydrate | Sigma-Aldrich | 258105 | pH 7.2 |
| Labeling solution | |||
| KOH | Fisher Scientific | P250-1 | 115 mM |
| HEPES | Sigma-Aldrich | H4034 | 10 mM |
| Calcium hydroxide | Sigma-Aldrich | 239232 | 2 mM |
| MES hydrate | Sigma-Aldrich | 258105 | pH 7.2 |
| TMR stock solution | |||
| Tetramethylrhodamine-5-maleimide (TMR) | Molcular Probes by Life Technologies | T6027 | 5 mM in DMSO |
| Anap stock solution | |||
| Anap | ABZENA (TCRS) | Custom synthesis TCRS-170 | 1 mM in nuclease-free water and 1% NaOH 1 N |
| Name | Company | Catalog Number | Comments |
| Material/Equipment | |||
| pAnap | Addgene | 48696 | |
| High Performance Oocyte Clamp | Dagan Corporation | CA-1B | |
| Gpatch Acquisition software | Department of Anesthesiology, University of California, Los Angeles | ||
| Analysis software | Department of Anesthesiology, University of California, Los Angeles | ||
| Recording Chamber | Custom machined | ||
| Photo diode detection system | Dagan Corporation | PhotoMax-200/PIN | |
| Electrical shutter driver | UNIBLITZ | VCM-D1 |
参考文献
- Mannuzzu, L. M., Moronne, M. M., Isacoff, E. Y. Direct physical measure of conformational rearrangement underlying potassium channel gating. Science. 271 (5246), 213-216 (1996).
- Kalstrup, T., Blunck, R. Dynamics of internal pore opening in K(V) channels probed by a fluorescent unnatural amino acid. Proc Natl Acad Sci U S A. 110 (20), 8272-8277 (2013).
- Xiao, H., Schultz, P. G. At the Interface of Chemical and Biological Synthesis: An Expanded Genetic Code. Cold Spring Harb Perspect Biol. 8 (9), (2016).
- Blunck, R. Chapter 9. Handbook of Ion Channels. , CRC Press. 113-133 (2015).
- Chatterjee, A., Guo, J., Lee, H. S., Schultz, P. G. A genetically encoded fluorescent probe in mammalian cells. J Am Chem Soc. 135 (34), 12540-12543 (2013).
- DeBerg, H. A., Brzovic, P. S., Flynn, G. E., Zagotta, W. N., Stoll, S. Structure and Energetics of Allosteric Regulation of HCN2 Ion Channels by Cyclic Nucleotides. J Biol Chem. 291 (1), 371-381 (2016).
- Shen, B., et al. Genetically encoding unnatural amino acids in neural stem cells and optically reporting voltage-sensitive domain changes in differentiated neurons. Stem Cells. 29 (8), 1231-1240 (2011).
- Aman, T. K., Gordon, S. E., Zagotta, W. N. Regulation of CNGA1 Channel Gating by Interactions with the Membrane. J Biol Chem. 291 (19), 9939-9947 (2016).
- Haddad, G. A., Blunck, R. Mode shift of the voltage sensors in Shaker K+ channels is caused by energetic coupling to the pore domain. J Gen Physiol. 137 (5), 455-472 (2011).
- Batulan, Z., Haddad, G. A., Blunck, R. An intersubunit interaction between S4-S5 linker and S6 is responsible for the slow off-gating component in Shaker K+ channels. J Biol Chem. 285 (18), 14005-14019 (2010).
- Kusch, J., et al. How subunits cooperate in cAMP-induced activation of homotetrameric HCN2 channels. Nat Chem Biol. 8 (2), 162-169 (2012).
- Lee, H. S., Guo, J., Lemke, E. A., Dimla, R. D., Schultz, P. G. Genetic incorporation of a small, environmentally sensitive, fluorescent probe into proteins in Saccharomyces cerevisiae. J Am Chem Soc. 131 (36), 12921-12923 (2009).
- Summerer, D., et al. A genetically encoded fluorescent amino acid. Proc Natl Acad Sci U S A. 103 (26), 9785-9789 (2006).
- Kalstrup, T., Blunck, R. Reinitiation at non-canonical start codons leads to leak expression when incorporating unnatural amino acids. Sci Rep. 5, 11866(2015).
- Liu, H., Naismith, J. H. An efficient one-step site-directed deletion, insertion, single and multiple-site plasmid mutagenesis protocol. BMC Biotechnol. 8, 91(2008).
- Beckert, B., Masquida, B. Synthesis of RNA by in vitro transcription. Methods Mol Biol. 703, 29-41 (2011).
- Goldin, A. L. Maintenance of Xenopus laevis and oocyte injection. Methods Enzymol. 207, 266-279 (1992).
- Rudokas, M. W., Varga, Z., Schubert, A. R., Asaro, A. B., Silva, J. R. The Xenopus oocyte cut-open vaseline gap voltage-clamp technique with fluorometry. J Vis Exp. (85), (2014).
- Zhao, J., Blunck, R. The isolated voltage sensing domain of the Shaker potassium channel forms a voltage-gated cation channel. Elife. 5, (2016).
- Posson, D. J., Ge, P., Miller, C., Bezanilla, F., Selvin, P. R. Small vertical movement of a K+ channel voltage sensor measured with luminescence energy transfer. Nature. 436 (7052), 848-851 (2005).
- Chanda, B., Asamoah, O. K., Blunck, R., Roux, B., Bezanilla, F. Gating charge displacement in voltage-gated ion channels involves limited transmembrane movement. Nature. 436 (7052), 852-856 (2005).
- Taraska, J. W., Puljung, M. C., Zagotta, W. N. Short-distance probes for protein backbone structure based on energy transfer between bimane and transition metal ions. Proc Natl Acad Sci U S A. 106 (38), 16227-16232 (2009).
- Baker, B. J., et al. Genetically encoded fluorescent sensors of membrane potential. Brain Cell Biol. 36 (1-4), 53-67 (2008).
- Miranda, P., et al. State-dependent FRET reports calcium- and voltage-dependent gating-ring motions in BK channels. Proc Natl Acad Sci U S A. , (2013).
- Sisido, M., Ninomiya, K., Ohtsuki, T., Hohsaka, T. Four-base codon/anticodon strategy and non-enzymatic aminoacylation for protein engineering with non-natural amino acids. Methods. 36 (3), 270-278 (2005).
- Hohsaka, T., Ashizuka, Y., Murakami, H., Sisido, M. Five-base codons for incorporation of nonnatural amino acids into proteins. Nucleic Acids Res. 29 (17), 3646-3651 (2001).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved