JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
전압 클램프 형광 측정법
요약
이 기사에서는 이온 채널에서 구조적 재배치를 조사하기 위해 말레이 미드 염료 대신 형광 비정상적인 아미노산 (fUAA)이 사용되는 기존의 VCF (Voltage-Clamp Fluorometry)의 향상에 대해 설명합니다. 이 절차에는 Xenopus oocyte DNA 주입, RNA / fUAA 동시 주입, 동시 전류 및 형광 측정이 포함됩니다.
초록
Voltage-Clamp Fluorometry (VCF)는 형광 및 전류의 실시간 측정이 국소 재배치 및 전지구 기능에 대해 동시에보고하는 전자 제 멤브레인 단백질의 구조 및 기능을 조사하기위한 기술이었습니다 1 . 저온 전자 현미경 또는 X 선 결정학과 같은 고해상도 구조 기술은 관심있는 단백질의 정적 이미지를 제공하지만 VCF는 구조적 재배치 (형광)를 동적 기능 데이터 (전기 생리학)에 연결할 수있는 동적 구조 데이터를 제공합니다. 최근까지 내재적 인 시스테인을 포함하여 접근 가능한 모든 시스테인이 분류 될 것이기 때문에 단백질의 부위 지시 형광 표지에 사용 된 티올 반응 화학은 접근법의 범위를 제한했다. 따라서 내인성 시스테인이없는 단백질을 만들어야했습니다. 표지는 또한 세포 외로부터 접근 가능한 사이트로 제한되었다측면. 이것은 직각 tRNA 및 tRNA 합성 효소 쌍을 사용하여 정지 코돈 억제에 대한 반응으로 작은 형광성 프로브를 구체적으로 통합하기 위해 형광 비 자연 아미노산 ( Fluorescent Unnatural Amino Acids , fUAA)을 사용하여 변경되었습니다. VCF 개선은 DNA 주입 (tRNA / synthetase 쌍)의 2 단계 주입 절차와 RNA / fUAA 동시 주입을 필요로합니다. 이제 세포 내 및 묻힌 부위 모두에 라벨을 붙일 수 있으며 VCF의 사용이 크게 늘어났습니다. VCF 기술은 광범위한 단백질 연구에 매력적이며, 더 중요한 것은 많은 세포질 조절 메커니즘을 연구 할 수 있다는 것입니다.
서문
다양한 화학적 및 물리적 특성을 지닌 200 개가 넘는 비 천연 아미노산이 대장균 , 효모 및 포유 동물 세포에서 단백질로 유 전적으로 도입되었습니다 3 . 비 천연 아미노산은 직교 조작 된 tRNA / 신테 타제 쌍을 통해 특정 종결 코돈에 반응하여 도입된다. 단백질을 수정하는 유전자 접근법은 단백질 구조와 기능에 대한 중요한 통찰력을 제공합니다. 여기서는 형광 UAA와 함께 전압 - 클램프 형광 측정 (VCF)을 사용하기위한 프로토콜을 제시합니다.
VCF에서 형광 프로브 주위에 위치하는 기능적 데이터와 구조적 재구성을 동시에 관찰하면 밀리 초 분해능 1의 동적 정보를 얻을 수 있습니다. 형광 프로브는 단백질의 국부적 인 움직임에 따라 급냉 상태를 변경합니다. 단지 1-2Å의 움직임만으로도 형광의 현저한 변화를 유도 할 수 있습니다강도 4 . 표적 단백질에서 관심 부위의 동정 후, 부위는 점 돌연변이에 의해 돌연변이된다. 고전적으로, 잔기는 시스테인으로 돌연변이되었지만, 지금은 황색 정지 코돈 (TAG)이 유전 fUAA 도입을 위해 도입되었다. 그 단백질은 시험 관내에서 전사된다.
다른 발현 시스템 ( 예 : 포유 동물 세포) 5 , 6 , 7 , Xenopus의 난 모세포는 구조 기능 연구에 더 큰 크기 때문에 더 쉽게 조작하고 높은 형광 강도 (더 fluorophores)로 이어질 수 있으므로, 노이즈 비율. 또한, Xenopus 난 모세포는 내인성 단백질 2 , 8 의 배경이 낮고 동물의 극 보호막에 어두운 색소가있어 t그는 세포질이다. Xenopus 난 모세포를 외과 적으로 제거하고 fUAA에 특이적인 직교 tRNA / tRNA 합성 효소 쌍을 암호화하는 DNA를 난 모세포의 핵으로 주입한다. 6-24 시간의 배양 시간 후, 단백질 RNA를 fUAA와 함께 난 모세포의 세포질에 주입하고 2-3 일간 배양한다. fUAA (photobleaching)의 손상을 방지하기 위해 적색광 하에서 형광체 여기를 피하기 위해 Anap을 포함한 절차를 수행해야합니다.
난 모세포는 직립 형광 현미경에 장착 된 컷 오픈 oocyte 전압 클램프 설정에서 연구하고, 전류와 형광 변화는 동시에 9 , 10 기록됩니다. 대안으로, 2- 전극 전압 클램프 ( 1) 또는 패치 - 클램프 구성 (11) 이 사용될 수있다. 형광은 낮은 RMS 잡음을 갖는 적절한 파장에 의해 여기되고 높은 증폭을 갖는 증폭기에 연결된 포토 다이오드를 사용하여 기록 된 방사.
전압 클램프 형광 측정법에 형광 비 천연 아미노산 (fUAAs)을 사용하면 몇 가지 장점이 있습니다. 하나는 막 단백질의 세포질 측에 대한 접근이다. 많은 조절 과정이 여기에 있습니다 ( 예 : Ca 2+ 또는 뉴클레오타이드 결합 부위, 전압 게이 티드 이온 채널의 빠른 및 폐쇄 상태 불 활성화, 기공 개방, 모듈 커플 링). 이 모든 공정에 형광 물질 표시가 가능합니다.
또 다른 이점은 작은 크기의 탐침으로 단백질의 방해가 적다는 것입니다. 지금까지, fUAA를위한 두 개의 직교 tRNA / tRNA 합성 효소 쌍이 조작 된 12 , 13 , 3- (6- 아세틸 나프탈렌 -2- 일 아미노) -2- 아미노 프로판 산 (Anap)은 Xenopus 난 모세포에서 사용 된 유일한 fUAA이다 도 2에 도시 된 바와 같이 ,"xref"> 8. Anap은 272.3 g / mol의 분자량을 지닌 환경 적으로 민감한 형광 단이며 트립토판 12 보다 약간 크다 ( 그림 1A, 1B ). 크기가 작기 때문에 링커 (일반적으로 500 g / mol 이상)를 통해 부착 된 기존 형광체와 비교하여 형광체가 입체 효과를 거의 일으키지 않습니다. 또한, Anap의 경우, 형광체는 시스테인에 연결된 것보다 단백질 백본에 더 가까이 위치하므로 결과적으로 Anap은 더 국부적 인 재배치를 탐색합니다. 마지막으로, UAA-VCF에서 부위 특이 적 표지를 보장하기 위해 기존의 VCF에서 내인성 시스테인을 제거함으로써 (i) 단백질을 (거의) 본래의 상태로두고 (ii) VCF를 적용 할 수있게한다 기능이 시스테인 치환에 의해 변경 될 수있는 더 넓은 범위의 단백질을 연구한다.

그림 1 : Anap and Fluorescence Spectra. ( A ) Anap의 화학 구조. ( B ) 1nM 아나프에 대한 표준화 된 흡수 스펙트럼 및 방출 스펙트럼, 아나 프 형광의 용매 소수성에 대한 민감성을 입증 함. 방출 스펙트럼은 350 nm에서 여기하여 얻은 것이다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
형광 UAA의 단점은 aminoacylated tRNA의 양이 부족하면 stop codon readthrough, translational reinitiation, C-terminal truncated proteins 또는 내인성 aminoacylation과의 crosstalk로 인해 단백질의 이종 집단이 생길 수 있다는 것입니다. 이러한 누출 발현은 fUAA와 tRNA / tRNA 합성 효소 쌍이없는 경우 항상 검사해야합니다. 우리는 트랜스의 문제를 다루었습니다.이전에 N 말단 삽입 부위에 대해 이성적 재발 및 그것을 막는 방법 14 . 그러나, fUAA, tRNA 및 tRNA 합성 효소가 포화 된 양으로 존재할 때, 누출 발현 가능성은 낮다.
fUAA-VCF와 재래식 VCF의 주요 절차상의 차이점은 난 모세포의 주입과 취급이다. tRNA 및 tRNA 합성 효소 (pAnap)를 코딩하는 DNA의 주입 후에 단백질 mRNA와 함께 주입되거나 양자 택일로 아세 톡시 메틸 (AM) 에스테르로서 배양 용액에 첨가되는 아나 (Anap)의 도입이 뒤 따른다.
프로토콜
개구리 조작은 캐나다 지침에 따라 수행되었으며 몬트리올 대학의 윤리위원회 (CDEA, 프로토콜 # 15-042)의 승인을 받았습니다.
1. fUAA 설립을위한 mRNA 준비
- 구조 변화가 발생할 것으로 예상되는 단백질의 관심 사이트를 선택하십시오. 이 지역의 아미노산을 선택하여 fUAA로 대체하십시오.
참고 : 위치 선택은 예상되는 구조적 재 배열을 기반으로합니다. 고해상도 구조가 존재하고 예상되는 움직임에 대한 가설이있는 경우, 화학적 환경이 변경 될 수 있도록 배치해야합니다. 이는 유전 상수의 변화 (소수성 대 친수성 환경) 또는 다른 아미노산에 의한 급냉 가능성이 있습니다. 가장 좋은 소독제는 트립토판입니다. Anap은 하나의 상태 (van der Waals 반경의 중복)에서 quencher와 접촉해야하고다른 하나. 고해상도 구조 또는 모델이 없으면 관심 영역을 스캔해야합니다. 두 경우 모두 표현과 형광 신호를 얻을 확률을 높이기 위해 몇 개의 가까운 위치를 선택하는 것이 좋습니다. 단백질 성숙 및 / 또는 기능을하는 동안 입체 효과를 최소화하기 위해 크고 방향성이있는 아미노산 (Phe, Trp, Tyr)을 대체 할 수 있습니다. 그러나 저자들은 치환 된 아미노산에 관계없이 fUAA 삽입에 대한 관심 부위를 스캔하는 것이 생산성이 높다는 것을 경험했다. - 사이트 지정 돌연변이 유발 15을 사용하여 선택한 사이트에서 호박색 정지 코돈 (TAG)을 삽입합니다. 해당 단백질이 호박색 정지 코돈 (TAG)에서 끝나지 않도록하십시오. 그렇다면 다른 하나 (황토 또는 오팔 정지 코돈)로 돌연변이하십시오. DNA를 증폭, 분리 및 염기 서열 화하십시오. 시험관 전사 16 에서 단백질 mRNA를 얻고 20 ° C 또는 80 ° C에서 mRNA를 저장하십시오.
- 외과 적으로 Xenopus laevis 개구리에서 Stage V 또는 VI oocytes를 얻고 이전에 설명한 17 과 같이 콜라게나 제를 사용하여 제거하십시오.
- 승인 된 동물 프로토콜 (여기 : 3- 아미노 안식향산 에틸 에스테르)에 따라 적절한 마취제로 개구리를 마취합니다. 발끝 (완치 반사의 상실)에 완치에 반응하지 못하면 수술을 위해 적절하게 마취됩니다.
- 즉시 마취 용액에서 개구리를 제거하고 담수로 피부를 깨끗이 헹구십시오. 이 헹굼은 흡수되지 않은 화학 물질을 피부 표면에서 제거하여 동물이 마취의 더 깊은 수준으로 떨어지는 것을 방지합니다.
- 한쪽에서 난소 노드를 외과 적으로 제거하고 조심스럽게 두 포셉을 사용하여 노드를 엽니 다. 20-30 분 동안 1 % (w / v) 콜라게나 제 함유 "표준 난 모세포 용액"(SOS)에서 난 모세포를 항온 배양하십시오.탈모증시킨다. SOS 용액으로 세 번 씻으십시오.
- 크고 건강한 난 모세포를 개별적으로 선택하고 주입 전 최소 4 시간 동안 항생제 (100 U / mL 페니실린, 100 μg / mL 스트렙토 마이신, 10 mg / 100 mL 카나마이신)와 5 % 말 혈청을 보충 한 바스 용액 .
참고 : 중간에 4 개월 지연된 2 ~ 4 회 수술 후, Xenopus laevis 는 3- 아미노 안식향산 에틸 에스테르와의 장기 배양 (> 1 시간)에 의해 안락사됩니다.
- DNA의 핵 주입을 위해 핵에 도달 할 수 있고 난 모세포 손상을 피하기 위해 길고 얇은 주입 팁을 준비하십시오. 주입 팁에 오일을 채우고 nanoinjector 장치에 장착하십시오.
- 나노 인젝터를 스테레오 현미경으로 설치하고 포 셉을 사용하여 팁 끝 부분을 부순다. 팁 끝 부분에 기포가 갇히지 않을 때까지 오일을 꺼내십시오.
- 1 μL의 0.1 μg /입체경 아래의 파라 필름에 NaOH (1N NaOH의 1 %)가 포함 된 핵산 분해 효소가없는 물을 넣고 DNA로 주입 팁을 채 웁니다.
- 항생제가 보충 된 Barth의 솔루션을 포함하는 메쉬 코팅 주입 접시에 40 oocytes을 전송합니다.
참고 : 메쉬 코팅 된 주입 접시를 만들려면 폴리스티렌 페트리 접시를 채우기 위해 800 μm 나일론 메쉬의 적절한 크기의 조각을 잘라냅니다. 중앙에 클로로포름을 넣고 그 위에 메시를 놓습니다. 플라스틱이 놓일 때까지 메쉬를 평평하게 유지하십시오. - oocyte nucleus가 동물 (어두운) 극에 위치하기 때문에, 주입 극을 동물 극의 중심에 맞추고 팁이 동물 반구의 중심 근처에 도달하도록 부 풀리십시오 (또는 RNA 주입에 비해 2-3 배 깊이) ). 9.2 nL의 pAnap을 각 난 모세포의 핵에 주입하십시오. 팁이 얇고 주입량이 적 으면 주입이 불규칙하거나 팁이 막힐 수 있습니다. 때때로 공기 주입식으로 주사가 작동하는지 확인하십시오..
참고 : DNA가 핵에 제대로 주입되는지 여부는 불확실합니다. 따라서 oocytes의 10 ~ 40 %가 tRNA / synthetase 쌍을 발현하지 않을 것으로 기대합니다. 자세한 내용은 토론을 참조하십시오. - 항생제와 5 % 말 혈청 (HS)을 보충 한 2 mL 바스 (Barth) 용액에 18 o C의 난 모세포를 넣고 6 ~ 24 시간 동안 Anap 특이 적 tRNAs와 tRNA- 신테 타제의 강력한 발현을 허용하십시오.
참고 : DNA 배양 시간은 RNA 주입 전 며칠 동안 지속될 수 있지만 발현은 증가하지 않습니다. - RNA 주입을 위해 나노 인젝터를 준비하십시오 (2.2 단계와 같지만 주입 팁은 DNA 주입만큼 얇을 필요는 없습니다). 이 시점에서 적색 빛 아래서 만 작업하면 Anap의 광 변색을 방지 할 수 있습니다.
- parafilm의 조각에 1-2 μg / μL mRNA의 1 μL와 1 MM Anap 1 μL를 직접 섞고 혼합 솔루션으로 주사 팁을 기입하십시오. 식물에서 멤브레인 바로 밑으로 퍼지다.(밝은) 기둥과 각 pAnap 주입 oocyte에 46 nL을 주입.
참고 : 필요한 mRNA 농도는 관심있는 단백질에 따라 다릅니다. - Barth의 용액 보충 항생제와 18 % 5 % 말 혈청에서 2 ~ 3 일 동안 박스에서 빛으로부터 보호하거나 알루미늄 호일로 포장 한 난 모세포를 부화시킵니다. 신선한 Barth 용액으로 매일 교환하고 오염을 피하기 위해 죽은 난 모세포를 제거하십시오.
- 이전에 설명한대로 컷 오픈 oocyte 전압 클램프 장비를 설치하십시오.
- oocyte 및 형광 측정 ( 그림 2c )을 수행하기 위해 현미경을 배치하기위한 표준 입체경 사이에 그것을 움직일 수있는 슬라이더에 녹음 챔버를 설치하여 직립 형광 현미경에 전기 생리학 기록 시스템을 탑재하십시오.
참고 : 절단 오픈 oocytes에 대한 챔버의 기하학은 정상적인 tra를 사용하여 적합하지 않습니다조작하는 동안 조명에 nsmitted 빛. 따라서 적색 필터가있는 "구즈넥 (goseneck)"할로겐 램프를 사용하여 상단에서 옆으로 조명합니다. 현미경의 응축기는 전기 생리학 챔버의 스테이지를 낮추기위한 공간을 제거 할 수 있습니다. - 형광 현미경 ( 그림 2a )의 C - 마운트 출구 포트에 photodiode 감지 시스템을 연결합니다. 광전 판독 값을 디지털 신호 프로세서 (DSP, 아날로그 / 디지털 - 디지털 / 아날로그 변환기)의 두 번째 입력 채널에 연결하십시오.
- 형광 여기를위한 광원으로 100 W, 12 V 할로겐 램프를 사용하십시오.
참고 : 또는, Hg 버너를 사용할 수도 있지만 레코딩 중에 너무 빠르게 빛이 번지는 것을 방지하려면 강도를 줄여야합니다. LED 조명은 각 LED가 여기 범위 ( 예 : Anap의 경우 ~ 350 nm)에서 중요한 강도를 나타내는 경우에만 권장됩니다. 대부분의 백색 LED는 UV 스펙트럼까지 멀리 도달하지 않습니다. - Ins여기 광원과 현미경 사이에 전기적으로 제어되는 셔터를 설치하고 제어 장치 (일반적으로 TTL 펄스)를 DSP의 디지털 출력에 연결합니다. 기록 소프트웨어에서 TTL 펄스 시간을 측정하고 (제조업체 설명서 참조) 녹음 시작 전 ~ 100ms 사이에 셔터가 열립니다. 이러한 방식으로, 열림 과정 중 진동이 녹음을 방해하지 않습니다. 시간은 셔터 속도와 진동에 따라 다릅니다. 그림 4 와 같이 녹음 종료 5ms 전에 펄스를 종료하십시오. 이 방법으로 총 형광 값도 기록됩니다.
- 필터 큐브 터렛에 적절한 필터 큐브 (여기 필터, 다이크로 익 미러 및 방출 필터)를 삽입하십시오. Anap의 경우 Ex : 377/50 nm bandpass, dichroic 409 nm longpass 및 Em : 470/40 nm bandpass를 사용하십시오.
- 이전에 설명한대로 컷 오픈 oocyte 전압 클램프에 대한 준비 단계를 따르십시오 18 (한천 다리 준비, oocyte 장착, 사포닌 permeabilization). 그러나 녹음하기 전에 형광 물질 표백을 피하기 위해 항상 적색 빛 아래에서 작업하십시오. 난 모세포를 놓을 때, 동물 극이 위쪽을 향하게하는지 확인하십시오. 동물성 극 멤브레인 아래의 색소 침착은 cytosol에서 유래하는자가 형광에 대해 차폐되므로 백그라운드 형광을 감소시킵니다.
- 챔버를 현미경 위로 밀고 4X 대물 렌즈를 사용하여 초점을 맞 춥니 다.
- 전압 감지 V1 전극 (3 M KCl)으로 oocyte를 주입하고 40X 수침 (NA 0.8 - 0.9) 대물 렌즈로 전환하십시오. 위쪽을 향하고있는 동물 극에 초점을 맞 춥니 다.
- 빨간색 표시등을 끕니다. 필터 큐브 터렛과 포토 다이오드에 연결된 광 출구 포트를 돌려 오른쪽 필터 큐브를 선택합니다. halog 켜기en 램프를 켜고 2 ~ 5 초 동안 셔터를 잠시 열어서 난 모세포에서 유래 한 배경 형광 강도를 읽습니다. 설명 된 설정 값은 Anap에 대해 약 50-200pA가되어야합니다.
- 클램프를 켜고 목욕 / 가드 스위치를 활성화 상태로 전환하고 막판의 노브를 돌려 멤브레인 전위 (V1 - V2)를 명령 전위로 조정하십시오.
- 녹음 소프트웨어에서 보유 잠재력, 단계 프로토콜, 펄스 수 및 길이 등을 선택하십시오. 전압 의존적 인 전류와 Anap 형광 강도를 기록하십시오.
- 동일한 단백질의 두 위치를 동시에 모니터하기 위해 세포 외 접근 가능한 아미노산을 시스테인으로 돌연변이시키고 다른 시스테인을 제거하여 티올 화학으로 특이적인 표지를 확보하십시오.
- 2.1-2.5 단계를 수행하십시오.
- VCF 녹음하기 전에, 15 분 (또는 다른 염료에 대한 라벨 솔루션에 TMR - maleimide 5 μm의에서 oocytes를 품어Anap과 비교하여 비 중첩 스펙트럼으로).
- 과잉 염료를 제거하기 위해 세 번 라벨링 솔루션으로 oocytes을 씻으십시오.
- 4.1-4.6 단계를 수행하십시오.
- 필터 큐브 터렛에 TMR (여기 필터, 이색 성 거울 및 방출 필터)에 적합한 필터 큐브를 삽입합니다. 필터 터렛을 돌려 TMR 필터 큐브로 전환하십시오.
- 4.4 단계에서 Anap에 대해 설명한대로 TMR의 배경 형광을 읽습니다.
참고 : thiol-chemistry로 표지하면 막의 비특이적 인 표지화로 인해 높은 백그라운드 형광이 발생합니다. 따라서 TMR 백그라운드 형광은 증폭기를 포화시킬 수 있습니다 (> 2,000 pA). 이 경우 광도를 줄이지 말고 포토 다이오드에 오프셋 전류를 추가하여 배경 형광을 뺍니다. 시중에서 판매되는 시스템에서는 검출기 시스템의 "샘플 앤 홀드"기능을 사용하십시오. 실험실 j에서 배경 형광 값 (10X 감도 필터 사용)을 확인하십시오.이 값은 기록되지 않습니다 (채도). - 4.6 단계와 같이 전압 의존적 전류와 TMR 형광 강도를 동시에 기록하십시오.
2. 난 모세포 준비 및 주입
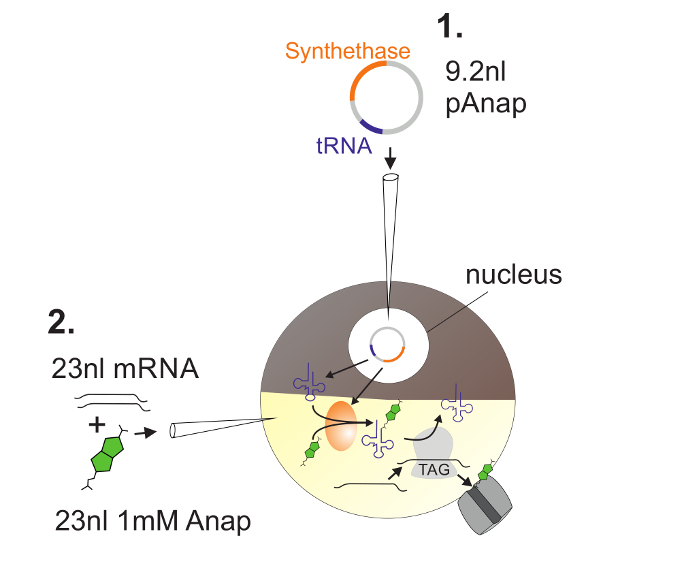
그림 2 : Anap Incorporation을 위한 Xenopus 난 모세포에 DNA와 RNA 주입의 예.
첫째, pAnap은 Xenopus 난 모세포의 핵에 주입됩니다 ( 1 ). 6 ~ 24 시간 후, Anap과 채널 RNA는 식물성 극으로 주입됩니다 ( 2 ). Anap은 pAnap에 의해 코딩되는 aminoacyl-tRNA synthetase에 의해 amber stop anti-codon을 가진 tRNA로 직각으로 아미노산 화 될 것이다. 이 방법으로 aminoacylated Anap-tRNA는 채널에 삽입 된 앰버 정지 코돈에서 리보솜에 의해 인식됩니다l RNA는 정지 코돈의 억제와 Anap의 삽입을 초래한다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
3. VCF 설정

도 3 < / strong> : VCF 설정. ( A ) 현미경 내부의 빛 경로를 보여주는 VCF 설정의 측면도. 필터 큐브는 여기 필터, 다이크로 익 미러 및 방출 필터를 포함합니다. ( B ) 선택된 난 모세포 챔버의 치수는 상부 챔버 반경 (1), 하부 챔버 길이 (2)는 5.5cm, 하부 챔버 폭 (3)은 1.4cm, 중간 챔버 폭 (4)은 1.7cm입니다. ( C ) VCF 설정의 앞면. 왼쪽의 첫번째 안구는 cut-open 전압 클램프 챔버에 난 모세포를 장착하고 투과성을 높이기위한 것입니다. 그런 다음, 챔버는 오른쪽 두 번째 안구에서 현미경으로 미끄러졌습니다. 여기에서, V1 전극은 4X 대물 렌즈를 사용하여 난 모세포에 삽입되고 형광은 수중 40X 대물 렌즈를 사용하여 기록됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
5. 2 색 VCF
결과
그림 4 는 pAnap과 Anap 존재 하에서 빠른 불활 화 제거 (IR), L382stop-W434F가있는 쉐이커 채널을 표현하는 난 모세포에서 얻은 VCF 기록의 예를 보여줍니다. W434F 돌연변이는 일시적인 게이팅 전하 이동 (게이팅 전류)을 측정 할 수있는 이온 칼륨 전류를 차단합니다. 감극시 게이팅 전류 (상부 트레이스)와 Anap 형광 강도 변화 (하부 트레이스)의 동시 기록은 Anap을 L382 ?...
토론
tRNA 합성 효소와 함께 연속적으로 전사되는 tRNA의 생체 내 aminoacylation은 형광 측정을위한 높은 발현 수준을 얻는 것을 가능하게합니다. 효율적인 fUAA 도입을 위해서는 pAnap이 핵에 정확하게 주입되는 것이 중요합니다. 핵의 정확한 위치에 대한 불확실성 때문에 DNA 주입의 10-40 %가 실패 할 것으로 예상되어 비 - 발현 (또는 누출 - 발현) 난 모세포가 발생합니다. 그러므로 Anap과 pAnap가없는 경우...
공개
저자는 공개 할 것이 없습니다.
감사의 말
pAnap은 Peter Schultz 박사 (Scripps Research Institute)의 친절한 선물이었습니다. 이 연구는 보건 연구 보조금 MOP-102689 및 MOP-136894 (RB에 대한) 및 캐나다 재단 혁신 기금 950-225005에 의해 자금 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| Solutions | |||
| Barth's solution | |||
| NaCl | Sigma-Aldrich | S7653 | 90 mM |
| KCl | Fisher Scientific | BP366-500 | 3 mM |
| MgSO4 | Sigma-Aldrich | M-9397 | 0.82 mM |
| CaCl2 | Sigma-Aldrich | C-7902 | 0.41 mM |
| Ca(NO3)2 | Sigma-Aldrich | C-1396 | 0.33 mM |
| HEPES | Sigma-Aldrich | H4034 | 5 mM |
| NaOH hydrate | BDH | BDH7225-4 | pH 7.6 |
| Penicilin | Invitrogen | 15140122 | 100 U/mL |
| Streptomycin | Invitrogen | 15140122 | 100 µg/mL |
| Kanamycin | Invitrogen | 15160054 | 10 mg/100mL |
| Horse Serum (HS) | Invitrogen | 16050122 | 5% |
| SOS Standard Oocyte Solution | |||
| NaCl | Sigma-Aldrich | 746398 | 102 mM |
| KCl | Sigma-Aldrich | 746436 | 3 mM |
| MgCl2 | Sigma-Aldrich | M9272 | 1 mM |
| HEPES | Sigma-Aldrich | H4034 | 5 mM |
| External recording solution | |||
| N-methyl-D-glucamine (NMDG) | Alfa Aesar | L14282 | 115 mM |
| HEPES | Sigma-Aldrich | H4034 | 10 mM |
| Calcium hydroxide | Sigma-Aldrich | 239232 | 2 mM |
| MES hydrate | Sigma-Aldrich | 258105 | pH 7.2 |
| Internal recording solution | |||
| N-methyl-D-glucamine (NMDG) | Alfa Aesar | L14282 | 115 mM |
| HEPES | Sigma-Aldrich | H4034 | 10 mM |
| Ethylenediamine Tetraacetic Acid (EDTA) | Fisher Scientific | E478-500 | 2 mM |
| MES hydrate | Sigma-Aldrich | 258105 | pH 7.2 |
| Labeling solution | |||
| KOH | Fisher Scientific | P250-1 | 115 mM |
| HEPES | Sigma-Aldrich | H4034 | 10 mM |
| Calcium hydroxide | Sigma-Aldrich | 239232 | 2 mM |
| MES hydrate | Sigma-Aldrich | 258105 | pH 7.2 |
| TMR stock solution | |||
| Tetramethylrhodamine-5-maleimide (TMR) | Molcular Probes by Life Technologies | T6027 | 5 mM in DMSO |
| Anap stock solution | |||
| Anap | ABZENA (TCRS) | Custom synthesis TCRS-170 | 1 mM in nuclease-free water and 1% NaOH 1 N |
| Name | Company | Catalog Number | Comments |
| Material/Equipment | |||
| pAnap | Addgene | 48696 | |
| High Performance Oocyte Clamp | Dagan Corporation | CA-1B | |
| Gpatch Acquisition software | Department of Anesthesiology, University of California, Los Angeles | ||
| Analysis software | Department of Anesthesiology, University of California, Los Angeles | ||
| Recording Chamber | Custom machined | ||
| Photo diode detection system | Dagan Corporation | PhotoMax-200/PIN | |
| Electrical shutter driver | UNIBLITZ | VCM-D1 |
참고문헌
- Mannuzzu, L. M., Moronne, M. M., Isacoff, E. Y. Direct physical measure of conformational rearrangement underlying potassium channel gating. Science. 271 (5246), 213-216 (1996).
- Kalstrup, T., Blunck, R. Dynamics of internal pore opening in K(V) channels probed by a fluorescent unnatural amino acid. Proc Natl Acad Sci U S A. 110 (20), 8272-8277 (2013).
- Xiao, H., Schultz, P. G. At the Interface of Chemical and Biological Synthesis: An Expanded Genetic Code. Cold Spring Harb Perspect Biol. 8 (9), (2016).
- Blunck, R. Chapter 9. Handbook of Ion Channels. , 113-133 (2015).
- Chatterjee, A., Guo, J., Lee, H. S., Schultz, P. G. A genetically encoded fluorescent probe in mammalian cells. J Am Chem Soc. 135 (34), 12540-12543 (2013).
- DeBerg, H. A., Brzovic, P. S., Flynn, G. E., Zagotta, W. N., Stoll, S. Structure and Energetics of Allosteric Regulation of HCN2 Ion Channels by Cyclic Nucleotides. J Biol Chem. 291 (1), 371-381 (2016).
- Shen, B., et al. Genetically encoding unnatural amino acids in neural stem cells and optically reporting voltage-sensitive domain changes in differentiated neurons. Stem Cells. 29 (8), 1231-1240 (2011).
- Aman, T. K., Gordon, S. E., Zagotta, W. N. Regulation of CNGA1 Channel Gating by Interactions with the Membrane. J Biol Chem. 291 (19), 9939-9947 (2016).
- Haddad, G. A., Blunck, R. Mode shift of the voltage sensors in Shaker K+ channels is caused by energetic coupling to the pore domain. J Gen Physiol. 137 (5), 455-472 (2011).
- Batulan, Z., Haddad, G. A., Blunck, R. An intersubunit interaction between S4-S5 linker and S6 is responsible for the slow off-gating component in Shaker K+ channels. J Biol Chem. 285 (18), 14005-14019 (2010).
- Kusch, J., et al. How subunits cooperate in cAMP-induced activation of homotetrameric HCN2 channels. Nat Chem Biol. 8 (2), 162-169 (2012).
- Lee, H. S., Guo, J., Lemke, E. A., Dimla, R. D., Schultz, P. G. Genetic incorporation of a small, environmentally sensitive, fluorescent probe into proteins in Saccharomyces cerevisiae. J Am Chem Soc. 131 (36), 12921-12923 (2009).
- Summerer, D., et al. A genetically encoded fluorescent amino acid. Proc Natl Acad Sci U S A. 103 (26), 9785-9789 (2006).
- Kalstrup, T., Blunck, R. Reinitiation at non-canonical start codons leads to leak expression when incorporating unnatural amino acids. Sci Rep. 5, 11866 (2015).
- Liu, H., Naismith, J. H. An efficient one-step site-directed deletion, insertion, single and multiple-site plasmid mutagenesis protocol. BMC Biotechnol. 8, 91 (2008).
- Beckert, B., Masquida, B. Synthesis of RNA by in vitro transcription. Methods Mol Biol. 703, 29-41 (2011).
- Goldin, A. L. Maintenance of Xenopus laevis and oocyte injection. Methods Enzymol. 207, 266-279 (1992).
- Rudokas, M. W., Varga, Z., Schubert, A. R., Asaro, A. B., Silva, J. R. The Xenopus oocyte cut-open vaseline gap voltage-clamp technique with fluorometry. J Vis Exp. (85), (2014).
- Zhao, J., Blunck, R. The isolated voltage sensing domain of the Shaker potassium channel forms a voltage-gated cation channel. Elife. 5, (2016).
- Posson, D. J., Ge, P., Miller, C., Bezanilla, F., Selvin, P. R. Small vertical movement of a K+ channel voltage sensor measured with luminescence energy transfer. Nature. 436 (7052), 848-851 (2005).
- Chanda, B., Asamoah, O. K., Blunck, R., Roux, B., Bezanilla, F. Gating charge displacement in voltage-gated ion channels involves limited transmembrane movement. Nature. 436 (7052), 852-856 (2005).
- Taraska, J. W., Puljung, M. C., Zagotta, W. N. Short-distance probes for protein backbone structure based on energy transfer between bimane and transition metal ions. Proc Natl Acad Sci U S A. 106 (38), 16227-16232 (2009).
- Baker, B. J., et al. Genetically encoded fluorescent sensors of membrane potential. Brain Cell Biol. 36 (1-4), 53-67 (2008).
- Miranda, P., et al. State-dependent FRET reports calcium- and voltage-dependent gating-ring motions in BK channels. Proc Natl Acad Sci U S A. , (2013).
- Sisido, M., Ninomiya, K., Ohtsuki, T., Hohsaka, T. Four-base codon/anticodon strategy and non-enzymatic aminoacylation for protein engineering with non-natural amino acids. Methods. 36 (3), 270-278 (2005).
- Hohsaka, T., Ashizuka, Y., Murakami, H., Sisido, M. Five-base codons for incorporation of nonnatural amino acids into proteins. Nucleic Acids Res. 29 (17), 3646-3651 (2001).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유