È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Fluorescenza a tensione di serraggio in
In questo articolo
Riepilogo
In questo articolo si descrive un miglioramento della convenzionale fluorometria a tensione di tensione (VCF) in cui vengono utilizzati fluorocinosi aminoacidi innaturali (fUAA) anziché coloranti maleimidi, per sondare i riarrangiamenti strutturali nei canali ionici. La procedura prevede l'iniezione di DNA Xenopus oocyte, la coinjection RNA / fUAA e le misure contemporanee di corrente e fluorescenza.
Abstract
La fluorometria a tensione di tensione (VCF) è stata la tecnica di scelta per studiare la struttura e la funzione delle proteine di membrana elettrogene dove le misurazioni in tempo reale di fluorescenza e correnti denunciano simultaneamente i riarrangiamenti locali e la funzione globale, rispettivamente 1 . Mentre le tecniche strutturali ad alta risoluzione come la microscopia crio-elettrona o la cristallografia a raggi X forniscono immagini statiche delle proteine di interesse, VCF fornisce dati strutturali dinamici che ci permettono di collegare i riarrangiamenti strutturali (fluorescenza) ai dati funzionali dinamici (elettrofisiologia). Fino a poco tempo fa, la chimica tiol-reattiva utilizzata per l'etichettatura fluorescente di proteine del sito ha limitato l'ambito dell'approccio perché tutte le cisteine accessibili, comprese quelle endogene, saranno etichettate. È stato pertanto necessario costruire proteine prive di cisteine endogene. L'etichettatura è stata anche limitata ai siti accessibili dall'estracellularelato. Ciò è cambiato con l'uso di fluidi naturali aminoacidi (fUAA) per incorporare specificamente una piccola sonda fluorescente in risposta alla soppressione del codone di arresto usando una coppia di tRNA e tRNA sintetasi ortogonale 2 . Il miglioramento del VCF richiede solo una procedura di iniezione in due fasi di iniezione del DNA (coppia tRNA / sintetasi) seguita da coinjection di RNA / fUAA. Ora, l'etichettatura di entrambi i siti intracellulari e sepolti è possibile, e l'uso di VCF è aumentato in modo significativo. La tecnica VCF diventa quindi attraente per studiare una vasta gamma di proteine e, soprattutto, consente di indagare numerosi meccanismi di regolazione citosolica.
Introduzione
Oltre 200 aminoacidi innaturali di varie proprietà chimiche e fisiche sono state incorporate geneticamente in proteine in E. coli , cellule di lieviti e mammiferi 3 . L'aminoacido innaturale è incorporato in risposta a un codone di arresto specifico tramite una coppia tRNA / sintetasi ingegnerizzata ortogonale. L'approccio genetico per modificare le proteine ha fornito preziosi approfondimenti sulla struttura e funzione delle proteine. Qui presentiamo un protocollo per l'utilizzo della Fluorometria a Voltage-Clamp (VCF) in combinazione con un UAA fluorescente.
In VCF, l'osservazione simultanea di dati funzionali e riarrangiamenti strutturali localizzati intorno alla sonda fluorescente (~ 5 Å) ci consente di ottenere informazioni dinamiche con risoluzione milliseconda 1 . Le sonde fluorescenti alterano lo stato di spegnimento sul movimento localizzato della proteina. Un movimento di soli 1-2 Å è sufficiente a condurre a significativi cambiamenti nella fluorescenzaIntensità 4 . Dopo l'identificazione del sito di interesse nella proteina bersaglio, il sito è mutato dalla mutazione di punti. Classicamente, il residuo era stato mutato a una cisteina, mentre adesso è stato introdotto un codone di arresto ambrato (TAG) per l'incorporazione genica fUAA. La proteina viene poi trascritta in vitro .
Mentre altri sistemi di espressione possono essere utilizzati ( ad esempio, le cellule di mammiferi) 5 , 6 , 7 , gli oociti di Xenopus sono preferiti per gli studi di funzionalità della struttura a causa della loro dimensione più grande, portando a una manipolazione più facile e ad una maggiore intensità di fluorescenza (più fluorofori) Rapporto rumore. Inoltre, gli oociti di Xenopus hanno un basso livello di background dalle proteine endogene 2 , 8 e dalla pigmentazione scura sugli schermi polari animali contro la fluorescenza di fondo da tLui citosol. Gli oociti Xenopus vengono rimossi chirurgicamente e il DNA che codifica la coppia ortogonale tRNA / tRNA-sintetasi specifica per la fUAA viene iniettato nel nucleo degli oociti. Dopo un tempo di incubazione di 6-24 ore, l'RNA di proteina viene coinsidito con il fUAA nel citosolo degli oociti, seguito da un periodo di incubazione di 2-3 giorni. Per prevenire eventuali danni alla fUAA (fotoelettrodo), le procedure con Anap devono essere eseguite sotto luce rossa per evitare l'eccitazione del fluoroforo.
Gli oociti sono studiati su un dispositivo di tensione-bloccaggio di oocyte a taglio aperto, che viene montato su un microscopio a fluorescenza verticale e vengono registrati contemporaneamente 9 e 10 cambiamenti di corrente elettrica e fluorescenza. In alternativa, possono essere impiegate configurazioni di tensione 1 a bobina a due elettrodi o configurazioni di patch-clamp 11 . La fluorescenza è eccitata da lunghezze d'onda appropriate con rumore RMS basso e Emissione registrata utilizzando un fotodiodo collegato ad un amplificatore ad alta amplificazione.
Ci sono diversi vantaggi dell'utilizzo di aminoacidi innaturali fluorescenti (fUAA) in fluorometria a tensione. Uno è l'accesso al lato citosolico delle proteine della membrana; Si trovano qui molti processi regolatori ( ad esempio, siti di legame di Ca 2+ o nucleotidi, inattivazione rapida e in stato di stato dei canali ionici a tensione, apertura dei pori, accoppiamento dei moduli). Tutti questi processi sono ora accessibili per l'etichettatura fluorescente.
Un altro vantaggio è la piccola dimensione della sonda che porta a meno disturbi della proteina. Finora, due coppie ortogonali di sintetasi tRNA / tRNA per fUAA sono state progettate 12 , 13 , dove l'acido 3- (6-acetilnaftalen-2-ilamino) -2-aminopropanoico (Anap) è l'unica fUAA che è stata utilizzata negli oociti Xenopus 2 ,"Xref"> 8. Anap è un fluoroforo sensibile all'ambiente con un peso molecolare di 272,3 g / mol ed è solo leggermente più grande del triptofano 12 ( figure 1A, 1B ). A causa della sua dimensione ridotta, probabilmente saranno meno gli effetti sterici da parte del fluoroforo rispetto ai fluorofori convenzionali collegati tramite un linker (tipicamente più di 500 g / mol). Inoltre, nel caso di Anap, il fluoroforo si trova più vicino alla base proteica rispetto a quelli legati alle cisteine, e di conseguenza, Anap sta sondando ulteriori riarrangiamenti localizzati. Infine, la rimozione di cisteine endogene nel VCF convenzionale per garantire che l'etichettatura specifica del sito non sia più un requisito in UAA-VCF e quindi (i) lascia le proteine in (quasi) il loro stato nativo e (ii) consente di applicare VCF Per studiare una gamma più ampia di proteine in cui la funzione può essere alterata dalla sostituzione di cisteina.

Figura 1 : Anap e Fluorescenza Spectra. ( A ) Struttura chimica di Anap. ( B ) Spettri di assorbimento normalizzati e spettri di emissione per 1 nM Anap, dimostrando la sensibilità della fluorescenza Anap all'idropobicità del solvente. Gli spettri di emissione sono stati ottenuti emozionanti a 350 nm. Clicca qui per visualizzare una versione più grande di questa figura.
Uno svantaggio dell'utilizzo di UAA fluorescenti è che una popolazione eterogenea di proteine può derivare dalla riduzione del codone di arresto, dalla reinitiazione della traslazione, dalle proteine troncate del C-terminale o dalla crisi con l'aminoacilazione endogena se la quantità di tRNA aminoacilati è scarsa. Tale espressione di perdita deve sempre essere controllata in assenza della fUAA e della coppia di sintesi tRNA / tRNA. Abbiamo affrontato la questione del transReinitiation lancinante e come eluderlo per i siti di inserimento del terminale N in precedenza 14 . Tuttavia, quando la fUAA, la tRNA e la tRNA sintetasi sono presenti in quantità saturate, rimane solo una bassa probabilità di espressione di perdita.
La differenza procedurale tra fUAA-VCF e VCF convenzionale è l'iniezione e la manipolazione degli ovociti; L'iniezione di DNA che codifica il tRNA e la sintetasi tRNA (pAnap) è seguita dall'introduzione di Anap, che viene co-iniettato con la proteina mRNA o aggiunta alternativamente alla soluzione di incubazione come estere acetossimetil (AM).
Access restricted. Please log in or start a trial to view this content.
Protocollo
Le manipolazioni di rana sono state eseguite secondo le linee guida canadesi e sono state approvate dal comitato etico (CDEA, protocollo n. 15-042) dell'Università di Montréal.
1. Preparazione di mRNA per l'integrazione fUAA
- Scegli un sito di interesse per la proteina in cui si prevede che si verifichino cambiamenti conformazionali. Selezionare un aminoacido in questa regione da sostituire per la fUAA.
NOTA: La scelta della posizione è basata sui riarrangiamenti strutturali che si prevede. Se esiste una struttura ad alta risoluzione e un'ipotesi dei movimenti attesi, l'anap dovrebbe essere posto in modo tale che l'ambiente chimico possa alterarsi; Questo potrebbe essere un cambiamento nella costante dielettrica (ambiente idrofobico o idrofilo) o, più probabilmente, spegnimento da un altro aminoacido. I migliori quenchers sono triptofani. Anap deve essere in contatto con il quencher in uno stato (sovrapposizione dei raggi van-der-Waals) e libera da essol'altro. Se non esistono strutture o modelli ad alta risoluzione, si dovrebbe analizzare la regione di interesse. In entrambi i casi, è consigliabile selezionare più località vicine per aumentare la probabilità di ottenere l'espressione e il segnale di fluorescenza. Per ridurre al minimo gli effetti sterici durante la maturazione e / o la funzione proteica, si può scegliere di sostituire gli amminoacidi grandi e aromatici (Phe, Trp, Tyr). Gli autori, tuttavia, hanno sperimentato che la scansione di una regione di interesse per l'inserzione fUAA indipendentemente dall'amminoacido sostituito, è più produttivo. - Inserisci un codone di stop ambra (TAG) nel sito selezionato utilizzando la mutagenesi del sito 15 . Assicurarsi che la proteina di interesse non finisca su un codone di stop ambra (TAG). In caso affermativo, mutare a un altro (codone di stop ocra o opale). Amplificare, isolare e sequenziare il DNA. Ottenere proteina mRNA con la trascrizione in vitro 16 e memorizzare l'mRNA a 20 ° C o 80 ° C.
- Chirurgicamente ottenere ovociti di fase V o VI da rane di Xenopus laevis e defolliculare con collagenasi come descritto in precedenza 17 .
- Anestetizzare le rane con un appropriato anestetico secondo il protocollo animale approvato (qui: estere etilico dell'acido 3-aminobenzoico). Quando non riescono a rispondere a un pizzico dolce a una punta di punta (perdita di riflesso di ritiro), allora sono opportunamente anestetizzati per la chirurgia.
- Rimuovere immediatamente le rane dalla soluzione anestetica e sciacquare accuratamente la pelle con acqua dolce. Questo risciacquo impedirà all'animale di cadere in livelli più profondi di anestesia rimuovendo la chimica non abbonata dalla superficie cutanea.
- Rimuovere i nodi delle ovaie da un lato chirurgicamente e con attenzione aprire i nodi utilizzando due pinze. Incubare ed agitare gli oociti in "soluzione standard oocitaria" (SOS) contenente collagenasi 1% (w / v) per 20-30 minPer defollicare. Lavare tre volte con la soluzione SOS.
- Selezionare singolarmente oociti grandi e sani e incubarli nella soluzione di Barth integrata con antibiotici (100 U / mL penicillina, 100 μg / mL streptomicina, 10 mg / 100 mL kanamicina) e 5% cavallo di cavallo a 18 ͦC per almeno 4 ore prima dell'iniezione .
NOTA: Dopo 2 - 4 interventi chirurgici con un ritardo di 4 mesi tra, Xenopus laevis viene eutanizzato da incubazione prolungata (> 1 h) con l'estere etilico dell'acido 3-aminobenzoico.
- Per l'iniezione nucleare del DNA, preparare una punta di iniezione lunga e sottile per poter raggiungere il nucleo e per evitare di danneggiare l'oocyte. Riempire la punta di iniezione con olio e montarla sul dispositivo nano iniettore.
- Installare il nano-iniettore sotto un microscopio stereo e utilizzare pinze per rompere l'estremità della punta. Estrarre l'olio finché non ci sono bolle d'aria intrappolate all'interno della punta della punta.
- Inserire 1 μl di 0,1 μg /# 181; L pAnap in acqua senza nucleasi contenente NaOH (1% di 1 N NaOH) su un pezzo di parafilm sotto uno stereoscopio e riempire la punta di iniezione con il DNA.
- Trasferire 40 oociti in un contenitore iniettato a maglia contenente la soluzione di Barth integrata con antibiotici.
NOTA: Per fare il piatto iniettato rivestito in rete, tagliare un pezzo di 800 micron di nylon adeguatamente dimensionato per riempire un piatto di polistirolo Petri. Aggiungere il cloroformio al centro e quindi mettere la maglia in cima. Tieni la maglia fino a quando non arriverà la plastica. - Poiché il nucleo di oocyte si trova nel polo animale (scuro), puntare la punta di iniezione al centro del polo animale e impalare tale che la punta raggiunga il centro dell'emisfero animale (o 2-3 volte la profondità rispetto all'infezione RNA ). Iniettare 9,2 nL di pAnap nel nucleo di ciascun oocyte. La punta sottile e il piccolo volume di iniezione possono causare iniezione irregolare o punta bloccata. Controllare occasionalmente se l'iniezione funziona iniettando nell'aria.
NOTA: se il DNA viene iniettato correttamente nel nucleo è incerto. Si aspetta quindi 10 - 40% degli oociti per non esprimere la coppia tRNA / sintetasi. Vedi Discussione per ulteriori elaborazioni. - Incubare gli oociti in 2 mL di soluzione di Barth integrata con antibiotici e 5% cavallo di cavallo (HS) a 18 ° C per 6-24 h per consentire una robusta espressione di tAPRs specifici Anap e sintetasi tRNA.
NOTA: il tempo di incubazione del DNA può durare diversi giorni prima dell'iniezione di RNA, ma non aumenta l'espressione. - Preparare il nano-iniettore per l'iniezione RNA (come al punto 2.2, ma la punta di iniezione non deve essere sottile come per l'iniezione del DNA). Lavorare solo sotto la luce rossa da questo punto per impedire la fotolibrazione dell'Anap.
- Mescolare 1 μL di 1 mM Anap con 1 μL di mRNA 1-2 μg / μL direttamente su un pezzo di parafilm e riempire la punta di iniezione con la soluzione mista. Impala appena sotto la membrana nel vegetale(Luminoso) ed iniettare 46 nL in ogni oocyte iniettato da pAnap.
NOTA: La concentrazione di mRNA necessaria dipende dalla proteina di interesse. - Incubare gli oociti protetti dalla luce in una scatola o avvolti in foglio di alluminio, nella soluzione di Barth ha completato gli antibiotici e il 5% cavallo di cavallo a 18 ° C per 2-3 giorni. Scambiare con la soluzione fresca di Barth ogni giorno e rimuovere gli oociti morti per evitare la contaminazione.
- Installare l'apparecchiatura di tensione-taglio del oocyte a taglio aperto come descritto in precedenza 18 .
- Montare il sistema di registrazione elettrofisiologica su un microscopio a fluorescenza verticale installando la camera di registrazione su un dispositivo di scorrimento che consente di spostarlo tra lo stereoscopio standard per mettere l'oocyte e il microscopio per eseguire le misurazioni della fluorescenza ( Figura 2c ).
NOTA: La geometria della camera per oociti taglienti non è adatta per l'uso del normale traIlluminata per illuminazione durante la manipolazione. Pertanto, una lampada alogena "gooseneck" con filtro rosso viene utilizzata per illuminare lateralmente dall'alto. Il condensatore del microscopio può essere rimosso fai spazio per abbassare lo stadio per la camera elettrofisiologica. - Collegare un sistema di rilevamento fotodiodo alla porta di uscita del microfono a fluorescenza ( figura 2a ). Collegare la lettura fotocirata a un secondo canale di ingresso nel processore di segnale digitale (DSP, convertitore analogico / digitale / digitale / analogico).
- Usare una lampada alogena da 100 W, 12 V come sorgente luminosa per l'eccitazione della fluorescenza.
NOTA: In alternativa, i bruciatori Hg possono essere utilizzati, ma devono essere ridotti in intensità per impedire una rapida eliminazione della foto durante le registrazioni. L'illuminazione del LED è consigliata solo se i rispettivi LED indicano un'intensità significativa nell'intervallo di eccitazione ( ad esempio, ~ 350 nm per Anap). La maggior parte dei LED bianchi non raggiunge molto lo spettro UV. - insUn otturatore controllato elettricamente tra sorgente luminosa di eccitazione e microscopio e collegare il suo controllo (tipicamente TTL-impulso) ad un'uscita digitale del DSP. Immettere l'impulso TTL nel software di registrazione (vedere la documentazione del produttore), in modo tale che l'otturatore si apra ~ 100 ms prima dell'inizio della registrazione. In questo modo, qualsiasi vibrazione durante il processo di apertura non interferisce con la registrazione. Il tempo dipende dalla velocità e dalla vibrazione dell'otturatore. Terminare l'impulso 5ms prima della fine della registrazione come mostrato in Figura 4 . In questo modo viene anche registrato il valore della fluorescenza totale.
- Inserire un cubo di filtro appropriato (filtro di eccitazione, specchio dicroico e filtro di emissione) nella torretta del filtro del cubo. Per Anap, utilizzare la banda passante Ex: 377/50 nm, il longpass dicroico 409 nm e la banda passante Em: 470/40 nm.
- Seguire le fasi di preparazione per il morsetto di tamponamento oocyte aperto come descritto in precedenza e visualizzato 18 (preparazione del ponte agar, montaggio dell'oocyte, permeabilizzazione della saponina). Tuttavia, lavorare sotto la luce rossa in qualsiasi momento per evitare di sbiancare il fluoroforo prima delle registrazioni. Quando si posiziona l'oocyte, accertarsi che il polo animale sia rivolto verso l'alto. La pigmentazione sotto la membrana polare animale protegge contro l'autofluorescenza proveniente dal citosolo e quindi riduce la fluorescenza di fondo.
- Far scivolare la camera al microscopio e mettere a fuoco con un obiettivo 4X.
- Impalare l'oocyte con l'elettrodo V1 a sensore di tensione (3 M KCl), passare all'obiettivo 40X d'immersione in acqua (NA 0,8 - 0,9). Focus sul polo animale rivolto verso l'alto.
- Spegnere la luce rossa. Selezionare il cubo filtro destro ruotando la torretta del filtro del cubo e la porta di uscita ottica collegata al fotodiodo. Accendi il halogEn lampada ad alta intensità e brevemente si accende l'otturatore aperto per 2-5 s per leggere l'intensità di fluorescenza di fondo originata dall'oocyte. Con l'impostazione descritta il valore dovrebbe essere di circa 50-200 pA per Anap.
- Accendere il morsetto, azionare l'interruttore di protezione / protezione in attivo e regolare il potenziale della membrana (V1 - V2) al potenziale di comando ruotando la manopola sullo schermo principale.
- Selezionare il potenziale di tenuta, il protocollo del passo, il numero e la lunghezza degli impulsi ecc. Nel software di registrazione. Registrare correnti di tensione dipendenti e intensità di fluorescenza Anap.
- Per monitorare simultaneamente due posizioni della stessa proteina, mutare un aminoacido extracellulare e accessibile in cisteina e rimuovere altre cisteine per garantire l'etichettatura specifica con la tiol-chimica.
- Eseguire il passo 2.1-2.5.
- Prima delle registrazioni VCF, incubare oociti in 5 μM TMR-maleimide nella soluzione di etichettatura per 15 minuti (o altri colorantiCon spettri non sovrapposti rispetto ad Anap).
- Lavare gli oociti con la soluzione di etichettatura tre volte per rimuovere la tintura in eccesso.
- Eseguire il passo 4.1-4.6.
- Inserire un cubo filtro appropriato per TMR (filtro di eccitazione, specchio dicroico e filtro di emissione) nella torretta del filtro del cubo. Passare al cubo del filtro TMR ruotando la torretta del filtro.
- Leggere la fluorescenza di sfondo per TMR come descritto per Anap nel punto 4.4.
NOTA: L'etichettatura con la tiolchimica provoca un'elevata fluorescenza di fondo a causa di etichettatura non specificata nella membrana. Pertanto, la fluorescenza di sfondo TMR può saturare l'amplificatore (> 2.000 pA). In tal caso, non diminuire l'intensità della luce, ma semplicemente sottrarre la fluorescenza di sfondo aggiungendo una corrente di offset al fotodiodo. Nei sistemi commercialmente disponibili, utilizzare la funzione "sample-and-hold" sul sistema di rilevazione. Si noti il valore della fluorescenza di fondo (utilizzando un filtro di densità neutro 10X) in un laboratorio jCome questo valore non verrà registrato (saturazione). - Registrare correnti di tensione e intensità di fluorescenza TMR contemporaneamente come nel punto 4.6.
2. Preparazione e iniezione di oociti
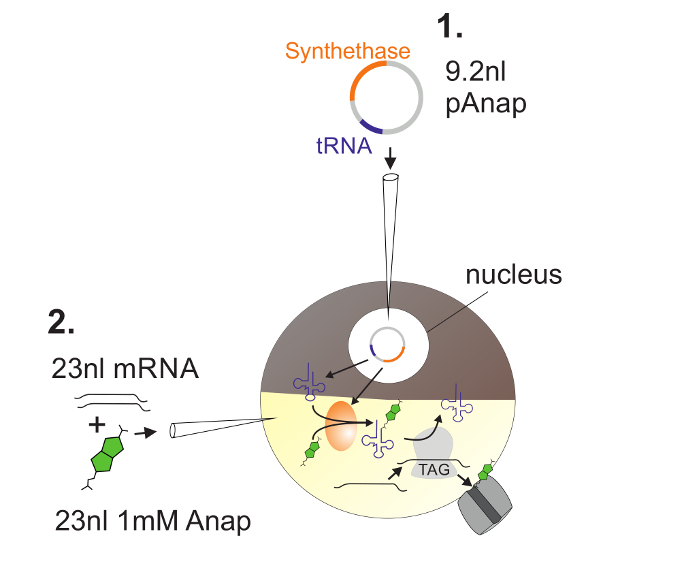
Figura 2 : Illustrazione dell'iniezione del DNA e dell'RNA in Oociti di Xenopus per l'incorporazione di Anap.
In primo luogo, pAnap viene iniettato nel nucleo del oocito Xenopus ( 1 ). Dopo 6-24 h, Anap e il canale RNA vengono coinettati nel polo vegetale ( 2 ). Anap sarà ortogonalmente aminoacilato con il tRNA portante un anti-codone di stop ambra, dalla sintetasi aminoacil-tRNA che è codificata da pAnap. In questo modo, gli aminoacilati Anap-tRNAs sono riconosciuti dal ribosoma al codone di stop ambra inserito nella cassaL RNA, con conseguente soppressione del codone di arresto e inserimento di Anap. Clicca qui per visualizzare una versione più grande di questa figura.
3. Impostazione VCF

Figura 3 < / Strong> : impostazione VCF. ( A ) Vista laterale della configurazione VCF che mostra il percorso della luce all'interno del microscopio. Il cubo filtro contiene un filtro di eccitazione, uno specchio dicroico e un filtro di emissione. ( B ) Le dimensioni della camera di oocyte selezionate sono di 3,4 cm per il raggio superiore della camera (1), 5,5 cm per la lunghezza della camera inferiore (2), 1,4 cm per la larghezza della camera inferiore (3) e 1,7 cm per la larghezza della camera centrale (4). ( C ) Vista frontale della configurazione VCF. Il primo ocularo a sinistra è quello di montare l'oocyte nella camera di serraggio della tensione di taglio e per la permeabilizzazione. Poi, la camera viene scivolata sotto il microscopio al secondo occhio a destra. Qui l'elettrodo V1 viene inserito nell'oocyte utilizzando l'obiettivo 4X e la fluorescenza viene registrata usando l'obiettivo di immersione acqua 40X. Clicca qui per visualizzare una versione più grande di questa figura.
5. VCF a due colori
Access restricted. Please log in or start a trial to view this content.
Risultati
La figura 4 mostra un esempio di registrazioni VCF ottenute da un canale Shaker espresso da oocyte con rimozione rapida di inattivazione (IR), L382stop-W434F in presenza di pAnap e Anap. La mutazione W434F blocca le correnti di potassio ioniche, che consente di misurare gli spostamenti di carica del gating transitorio (correnti gating). Le registrazioni simultanee delle correnti di gating (traccia superiore) e le variazioni dell'intensità di fluorescenza Anap (tracc...
Access restricted. Please log in or start a trial to view this content.
Discussione
L'aminoacilazione in vivo di tRNAs che vengono continuamente trascritti insieme alla sintetasi tRNA, consente di ottenere livelli elevati di espressione per le misurazioni di fluorescenza. Per l'efficace incorporazione fUAA, è fondamentale che pAnap sia iniettato correttamente nel nucleo. A causa dell'incertezza della posizione esatta del nucleo, 10-40% delle iniezioni del DNA dovrebbero fallire, causando oociti non esprimenti (o esprimendo perdite). Pertanto, è importante controllare l'espres...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno niente da rivelare.
Riconoscimenti
PAnap è stato un regalo gentile del Dr. Peter Schultz (Scripps Research Institute). Questo lavoro è stato finanziato dagli istituti canadesi per i finanziamenti di ricerca sanitaria MOP-102689 e MOP-136894 (a RB) e la Fondazione canadese per l'innovazione 950-225005.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Solutions | |||
| Barth's solution | |||
| NaCl | Sigma-Aldrich | S7653 | 90 mM |
| KCl | Fisher Scientific | BP366-500 | 3 mM |
| MgSO4 | Sigma-Aldrich | M-9397 | 0.82 mM |
| CaCl2 | Sigma-Aldrich | C-7902 | 0.41 mM |
| Ca(NO3)2 | Sigma-Aldrich | C-1396 | 0.33 mM |
| HEPES | Sigma-Aldrich | H4034 | 5 mM |
| NaOH hydrate | BDH | BDH7225-4 | pH 7.6 |
| Penicilin | Invitrogen | 15140122 | 100 U/mL |
| Streptomycin | Invitrogen | 15140122 | 100 µg/mL |
| Kanamycin | Invitrogen | 15160054 | 10 mg/100mL |
| Horse Serum (HS) | Invitrogen | 16050122 | 5% |
| SOS Standard Oocyte Solution | |||
| NaCl | Sigma-Aldrich | 746398 | 102 mM |
| KCl | Sigma-Aldrich | 746436 | 3 mM |
| MgCl2 | Sigma-Aldrich | M9272 | 1 mM |
| HEPES | Sigma-Aldrich | H4034 | 5 mM |
| External recording solution | |||
| N-methyl-D-glucamine (NMDG) | Alfa Aesar | L14282 | 115 mM |
| HEPES | Sigma-Aldrich | H4034 | 10 mM |
| Calcium hydroxide | Sigma-Aldrich | 239232 | 2 mM |
| MES hydrate | Sigma-Aldrich | 258105 | pH 7.2 |
| Internal recording solution | |||
| N-methyl-D-glucamine (NMDG) | Alfa Aesar | L14282 | 115 mM |
| HEPES | Sigma-Aldrich | H4034 | 10 mM |
| Ethylenediamine Tetraacetic Acid (EDTA) | Fisher Scientific | E478-500 | 2 mM |
| MES hydrate | Sigma-Aldrich | 258105 | pH 7.2 |
| Labeling solution | |||
| KOH | Fisher Scientific | P250-1 | 115 mM |
| HEPES | Sigma-Aldrich | H4034 | 10 mM |
| Calcium hydroxide | Sigma-Aldrich | 239232 | 2 mM |
| MES hydrate | Sigma-Aldrich | 258105 | pH 7.2 |
| TMR stock solution | |||
| Tetramethylrhodamine-5-maleimide (TMR) | Molcular Probes by Life Technologies | T6027 | 5 mM in DMSO |
| Anap stock solution | |||
| Anap | ABZENA (TCRS) | Custom synthesis TCRS-170 | 1 mM in nuclease-free water and 1% NaOH 1 N |
| Name | Company | Catalog Number | Comments |
| Material/Equipment | |||
| pAnap | Addgene | 48696 | |
| High Performance Oocyte Clamp | Dagan Corporation | CA-1B | |
| Gpatch Acquisition software | Department of Anesthesiology, University of California, Los Angeles | ||
| Analysis software | Department of Anesthesiology, University of California, Los Angeles | ||
| Recording Chamber | Custom machined | ||
| Photo diode detection system | Dagan Corporation | PhotoMax-200/PIN | |
| Electrical shutter driver | UNIBLITZ | VCM-D1 |
Riferimenti
- Mannuzzu, L. M., Moronne, M. M., Isacoff, E. Y. Direct physical measure of conformational rearrangement underlying potassium channel gating. Science. 271 (5246), 213-216 (1996).
- Kalstrup, T., Blunck, R. Dynamics of internal pore opening in K(V) channels probed by a fluorescent unnatural amino acid. Proc Natl Acad Sci U S A. 110 (20), 8272-8277 (2013).
- Xiao, H., Schultz, P. G. At the Interface of Chemical and Biological Synthesis: An Expanded Genetic Code. Cold Spring Harb Perspect Biol. 8 (9), (2016).
- Blunck, R. Chapter 9. Handbook of Ion Channels. , CRC Press. 113-133 (2015).
- Chatterjee, A., Guo, J., Lee, H. S., Schultz, P. G. A genetically encoded fluorescent probe in mammalian cells. J Am Chem Soc. 135 (34), 12540-12543 (2013).
- DeBerg, H. A., Brzovic, P. S., Flynn, G. E., Zagotta, W. N., Stoll, S. Structure and Energetics of Allosteric Regulation of HCN2 Ion Channels by Cyclic Nucleotides. J Biol Chem. 291 (1), 371-381 (2016).
- Shen, B., et al. Genetically encoding unnatural amino acids in neural stem cells and optically reporting voltage-sensitive domain changes in differentiated neurons. Stem Cells. 29 (8), 1231-1240 (2011).
- Aman, T. K., Gordon, S. E., Zagotta, W. N. Regulation of CNGA1 Channel Gating by Interactions with the Membrane. J Biol Chem. 291 (19), 9939-9947 (2016).
- Haddad, G. A., Blunck, R. Mode shift of the voltage sensors in Shaker K+ channels is caused by energetic coupling to the pore domain. J Gen Physiol. 137 (5), 455-472 (2011).
- Batulan, Z., Haddad, G. A., Blunck, R. An intersubunit interaction between S4-S5 linker and S6 is responsible for the slow off-gating component in Shaker K+ channels. J Biol Chem. 285 (18), 14005-14019 (2010).
- Kusch, J., et al. How subunits cooperate in cAMP-induced activation of homotetrameric HCN2 channels. Nat Chem Biol. 8 (2), 162-169 (2012).
- Lee, H. S., Guo, J., Lemke, E. A., Dimla, R. D., Schultz, P. G. Genetic incorporation of a small, environmentally sensitive, fluorescent probe into proteins in Saccharomyces cerevisiae. J Am Chem Soc. 131 (36), 12921-12923 (2009).
- Summerer, D., et al. A genetically encoded fluorescent amino acid. Proc Natl Acad Sci U S A. 103 (26), 9785-9789 (2006).
- Kalstrup, T., Blunck, R. Reinitiation at non-canonical start codons leads to leak expression when incorporating unnatural amino acids. Sci Rep. 5, 11866(2015).
- Liu, H., Naismith, J. H. An efficient one-step site-directed deletion, insertion, single and multiple-site plasmid mutagenesis protocol. BMC Biotechnol. 8, 91(2008).
- Beckert, B., Masquida, B. Synthesis of RNA by in vitro transcription. Methods Mol Biol. 703, 29-41 (2011).
- Goldin, A. L. Maintenance of Xenopus laevis and oocyte injection. Methods Enzymol. 207, 266-279 (1992).
- Rudokas, M. W., Varga, Z., Schubert, A. R., Asaro, A. B., Silva, J. R. The Xenopus oocyte cut-open vaseline gap voltage-clamp technique with fluorometry. J Vis Exp. (85), (2014).
- Zhao, J., Blunck, R. The isolated voltage sensing domain of the Shaker potassium channel forms a voltage-gated cation channel. Elife. 5, (2016).
- Posson, D. J., Ge, P., Miller, C., Bezanilla, F., Selvin, P. R. Small vertical movement of a K+ channel voltage sensor measured with luminescence energy transfer. Nature. 436 (7052), 848-851 (2005).
- Chanda, B., Asamoah, O. K., Blunck, R., Roux, B., Bezanilla, F. Gating charge displacement in voltage-gated ion channels involves limited transmembrane movement. Nature. 436 (7052), 852-856 (2005).
- Taraska, J. W., Puljung, M. C., Zagotta, W. N. Short-distance probes for protein backbone structure based on energy transfer between bimane and transition metal ions. Proc Natl Acad Sci U S A. 106 (38), 16227-16232 (2009).
- Baker, B. J., et al. Genetically encoded fluorescent sensors of membrane potential. Brain Cell Biol. 36 (1-4), 53-67 (2008).
- Miranda, P., et al. State-dependent FRET reports calcium- and voltage-dependent gating-ring motions in BK channels. Proc Natl Acad Sci U S A. , (2013).
- Sisido, M., Ninomiya, K., Ohtsuki, T., Hohsaka, T. Four-base codon/anticodon strategy and non-enzymatic aminoacylation for protein engineering with non-natural amino acids. Methods. 36 (3), 270-278 (2005).
- Hohsaka, T., Ashizuka, Y., Murakami, H., Sisido, M. Five-base codons for incorporation of nonnatural amino acids into proteins. Nucleic Acids Res. 29 (17), 3646-3651 (2001).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon