このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
レンチウイルスベクター人間にエピゲノム編集ツールの効率的な配信用誘導多能性幹細胞由来の疾患モデル
要約
編集を表す DNA エピゲノム強力な治療アプローチを対象としました。このプロトコルは、生産、精製、ひと誘導多能性幹細胞 (こと) のエピゲノム編集アプリケーションに CRISPR dCas9 DNMT3A 遺伝子をかくまっているオール ・ イン ・ ワン レンチウイルスベクターの濃度について説明します-ニューロンを派生しました。
要約
による細胞の使用は、人間の神経変性疾患の研究に貴重なアプローチを表します。ここでは、α-シヌクレイン遺伝子 (SNCA) 座の 3 倍にパーキンソン病 (PD) 患者由来 hiPSCs の差別化のための最適化されたプロトコルについて述べる-関連するドーパミン作動性ニューロン集団。SNCAの高レベルが特にこれらのSNCA表現の規制をターゲット、PD の新規治療法を確立する必要は満たされていない討認識の開発の原因となることを示している証拠の蓄積我々最近 epigenetically SNCAイントロン 1 規制地域でメチル化レベルを富ませることによってSNCAの転写を調節する CRISPR/dCas9-DNA メチル化-ベースのシステムを開発しました。死んでから成るシステムを提供する Cas9 の (非アクティブ) バージョン (dCas9) は、DNA メチル基転移酵素酵素 3 a の触媒ドメインと融合 (DNMT3A)、レンチ ウイルスのベクトルを使用しています。このシステムは、 SNCA遺伝子座の 3 倍のセルに適用されます、 SNCAmRNA および蛋白質レベルSNCAイントロン 1 のターゲット DNA のメチル化を約 30% 削減します。SNCA レベルの微調整のダウンレギュレーションは、病気に関連した細胞表現を救出します。現在のプロトコルの神経前駆細胞 (Npc) と確立とSNCAのメチル化プロファイルの評価ピロシーケンス法の検証 hiPSCs を区別するための手順を説明を目指しますイントロン 1.詳細にこれらの実験で使用するレンチ-CRISPR/dCas9 システムのアウトラインを作成するには、このプロトコル記述を生成し、浄化、レンチウイルスベクターを集中およびエピゲノムとゲノムの編集アプリケーションを使用して自分の適性を強調表示する方法hiPSCs と Npc。プロトコルは、容易に適応可能であるし、in vitro および in vivo のアプリケーションのための高価レンチを生成する使用ことができます。
概要
複数のエピゲノム編集プラットフォームは、最近遺伝子式1,2を制御する領域の任意の DNA シーケンスをターゲットとして開発されたが。作成したエピゲノム編集ツールは、(i) の転写を調節する、(ii) 変更ヒストン翻訳後修飾、(iii) DNA のメチル化の変更、(iv) 規制要素の相互作用を調節するのに設計されています。非アクティブ化された (死んで) Cas9 に転写・ クロマチン修飾子をアンカーへのアプローチ (dCas9) 発生以前から亜鉛などの先進のエピゲノム編集プラットフォーム指蛋白質 (ZFPs) と転写活性化因子のようなエフェクタ (テイルズ)強力な転写エフェクター ドメインをかくまっている (ED) の設計の DNA 結合ドメイン (DBD)3を融合しました。活性化や抑圧など所望の表現型の結果は、内在性の遺伝子座 (図 1) に固定されてエフェクター分子によって定義されます。プログラム可能な転写活性化因子を作成するには、dCas9/gRNA モジュールは VP164,5,6 (図 1 a)、Pol II と一般転写機械をリクルートするウイルス活性化ドメインにリンクされます。このシステムの変更は、VP64、VP16 ドメインの四量体よりも強力な活性化率5,6を提供する含まれています。システムは、正常にプロモーターと規制要素をターゲットしてコーディングおよび非翻訳領域をアクティブに採用されています。重要なは、にもかかわらず、VP64 分子は、ターゲット領域のクロマチン構造を直接変更しないでください、それはクロマチンの修飾子は H3 ・ H4 アセチル化と H3 K4 di としてを含むアクティブ (ユークロマチン) マークの沈着の結果をバインドを募集/tri-メチル化5,6。VP64、に加えて人間の NF-κ B の複合体の p65 サブユニットを dCas9/gRNA モジュール7に繋留されています。興味深いことに、強い遺伝子発現誘導の結果地域上流転写開始部位 (TSSs) とプロモーター内にこれらのエフェクターのテザリングします。それにもかかわらず、VP64、p65 のエフェクタにリンクされている遠位エンハンサー7、8TSSs の下流側にある地域も賦活効果を発揮できます。堅牢な転写応答を引き出す複数の dCas9 VP64 または dCas9 p65 の融合が単一ターゲットの軌跡9,10に募集する必要があります。DCas9 VP64 融合対応11比較活性化強力な機能で単一 dCas9 gRNA 複合体、SunTag などによって複数のエフェクター ドメインを採用する次世代の活性剤の最近の開発をもたらしたなど,12. 転写制御の改善、VP64、p65、Rta (VPR)、dCas913 (図 1 a) の C 末端に γ-ヘルペス ウイルスの転写活性化ドメインの融合によって取得されました。同様の CRISPR/dCas9 システムは、ターゲット固有の抑圧 (図 1 b) に開発されています。
内因性遺伝子の抑圧は、さまざまなメカニズム (図 1 b) を人工リプレッサーの融合で実現できます。(なくてもエフェクター ドメイン/s) は、リプレッサー DBD にリンク CRISPR/dCas9 システムが遺伝子発現プロモーターや上流/下流 TSS 地域3,6 につながれたときに沈黙が効率的にそれが実証されています、14。転写に及ぼす影響は、転写因子結合と RNA ポリメラーゼ加工立体干渉が原因です。それにもかかわらず、単独で立体障害によって遺伝子発現抑制が頻繁に堅牢な沈黙のために十分より包括的なアプローチが必要です。転写リプレッサー ドメイン (Trd) ヒストン修飾子を運ぶ CRISPR/dCas9 システムに基づくサイレンサーの次世代の最近の開発 (H3 K9 ディ-/トリ メチル化、H3 K27 ディ-/tri-メチル化;H3-K36 ・ ディ ・-/トリ メチル化、脱アセチル化 H3 ・ H4)、およびエピジェネティックなサイレンシング効果4,5,15,16より堅牢なツールの建設につながった (CpG) DNA メチル化 17,18,19,20。それは、DNA にこれらのエピジェネティックな修飾子の募集は通常より強力なサイレンシング結果21,22に生成より閉鎖と凝縮したクロマチンの形成につながることを実証されています。つながれてで使用される最も一般的サイレンシング ドメインは Krüppel 関連付けられているボックス (ボブ)4,5です。要因の採用は、クロマチンの変化に対応する実証されています。それにもかかわらず、これらの変更のメカニズムはまだ解明16,17,18をします。最近では、それは DNA に KRAB の局在化はヒストン メチルトランスフェラーゼ SETDB1 のヒストンの脱アセチル化 (HDAC) NuRD 複合体で、これらの相互作用の形成を仲介する可能性が示唆されたアセンブリを促進することが示されています。chromatin の凝縮が、転写のサイレンシング3,13。別のアプローチとしてエフェクター ドメインはつながれてカスタム エピジェネティックなサイレンシング蛋白質を作成するために融合されます。このシステムは、抑圧的な DNA のマークやヒストンの修正に直接触媒します。
最近、DNMT3A 酵素につながれた合成の CRISPR/dCas9 システムの使用用途が変更されて転写不活性化のため。DNMT3A 内因性遺伝子プロモーターと他の規制地域 (図 1 b)18,20異質染色質の形成を通して転写抑制を発揮する DNA のメチル化を触媒します。マクドナルドら18とボイタら20エピゲノム遺伝子サイレンシングまたはプラスミド配信 dCas9 DNMT3A の融合システムが高めることができる強力デモ抑圧用 DNA のメチル化することができますを報告する最初の作家であったTSS18,20前後のシトシンのメチル化。マクドナルドと同僚戦略の雇用により、大幅に低減 (約 40%) されることを示した腫瘍抑制遺伝子CDKN2A mRNA レベル18です。同様に、バッハやIL6ST遺伝子の非メチル化プロモーター領域をターゲット遺伝子発現20二重減少と相関している増加の CpG メチル化を示しています。私たちの研究室は最近SNCA過剰発現 (図 2)23の病理結果を減衰のため DNA のメチル化の使用を再利用します。戦略に基づくSNCAイントロン 1 領域内での DNA メチル化の選択的強化以前 PD と痴呆 Lewy ボディ (DLB) 脳24,25、hypomethylated に報告されました。 26。この低いメチル化は、 SNCAの過剰発現は、治療的介入24,27,28にとって魅力的なターゲットを提供していますにリンクされています。我々 は最近、 SNCAイントロン 1 のSNCA 3 倍23PD 患者から得られたこと由来ドーパミン Npc の領域 DNA のメチル化の低レベルを示した。この実験モデルの利点は、Npc の文化で確実伝達またはさらに酸化を含む細胞の表現型を仲介する遺伝的要因を識別するために有効なスクリーニングを可能に成熟したニューロンに区別できることストレス、アポトーシス29。さらに、このモデル システムにより患者の症状発現前に生じた発達のイベントを要約する科学者です。さらに、Npc のこと由来は遺伝子発現に関連する細胞・分子経路をテストするための素晴らしいツールを表します。重要なは、CRISPR/Cas9-エピゲノム技術と組み合わせること由来の Npc では、多くの神経変性疾患の「次世代治療薬」の開発が非常に容易します。
SNCA 式の病理組織学的レベルを減らすためには、最近 dCas9 DNMT3A 融合タンパク質と具体的ターゲット CpG のメチル化SNCAイントロン 1 (図 2 a) 内に gRNA を運ぶレンチ ベースのシステムを開発した23。このプロトコルは、レンチウイルスベクター (LV) の設計・製作の詳細について説明します。LVs を表す CRISPR/dCas9 コンポーネントを提供するいくつかの理由のための効果的な手段、すなわち (i) かさばる DNA を運ぶ能力を挿入します、(ii)30 分割と非分裂細胞を含む細胞の広い範囲の高効率、および (iii) 最小限の細胞毒性や免疫反応を誘導する能力を。最近、 SNCA遺伝子座の 3 倍の患者から LV システムをによる由来ドーパミン作動性ニューロンに適用してエピゲノム編集の配信の LVs の治療の可能性を示すメチル化ツール23 (図 2B)。確かに、LV gRNA/dCas9 DNMT3A システムSNCAイントロン 1 領域における DNA メチル化の大幅な増加が発生します。この増加は、 SNCA mRNA および蛋白質23のレベルの減少で対応しています。また、 SNCAダウンレギュレーションSNCA 3 倍/による由来ドーパミン作動性ニューロン (例えば、ミトコンドリア ROS 生産と細胞生存率)23で PD に関連した表現を救出します。重要なは、我々 は実証 LV-gRNA-dCas9-DMNT3A システムによるSNCA発現の減少は、を行った PD 患者からによる由来ドーパミン作動性ニューロンの特徴である表現型を反転が可能SNCA 3 倍に、ミトコンドリア ROS 生産と細胞生存率23など。このプロトコルの目的は、生産のプロトコルおよび濃度ウイルス製剤の高 tittered を生成するための最適化された LV プラットフォームの概要を説明するには 1) と 2) hiPSCs の成熟したドーパミンになるパターンの Npc への分化を説明するにはニューロン31,32とSNCAイントロン 1 内の対象領域のメチル化レベルの評価。
レンチ ウイルス プラットフォームすなわちアデノ随伴ベクトル (通気)、大きい遺伝的挿入33,34に対応する元の能力である最も人気のあるベクトル プラットフォーム上の大きな利点があります。通気は有意に高い利回りで生成することができますが、低包装容量を所有 (< 4.8 kb)、オール イン ワン CRISPR/Cas9 システムを提供するための使用を損なうこと。したがって、それは LVs の CRISPR/dCas9 ツールの配信に関連するアプリケーションのプラットフォームの選択になるようです。したがって、ここで説明したプロトコルは細胞や臓器にエピゲノム編集コンポーネントを効果的に配信することを望む研究者にとって貴重なツールになります。プロトコルをさらにベクトル式カセット30,35内の要素の cis 諸国における変更を介してベクトルの生産と表現能力を増すための戦略の概要を説明します。戦略は基づいています小説にシステム開発し私たちの研究室で勉強し、1010ウイルス ユニット (VU)/mL30,35の範囲でウイルス粒子を生成する能力を強調表示します。
プロトコル
1. システム設計・ ウイルス制作
- プラスミド設計・施工
注:生産を使用して、すべての 1 つの LV-gRNA-dCas9-DNMT3A ベクターの構築を実行- と Ortinski ら30刊式最適化式カセット。ベクトル カセット運ぶ転写因子 Sp1 と最新の削除、無変換内の認識サイトの繰り返し (U3') 3'-長いターミナルの繰り返し (LTR) (図 2 a)30,36の領域。ベクター バックボーンを提供し、CRISPR/Cas930,35を表現で効果的な発見されています。- SpCas9 の非アクティブ化 (死者) バージョンを入手 (dCas9) 経由でサイト指示された突然変異誘発 (データは示されていない)。PBK30129上井病原フラグメント (図 3) 間の交換によってアクティブな Cas9 とそれぞれ、D10A と H840A 突然変異は酵素のアレイと RuvC の触媒ドメインでをかくまっているクローンを交換してください。
- DNMT3A の触媒ドメインに由来する pdCas9 DNMT3A eGFP (材料の表を参照してください)、DNMT3A の増幅によって病原-429/R 5 '- GAGCGGATCCCCCTCCCG - 3' 病原-429/L 5' - CTCTCCACTGCCGGATCCGG - の部分 3' (図 3)。DNMT3A を含む領域を増幅する次の条件を使用して、: (1) 95 ° C、60 s、10 (2) 95 ° C (3) 60 ° C、20 s s、60 s. 繰り返し条件 2 に 4 30 x (4) 68 ° C。最終延長 3 分 68 ° C を使用し、4 ° C を保持
- DCas9 を運ぶ修正 pBK301 ベクトルの病原のサイトに病原制限の酵素によって消化 DNMT3A フラグメントをクローンします。直接サンガーによるクローニングを確認シーケンス。DCas9 DNMT3A-p2a ピューロマイシン transgene をもたらしたプラスミドに港湾に注意してください。プラスミッドは人間の U6 プロモーター (図 3) から gRNA 足場を表しています。
- ピューロマイシン レポーターの遺伝子を緑色蛍光タンパク質 (GFP) dCas9 DNMT3A p2a GFP を作成するに置き換えます。FseI と dCas9 DNMT3A-p2a Puro プラスミドを消化します。ゲル精製法によるベクトル フラグメントを浄化します。PBK201a の消化によって挿入の準備 (pLenti-GFP) FseI で。FseI フラグメントのベクターにクローンを作成します。結果プラスミド pBK539 dCas9 DNMT3A p2a GFP 遺伝子 (図 3) を隠し持っています。
- HEK 293 t 細胞を培養とトランスフェクション細胞をめっき
注:人間の萌芽期の腎臓 293 t (HEK 293 t) 細胞を培養している完全な高血糖でダルベッコ変法イーグル培地 (DMEM; 10% ウシ血清、1 x 抗生物質抗真菌薬, ピルビン酸ナトリウム x 1, 1 x 非必須アミノ酸、2 mM L グルタミン) 5 %37 ° C で CO2。 プロトコルの再現性、異なる多く/バッチへの切り替え時の子牛の血清をテストする勧めします。6 15 cm までレンチ ウイルスの生産のためのプレートが必要です。- 低通過セルを使用して、新しい文化を (20 の通路より低い) 開始。セルが 90-95% の合流点に達すれば、メディアを吸引し、リン酸緩衝生理食塩水 (PBS) x 滅菌 1 で優しく洗ってください。
- トリプシン-EDTA (0.05%) 2 mL を追加します。37 ° c 3-5 分間加温します。解離試薬を不活化できる、完全な高グルコース DMEM と 10 mL の血清ピペット ピペット 10 x-15 x 4 x 10 6/mL の細胞の単一細胞懸濁液を作成する 8 mL を追加します。
- Transfections の 0.2% のゼラチンと 15 cm プレートをコートします。高グルコース培地の 22.5 mL 追加し、細胞懸濁液 (合計 〜 1 107セル/プレート x) の 2.5 mL を追加することによって、細胞を播きます。70%-80% の合流点に到達するまでは、5% CO2と 37 ° C で版を孵化させなさい。
- HEK 293 t 細胞をトランスフェクション
- ・ ラガバンとカンター35によると BES 緩衝液掲示板 × 2 と 1 M CaCl2を準備します。0.22 μ m のフィルターを介してそれらを渡すことによって、ソリューションをフィルター、4 ° C で保管トランスフェクション ミックスは、細胞への添加前に明確にすること。ミックスは、インキュベーション中に曇りになると、新鮮な 2 x BBS を準備 (pH = 6.95)。
- 示すように 4 つのプラスミド プラスミド ミックスを準備する使用 (次のミックスは 1 つ 15 cm プレートの十分な): CRISPR/dCas9-転送ベクトルの 37.5 μ g (pBK492 [DNMT3A Puro NO gRNA] または pBK539 [DNMT3A GFP なし gRNA]);pBK240 の 25 μ g (psPAX2);pMD2.G; 12.5 μ gpRSV rev (図 4 a) の 6.25 μ g。濃度に基づくプラスミドの体積を計算し、15 mL の円錐管に必要な量を追加します。1 M CaCl2 312.5 μ L を追加し、1.25 mL に最終巻をもたらす、ボルテックス ミックス中 2 の x BBS ソリューションの 1.25 mL 滅菌 dd H2を o. 軽くを使用して追加。室温で 30 分間インキュベートします。セルは、彼らが 70-80% の合流 transfection のため準備ができています。
- メディアを吸引し、血清せず作りたての高グルコース DMEM の 22.5 mL と置き換えます。各 15 cm プレートに滴下トランスフェクション混合物を 2.5 mL を追加します。渦巻き模様のプレートと 5% CO2 2-3 h の 37 ° C で孵化させなさい。
- 3 h 後 2.5 mL (10%) を追加します。プレートごと血清の 5% CO2と 37 ° C で一晩インキュベートし、。
- 日 1、トランスフェクション後、あることを確認するために細胞を観察なしまたは最小限の細胞死と細胞がコンフルエント文化 (100%) を形成しました。
- メディアを変更するには、各プレートに作りたての高グルコース DMEM と 10% 血清 25 mL を追加します。
- 5% CO2 48 h の 37 ° C で孵化させなさい。
- ウイルスの収穫
- すべての transfected セルから上澄みを収集し、50 mL の円錐管にそれらをプールします。400-450 x gで 10 分間遠心フィルター 0.45 μ m の真空フィルター ユニットを介して上清です。濾過後、上清は短期記憶 (最大 4 日) のための 4 ° C で保存できます。長期的なストレージの因数を準備し、-80 ° C で保存
注:Nonconcentrated ウイルス製剤が予想される ~ 10 x 2 3 x 107 vu/mL に7 (力価測定 1.5 を参照してください)。複数凍結融解サイクル機能価で 10% – 20% の損失になりますので、使い捨ての因数を準備することを強くお勧めします。
- すべての transfected セルから上澄みを収集し、50 mL の円錐管にそれらをプールします。400-450 x gで 10 分間遠心フィルター 0.45 μ m の真空フィルター ユニットを介して上清です。濾過後、上清は短期記憶 (最大 4 日) のための 4 ° C で保存できます。長期的なストレージの因数を準備し、-80 ° C で保存
- ウイルス粒子の濃度
注:浄化、ショ糖勾配ステップとショ糖のクッション ステップを含む二段二重ショ糖法を行った (図 4 b)- ショ糖密度勾配を作成するには、次の順序で円錐形遠心チューブを用意: 1 × PBS、0.5 mL DMEM、DMEM、30% のショ糖の 1 mL、2 mL の 1x PBS で 20% ショ糖の 60% のショ糖の 70% のショ糖の 0.5 mL。
- 慎重に、セクション 1.4, によると収集した上清をグラデーションに追加します。4 15 cm プレートから収集した総量は 100 mL なのでウイルス上清を処理するのに六つの遠心管を使用します。
- 各遠心管中でウイルス上清を均等に分散します。遠心分離中にチューブの破損を避けるため、その総容量の少なくとも 4 分の 3 を遠心管を記入します。1x PBS で管のバランスをとる。遠心分離機の 17 ° C で 2 時間 70,000 x gでサンプル
注:加速と減速の手順中にショ糖層を維持するためにゆっくり加速・減速ローター 0 から 200 g x、0xg200 から、スピンの最初と最後の 3 分間それぞれ超遠心機を許可します。 - 優しくきれいな管 (図 4 b) に 30-60% ショ糖分数を収集します。容量 100 mL に PBS (冷たい) アップ x 1 を追加します。複数回のピペッティングで混ぜます。
- 慎重にチューブに 20% (1 x PBS) でショ糖の 4 mL を追加してショ糖クッションにウイルスの準備を分類します。ピペッティング 〜 20-25 まで各管ごとにウイルス解決策の mL。その内容のボリュームがチューブあたり 4 分の 3 未満の場合 1 × PBS でチューブを入力します。チューブの慎重にバランスをとる。17 ° C で 2 時間 70,000 × gで遠心します。上清を空にし、残りを許可するペーパー タオルの管を反転排出する液体。
- すべての液体を削除するには、慎重に残りの液体を吸引します。なお、この段階で、ウイルスを含むペレットは、ほとんど半透明の小さな白い斑点として表示されます。ペレットを再懸濁します初の管に 1x PBS の 70 μ L を追加します。徹底的にピペットの懸濁液、すべての餌を再停止されるまで、次のチューブに転送。
- 1 × PBS とミックスする前に追加の 50 μ L でチューブを洗浄します。注意してください、この段階で最終的な懸濁液の量は 〜 120 μ L と若干乳白色が表示されます。明確な懸濁液を得るためには、10,000 x gで 60 s の遠心分離を続行します。上清を新しいチューブに転送、5 μ 因数を作る、-80 ° C で保存
注:レンチウイルスベクターの準備は、凍結融解の繰り返しに敏感です。さらに、残りの手順が培養封じ込めで行うかで指定されたエリア (図 4 b) バイオ セーフティ規格の適切なレベルでされている面で修飾されてお勧めします。
- ウイルス抗体価の定量
注:ウイルス抗体価の推定 (p24ギャグELISA) p24 酵素免疫測定法 (ELISA) 法を用いて、国立衛生研究所 (NIH) エイズ ワクチン プログラムによると HIV 1 p24 抗原をキャプチャ試金のためのプロトコル若干の変更。- 96 ウェル プレート 3 の井戸を洗う 0.05% Tween 20 冷 1 x PBS (PBS-T) 200 μ L を使用して x。
- プレートをコートで 1:1,500 1x PBS で希釈したモノクローナル抗 p24 抗体の 100 μ L を使用します。4 ° C で一晩プレートを孵化させなさい
- ブロッキング試薬 (1% ウシ血清アルブミン [BSA] 1 × PBS) を準備し、非特異的な結合を避けるためにも 200 μ L を追加します。200 μ L の PBS-T を使用して、室温で少なくとも 1 時間も 3 倍を洗浄します。
- サンプル準備を進めます。集中のベクトル準備を使用する場合 (最終濃度 10%) で 1 μ L のサンプル、dd H20, 89 μ およびトリトン X-100 の 10 μ L を使用して 1: 100 でベクトルを希釈します。Nonconcentrated の準備のため 1:10 でサンプルを希釈します。
- (開始の集中は 5 ng/mL) 二重シリアル希釈を使用して HIV-1 規格を取得します。
- 1:10, 000 を取得する 0.2% Tween 20 と 1 %bsa 添加 RPMI 1640 年に集中してサンプル (1.6.4 の手順で準備) を希釈 1:50, 000、および 1:250,000 希薄。同様に、1: 500、1:2 を確立する 0.2% Tween 20 と 1 %bsa 添加 RPMI 1640 年 (1.6.4 の手順で準備) nonconcentrated サンプルを希釈 500、および 1:12,500 希薄。
- トリプリケートでプレートのサンプルおよび標準を追加します。4 ° C で一晩インキュベートします。
- 次の日、洗浄 6 x。
- RPMI 1640、10% 牛胎児血清 (FBS)、0.25 %bsa、2% に正常マウス血清 (NMS) の縮尺で希釈したウサギ ポリクローナル抗 p24 抗体の 100 μ L を追加し、37 ° C 4 時間インキュベートします。
- 井戸 6 x。ヤギ抗うさぎ西洋ワサビペルオキシダーゼ 5% ヤギ血清、2 %nms、0.25 %bsa、0.01% 添加 RPMI 1640 年に 1: 10,000 で希釈した IgG を追加トゥイーン 20。37 ° C 1 時間インキュベートします。
- 井戸 6 x。TMB ペルオキシダーゼ基質を追加し、15 分間室温でインキュベートします。
- 反作用を停止し、1 N の HCl の 100 μ L を追加します。マイクロ プレート リーダーで測定吸光度 450 nm。
- 蛍光レポーター強度の測定
- ウイルスの懸濁液を使用すると、1 × PBS で (10-1 10-5) から十倍のシリアル希薄を取得できます。
- 6 ウェル プレートの各ウェルに 5 × 105 HEK 293 t 細胞をプレートします。セルに各ウイルスの希釈の 10 μ L を適用し、, 5% CO2 48 h の 37 ° C で。
- 蛍光活性化セル (FACS) 分析を次のように並べ替えを続行: 0.05% トリプシン-EDTA 溶液 200 μ L を追加することによって細胞をデタッチします。37 ° C、5 分で細胞をインキュベートし、(血清) を DMEM 培地 2 mL にそれらを再懸濁します。15 mL の円錐管に 4 ° C で 400 × gで遠心するサンプルを収集します。500 μ L の冷 1x PBS でペレットを再懸濁します。
- 4% パラホルムアルデヒド (PFA) 500 μ L を追加することによって、細胞を修正し、室温で 10 分間インキュベートします。
- 4 ° C で 400 × gで遠心し、1 mL の 1x PBS でペレットを再懸濁します。FACS の計測器を使用して Ortinski ら30GFP 発現を分析します。ウイルスの機能抗体価を調べるには、次の数式を使用します。

ここは
Tg = GFP 陽性細胞数
Tn セルの合計数 =
N = 導入されたセルの合計数
V = (マイクロリットル) の伝達に使用されるボリューム。
- GFP 陽性細胞を数える
注:伝達の採用されている感染症 (MOI) の多様性を決定します。モアの広い範囲をテスト (MOI MOI に 1 = = 10)。- シード 3 × 105 6 ウェル プレートの各ウェルあたり 4 × 105 HEK 293 t 細胞。
- セルに達するとき > 80% の合流点、関心の MOI でベクターを使用してそれらを変換します。
- 5% CO2、37 ° C で、1-7 日間の細胞における GFP 信号を監視します。
- GFP 陽性細胞の数をカウントします。蛍光顕微鏡を採用 (0.1 プラン 4 倍対物レンズ 40 倍の倍率、エヌ ・ エイ) GFP フィルターを使用して (励起波長 = 470 nm、発光波長 = 525 nm)。GFP 陰性細胞の対照群を設定するのには、untransduced セルを使用します。
- ウイルスの機能抗体価を決定する次の式を採用してください。
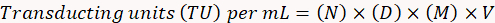
ここは
N = GFP 陽性細胞数
D = 希釈係数;
M = 倍率。
V = ウイルス伝達の使用のボリューム。
注: この例に続いて結果を計算: 10 GFP 陽性細胞 (N) 10-4 (1:10, 000) 10 μ L のサンプル (V) の 20 倍の倍率 (M) で希釈 (D) でカウントの 1 ミリリットルあたり TU (10 x 10 の4) になります (20) x (10) x (100) x = 2 x 108vu/mL。
2. ドーパミン作動性神経前駆細胞の分化
- 養殖 hiPSCs
注: SNCA 遺伝子座、ND34391 の 3 倍の患者から hiPSCs 国立研究所の神経疾患と脳卒中 (NINDS) カタログ (材料表を参照) から得られました。- フィーダー無料 ESC iPSC 培地のフィーダーに依存しない条件下での hiPSCs の文化 (材料の表を参照してください) 基本的なマトリックスの hESC 修飾膜 (BMM) に-塗布鋼板 (材料の表を参照してください)。1 ml DMEM/F12 の合流のコロニーを洗って、解離試薬 1 mL を追加 (材料の表を参照)、室温で 3 分間インキュベートし、。
- 解離試薬を吸引し、フィーダー無料 ESC iPSC 培養培地 1 mL を追加します。
- セル リフターを使用してプレートをこすりし、ホウケイ酸塩のピペットを使用して x ピペッティング 4 x-5 でフィーダー無料 ESC iPSC 培地の 11 mL のコロニーを再懸濁します。
- プレートの BMM コーティング プレート上にコロニー懸濁液 2 mL、5% CO2と 37 ° C でプレートを配置します。毎日中型の変更を行い、分割セルごとに 5-7 日。
- ドーパミン作動性神経前駆細胞への分化
注:HiPSCs のドーパミン作動性神経前駆細胞 (MD Npc) への分化は、わずかな変更31,32 (製造業者の指示に従って市販の神経誘導中型のプロトコルを使用して実行されます。材料のテーブルを見なさい)。分化の最初の日は0 日と見なされます。高品質 hiPSCs は、効率的な神経分化に必要です。MD Npc の誘導、細胞胚様体 (EB)-ベースのプロトコル。- HiPSCs の分化を開始する前に準備マイクロウェル培養プレート (材料の表を参照してください)、製造元の指示に従って。
- マイクロウェル培養プレートの準備ができたら、10 μ M を添加した神経誘導培地 (NIM; 参照テーブルの材料) の 1 つの mL を追加 Y-27632。
- プレートを使用する準備ができるまで置いておきます。
- DMEM/f12 キーで hiPSCs を洗って、細胞剥離液の 1 mL を追加 (材料の表を参照)、5% CO2と 37 ° C で 5 分間インキュベートし、。
- DMEM/f12 キーで単一細胞を再懸濁します、5 分 300 × gで遠心分離機のそれら。
- 慎重に、上清を吸引し、NIM + 10 μ M で細胞を再懸濁します Y-27632 3 x 106セル/mL の最終的な集中を取得します。
- マイクロウェル培養プレートのよく、単一単一細胞懸濁液の 1 mL を追加し、3 分間 100 x gでプレートを遠心します。
- マイクロウェル、間細胞の均一な配分を確保するために、顕微鏡下でプレートを調べ、5% CO2と 37 ° C で細胞をインキュベートします。
- 1-4 日、毎日部分的な中型の変更を実行します。
- 1 mL ピペットを使用して、媒体の 1.5 mL を取り外して廃棄します。ゆっくりと、Y-27632 せず新鮮なニムの 1.5 mL を追加します。
- 4 日目まで 2.2.10 のステップを繰り返します。
- 5 日目、コート BMM.と 6 ウェル プレートのウェル 1 個
- 37 μ m リバーシブル ストレーナーを配置 (材料表参照) 50 mL コニカル チューブ (廃棄物) の上に。リバーシブルのストレーナ上向きの矢印をポイントします。
- マイクロウェル培養プレートから形成された EBs を乱すことがなくメディアを削除します。
- DMEM/f12 キーの 1 mL を加えると速やかにホウケイ酸ピペットと EBs を収集し、こし器を通してそれらをフィルタ リングします。
- すべての EBs がマイクロウェル培養プレートから削除されるまで、手順 2.2.15。
- 新しい 50 mL の円錐管にストレーナを反転し、EBs のすべてを収集する NIM の 2 mL を追加します。
- ホウケイ酸ピペットを使用して BMM コーティング プレートの単一ウェルに EB 懸濁液 2 mL をプレートします。5% CO2と 37 ° C で EBs を孵化させなさい。
- 6 日目NIM + 200 ng/mL SHH の 2 mL を準備 (材料の表を参照してください)、毎日媒体の変更が実行します。
- 8 日目、神経誘導の割合を確認します。
- 取り付けられているすべての EBs をカウントし、具体的には、神経ロゼットで満たされているそれぞれの個別 EB 数を決定します。次の数式を使用してニューラル ロゼット誘導を定量化します。
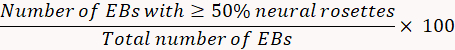
注:神経の誘導がある場合 < 75%、神経ロゼット選択できない場合があります効率的。 - 12 日目、250 mL N2B27 中の neurobasal 中、DMEM/F12 媒体、GlutaMAX、2.5 mL 2.5 mL NEAA、N2 サプリメント、ビタミン A なし B27 の 5 mL の 2.5 mL の 119 mL、ゲンタマイシン (50 mg/mL) の 250 μ L の 119 ml を準備します。、および 19.BSA (7 mg/mL) の 66 μ L。
- 完全 N2B27 培地 50 mL を準備するには、3 μ M を追加 CHIR99021、2 μ M SB431542、20 ng/mL bFGF、20 ng/mL EGF と 200 ng/mL SHH。
注:準備完了したことが重要です使用前右に、中。 - 神経のロゼットを含む井戸から培地を吸引、DMEM/f12 キーの 1 つの mL で洗います。
- 広告ニューラル ロゼット選択試薬 1 mL (材料の表を参照してください), 5% CO2 1 h の 37 ° C でと。
- 選択試薬を削除し、ロゼット クラスターを直接目指す 1 mL pipettor を使用して、します。
- 15 mL の円錐管に懸濁液を追加し、手順 2.2.25 と 2.2.26 ニューラル ロゼット クラスターの大部分を収集されているを繰り返します。
注:1変細胞の種類による汚染を避けるためには、overselect はありません。 - 350 x gで 5 分間吸引ロゼット懸濁液上清を遠心し、N2B27 + 200 ng/mL SHH ニューラル ロゼットを再懸濁します。BMM コーティングよく神経ロゼットの懸濁液を追加し、5% CO2と 37 ° C でプレートを孵化させなさい。
- 13-17 日、完成した N2B27 の媒体を使用して毎日中型の変更を実行します。文化が 80-90% 合流セルを通路します。
- セルを分割するには、BMM コーティング プレートを準備します。
- 1 mL DMEM/f12 キーでセルを洗浄して、培地を吸引、解離試薬 1 mL を追加 (材料の表を参照してください)。
- 37 ° C で 5 分間インキュベート DMEM/f12 キー、1 mL を追加し、上下にピペッティングにより接続されている細胞を取り除きます。15 mL の円錐管に NPC サスペンションを収集します。300 × gで 5 分間遠心します。
- 上清を吸引し、200 ng/mL SHH + 完全な N2B27 の 1 mL の細胞を再懸濁します。
- セルをカウント × 105セル/cm21.25 の密度でそれらをプレートし、5% CO2と 37 ° C でセルを孵化させなさい。
- 毎日、メディアを変更完全な N2B27 + 200 ng/mL SHH を使用して。
注:この一節を Npc が通路 (P) としてと見なされます 0。SHH は、P2 で N2B27 中から撤回することができます。 - 80%-90% の合流点に達すればセルを通路します。
- この段階で細胞が免疫細胞化学と qPCR による Nestin と FoxA2 マーカーを表現することを確認します。このプロトコルは、Nestin と FoxA2 マーカーの 85% 二重陽性細胞の生成に します。
- セルを通過するには、2.2.31–2.2.36 の手順を繰り返します。
- P2 の通路から始まるセルをフリーズします。細胞の凍結の手順 2.2.31–2.2.36 を繰り返し、2 x 106 4 x 106セル/ml 使用培地の凍結冷たい神経前駆細胞ペレットを再懸濁します (材料の表を参照してください)。
- 各クリオバイアルに細胞懸濁液の 1 mL を移すし、標準的な低速速度制御の冷却システムを用いた細胞を凍結します。長期保存のためには、液体窒素で細胞を維持します。
- MD Npc を融解
- BMM コーティング プレートを準備して完全な N2B27 をウォーム アップします。暖かい DMEM/F12 の 10 mL を 15 mL の円錐管に追加します。場所、37 ° C の熱ブロック 2 分をクリオバイアル。
- DMEM/f12 キーを含んでいる管に、クリオバイアルからセルを転送します。300 × gで 5 分間遠心します。
- 上清を吸引、N2B27、2 mL の細胞を再懸濁します、BMM コーティング プレートのウェル 1 個に細胞懸濁液を追加します。5% CO2と 37 ° C でセルを孵化させなさい。
- BMM コーティング プレートを準備して完全な N2B27 をウォーム アップします。暖かい DMEM/F12 の 10 mL を 15 mL の円錐管に追加します。場所、37 ° C の熱ブロック 2 分をクリオバイアル。
3. MD Npc とメチル化の分析の伝達
-
MD Npc の伝達
- MOI で LV gRNA/dCas9 DNMT3A ベクトル 70% 合流点で MD Npc を変換 = 2。N2B27 中 16 h posttransduction を交換してください。
- 5 μ g/mL ピューロマイシン、変換後 48 h で N2B27 を追加します。N2B27 プラス ピューロマイシン安定した MD NPC ラインを取得するに 3 週間培養します。細胞は (DNA、RNA、タンパク質解析、表現型特性23、凍結と継) 下流のアプリケーションの準備ができています。
-
SNCA イントロン 1 のメチル化プロファイルの評価
- 安定して導入された各セルからの DNA の抽出 DNA 抽出法を用いたライン キット (材料の表を参照してください)。
- 重亜硫酸塩の変換は、市販を使用してを実行する DNA の使用 800 ng キット (材料の表を参照してください)。重亜硫酸塩の変換後 20 ng/μ L の重亜硫酸塩の変換された DNA を溶出します。
-
パイロシークエンス分析のための PCR
- ヌクレアーゼ フリー チューブ PCR マスター ミックスを準備します。各反作用のため逆プライマー (10 μ M)、前方のプライマー (10 μ M)、MgCl2 (25 mM) CoralLoad 集中、2 x PCR マスター ミックスの 10 μ L、5 の 4 μ L x 10 の 2 μ L の 1.6 μ L の 0.4 μ L の 0.4 μ L を使用して DNA の 1 μ L、Q ソリューション x、、ヌクレアーゼ フリー水の 0.6 μ L。
- たちにリアクション プレートを転送し、次の条件を使用して PCR を行う: 95 ° C、15 分、30 94 ° C の 50 サイクル s 56 ° C、30 s、および 30 のための 72 ° C s 72 ° C で最終 10 分拡張ステップとSNCA イントロン 1 のパイロシークエンス用プライマーは、表 1図 7 aと補足図 1に表示されます。
- 増幅後エチジウム ブロマイド染色、次アガロース電気泳動で PCR の製品の 2 μ L を使用して amplicons を視覚化します。
- パイロシークエンス試金は非メチル化 (U) の混合物を使用して検証および次の比、すなわち 100U:0M、75U:25M、50U:50M、25U:75M、および 0U:100M (材料の表を参照) で (M) の重亜硫酸塩の変換された Dna をメチル化します。
- パイロシークエンス パイロシークエンス試薬 (材料の表を参照) を使用してを行いパイロシークエンス ソフトウェアを使用して各 CpG サイトのメチル化値を計算します。詳細なピロシーケンスプロト Bassil ら37を参照してください。
結果
比べて、素朴な GFP、LV-dCas9-DNMT3A-GFP/Puro ベクトルの生産キャプションの検証
P24ギャグ素朴な GFP/プーロのカウンター パートと LV-dCas9-DNMT3A-GFP/プーロの物理的な抗体価を比較する ELISA を行った。ここに概説されたプロトコルを使用して生成されたベクトルの物理的な利回り匹敵する代表的な結果は、
ディスカッション
LVs は、遺伝性疾患の中で特に、エピゲノムの編集のための最適な手段として浮上し始めている、主に能力 (i) に対応するため大規模な DNA ペイロード、および (ii) 効率的に変換分割の広い範囲と非分裂細胞。LVs の大型包装効果が特大 CRISPR/dCas9 システムのパッケージを含むアプリケーションは特に有益です。このような観点からは、LVs は、オール イン ワン CRISPR/Cas9 システムの配信のための?...
開示事項
デューク大学は、この研究に関連する仮特許出願を提出しました。
謝辞
この作品はカーン ニューロ開発賞受賞 (の OC) と国家機関の健康/国立研究所の神経疾患脳卒中 (NIH/NINDS) 部分で資金が供給された (R01 NS085011 の OC に)。
資料
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| 0.22 μM filter unit, 1 L | Corning | 430513 | |
| 0.45-μm filter unit, 500 mL | Corning | 430773 | |
| 100 mm TC-Treated Culture Dish | Corning | 430167 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 50 mL conical centrifuge tubes | Corning | 430291 | |
| 6-well plates | Corning | 3516 | |
| Aggrewell 800 | StemCell Technologies | 34811 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| BD FACS | Becton Dickinson | 338960 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| Eppendorf Cell Imaging Slides | Eppendorf | 30742060 | |
| High-binding 96-well plates | Corning | 3366 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Reversible Strainer | StemCell Technologies | 27215 | |
| SW32Ti rotor | Beckman Coulter | 369650 | |
| VWR® Disposable Serological Pipets, Glass, Nonpyrogenic | VWR | 93000-694 | |
| VWR® Vacuum Filtration Systems | VWR | 89220-694 | |
| xMark™ Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| Accutase | StemCell Technologies | 7920 | |
| Anti-Adherence Rinsing Solution | StemCell Technologies | 7010 | |
| Anti-FOXA2 Antibody | Abcam | Ab60721 | |
| Anti-Nestin Antibody | Abcam | Ab18102 | |
| Antibiotic-antimycotic solution, 100x | Sigma Aldrich | A5955-100ML | |
| B-27 Supplement (50x), minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| BES | Sigma Aldrich | B9879 - BES | |
| Bovine Albumin Fraction V (7.5% solution) | Thermo Fisher Scientific | 15260037 | |
| CHIR99021 | StemCell Technologies | 72052 | |
| Corning Matrigel hESC-Qualified Matrix | Corning | 08-774-552 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| DMEM-F12 | Lonza | 12-719 | |
| DMEM, high glucose media | Gibco | 11965 | |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | |
| EpiTect PCR Control DNA Set | Qiagen | 596945 | |
| EZ DNA Methylation Kit | Zymo Research | D5001 | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Gentamicin | Thermo Fisher Scientific | 15750078 | |
| Gentle Cell Dissociation Reagent | stemCell Technologies | 7174 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Human Recombinant bFGF | StemCell Technologies | 78003 | |
| Human Recombinant EGF | StemCell Technologies | 78006 | |
| Human Recombinant Shh (C24II) | StemCell Technologies | 78065 | |
| MEM Non-Essential Amino Acids Solution (100x) | Thermo Fisher Scientific | 11140050 | |
| mTeSR1 | StemCell Technologies | 85850 | |
| N-2 Supplement (100x) | Thermo Fisher Scientific | 17502001 | |
| Neurobasal Medium | Thermo Fisher Scientific | 21103049 | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| PyroMark PCR Kit | Qiagen | 978703 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| SB431542 | StemCell Technologies | 72232 | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| STEMdiff Neural Induction Medium | StemCell Technologies | 5835 | |
| STEMdiff Neural Progenitor Freezing Medium | StemCell Technologies | 5838 | |
| TaqMan Assay FOXA2 | Thermo Fisher Scientific | Hs00232764 | |
| TaqMan Assay GAPDH | Thermo Fisher Scientific | Hs99999905 | |
| TaqMan Assay Nestin | Thermo Fisher Scientific | Hs04187831 | |
| TaqMan Assay OCT4 | Thermo Fisher Scientific | Hs04260367 | |
| TaqMan Assay PPIA | Thermo Fisher Scientific | Hs99999904 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| Y27632 | StemCell Technologies | 72302 | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Goat serum, Sterile, 10 mL | Sigma | G9023 | Working concentration 1:1000 |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Normal mouse serum, Sterile, 500 mL | Equitech-Bio | SM30-0500 | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| pMD2.G | Addgene | 12253 | |
| pRSV-Rev | Addgene | 52961 | |
| psPAX2 | Addgene | 12259 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsmBI | New England Biolabs | R0580S | |
| BsrGI | New England Biolabs | R0575S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
参考文献
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Gaj, T., Gersbach, C. A., Barbas, C. F. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology. 31 (7), 397-405 (2013).
- Thakore, P. I., Black, J. B., Hilton, I. B., Gersbach, C. A. Editing the epigenome: technologies for programmable transcription and epigenetic modulation. Nature Methods. 13 (2), 127-137 (2016).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Gilbert, L. A., et al. Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. Cell. 159 (3), 647-661 (2014).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Chavez, A., et al. Highly efficient Cas9-mediated transcriptional programming. Nature Methods. 12 (4), 326-328 (2015).
- Horlbeck, M. A., et al. Nucleosomes impede Cas9 access to DNA in vivo and in vitro. eLife. 5, (2016).
- Chavez, A., et al. Comparison of Cas9 activators in multiple species. Nature Methods. 13 (7), 563-567 (2016).
- Zhou, H., et al. In vivo simultaneous transcriptional activation of multiple genes in the brain using CRISPR-dCas9-activator transgenic mice. Nature Neuroscience. 21 (3), 440-446 (2018).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell. 159 (3), 635-646 (2014).
- Konermann, S., et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517 (7536), 583-588 (2015).
- Holtzman, L., Gersbach, C. A. Editing the Epigenome: Reshaping the Genomic Landscape. Annual Review of Genomics and Human Genetics. , (2018).
- Perez-Pinera, P., et al. Synergistic and tunable human gene activation by combinations of synthetic transcription factors. Nature Methods. 10 (3), 239-242 (2013).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nature Methods. 12 (12), 1143-1149 (2015).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- McDonald, J. I., et al. Reprogrammable CRISPR/Cas9-based system for inducing site-specific DNA methylation. Biology Open. 5 (6), 866-874 (2016).
- Huang, Y. H., et al. DNA epigenome editing using CRISPR-Cas SunTag-directed DNMT3A. Genome Biology. 18 (1), 176 (2017).
- Vojta, A., et al. Repurposing the CRISPR-Cas9 system for targeted DNA methylation. Nucleic Acids Research. 44 (12), 5615-5628 (2016).
- Razin, A., Kantor, B. DNA methylation in epigenetic control of gene expression. Progress in Molecular and Subcellular Biology. 38, 151-167 (2005).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18786-18791 (2009).
- Kantor, B., et al. Downregulation of SNCA Expression by Targeted Editing of DNA Methylation: A Potential Strategy for Precision Therapy in PD. Molecular Therapy. , (2018).
- Jowaed, A., Schmitt, I., Kaut, O., Wullner, U. Methylation regulates alpha-synuclein expression and is decreased in Parkinson's disease patients' brains. Journal of Neuroscience. 30 (18), 6355-6359 (2010).
- Wang, Y., et al. A DNA methyltransferase inhibitor, 5-aza-2'-deoxycytidine, exacerbates neurotoxicity and upregulates Parkinson's disease-related genes in dopaminergic neurons. CNS Neuroscience & Therapeutics. 19 (3), 183-190 (2013).
- Matsumoto, L., et al. CpG demethylation enhances alpha-synuclein expression and affects the pathogenesis of Parkinson's disease. PLOS One. 5 (11), e15522 (2010).
- Desplats, P., et al. Alpha-synuclein sequesters Dnmt1 from the nucleus: a novel mechanism for epigenetic alterations in Lewy body diseases. Journal of Biological Chemistry. 286 (11), 9031-9037 (2011).
- Ai, S. X., et al. Hypomethylation of SNCA in blood of patients with sporadic Parkinson's disease. Journal of the Neurological Sciences. 337 (1-2), 123-128 (2014).
- Tagliafierro, L., Chiba-Falek, O. Up-regulation of SNCA gene expression: implications to synucleinopathies. Neurogenetics. 17 (3), 145-157 (2016).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Molecular Therapy - Methods & Clinical Development. 5, 153-164 (2017).
- Tagliafierro, L., et al. Genetic analysis of alpha-synuclein 3' untranslated region and its corresponding microRNAs in relation to Parkinson's disease compared to dementia with Lewy bodies. Alzheimer’s & Dementia. 13 (11), 1237-1250 (2017).
- Tagliafierro, L., Zamora, M. E., Chiba-Falek, O. Multiplication of the SNCA locus exacerbates neuronal nuclear aging. Human Molecular Genetics. , (2018).
- Kantor, B., McCown, T., Leone, P., Gray, S. J. Clinical applications involving CNS gene transfer. Advances in Genetics. 87, 71-124 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Advances in Genetics. 87, 125-197 (2014).
- Vijayraghavan, S., Kantor, B. A Protocol for the Production of Integrase-deficient Lentiviral Vectors for CRISPR/Cas9-mediated Gene Knockout in Dividing Cells. Journal of Visualized Experiments. (130), e56915 (2017).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Molecular Therapy. 16 (12), 1968-1976 (2008).
- Bassil, C. F., Huang, Z., Murphy, S. K. Bisulfite pyrosequencing. Methods in Molecular Biology. 1049, 95-107 (2013).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Research. 43 (13), 6450-6458 (2015).
- Nishimasu, H., et al. Crystal Structure of Staphylococcus aureus Cas9. Cell. 162 (5), 1113-1126 (2015).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. Journal of Virology. 68 (4), 2632-2648 (1994).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. Journal of Virology. 71 (8), 6113-6127 (1997).
- Goffin, V., et al. Transcription factor binding sites in the pol gene intragenic regulatory region of HIV-1 are important for virus infectivity. Nucleic Acids Research. 33 (13), 4285-4310 (2005).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. Journal of Biological Chemistry. 280 (22), 21545-21552 (2005).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Molecular Therapy. 19 (3), 547-556 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved