Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Лентивирусные вектор платформа для эффективной доставки Epigenome-инструменты для редактирования в человека индуцированных плюрипотентных стволовых клеток болезни моделей
В этой статье
Резюме
Целевые epigenome ДНК, редактирования представляет собой мощный терапевтический подход. Этот протокол описывает производства, очищения и концентрации все-в-одном лентивирусные векторы, укрывательство трансген ТРИФОСФАТЫ dCas9-DNMT3A для epigenome редактирование приложений в человеческое индуцированных плюрипотентных стволовых клеток (hiPSC)-производных нейронов.
Аннотация
Использование производных hiPSC клеток представляет собой ценный подход к изучению человека нейродегенеративных заболеваний. Здесь мы описываем оптимизированный протокол для дифференциации hiPSCs, производного от пациента с постулатов Локус гена (SNCA) альфа synuclein в болезнь Паркинсона (PD)-отношение дофаминергической нейрональных популяций. Накопление доказательств показывает, что высокий уровень SNCA каузативных для развития PD. Признавая неудовлетворенных необходимо создать новые терапевтические подходы для PD, особенно направленными на регуляцию экспрессии SNCA , мы недавно разработанных ТРИФОСФАТЫ/dCas9-ДНК Метилировани-на базе системы эпигеномно модулировать транскрипции SNCA , обогащая уровень метилирования в регионе нормативных Интрон 1 SNCA . Чтобы доставить система, состоящая из мертвых (отключен) версия Cas9 (dCas9) сливается с каталитического домен 3A фермента ДНК метилтрансфераза (DNMT3A), используется лентивирусные вектор. Эта система применяется к ячейкам с постулатов Локус SNCA и уменьшает SNCA-мРНК и уровни белка, около 30% через целевые метилирования Интрон SNCA 1. Доработаны Даунрегуляция SNCA уровней спасает сотовой фенотипов, связанных с болезнью. В текущем протоколе мы стремимся описать пошаговую процедуру для дифференциации hiPSCs в нейронных прогениторных клеток (НПС) и создание и проверка пиросеквенирования анализов для оценки профиля метилирования в SNCA Интрон 1. Более подробно изложить человека ТРИФОСФАТЫ/dCas9 система, используемая в этих экспериментах, этот протокол описывает, как производить, очищают и сконцентрировать лентивирусные векторы и подчеркнуть их пригодности для epigenome - и -изменения генома приложений, использующих hiPSCs и НПС. Протокол может быть легко адаптирована и могут быть использованы для производства высокого титра lentiviruses для приложений в vitro и in vivo.
Введение
Epigenome редактирования платформ недавно была разработана для любых последовательностей ДНК в регионах, которые контролируют ген выражение1,2. Созданные инструменты редактирования epigenome предназначены для (i) регулируют транскрипцию, (ii) изменять Посттрансляционная гистона модификации, (iii) изменения метилирования и (iv) модулировать регуляторные элементы взаимодействия. Подход к якорь транскрипция/хроматина модификаторы списанного (мертвый) Cas9 (dCas9) поднял из ранее развитых epigenome редактирования платформ, таких как цинк палец белки (ZFPs) и транскрипции активатор как эффекторов (сказки), укрывательство мощным transcriptional эффекторных домен (Эд) сливается с дизайном ДНК связывающий домен (DBD)3. Результаты желаемого фенотип, такие как активация или репрессий определяется эффекторные молекулы, привязанный к эндогенной локусов (рис. 1). Для создания программируемой transcriptional активаторы, dCas9/gRNA модули связаны с VP164,5,6 (рис. 1а), вирусные активации домена, новобранцев Pol II и механизмы общего транскрипции. Модификации этой системы включает VP64, Тетрамер VP16 доменов, обеспечивая еще более надежной активации ставка5,6. Система успешно работает для того чтобы активировать кодирования и некодирующей регионов путем промоутеров и нормативных элементов. Важно отметить, что даже несмотря на то, что VP64 молекулы изменить структуру хроматина в целевом регионе непосредственно, он новобранцев хроматина модификаторы, которые связывают результаты осаждения активный (euchromatin) знаков, включая как H3/H4 ацетилирования и H3-K4 di / Tri метилирования5,6. В дополнение к VP64 субъединица p65 человека NF-κB комплекса были привязаны к dCas9/gRNA модуль7. Интересно привязывая Эти эффекторы в регионы вверх по течению транскрипции начала сайтов (тссс) и в пределах промоутеров приводит к сильным гена индукции. Тем не менее VP64 и p65 эффекторов можно также оказывают activatory эффекты во время связаны с регионов, расположенных ниже по течению, ТССС и дистальной усилители7,8. Для получения более надежной транскрипционный анализ ответа, несколько dCas9-VP64 или dCas9-p65 сплавливания нужно набираться для одной цели Локус9,10. Таким образом недавнее развитие активаторы следующего поколения, которые набору несколько доменов эффекторных одного dCas9-gRNA комплексом, таких как SunTag, привело к сильной активации возможности по сравнению с dCas9-VP64 фьюжн партнерам11 , 12. Улучшение transcriptional активации были получены через слияние VP64, Р65 и Rta (VPR), transactivation домена от гамма Герпесвирусы, C-конечная dCas913 (рис. 1A). Подобные системы ТРИФОСФАТЫ/dCas9 были разработаны для конкретных целевых объектов репрессий (рис. 1B).
Эндогенные гена репрессий с инженерии репрессор сплавливания можно добиться различных механизмов (рис. 1B). Было продемонстрировано, что ТРИФОСФАТЫ/dCas9 систем, связанных с репрессор DBD (даже без домена эффекторных/s), можно эффективно замолчать экспрессии генов в то время как привязанный к промоутер или вверх/вниз по течению TSS регионы3,6 ,14. Влияние на транскрипции обусловлено их пространственной помех привязки фактор транскрипции и обработки РНК-полимеразы. Тем не менее необходимы более всеобъемлющие подходы, как ген репрессии их пространственной помех только часто не достаточно для надежной глушителей. Последние разработки следующего поколения глушители на основе ТРИФОСФАТЫ/dCas9 систем перевозящих transcriptional репрессор домены (TRDs), модификаторы гистонов (H3-K9 ди-/ tri метилирования, H3-K27 ди-/ tri метилирования; H3-K36 ди-/ tri метилирования, H3/H4 диацетилморфина) и привело к строительству эпигеномные инструментов, позволяя более надежной, глушителей эффекты4,5,,1516, метилирование ДНК (CpG) 17,18,19,20. Было продемонстрировано, что набор эти эпигеномные модификаторов для ДНК может привести к формированию более закрытой и конденсированных хроматина, который обычно генерируют более мощным глушителей исход21,22. Наиболее часто глушителей домен, используется с DBDs — это связанные Krüppel box (краб)4,5. Набор фактора была продемонстрирована в соответствие с изменениями chromatin; Тем не менее механизмы этих изменений еще не должны быть проясненного16,17,18. Недавно было показано, что локализация краб ДНК может содействовать Ассамблея метилтрансфераза гистона SETDB1 и гистонов диацетилморфина (HDAC) NuRD комплексов, предлагая возможность того, что эти взаимодействия посредником формирования конденсацию хроматина и transcriptional глушителей3,13. В качестве альтернативного подхода эффекторные домены можно сливается с DBDs для создания пользовательских эпигеномные глушителей белка. Эта система непосредственно катализирует репрессивных ДНК знаки или изменения гистона.
Недавно использование синтетических ТРИФОСФАТЫ/dCas9 систем, привязанный к DNMT3A фермента был многоцелевых transcriptional дезактивации. DNMT3A катализирует метилирование ДНК, которая оказывает транскрипционный анализ репрессий на протяжении формирования гетерохроматин эндогенного генным стимуляторам и других регулирующих регионов (рис. 1B)18,20. McDonald et al.18 и войта et al.20 были первые авторы сообщить что метилирование ДНК может использоваться для epigenome-генов или репрессий, демонстрируя, что доставлено плазмида dCas9-DNMT3A системы фьюжн мощно может повысить метилирование цитозина вокруг18,ТП20. Макдональд и coworkers продемонстрировал, что занятости стратегия может привести к значительному сокращению (около 40%) в опухоль супрессирующем Гене CDKN2A мРНК уровнях18. Аналогичным образом ориентация региона unmethylated промоутера генов, Бах или IL6ST показывает увеличение метилирования CpG, что был коррелирует с сокращением вдвое выражение гена20. Наша лаборатория недавно многоцелевых использование метилирование ДНК для ослабления патологических результатов SNCA гиперэкспрессия (рис. 2)23. Стратегия основана на избирательное повышение метилирование ДНК в регионе Интрон 1 SNCA , как ранее сообщалось, чтобы быть hypomethylated в PD и деменции с Леви органов (ДЛБ) мозги24,25, 26. Этот hypomethylation связан с SNCA гиперэкспрессия, таким образом предлагая привлекательной мишенью для терапевтического вмешательства24,27,28. Недавно мы показали низкий уровень метилирования ДНК в SNCA Интрон 1 региона в hiPSC производные дофаминергической НИПы, полученную от PD пациента с SNCA постулатов23. Преимуществом этой экспериментальной модели является что NPC может быть надежно распространен в культуре или далее дифференцированной в зрелых нейронов, позволяя эффективный скрининг для выявления генетических факторов, которые посредником сотовой фенотипов, включая окислительного стресс и апоптоз29. Кроме того эта модель система позволяет ученым охарактеризовать развития событий, которые произошли до появления симптомов у больных. Кроме того hiPSC производные НИПы представляют собой прекрасный инструмент для тестирования клеточном и молекулярном пути, связанные с выражением генов. Важно отметить, что hiPSC производные НПС, в сочетании с технологией ТРИФОСФАТЫ/Cas9-epigenome искусство может значительно облегчить развитие «next-generation наркотиков» для многих нейродегенеративных заболеваний.
Чтобы уменьшить патологического уровня SNCA выражение, мы недавно разработали системы, основанной на человека, dCas9-DNMT3A синтез белка и gRNA специально целевой метилирования CpG внутри SNCA Интрон 1 (рисунок 2A)23. Этот протокол будет описывать лентивирусные вектор (LV) проектирование и производство в деталях. LVs являются эффективным средством доставки компонентов ТРИФОСФАТЫ/dCas9 по нескольким причинам, а именно: (i) их способности выполнять громоздкие ДНК вставляет, (ii) высокой эффективности преобразователя широкий спектр клеток, включая клетки деления и nondividing30 и (iii) их способность вызывать минимальное цитотоксических и иммуногенные ответы. Недавно мы обратились LV системы hiPSC производные дофаминергические нейроны от пациента с постулатов Локус SNCA и продемонстрировал терапевтический потенциал LVs для доставки epigenome редактирования метилирования инструменты23 ( Рисунок 2B). Действительно LV-gRNA/dCas9-DNMT3A системы вызывает значительное увеличение метилирование ДНК в регионе Интрон 1 SNCA . Этот рост соответствует снижением уровня мРНК и белка SNCA 23. Кроме того SNCA Даунрегуляция спасает PD-связанных фенотипы в SNCA постулатов/hiPSC производные дофаминергических нейронов (например, митохондриальной ROS производства и клеток жизнеспособности)23. Важно отметить, что мы показали, что сокращение в выражении SNCA LV-gRNA-dCas9-DMNT3A системы способны обратить вспять фенотипов, которые характерны для hiPSC производные дофаминергические нейроны от PD пациента, который нес SNCA постулатов, например митохондриальной ROS производства и клеток жизнеспособности23. Целью настоящего Протокола является 1) чтобы наметить протокол производства и концентрации LV платформу для создания высокой tittered вирусных препаратов и 2) описывают дифференцирование hiPSCs в NPC, рисунком стать зрелой дофаминергической нейроны31,32 и характеристика уровень метилирования целевого региона в рамках SNCA Интрон 1.
Лентивирусные платформы имеют значительное преимущество над самой популярной платформы вектор, а именно аденоассоциированный векторов (AAVs), который является бывший возможность разместить большие генетические вставки33,34. AAVs могут быть собраны в значительно более высокие урожаи, но обладают низким упаковки емкостью (< 4.8 КБ), ущерба для их использования для доставки все-в-одном ТРИФОСФАТЫ/Cas9 систем. Таким образом оно кажется, что рН будет выбор платформы приложений, участвующих в доставке ТРИФОСФАТЫ/dCas9 инструментов. Таким образом протокол, изложенные здесь будет ценным инструментом для исследователей, желающих эффективно передавать epigenome редактирования компонентов клеток и органов. Протокол также излагается стратегия для увеличения производства и выражения возможности векторов через изменения в СНГ элементов в вектор выражения кассеты30,35. Стратегия основана на романе система разработана и учился в нашей лаборатории и подчеркивается его способность производить вирусных частиц в диапазоне 1010 вирусных единиц (VU) / мл30,35.
Access restricted. Please log in or start a trial to view this content.
протокол
1. системы проектирования и производства вирус
- Плазмиды проектирование и строительство
Примечание: Строительство вектор LV-gRNA-dCas9-DNMT3A все-в-одном осуществляется с помощью производства- и оптимизированный выражение выражение кассеты, опубликованные Ортинский et al.30. Вектор кассеты носит повторение признание сайта транскрипционный фактор Sp1 и удаления состояние искусства в непереведенные (U3') региона терминал 3'-Лонг повторить30,(LTR) (рисунок 2A)36. Было установлено, что вектор позвоночника эффективно доставлять и выражая ТРИФОСФАТЫ/Cas930,35.- Получить версию деактивировать (мертвых) SpCas9 (dCas9) через сайт Направленный мутагенез (данные не показаны). Замените клон, укрывательство D10A и H840A мутации в доменах каталитического HNH и RuvC фермента, соответственно, с активной Cas9 в pBK30129 путем обмена между фрагментами АИОД-BamHI (рис. 3).
- Получить домен каталитического DNMT3A от pdCas9-DNMT3A-eGFP (см. Таблицу материалы) путем усиления DNMT3A часть BamHI-429/R 5 '- GAGCGGATCCCCCTCCCG - 3' BamHI-429/Л 5' - CTCTCCACTGCCGGATCCGG - 3' (рис. 3). Для того чтобы усилить региона, содержащие DNMT3A, используйте следующие условия: (1) 95 ° C 60 s, (2) 95 ° C 10 s, (3) 60 ° C для 20 s, (4) 68 ° C для 60 s. повторить условия 2-4 30 x. Для окончательного расширения используйте 68 ° C 3 мин и удерживайте 4 ° C.
- Клон DNMT3A фрагмент, усваивается энзима ограничения BamHI, на BamHI сайт модифицированных pBK301 вектора, перевозящих dCas9. Проверьте клонирования прямой Сэнгером последовательности. Обратите внимание, что привело плазмида затаивает трансген dCas9-DNMT3A-p2a-puromycin. Плазмида выражает gRNA лески от человека U6 промоутер (рис. 3).
- Замените puromycin Репортер ген Зеленый флуоресцентный белок (ГПУП) для создания dCas9-DNMT3A-p2a-GFP. Дайджест dCas9-DNMT3A-p2a-Puro плазмида с ФГОУ. Очищайте фрагмента вектор, с помощью метода очистки гель. Подготовьте insert, переваривание pBK201a (pLenti-GFP) с ФГОУ. Клонирование фрагмента ФГОУ в вектор. Результате плазмида pBK539 гавани dCas9-DNMT3A-p2a-GFP трансген (рис. 3).
- Культивирование клеток ГЭС 293T и покрытие клетки для transfection
Примечание: Человеческих эмбриональных почки 293T (ГЭС 293T) клетки культивировали в полной высокой глюкозы Дульбекко модифицированная орла среднего (DMEM; 10% говядину телячьей сыворотки, 1 x антибиотик противогрибковое, 1 x пируват натрия, 1 x заменимая аминокислота, 2 мм L-глютамин) при 37 ° C с 5% CO 2. для воспроизводимости протокола, рекомендуется проверить телячьей сыворотки при переключении на другой много/партии. До шести 15 см пластины необходимы для лентивирусные производства.- Используйте низкий проход клетки для начала новой культуры (ниже, чем проезд 20). После того, как клетки достигают 90%-95% слияния, аспирационная СМИ и осторожно промыть стерильной 1 x фосфат амортизированное saline (PBS).
- Добавить 2 мл трипсина-ЭДТА (0,05%) и проинкубируйте его при 37 ° C для 3-5 мин. Для инактивации диссоциации реагента, добавьте 8 мл полной DMEM высокой глюкозы и x-15 пипетки 10 x с 10 мл Серологические Пипетки для создания одной ячейки подвеска 4 х 106 клеток/мл.
- Для transfections пальто 15 см пластины с 0,2% желатина. Добавьте 22,5 мл среды высокого глюкозы и семян клетки путем добавления 2,5 мл суспензии клеток (всего ~ 1 x 107 клеток/плита). Инкубируйте пластины при 37 ° C с 5% CO2 , вплоть до 70%-80% слияния.
- Transfection клетки ГЭС 293T
- Готовить 2 x BES-амортизированное решение BBS и CaCl 1 M2, по данным Vijayraghavan и Кантор35. Фильтр решения, передавая их через фильтр 0,22 мкм и хранить их на 4 ° C. Трансфекция смеси должно быть ясно перед его добавлением к клеткам. Если смесь становится мутным во время инкубации, подготовьте свежие 2 x BBS (рН = 6.95).
- Подготовить смесь плазмида, использовать четыре плазмид, как указано (следующая смесь является достаточным для одной плиты 15 см): 37,5 мкг ТРИФОСФАТЫ/dCas9-передачи вектора (pBK492 [DNMT3A-Puro-NO-gRNA] или pBK539 [DNMT3A-GFP-NO-gRNA]); 25 мкг pBK240 (psPAX2); 12,5 мкг pMD2.G; 6.25 мкг pRSV-rev (рис. 4A). Рассчитать объем плазмид, основанный на концентрации и добавьте необходимые количества в коническую пробирку 15 мл. 312.5 мкл CaCl 1 М2 и принести окончательный объем до 1,25 мл, используя стерильные dd-H2O. осторожно добавить 1,25 мл раствора 2 x BBS во время vortexing смеси. Инкубируйте 30 мин при комнатной температуре. Клетки готовы к трансфекции после того, как они являются 70%-80% притока.
- Аспирационная СМИ и заменить его с 22,5 мл свежеприготовленные высокой глюкозы DMEM без сыворотки. Добавьте 2.5 мл смеси трансфекции каплям на каждой пластине 15 см. Вихрем пластины и инкубировать при 37 ° C с 5% CO2 на 2-3 ч.
- После 3 h добавьте 2,5 мл (10%) сыворотки на пластину и инкубировать на ночь при 37 ° C с 5% CO2.
- На 1 день после трансфекции, наблюдать клетки, чтобы убедиться, что нет или минимальным клеточную смерть и что клетки образуется вырожденная культуры (100%).
- Измените СМИ, добавив 25 мл свежеприготовленные DMEM и 10% сыворотки высокой глюкозы в каждой плиты.
- Инкубируйте при 37 ° C с 5% CO2 в течение 48 часов.
- Урожай вируса
- Собирать супернатант от всех transfected клеток и объединить их в 50 мл конические трубы. Центрифуга в 400 – 450 x g 10 мин фильтр супернатант через блок вакуумного фильтра 0.45 мкм. После фильтрации супернатанта может храниться при температуре 4 ° C для кратковременного хранения (до 4 дней). Для длительного хранения подготовить аликвоты и хранить их в-80 ° C.
Примечание: Предполагается, что nonconcentrated вирусных препаратов являются ~ 2 x 107 до 3 x 107 vu/мл (обратитесь к разделу 1.5 для определения титра антител). Настоятельно рекомендуется подготовить аликвоты одноразового использования, поскольку несколько циклов замораживания оттаивания приведет к потере 10% – 20% в функциональных титры.
- Собирать супернатант от всех transfected клеток и объединить их в 50 мл конические трубы. Центрифуга в 400 – 450 x g 10 мин фильтр супернатант через блок вакуумного фильтра 0.45 мкм. После фильтрации супернатанта может храниться при температуре 4 ° C для кратковременного хранения (до 4 дней). Для длительного хранения подготовить аликвоты и хранить их в-80 ° C.
- Концентрация вирусных частиц
Примечание: Для очистки двухэтапный метод двойной сахароза, с участием шага градиентного сахарозы и сахароза подушки шаг выполняется (рис. 4В).- Чтобы создать градиент, сахароза, подготовить трубы конической ultracentrifugation в следующем порядке: 0,5 мл 70%-ая сахароза в однократном ПБС, 0,5 мл 60% сахарозы в среде DMEM, 1 мл 30% сахарозы в среде DMEM и 2 мл 20% сахарозы в однократном ПБС.
- Осторожно добавьте супернатанта, собранных согласно статье 1.4, градиента. Так как общий объем собранных из четырех 15 см пластины составляет 100 мл, используйте шесть ultracentrifugation трубы для обработки вирусный супернатант.
- Равномерное распределение вирусный супернатант среди каждой трубы ultracentrifugation. Чтобы избежать поломки труб в процессе центрифугирования, заполните ultracentrifugation трубы для по меньшей мере три четверти их общего объема емкости. Баланс трубки с ПБС. Центрифугуйте образцы на 70000 x g на 2 ч в 17 ° C.
Примечание: Поддерживать уровень сахарозы во время ускорения и замедления шаги, позволяют ультрацентрифуга для медленно ускорения и замедления ротора от 0 до 200 x g и от 200 до 0 x g во время первого и последнего 3 мин спина, соответственно. - Аккуратно Соберите 30% – 60% сахарозы фракций в чистой трубы (рис. 4В). Добавьте 1 x PBS (холодной) вверх к 100 мл общего объема. Смешайте закупорить несколько раз.
- Осторожно слоиться вирусной подготовки на подушке сахарозы, добавив 4 мл 20%-ая сахароза (в ПБС) на трубу. Продолжить, дозирования ~ 20-25 мл вирусный раствора в каждую пробирку. Заполните трубы с ПБС, если объем их содержание составляет менее чем три четверти в трубку. Тщательно сбалансировать трубы. Центрифуга на 70000 x g на 2 ч в 17 ° C. Инвертировать трубы на бумажные полотенца для разрешить оставшиеся и пустые супернатант жидкости стечь.
- Удалите все жидкости, осторожно аспирационных оставшуюся жидкость. Обратите внимание, что на данном этапе, едва видна, как небольшие пятна полупрозрачные гранулы, содержащие вирус. 70 мкл ПБС, к первой трубки Ресуспензируйте гранулы. Тщательно Пипетка подвеска и перенести его на следующий трубку до тех пор, пока все гранулы высокомобильна.
- Промойте трубы с дополнительной 50 мкл 1 x PBS и смесь как раньше. Обратите внимание, что на данном этапе объем окончательное приостановление ~ 120 мкл и появляется немного Млечный. Для получения четких подвеска, перейти с 60 центрифугирования s на 10000 x g. Супернатант передать новой трубки, сделать 5 мкл аликвоты и хранить их при температуре-80 ° C.
Примечание: Лентивирусные вектор препараты чувствительны к повторных циклов замораживания и оттаивания. Кроме того предполагается, что оставшиеся шаги в культуре ткани сдерживания или в специально отведенных местах, квалифицированных в том смысле, на адекватном уровне стандартов биобезопасности (Рисунок 4B).
- Количественная оценка вирусной титры
Примечание: Оценка вирусной титры выполняется с помощью метода p24 иммуноферментного анализа (ИФА) (p24gag ELISA) и согласно программе вакцины против СПИДа национальных институтов здравоохранения (НИЗ) протокол Assay захвата антиген p24 ВИЧ-1 с незначительные изменения.- Используйте 200 мкл 0,05% анимации 20 в холодной ПБС (PBS-T) для мытья скважин 96-луночных пластины 3 x.
- Чтобы покрыть пластину, используйте 100 мкл антител моноклонального анти p24 разбавляют в 1:1,500 в однократном ПБС. Инкубировать пластину на ночь при 4 ° C.
- Подготовить блокировки реагента (1% альбумина bovine сыворотки [BSA] в однократном ПБС) и 200 мкл метки для каждой скважины, чтобы избежать неспецифического связывания. Используйте 200 мкл PBS-T мыть хорошо 3 x для по крайней мере 1 час при комнатной температуре.
- Приступайте к подготовке образца. При работе с концентрированной вектор препараты, разбавляют вектор на масштаба 1: 100, используя 1 мкл пример, 89 мкл dd-H20 и 10 мкл X-100 Тритон (в конечной концентрации 10%). Для nonconcentrated подготовки развести образцы в 1:10.
- Получите стандартам ВИЧ-1, используя двойную серийный разрежения (Начальная концентрация составляет 5 нг/мл).
- Разбавьте концентрированный образцы (подготовленных на шаге 1.6.4) в RPMI 1640 дополнены 20 анимации и 1% BSA 0,2% для получения 1:10 000, 1:50 000 и растительного разведениях. Аналогичным образом, разбавить nonconcentrated образцы (подготовленных на шаге 1.6.4) в RPMI 1640 с 0,2% 20 анимации и 1% BSA установить 1: 500, 1:2, 500 и 1:12,500 растворов.
- Добавление образцов и стандартов на плите в triplicates. Инкубируйте на ночь при 4 ° C.
- Следующий день, промыть скважин 6 x.
- 100 мкл кролика polyclonal антитела анти p24, разбавляют в 1:1,000 в RPMI 1640, 10% плода бычьим сывороточным (ФБС), BSA 0,25% и 2% нормальной мыши сыворотки (NMS) и инкубировать при 37 ° C в течение 4 ч.
- Промыть лунки 6 x. Добавить козье анти кролик пероксидаза IgG, разбавляют в мэм в RPMI 1640 с 5% нормальной козьего сыворотки, 2% NMS, 0,25% BSA и 0,01% 20 анимации. Инкубируйте при 37 ° C в течение 1 ч.
- Промыть лунки 6 x. Добавление ТМБ пероксидазы субстрата и инкубации при комнатной температуре в течение 15 мин.
- Чтобы остановить реакции, добавьте 100 мкл 1 N HCl. В Считыватель микропланшетов измерять поглощение на 450 Нм.
- Измерение интенсивности флуоресцентные репортер
- Используйте вирусный подвеска для получения десятикратного серийный разрежения (от 10-1 -10-5) в однократном ПБС.
- Пластина 5 x 105 ГЭС 293T клеток в каждой скважине 6-ну плиты. Применить к ячейкам 10 мкл каждого вирусный разрежения и инкубировать при 37 ° C с 5% CO2 в течение 48 часов.
- Перейдите к активации флуоресценции клеток, сортировка (FACS) анализ следующим: отсоединение клетки путем добавления 200 мкл 0,05% раствор трипсина ЭДТА. Инкубировать клетки при 37 ° C за 5 мин и Ресуспензируйте их в 2 мл DMEM среднего (с сывороткой). Сбор образцов в 15 мл Конические трубки и центрифуги на 400 x g при 4 ° C. Ресуспензируйте гранулы в 500 мкл холодной ПБС.
- Исправьте клетки путем добавления 500 мкл параформальдегида 4% (PFA) и проинкубируйте втечение 10 мин при комнатной температуре.
- Центрифуга на 400 x g при 4 ° C и Ресуспензируйте гранулы в 1 мл ПБС. Анализируйте экспрессия гена GFP с помощью инструмента СУИМ, как описано в Ортинский et al.30. Чтобы определить функциональные титр вируса, используйте следующую формулу.

Здесь,
TG = количество GFP-положительных клеток;
TN = общее количество клеток;
N = общее количество transduced клеток;
V = объем, используемый для трансдукции (в микролитров).
- Подсчет GFP-положительных клеток
Примечание: Определите кратность инфекции (МВД), который используется для трансдукции. Тестировать широкий диапазон MOIs (от МВД = 1 мои = 10).- 5 семян 3 x 10-4 x 105 ГЭС 293T клеток на каждой скважине 6-ну плиты.
- Когда клетки достичь > 80% слияния, передают их с вектором на мой интерес.
- Инкубировать при 37 ° C с 5% CO2и контролировать GFP сигнала в клетках на 1 – 7 дней.
- Подсчитать количество GFP-положительных клеток. Использовать флуоресцентный микроскоп (план 4 x цель, 0.1 н.а., 40 кратном) с использованием GFP фильтра (длина волны возбуждения = 470 Нм, длина волны излучения = 525 Нм). Используйте untransduced клетки для задания управления популяция клеток GFP-отрицательные.
- Используют следующую формулу для определения функциональных титр вируса.
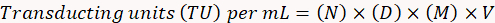
Здесь,
N = количество GFP-положительных клеток;
D = коэффициент разрежения;
M = коэффициент увеличения;
V = объем вирус используется для трансдукции.
Примечание: Вычислить результаты этого примера: за 10 GFP-положительных клеток (N) учитываются в разведении 10-4 (1:10, 000) при 20-кратном (M) в 10 мкл пример (V) (D), ту на миллилитр будет (10 х 104) x (20) x (10) х (100) = 2 x 108 vu/мл.
2. дифференциация дофаминергической нейронных прогениторных клеток
- Выращивание hiPSCs
Примечание: hiPSCs от пациента с постулатов Локус SNCA, ND34391, были получены от Национальный институт неврологических нарушений и инсульта (NINDS) каталог (см. таблицу материалы).- Культура hiPSCs в условиях фидер независимые в свободной подачи ESC-iPSC питательной среды (см. Таблицу материалы) на квалифицированных Госкомсанэпиднадзором базовой матрицы мембраны (BMM)-покрытием пластин (см. Таблицу материалы). Мыть вырожденная колоний с 1 мл раствора DMEM/F12, добавьте 1 mL реагента диссоциации (см. Таблицу материалы) и Инкубируйте 3 мин при комнатной температуре.
- Аспирационная диссоциации реагента и добавьте 1 mL фидер бесплатно ESC-iPSC питательной среды.
- Скрип пластину, используя погрузчик клетки и Ресуспензируйте колоний в 11 мл свободной подачи ESC-iPSC питательной среды, дозирования 4 x-5 x с использованием боросиликатного пипетки.
- Пластина 2 мл суспензии колонии на BMM-покрытием пластин и поместите пластины при 37 ° C с 5% CO2. Выполнять ежедневные изменения средних и разбить ячейки каждые 5 – 7 дней.
- Дифференциация в дофаминергической нейронных прогениторных клеток
Примечание: Дифференциация hiPSCs в дофаминергической нейронных прародитель клетки (MD NPC) осуществляется с помощью коммерчески доступных нейронной индукции средних протокола в соответствии с инструкциями производителей, с незначительными изменениями (32 )31, см Таблицу материалы). В первый день дифференциация рассматривается как день 0. Высокое качество hiPSCs, необходимые для эффективного нейронных дифференциации. Индукции MD NPC – performedusing embryoid тело (EB)-на основе протокола.- Перед началом дифференциация hiPSCs, подготовить микрорезервуар культуры плиты (см. Таблицу материалы) согласно инструкциям ее производителя.
- После подготовки пластину микрорезервуар культуры, добавьте 1 mL нейронной индукции среды (NIM; см. Таблицу материалы) с 10 мкм Y-27632.
- Отложите пластины до готовой к использованию.
- Мыть hiPSCs с DMEM/F12, добавить 1 мл раствора отряд клеток (см. Таблицу материалы) и инкубировать в течение 5 минут при 37 ° C с 5% CO2.
- Ресуспензируйте единичных клеток в среде DMEM/F12 и центрифуги их на 300 x g за 5 мин.
- Тщательно удалить супернатант и Ресуспензируйте клетки в NIM + 10 мкм Y-27632 для получения конечной концентрации 3 х 106 клеток/мл.
- Добавить 1 мл суспензии одноклеточных одного хорошо плиты культуры микрорезервуар и центрифуги пластину на 100 g x 3 мин.
- Изучить пластину под микроскопом, чтобы обеспечить равномерное распределение ячеек среди микрорезервуар и инкубации клеток при 37 ° C с 5% CO2.
- На дней 1 – 4выполните ежедневное частичное изменение средних.
- 1 мл микропипеткой, удалите 1,5 мл среды и выбросите. Медленно добавьте 1,5 мл свежего NIM без Y-27632.
- Повторите шаг 2.2.10 до дня 4.
- На 5 деньпальто одной скважиной 6-ну плиты с БММ.
- Место стрейнер реверсивные 37 мкм (см. Таблицу материалы) на вершине 50 мл Конические трубки (отходы). Точка стрелку реверсивные фильтр вверх.
- Удалите носитель с микрорезервуар культуры пластины не нарушая сформированные EBs.
- Добавьте 1 mL DMEM/F12 и оперативно собирать EBs с пипеткой боросиликатное и фильтровать их через сито.
- Повторите шаг 2.2.15 до тех пор, пока все EBs удаляются из пластины микрорезервуар культуры.
- Инвертировать фильтр над новой 50 мл Конические трубки и добавить 2 мл NIM собрать все EBs.
- Пластина 2 мл суспензии EB в одну скважину BMM-покрытием плиты с помощью пипетки боросиликатное. Инкубируйте EBs при 37 ° C с 5% CO2.
- На 6 день, подготовить 2 мл NIM + 200 нг/мл Тсс (см. Таблицу материалы) и выполнять ежедневные средние изменения.
- На 8 деньпроверьте процент нейронной индукции.
- Подсчет всех присоединенных EBs и, в частности, определить количество каждого индивидуального EB, которая заполняется с нейронных розеток. Количественно розетка нейронной индукции, по следующей формуле.
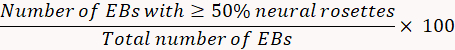
Примечание: Если нейронной индукции < 75%, выбор нейронных розетка может быть неэффективным. - День 12Подготовьте 250 мл N2B27 среды с 119 мл neurobasal средний, 119 мл в среде DMEM/F12, 2,5 мл GlutaMAX, 2,5 мл NEAA, 2,5 мл N2 дополнения, 5 мл B27 без витамина A, 250 мкл гентамицина (50 мг/мл) и 19. 66 мкл BSA (7 мг/мл).
- Для приготовления 50 мл полного среднего N2B27, добавить 3 мкм CHIR99021, 2 мкм SB431542, bFGF 20 нг/мл, 20 нг/мл EGF и 200 нг/мл ТСС.
Примечание: Важно подготовить заполненные средних прямо перед использованием. - Аспирационная среднего из скважин, содержащий нейронных розеток и мыть с 1 мл раствора DMEM/F12.
- Объявление 1 mL реагента нейронных розетка отбора (см. Таблицу материалы) и инкубировать при 37 ° C с 5% CO2 на 1 ч.
- Удалить выбор реагента и, используя 1 мл дозаторов, направлены непосредственно на кластерах розетка.
- Добавьте подвеска 15 мл Конические трубки и повторить шаги 2.2.25 и 2.2.26 до большинства кластеров нейронных розетки были собраны.
Примечание: Чтобы избежать загрязнения с nonneuronal клеток, не overselect. - Центрифуга розетка подвеска на 350 x g 5 мин аспирата супернатант и Ресуспензируйте нейронных розеток в N2B27 + 200 нг/мл ТСС. Добавьте нейронных розетка подвеска хорошо BMM-покрытием и инкубировать пластины при 37 ° C с 5% CO2.
- На дней 13 – 17выполните ежедневных средних изменений с использованием завершенных среднего N2B27. Проход клетки культуры при 80%-90% притока.
- Чтобы разбить ячейки, подготовьте BMM-покрытием плиты.
- Вымыть клетки с 1 мл раствора DMEM/F12, аспирационная среды и добавьте 1 mL реагента диссоциации (см. Таблицу материалы).
- Инкубировать в течение 5 минут при 37 ° C, добавляют 1 мл DMEM/F12 и выбить прилагаемый клетки, закупорить вверх и вниз. Соберите подвеска NPC в 15 мл Конические трубки. Центрифуга на 300 x g за 5 мин.
- Аспирационная супернатант и Ресуспензируйте клеток в 1 мл полного N2B27 + 200 нг/мл ТСС.
- Подсчитать количество ячеек и их пластины на плотности 1,25 x 105 клеток/см2, инкубации клеток при 37 ° C с 5% CO2.
- Изменение среднего каждый день, используя полный N2B27 + 200 нг/мл ТСС.
Примечание: В этом отрывке, НИПы, считаются проход (P) 0. Тсс могут быть выведены из N2B27 среды в точке P2. - Проход клетки, как только они достигают 80%-90% слияния.
- На данном этапе убедитесь, что клетки Экспресс маркеры Nestin и FoxA2 с помощью иммуноцитохимии и ПЦР. Этот протокол приводит к генерации 85% двойной положительных клеток на маркеры Nestin и FoxA2.
- Для пассированый клетки, повторите шаги 2.2.31–2.2.36.
- Заблокировать ячейки, начиная от прохода P2. Для замораживания клеток, повторите шаги 2.2.31–2.2.36 и Ресуспензируйте Пелле клеток в6 2 x 10-4 х 106 клеток/мл с помощью холодной нейронных прародитель замораживание среднего (см. Таблицу материалы).
- Перевести 1 мл суспензии клеток в каждой cryovial и заморозить клетки, с помощью стандартного замедлить скорость контролируемые системой охлаждения. Для длительного хранения держите клетки в жидком азоте.
- Размораживание MD РНУ
- Подготовить тарелку BMM-покрытием и теплое полное N2B27. Добавьте 10 мл теплой среде DMEM/F12 15 мл Конические трубки. Место cryovial в блоке 37 ° C тепла на 2 мин.
- Передать клетки от cryovial трубка, содержащая DMEM/F12. Центрифуга на 300 x g за 5 мин.
- Аспирационная супернатанта, Ресуспензируйте клетки в 2 мл N2B27 и добавьте суспензию клеток одной скважиной BMM-покрытием плиты. Инкубируйте клетки при 37 ° C с 5% CO2.
- Подготовить тарелку BMM-покрытием и теплое полное N2B27. Добавьте 10 мл теплой среде DMEM/F12 15 мл Конические трубки. Место cryovial в блоке 37 ° C тепла на 2 мин.
3. трансдукции MD НПС и анализ изменений метилирования дна
-
Трансдукция MD РНУ
- Передают MD НИПы в 70% слияния с LV-gRNA/dCas9-DNMT3A векторов в MOI = 2. Замените posttransduction средних 16 h N2B27.
- Добавьте N2B27 с 5 мкг/мл puromycin, 48 ч после трансдукции. Культура клетки на 3 недели в N2B27 плюс puromycin для получения стабильной линии MD NPC. Клетки будут готовы к течению приложений (ДНК, РНК, белков анализы, фенотипические характеристики23, замораживания и пассированый).
-
Характеристика метилирования профиля SNCA Интрон 1
- Извлечь ДНК из каждой стабильно transduced ячейки строки с помощью экстракции ДНК комплекта (см. Таблицу материалы).
- Использование 800 нг ДНК, чтобы выполнить преобразование бисульфита, использование коммерчески доступных комплект (см. Таблицу материалы). После преобразование бисульфита элюировать ДНК бисульфит преобразованы к 20 нг/мкл.
-
ПЦР для анализа пиросеквенирования
- Подготовка мастер смесь ПЦР в трубу нуклеиназы бесплатно. Для каждой реакции, используйте 0.4 мкл обратного праймера (10 мкм), 0,4 мкл вперед праймера (10 мкм), 1,6 мкл MgCl2 (25 мм), 2 мкл 10 x CoralLoad концентрата, 10 мкл 2 x PCR мастер смеси, 4 мкл 5 x Q-раствор, 1 мкл ДНК и 0,6 мкл воды, свободной от нуклеиназы.
- Передать Термоциклер пластину реакции и выполнять ПЦР с использованием следующих условий: 95 ° C в течение 15 мин, 50 циклов 94 ° C за 30 s, 56 ° C за 30 s и 72 ° C за 30 сек, с шагом расширение окончательного 10 мин при 72 ° C. Праймеры для пиросеквенирования SNCA Интрон 1 перечислены в таблице 1, рис. 7Aи дополнительная цифра 1.
- После усиления Визуализируйте ампликонами, используя 2 мкл продукта PCR бромидом ethidium окрашивание, следующие электрофорез геля агарозы.
- Пиросеквенирование проверяются с помощью смеси unmethylated (U) и метилированию (М) преобразование бисульфита ННО в следующих соотношений, а именно 100U:0M, 75U:25M, 50U:50M, 25U:75M и 0U:100M (см. Таблицу материалы).
- Проводить пиросеквенирования с помощью пиросеквенирования реагентов (см. Таблицу материалы) и рассчитать значения метилирования для каждого сайта CpG, используя пиросеквенирования программного обеспечения. Для подробного пиросеквенирования протокол обратитесь к Бэссил et al.37.
Access restricted. Please log in or start a trial to view this content.
Результаты
Проверка производства титры LV-dCas9-DNMT3A-GFP/Puro векторов, по сравнению с коллегой GFP наивно
Мы исполняли p24gag ELISA для сравнения между физической титры LV-dCas9-DNMT3A-GFP/Puro с коллегами GFP/Puro наивными. Представитель результаты, представл...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
LVs начали появляться как транспортное средство выбора для редактирования epigenome, особенно в контексте генетических заболеваний, главным образом из-за их способности (i) размещения больших нагрузок ДНК и (ii) эффективно передают широкий спектр деления и nondividing клетки. Крупногабаритная тара...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Университет Дьюка подал предварительной патентной заявки, связанные с настоящим исследованием.
Благодарности
Эта работа частично финансируется Кан нейротехнология развития премию (O.C.) и национальных институтов из здравоохранения/Национальный институт от неврологических расстройств и инсульта (NIH/NINDS) (R01 NS085011 для O.C.).
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| 0.22 μM filter unit, 1 L | Corning | 430513 | |
| 0.45-μm filter unit, 500 mL | Corning | 430773 | |
| 100 mm TC-Treated Culture Dish | Corning | 430167 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 50 mL conical centrifuge tubes | Corning | 430291 | |
| 6-well plates | Corning | 3516 | |
| Aggrewell 800 | StemCell Technologies | 34811 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| BD FACS | Becton Dickinson | 338960 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| Eppendorf Cell Imaging Slides | Eppendorf | 30742060 | |
| High-binding 96-well plates | Corning | 3366 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Reversible Strainer | StemCell Technologies | 27215 | |
| SW32Ti rotor | Beckman Coulter | 369650 | |
| VWR® Disposable Serological Pipets, Glass, Nonpyrogenic | VWR | 93000-694 | |
| VWR® Vacuum Filtration Systems | VWR | 89220-694 | |
| xMark™ Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| Accutase | StemCell Technologies | 7920 | |
| Anti-Adherence Rinsing Solution | StemCell Technologies | 7010 | |
| Anti-FOXA2 Antibody | Abcam | Ab60721 | |
| Anti-Nestin Antibody | Abcam | Ab18102 | |
| Antibiotic-antimycotic solution, 100x | Sigma Aldrich | A5955-100ML | |
| B-27 Supplement (50x), minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| BES | Sigma Aldrich | B9879 - BES | |
| Bovine Albumin Fraction V (7.5% solution) | Thermo Fisher Scientific | 15260037 | |
| CHIR99021 | StemCell Technologies | 72052 | |
| Corning Matrigel hESC-Qualified Matrix | Corning | 08-774-552 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| DMEM-F12 | Lonza | 12-719 | |
| DMEM, high glucose media | Gibco | 11965 | |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | |
| EpiTect PCR Control DNA Set | Qiagen | 596945 | |
| EZ DNA Methylation Kit | Zymo Research | D5001 | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Gentamicin | Thermo Fisher Scientific | 15750078 | |
| Gentle Cell Dissociation Reagent | stemCell Technologies | 7174 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Human Recombinant bFGF | StemCell Technologies | 78003 | |
| Human Recombinant EGF | StemCell Technologies | 78006 | |
| Human Recombinant Shh (C24II) | StemCell Technologies | 78065 | |
| MEM Non-Essential Amino Acids Solution (100x) | Thermo Fisher Scientific | 11140050 | |
| mTeSR1 | StemCell Technologies | 85850 | |
| N-2 Supplement (100x) | Thermo Fisher Scientific | 17502001 | |
| Neurobasal Medium | Thermo Fisher Scientific | 21103049 | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| PyroMark PCR Kit | Qiagen | 978703 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| SB431542 | StemCell Technologies | 72232 | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| STEMdiff Neural Induction Medium | StemCell Technologies | 5835 | |
| STEMdiff Neural Progenitor Freezing Medium | StemCell Technologies | 5838 | |
| TaqMan Assay FOXA2 | Thermo Fisher Scientific | Hs00232764 | |
| TaqMan Assay GAPDH | Thermo Fisher Scientific | Hs99999905 | |
| TaqMan Assay Nestin | Thermo Fisher Scientific | Hs04187831 | |
| TaqMan Assay OCT4 | Thermo Fisher Scientific | Hs04260367 | |
| TaqMan Assay PPIA | Thermo Fisher Scientific | Hs99999904 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| Y27632 | StemCell Technologies | 72302 | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Goat serum, Sterile, 10 mL | Sigma | G9023 | Working concentration 1:1000 |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Normal mouse serum, Sterile, 500 mL | Equitech-Bio | SM30-0500 | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| pMD2.G | Addgene | 12253 | |
| pRSV-Rev | Addgene | 52961 | |
| psPAX2 | Addgene | 12259 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsmBI | New England Biolabs | R0580S | |
| BsrGI | New England Biolabs | R0575S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
Ссылки
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Gaj, T., Gersbach, C. A., Barbas, C. F. 3rd ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology. 31 (7), 397-405 (2013).
- Thakore, P. I., Black, J. B., Hilton, I. B., Gersbach, C. A. Editing the epigenome: technologies for programmable transcription and epigenetic modulation. Nature Methods. 13 (2), 127-137 (2016).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Gilbert, L. A., et al. Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. Cell. 159 (3), 647-661 (2014).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Chavez, A., et al. Highly efficient Cas9-mediated transcriptional programming. Nature Methods. 12 (4), 326-328 (2015).
- Horlbeck, M. A., et al. Nucleosomes impede Cas9 access to DNA in vivo and in vitro. eLife. 5, (2016).
- Chavez, A., et al. Comparison of Cas9 activators in multiple species. Nature Methods. 13 (7), 563-567 (2016).
- Zhou, H., et al. In vivo simultaneous transcriptional activation of multiple genes in the brain using CRISPR-dCas9-activator transgenic mice. Nature Neuroscience. 21 (3), 440-446 (2018).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell. 159 (3), 635-646 (2014).
- Konermann, S., et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517 (7536), 583-588 (2015).
- Holtzman, L., Gersbach, C. A. Editing the Epigenome: Reshaping the Genomic Landscape. Annual Review of Genomics and Human Genetics. , (2018).
- Perez-Pinera, P., et al. Synergistic and tunable human gene activation by combinations of synthetic transcription factors. Nature Methods. 10 (3), 239-242 (2013).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nature Methods. 12 (12), 1143-1149 (2015).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- McDonald, J. I., et al. Reprogrammable CRISPR/Cas9-based system for inducing site-specific DNA methylation. Biology Open. 5 (6), 866-874 (2016).
- Huang, Y. H., et al. DNA epigenome editing using CRISPR-Cas SunTag-directed DNMT3A. Genome Biology. 18 (1), 176(2017).
- Vojta, A., et al. Repurposing the CRISPR-Cas9 system for targeted DNA methylation. Nucleic Acids Research. 44 (12), 5615-5628 (2016).
- Razin, A., Kantor, B. DNA methylation in epigenetic control of gene expression. Progress in Molecular and Subcellular Biology. 38, 151-167 (2005).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18786-18791 (2009).
- Kantor, B., et al. Downregulation of SNCA Expression by Targeted Editing of DNA Methylation: A Potential Strategy for Precision Therapy in PD. Molecular Therapy. , (2018).
- Jowaed, A., Schmitt, I., Kaut, O., Wullner, U. Methylation regulates alpha-synuclein expression and is decreased in Parkinson's disease patients' brains. Journal of Neuroscience. 30 (18), 6355-6359 (2010).
- Wang, Y., et al. A DNA methyltransferase inhibitor, 5-aza-2'-deoxycytidine, exacerbates neurotoxicity and upregulates Parkinson's disease-related genes in dopaminergic neurons. CNS Neuroscience & Therapeutics. 19 (3), 183-190 (2013).
- Matsumoto, L., et al. CpG demethylation enhances alpha-synuclein expression and affects the pathogenesis of Parkinson's disease. PLOS One. 5 (11), e15522(2010).
- Desplats, P., et al. Alpha-synuclein sequesters Dnmt1 from the nucleus: a novel mechanism for epigenetic alterations in Lewy body diseases. Journal of Biological Chemistry. 286 (11), 9031-9037 (2011).
- Ai, S. X., et al. Hypomethylation of SNCA in blood of patients with sporadic Parkinson's disease. Journal of the Neurological Sciences. 337 (1-2), 123-128 (2014).
- Tagliafierro, L., Chiba-Falek, O. Up-regulation of SNCA gene expression: implications to synucleinopathies. Neurogenetics. 17 (3), 145-157 (2016).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Molecular Therapy - Methods & Clinical Development. 5, 153-164 (2017).
- Tagliafierro, L., et al. Genetic analysis of alpha-synuclein 3' untranslated region and its corresponding microRNAs in relation to Parkinson's disease compared to dementia with Lewy bodies. Alzheimer’s & Dementia. 13 (11), 1237-1250 (2017).
- Tagliafierro, L., Zamora, M. E., Chiba-Falek, O. Multiplication of the SNCA locus exacerbates neuronal nuclear aging. Human Molecular Genetics. , (2018).
- Kantor, B., McCown, T., Leone, P., Gray, S. J. Clinical applications involving CNS gene transfer. Advances in Genetics. 87, 71-124 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Advances in Genetics. 87, 125-197 (2014).
- Vijayraghavan, S., Kantor, B. A Protocol for the Production of Integrase-deficient Lentiviral Vectors for CRISPR/Cas9-mediated Gene Knockout in Dividing Cells. Journal of Visualized Experiments. (130), e56915(2017).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Molecular Therapy. 16 (12), 1968-1976 (2008).
- Bassil, C. F., Huang, Z., Murphy, S. K. Bisulfite pyrosequencing. Methods in Molecular Biology. 1049, 95-107 (2013).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Research. 43 (13), 6450-6458 (2015).
- Nishimasu, H., et al. Crystal Structure of Staphylococcus aureus Cas9. Cell. 162 (5), 1113-1126 (2015).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. Journal of Virology. 68 (4), 2632-2648 (1994).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. Journal of Virology. 71 (8), 6113-6127 (1997).
- Goffin, V., et al. Transcription factor binding sites in the pol gene intragenic regulatory region of HIV-1 are important for virus infectivity. Nucleic Acids Research. 33 (13), 4285-4310 (2005).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. Journal of Biological Chemistry. 280 (22), 21545-21552 (2005).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Molecular Therapy. 19 (3), 547-556 (2011).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены