Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Lentivirale Vektor-Plattform für die effiziente Bereitstellung von Epigenom-editing-Tools in menschlichen induzierten pluripotenten Stammzellen abgeleitet Krankheitsmodelle
In diesem Artikel
Zusammenfassung
Gezielte DNA Epigenom Bearbeitung stellt eine leistungsstarke Therapieansatz. Dieses Protokoll beschreibt die Produktion, Reinigung und Konzentration der all-in-One Lentivirale Vektoren beherbergen das CRISPR-dCas9-DNMT3A Transgen für Epigenom-editing-Anwendungen in menschlichen induzierten pluripotenten Stammzellen (HiPSC)-Neuronen abgeleitet.
Zusammenfassung
Die Verwendung von HiPSC abgeleitete Zellen stellt einen wertvollen Ansatz zur menschlichen neurodegenerativen Krankheiten zu studieren. Hier beschreiben wir ein optimiertes Protokoll für die Differenzierung von HiPSCs abgeleitet von einem Patienten mit der Parkinson-Krankheit (PD) die Verdreifachung der Alpha-Synuclein Gens (SNCA) Locus-relevanten dopaminergen neuronalen Populationen. Beweise sammeln hat gezeigt, dass hohe Konzentrationen von SNCA ursächlich für die Entwicklung der PD erkennen die ungedeckten neue therapeutische Ansätze für PD, vor allem die Ausrichtung auf die Regulierung der SNCA Ausdruck, schaffen müssen wir vor kurzem entwickelt ein CRISPR/dCas9-DNA-Methylierung-basiertes System zur epigenetisch modulieren SNCA Transkription durch Methylierung Ebenen auf der SNCA Intron 1 regulatorischen Region zu bereichern. Das System, bestehend aus einem Toten liefern (deaktiviert) Version des Cas9 (dCas9) fusioniert mit der katalytischen Domäne des DNA-Methyltransferase Enzym 3A (DNMT3A), Lentivirale Vektor verwendet. Dieses System gilt für Zellen mit der Verdreifachung der SNCA -Locus und reduziert die SNCA-mRNA und Protein-Spiegel um ca. 30 % durch die gezielte DNA-Methylierung der SNCA Intron 1. Die fein abgestimmte Herabregulation der SNCA Ebenen rettet krankheitsbezogenen zelluläre Phänotypen. Das aktuelle Protokoll wollen wir eine schrittweise Anleitung zur Differenzierung von HiPSCs in neurale Vorläuferzellen (NPCs) und die Etablierung und Validierung von Pyrosequenzierung Assays für die Auswertung des Profils Methylierung in der SNCA beschreiben Intron 1. Um im Detail die Lentivirus-CRISPR/dCas9-System in diesen Experimenten verwendeten umreißen, beschreibt dieses Protokoll zu produzieren, zu reinigen und Lentivirale Vektoren zu konzentrieren und ihre Eignung für Epigenom und Genom-Bearbeitung Anwendungen mit hervorheben HiPSCs und NPCs. Das Protokoll ist leicht anpassbar und kann verwendet werden, um hohe Titer Lentiviren für in-vitro- und in-vivo Anwendungen zu produzieren.
Einleitung
Mehreren Epigenom-editing-Plattformen wurden vor kurzem entwickelt, um jede DNA-Sequenzen in den Regionen abzielen, die gen-Expression-1,-2zu kontrollieren. Die erstellten Epigenom-editing-Tools sollen (i) Transkription regulieren, (Ii) Änderungen posttranslationale Histon-Modifikationen, (Iii) ändern DNA-Methylierung und (iv) modulieren regulatorisches Element Interaktionen. Der Ansatz, die Transkription/Chromatin-Modifizierer auf eine deaktivierte (tot) Cas9 zu verankern (dCas9) von zuvor entwickelten Epigenom-editing-Plattformen, z. B. Zink angehoben finger Proteine (ZFPs) und Transkription Aktivator-ähnlichen Effektoren (Geschichten) beherbergen eine potente transkriptionelle Effektor-Domäne verschmolzen (ED) mit der gestalteten DNA-bindende Domäne (DBD)3. Die Ergebnisse der gewünschten Phänotyp wie Aktivierung oder Unterdrückung zeichnet sich durch die Effektor-Molekül an der endogenen Loci (Abbildung 1) verankert. Um programmierbare transcriptional Aktivatoren zu erstellen, sind dCas9/gRNA Module mit VP164,5,6 (Abbildung 1A), einer viralen Aktivierungsdomäne verknüpft, die Pol-II und die allgemeine Transkriptionsmaschinerie rekrutiert. Die Änderung dieses Systems hat VP64, ein Tetramer VP16 Domains, bietet eine noch robustere Aktivierung Rate5,6enthalten. Das System wurde erfolgreich eingesetzt, um Codierung und nichtkodierende Regionen zu aktivieren, indem Sie auf Förderer und regulatorischen Elemente. Wichtig ist, auch wenn VP64 Moleküle der Chromatinstruktur in der Zielregion nicht direkt ändern, es Rekruten Chromatin-Modifikatoren, die Ergebnisse im Ablagerung der aktiven (Euchromatin) Marken, unter anderem als H3/H4 Acetylierung und H3-K4 di binden / Tri-Methylierung5,6. Neben VP64 hat der p65-Untereinheit des menschlichen NF-κB-Komplexes zur dCas9/gRNA Modul7angebunden worden. Interessanterweise führt die Anbindehaltung von diese Effektoren für die Regionen stromaufwärts der Transkription Start Seiten (TSK) und innerhalb von Promotoren einen starken gen Induktion. Dennoch können VP64 und p65 Effektoren auch die activatory Wirkungen ausüben, während der Verbindung mit den Regionen flussabwärts von TSK und am distalen Geschmacksverstärker7,8. Eine robustere transcriptional Antwort zu entlocken, müssen mehrere dCas9-VP64 oder dCas9-p65 Fusionen in ein einzelnes Ziel Locus9,10eingestellt werden. So führte die jüngste Entwicklung der nächsten Generation Aktivatoren, die mehrere Effektor Domains durch einen einzigen dCas9-gRNA Komplex, wie SunTag, rekrutieren eine stärkere Aktivierung Fähigkeit im Vergleich zu dCas9-VP64 Fusion Kollegen11 , 12. eine verbesserte transkriptionelle Aktivierung erzielt wurde, durch die Verschmelzung von VP64, p65 und Rta (VPR), einer Domäne werden von Gamma-Herpesviren an den C-Terminus des dCas913 (Abb. 1A). Ähnliche CRISPR/dCas9-Systeme wurden für Target-spezifische Unterdrückung (Abbildung 1 b) entwickelt.
Endogenen gen Unterdrückung kann mit veränderter Repressor Fusionen durch eine Vielzahl von Mechanismen (Abbildung 1 b) erreicht werden. Es hat sich gezeigt, dass CRISPR/dCas9 Systeme, verbunden mit der Repressor DBD (auch ohne ein Effektor-Domäne/s), effizient Genexpression während angebunden, um einen Promotor oder upstream/downstream-TSS Regionen3,6 Schweigen kann ,14. Die Auswirkungen auf die Transkription wird durch die sterische Einmischung der Transkription Faktor Bindung und RNA-Polymerase Verarbeitung verursacht. Dennoch sind umfassendere Ansätze notwendig, da gen Unterdrückung durch sterische Behinderung allein oft nicht ausreicht für robuste zum Schweigen zu bringen. Die jüngste Entwicklung der nächsten Generation von Schalldämpfern auf Basis CRISPR/dCas9 Systeme tragen transkriptioneller Repressor Domänen (TRDs), Histon Modifikatoren (H3-K9 - di / Tri-Methylierung, H3-K27 di-/ Tri-Methylierung; H3-K36 di-/ Tri-Methylierung, H3/H4 Deacetylation), und führte zum Bau von epigenetischen Tools ermöglicht stabilere Effekte4,5,15,16, zum Schweigen zu bringen (CpG) DNA-Methylierung 17,18,19,20. Es wurde nachgewiesen, dass die Rekrutierung von diese epigenetischen Modifikatoren der DNA führen zur Bildung von mehr geschlossen und verkürzte Chromatin, die in der Regel ein potenter Stummschaltungs-Ergebnis21,22erzeugen. Die am häufigsten Stummschaltungs-Domäne mit DBDs verwendet ist der Krüppel-assoziierten Box (Krabbe)4,5. Die Einstellung des Faktors nachgewiesen Chromatin Änderungen entsprechen; Allerdings sind die Mechanismen dieser Modifikationen noch aufgeklärt16,17,18sein. Vor kurzem hat sich gezeigt, dass die Lokalisierung von KRAB DNA die Montage von Histon-Methyltransferase SETDB1 und die Histon Deacetylation (HDAC) NuRD-komplexe fördern kann, die Möglichkeit, die diese Interaktionen die Bildung von vermitteln Chromatin-Kondensation und transcriptional silencing3,13. Als ein alternativer Ansatz können Effektor Domänen, DBDs, erstellen Sie eine benutzerdefinierte epigenetische silencing Protein fusioniert werden. Dieses System katalysiert direkt repressive DNA-Spuren oder Histon-Modifikationen.
Vor kurzem hat die Verwendung von synthetischen CRISPR/dCas9-Systeme angebunden an das Enzym DNMT3A transkriptionelle Deaktivierung zweckentfremdet worden. DNMT3A katalysiert DNA-Methylierung, die transkriptionelle Repression während der Bildung von Heterochromatin auf endogene gen Promotoren und anderen regulatorischen Regionen (Abbildung 1 b)18,20ausübt. McDonald Et Al.18 und Vojta Et Al.20 waren die ersten Autoren berichten, dass DNA-Methylierung verwendet werden kann, für Epigenom-Gen-silencing oder Unterdrückung, nachzuweisen, dass das Plasmid geliefert dCas9-DNMT3A-Fusion-System potent verbessern können Cytosin Methylierung um die TSS-18,-20. McDonald und Kollegen gezeigt, dass die Beschäftigung der Strategie von einer signifikanten Reduktion (ca. 40 %) führen kann in einem Tumor-Suppressor-gen Level CDKN2A mRNA18. Ebenso zeigt das unmethylated Promotorregion BACH oder IL6ST Gene targeting erhöhter CpG Methylierung, die mit einem doppelten Rückgang der gen-Expression-20korreliert worden. Unser Labor hat vor kurzem die Verwendung von DNA-Methylierung für die pathologischen Ergebnisse der SNCA Überexpression (Abbildung 2)23mildernde umgewidmet. Die Strategie basiert auf selektiven Verstärkung in der DNA-Methylierung in der SNCA Intron 1 Region, wie es bereits berichtet wurde, zu hypomethyliert bei Morbus Parkinson und Demenz mit Lewy Körpern (DLB) Gehirn24,25, 26. Diese Hypomethylierung ist mit SNCA Überexpression, bietet somit ein attraktives Ziel für therapeutische Intervention24,27,28verbunden worden. Wir zeigten kürzlich einem niedrigen Niveau der DNA-Methylierung in der SNCA Intron 1 Region in HiPSC abgeleitet dopaminergen NPCs aus einem PD-Patienten mit der SNCA Verdreifachung23gewonnen. Der Vorteil dieser experimentellen Modells ist, dass die NPCs können robust in Kultur vermehrt oder weiter in Reife Neuronen ermöglichen eine effiziente Sortierung differenziert, genetische Faktoren zu identifizieren, die zelluläre Phänotypen, einschließlich oxidativen vermitteln Stress und Apoptose29. Darüber hinaus ermöglicht dieses Modellsystem Wissenschaftler, die entwicklungspolitische Ereignisse zu rekapitulieren, die vor dem Einsetzen der Symptome bei Patienten aufgetreten sind. Darüber hinaus vertreten HiPSC abgeleitet NPCs ein großartiges Werkzeug, um die zellulären und molekularen Wege zugeordnete Genexpression zu testen. Wichtig ist, können HiPSC abgeleitet NPCs in Kombination mit State-of-the-Art CRISPR/Cas9-Epigenom Technologie die Entwicklung der "Next-Generation-Drogen" für viele Neurodegenerative Erkrankungen erheblich erleichtern.
Um pathologische Verringerung der SNCA Ausdruck wir kürzlich entwickelt, ein Lentivirus basierendes System mit einem dCas9-DNMT3A Schmelzverfahren Protein und gRNA, speziell Ziel CpG Methylierung innerhalb der SNCA Intron 1 (Abbildung 2A)23. Dieses Protokoll wird Lentivirale Vektor (LV)-Design und Produktion im Detail beschreiben. LVs repräsentieren ein wirksames Mittel zur Bereitstellung von CRISPR/dCas9 Komponenten aus mehreren Gründen, nämlich (i) ihre Fähigkeit, sperrige DNA tragen einfügt, (Ii) einen hohen Wirkungsgrad von transducing eine Vielzahl von Zellen, einschließlich der trennende und nondividing Zellen30 , und (Iii) ihre Fähigkeit, minimale zytotoxische und immunogenen Reaktionen auslösen. Vor kurzem, wir das LV-System auf HiPSC abgeleitet dopaminergen Neuronen eines Patienten mit der Verdreifachung der SNCA Locus angewendet und demonstriert das therapeutische Potenzial von LVs für die Lieferung der Epigenom-Bearbeitung Methylierung Werkzeuge23 ( Abbildung 2 b). In der Tat bewirkt, dass eine LV-gRNA/dCas9-DNMT3A System eine deutliche Steigerung in der DNA-Methylierung bei der SNCA Intron 1 Region. Diese Erhöhung entspricht die Reduktion in der Höhe der SNCA mRNA und Protein23. Außerdem rettet SNCA Herabregulation PD-bezogene Phänotypen in der SNCA Verdreifachung/HiPSC-abgeleitete dopaminergen Neuronen (z. B. mitochondrialen ROS Produktion und Zelle Tragfähigkeit)23. Wichtig ist, haben wir bewiesen, dass der Rückgang der SNCA Ausdruck vom LV-gRNA-dCas9-DMNT3A-System ist in der Lage, die Umkehrung der Phänotypen, die charakteristisch sind für HiPSC abgeleitet dopaminergen Neuronen von einem PD-Patienten, die die durchgeführt SNCA Verdreifachung, z. B. mitochondrialen ROS Produktion und Zelle Lebensfähigkeit23. Das Ziel dieses Protokolls ist (1) das Protokoll der Produktion und Konzentration einer optimierten LV-Plattform für die Erzeugung von qualitativ tittered virale Vorbereitungen zu skizzieren und (2) die Differenzierung des HiPSCs in NPCs zu Reifen dopaminergen gemustert zu beschreiben Neuronen31,32 und die Charakterisierung der Methylierung Ebenen der gezielten Region innerhalb SNCA Intron 1.
Lentivirale Plattformen haben einen großen Vorteil über die beliebteste Plattform der Vektor, nämlich Adeno-assoziierten Vektoren (Flugabwehrpanzer), die das ehemalige Fähigkeit, größere genetische Einsätze33,34unterzubringen ist. Flugabwehrpanzer auf deutlich höhere Erträge generiert werden können, sondern besitzen eine niedrige Verpackungsleistung (< 4,8 kb), gefährden ihre Verwendung für die Bereitstellung von all-in-One CRISPR/Cas9 Systeme. So scheint es, dass die LVs die Plattform der Wahl in den Anwendungen an der Lieferung beteiligten CRISPR/dCas9 Werkzeuge wäre. Daher werden die hier beschriebene Protokoll ein wertvolles Werkzeug für die Forscher in dem Wunsch, effektiv Epigenom-Bearbeiten von Komponenten zu den Zellen und Organen liefern. Das Protokoll weiter skizziert die Strategie, um die Produktion und Ausdruck der Vektoren über eine Änderung in der GUS der Elemente innerhalb der Vektor Ausdruck Kassette30,35zu verbessern. Die Strategie basiert auf dem Roman System entwickelt und in unserem Labor untersucht und zeigt seine Fähigkeit auf virale Partikel im Bereich von 1010 virale Einheiten (VU) /mL30,35zu produzieren.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Entwicklung und Produktion von Virus
- Plasmid-Design und Konstruktion
Hinweis: Der Bau von einem all-in-One-LV-gRNA-dCas9-DNMT3A-Vektor erfolgt mithilfe einer Produktions- und Ausdruck optimiert Expressionskassette von Ortinski Et Al.30veröffentlicht. Die Vektor-Kassette trägt eine Wiederholung des Standortes Anerkennung der Transkriptionsfaktor Sp1 und eine State-of-the-Art-Löschung innerhalb der unübersetzte (U3 ") und Umgebung: eine 3' Long terminal repeat (LTR) (Abbildung 2A)30,36. Das Vektor-Rückgrat hat sich herausgestellt, liefern und mit dem Ausdruck CRISPR/Cas930,35wirksam.- Erhalten Sie die deaktivieren (Toten) Version des SpCas9 (dCas9) über Site-verwiesene Mutagenese (Daten nicht gezeigt). Ersetzen Sie den Klon beherbergen D10A und H840A Mutationen in Unsichern und RuvC katalytische Domänen des Enzyms, bzw. mit der aktiven Cas9 in pBK30129 durch den Austausch zwischen AgeI-BamHI-Fragmente (Abbildung 3).
- PdCas9-DNMT3A-eGFP die katalytische Domäne DNMT3A ableiten (siehe Tabelle der Materialien) durch die Verstärkung der DNMT3A portion BamHI-429/R 5 '- GAGCGGATCCCCCTCCCG - 3' BamHI-429/L-5' - CTCTCCACTGCCGGATCCGG - 3' (Abbildung 3). Um die Region mit DNMT3A zu verstärken, verwenden Sie die folgenden Bedingungen: (1) 95 ° C für 60 s, (2) 95 ° C für 10 s, (3) 60 ° C für 20 s, (4) 68 ° C für 60 S. Wiederholung Bedingungen 2 bis 4 30 X. Verwenden Sie für die letzte Erweiterung 68 ° C für 3 min und halten 4 ° c zu
- Klonen Sie das DNMT3A-Fragment in der BamHI-Website von der modifizierten pBK301-Vektor dCas9 tragen durch eine BamHI Restriktionsenzym verdaut. Stellen Sie sicher, das Klonen von direkten Sanger Sequenzierung. Beachten Sie, dass die entstandenen Plasmids dCas9-DNMT3A-p2a-Puromycin Transgen beherbergt. Das Plasmid drückt gRNA Gerüst aus menschlichen U6 Promotor (Abbildung 3).
- Ersetzen Sie das Puromycin Reportergen mit grün fluoreszierenden Proteins (GFP), dCas9-DNMT3A-p2a-GFP zu schaffen. DCas9-DNMT3A-p2a-Puro Plasmid mit FseI zu verdauen. Reinigen Sie das Vektor-Fragment mit einer Gel-Reinigung-Methode. Vorbereiten des Einsatzes von pBK201a zu verdauen (pLenti-GFP) mit FseI. FseI Fragment in den Vektor zu klonen. Die entstandenen Plasmid pBK539 birgt dCas9-DNMT3A-p2a-GFP Transgen (Abbildung 3).
- HEK-293T Zellen kultiviert und galvanischen Zellen zur Transfektion
Hinweis: Menschliche embryonale Nieren 293T (HEK-293T) Zellen kultiviert in komplette High-Glukose modifizierten Dulbecco Adlers Medium (DMEM; 10 % bovine Kälberserum, 1 X Antibiotikum Antimykotikums, 1 X Natrium Pyruvat, 1 X unwesentliche Aminosäure, 2 mM L-Glutamin) bei 37 ° C mit 5 % CO 2. für die Reproduzierbarkeit des Protokolls, wird es empfohlen, die Kälberserum testen beim Wechsel zu einer anderen Menge/Charge. Bis zu sechs 15 cm sind Platten für Lentivirale Produktion benötigt.- Verwenden Sie niedrig-Passage Zellen, um eine neue Kultur (niedriger als Durchgang 20) starten. Sobald die Zellen 90 – 95 % Zusammenfluss erreichen, aspirieren Sie die Medien und waschen Sie es sanft mit sterilen 1 x Phosphat-gepufferte Kochsalzlösung (PBS).
- Fügen Sie 2 mL Trypsin-EDTA (0,05 %) und bei 37 ° C für 3 – 5 min inkubieren. Um die Dissoziation Reagenz zu inaktivieren, hinzugeben Sie 8 mL komplette High-Glukose DMEM und Pipette 10 x-15 x mit einer serologischen 10 mL-Pipette, erstellen Sie eine einzelne Zelle Aussetzung der 4 x 106 Zellen/mL.
- Für die Transfektionen Mantel 15 cm Platten mit 0,2 % Gelatine. Fügen Sie 22,5 mL Medium High-Glukose und Samen der Zellen durch Zugabe von 2,5 mL Zellsuspension (insgesamt ~ 1 x 107 Zellen/Platte). Inkubieren Sie die Platten bei 37 ° C mit 5 % CO2 bis 70 – 80 % Zusammenfluss erreicht ist.
- Transfektion von HEK-293T Zellen
- 2 x BES-gepufferte Lösung BBS und 1 M CaCl2, nach Vijayraghavan und Kantor35vorbereiten. Filtern Sie die Lösungen nach Weitergabe durch einen 0,22 µm-Filter und speichern Sie diese 4 ° C. Die Transfektion Mischung muss klar vor seinen Zusatz zu den Zellen sein. Wenn die Mischung während der Inkubation trübe wird, bereiten frische 2 X BBS (pH = 6,95).
- Zur Vorbereitung des Plasmid-Mix verwenden die vier Plasmide wie aufgeführt (die folgende Mischung ist ausreichend für eine 15 cm Platte): 37,5 µg des Vektors CRISPR/dCas9-Transfer (pBK492 [DNMT3A-Puro-NO-gRNA] oder pBK539 [DNMT3A-GFP-NO-gRNA]); 25 µg des pBK240 (psPAX2); 12,5 µg des pMD2.G; 6,25 µg pRSV-Rev (Abb. 4A). Berechnen Sie das Volumen der Plasmide, die anhand der Konzentrationen und fügen Sie die benötigten Mengen in einem 15 mL konische Röhrchen. 312,5 µL 1 M CaCl2 hinzufügen und bringen das Endvolumen auf 1,25 mL, mit sterilen Dd-H2O. sanft 1,25 mL 2 X BBS Lösung beim Aufschütteln der Mischung hinzufügen. 30 min bei Raumtemperatur inkubieren. Die Zellen sind bereit zur Transfektion, sobald sie 70 – 80 % Zusammenfluss sind.
- Aspirieren Sie die Medien, und ersetzen Sie es mit 22,5 mL frisch zubereitete High-Glukose-DMEM ohne Serum. Fügen Sie 2,5 mL der Transfektion Mischung tropfenweise auf jede Platte 15 cm. Schwenken Sie die Platten und Inkubation bei 37 ° C mit 5 % CO2 für 2 – 3 h.
- Fügen Sie nach 3 h 2,5 mL (10 %) des Serums pro Platte und Inkubation über Nacht bei 37 ° C mit 5 % CO2.
- Am 1. Tag nach der Transfektion, beobachten die Zellen, um sicherzustellen, dass es keine oder nur minimale Zelltod und dass die Zellen eine konfluierende Kultur (100 %) gebildet.
- Wechseln Sie das Medium, durch Zugabe von 25 mL frisch zubereitete High-Glukose DMEM und 10 % Serum auf jede Platte.
- Inkubation bei 37 ° C mit 5 % CO2 für 48 h.
- Ernte des virus
- Sammeln Sie den Überstand von den transfizierten Zellen und bündeln sie in 50 mL konische Röhrchen. Zentrifuge bei 400 – 450 X g für 10 min. filtern den Überstand durch eine 0,45 µm-Vakuum-Filter-Einheit. Nach der Filtration kann der Überstand bei 4 ° C für kurzfristige Lagerung (bis zu 4 Tage) gehalten werden. Für die langfristige Lagerung Aliquote vorzubereiten und bei-80 ° c lagern
Hinweis: Die nonconcentrated viralen Vorbereitungen werden voraussichtlich ~ 2 x 107 3 x 107 Fe/ml (siehe Abschnitt 1.5 für Titer-Bestimmung). Es wird dringend empfohlen, Einweg-Aliquote vorzubereiten, da mehrere Gefrier-Tau-Zyklen ein 10 – 20 % Verlust in funktionale Titer führt.
- Sammeln Sie den Überstand von den transfizierten Zellen und bündeln sie in 50 mL konische Röhrchen. Zentrifuge bei 400 – 450 X g für 10 min. filtern den Überstand durch eine 0,45 µm-Vakuum-Filter-Einheit. Nach der Filtration kann der Überstand bei 4 ° C für kurzfristige Lagerung (bis zu 4 Tage) gehalten werden. Für die langfristige Lagerung Aliquote vorzubereiten und bei-80 ° c lagern
- Konzentration der Viruspartikel
Hinweis: Für die Reinigung durchgeführt, einer zweistufigen Doppel-Saccharose-Methode mit einer Saccharose Farbverlauf Schritt und ein Saccharose Kissen ist (Abbildung 4 b).- Erstellen Sie einen Farbverlauf von Saccharose, bereiten die konische Ultrazentrifugation Röhren in der folgenden Reihenfolge: 0,5 mL 70 % Saccharose in 1 X PBS, 0,5 mL 60 % Saccharose in DMEM, 30 % Saccharose in DMEM 1 mL und 2 mL 20 % Saccharose in 1 X PBS.
- Sorgfältig, Hinzufügen des Überstands, gemäß Ziffer 1.4, gesammelt auf den Farbverlauf. Da das Gesamtvolumen von vier 15 cm Platten gesammelt 100 mL ist, verwenden Sie sechs Ultrazentrifugation Röhren, um die virale überstand zu verarbeiten.
- Den virale Überstand unter jedes Ultrazentrifugation Rohr gleichmäßig zu verteilen. Um bei der Zentrifugation Rohr Bruch zu vermeiden, füllen Sie die Ultrazentrifugation Röhrchen mindestens drei Viertel ihrer gesamten Kapazität. Die Röhrchen mit 1 X PBS zu balancieren. Zentrifugieren Sie die Proben bei 70.000 X g 2 h bei 17 ° C.
Hinweis: Um die Saccharose-Schicht während der Beschleunigungs- und Verzögerungswerte Schritte zu gewährleisten, erlauben der Ultrazentrifuge langsam beschleunigen und Abbremsen des Rotors von 0 bis 200 x g und von 200 auf 0 X g während der ersten und letzten 3 min der Spin bzw.. - Sammeln Sie sanft 30 – 60 % Saccharose Brüche in saubere Rohre (Abbildung 4 b). 100 mL Gesamtvolumen 1 x PBS (kalt) oben hinzufügen. Mischen Sie, indem Sie mehrere Male pipettieren.
- Sorgfältig, Schichten der viralen Vorbereitung auf einem Kissen von Saccharose durch das Rohr 4 mL 20 % Saccharose (mit 1 X PBS-Puffer) hinzufügen. Weiter durch Pipettieren ~ 20 – 25 mL der viralen Lösung pro jedes Rohr. Füllen Sie die Rohre mit 1 X PBS, wenn die Lautstärke ihres Inhalts weniger als drei Viertel pro Röhre ist. Abgewogen Sie sorgfältig gegeneinander die Rohre. Zentrifuge bei 70.000 X g 2 h bei 17 ° C. Leeren des Überstands und invertieren die Rohre auf Küchenpapier erlauben die verbleibenden Flüssigkeit abtropfen lassen.
- Entfernen Sie die Flüssigkeit durch die restliche Flüssigkeit vorsichtig absaugen. Beachten Sie, dass bei diesem Schritt Pellets mit dem Virus als kleine durchscheinende Flecken kaum sichtbar sind. Das erste Rohr, das Pellet Aufschwemmen fügen Sie 70 µL 1 X PBS hinzu. Gründlich pipette die Aussetzung und überträgt es auf das nächste Rohr, bis alle Pellets Nukleinsäuretablette sind.
- Waschen Sie die Rohre mit einer zusätzlichen 50 µL 1 x PBS und Mischung als vor. Beachten Sie, dass bei diesem Schritt das Volumen der endgültigen Aufhebung ~ 120 µL ist und leicht milchig erscheint. Um eine klare Suspension zu erhalten, fahren Sie mit einem 60 s Zentrifugation bei 10.000 X g. Übertragen Sie den Überstand zu, zu einem neuen Schlauch, 5 µL-Aliquots, und speichern Sie diese bei-80 ° C.
Hinweis: Lentivirale Vektor Vorbereitungen sind empfindlich gegen die wiederholten Zyklen von Einfrieren und Auftauen. Darüber hinaus wird vorgeschlagen, dass die verbleibenden Schritte in Gewebekultur Containment getan oder in ausgewiesenen Bereichen qualifiziert in Bezug auf die angemessene Ebene der biologischen Sicherheit Standards (Abbildung 4 b).
- Quantifizierung der viralen Titer
Hinweis: Die Schätzung der viralen Titer erfolgt mit der p24 Enzym-linked Immunosorbentprobe Assay (ELISA) Methode (p24gag ELISA) und gemäß dem National Institute of Health (NIH) AIDS-Impfstoff-Programm für die HIV-1 p24 Antigen Capture-Assay mit Protokoll geringfügige Änderungen.- 200 µL von 0,05 % Tween 20 in kalten 1 X PBS (PBS-T) verwenden, um die Brunnen von einer 96-Well-Platte 3 waschen X.
- Verwenden Sie, um die Platte zu beschichten, 100 µL monoklonalen Anti-p24 Antikörper 1:1,500 in 1 X PBS verdünnt. Die Platte über Nacht bei 4 ° c inkubieren
- Bereiten Sie blockierende Reagenzien (1 % Rinderserumalbumin [BSA] mit 1 X PBS-Puffer) und fügen Sie 200 µL in jede Vertiefung, unspezifische Bindung zu vermeiden. Verwenden Sie 200 µL PBS-T, um das auch 3 X für mindestens 1 h bei Raumtemperatur zu waschen.
- Fahren Sie mit der Probenvorbereitung. Beim Arbeiten mit konzentrierter Vektor Präparate Verdünnen des Vektors bei 1: 100 mit 1 µL der Probe, 89 µL Dd-H20 und 10 µL Triton x-100 (auf eine Endkonzentration von 10 %). Verdünnen Sie für nonconcentrated Präparate die Proben bei 01:10.
- Erhalten Sie HIV-1-Standards mit einer doppelten serielle Verdünnung (ab Konzentration ist 5 ng/mL).
- Verdünnen der konzentrierten Proben (vorbereitet in Schritt 1.6.4) RPMI 1640 ergänzt mit 0,2 % Tween 20 und 1 % BSA zu 01:10, 000, 01:50, 000 und 1:250,000 Verdünnungen. In ähnlicher Weise Verdünnen der nonconcentrated Proben (in Schritt 1.6.4) RPMI 1640 ergänzt mit 0,2 % Tween 20 und 1 % BSA herstellen 1: 500, 1:2, 500 und 1:12,500 Verdünnungen.
- Fügen Sie die Proben und Normen auf der Platte in Triplicates. Über Nacht bei 4 ° c inkubieren
- Am nächsten Tag waschen die Brunnen 6 X.
- Fügen Sie 100 µL polyklonale Kaninchen Anti-p24 Antikörper, 1:1,000 in RPMI 1640, 10 % fetalen bovine Serum (FBS), 0,25 % BSA und 2 % normale Maus Serum (NMS), verdünnt und Inkubation bei 37 ° C für 4 h.
- Waschen Sie die Brunnen 6 X. Hinzufügen von Ziege-Anti-Kaninchen-Meerrettich-Peroxidase IgG verdünnt 1: 10.000 RPMI 1640 ergänzt mit normalem Ziegenserum 5 %, 2 % NMS, 0,25 % BSA und 0,01 % Tween 20. Inkubation bei 37 ° C für 1 h.
- Waschen Sie die Brunnen 6 X. TMB-Substrat Peroxidase hinzufügen und bei Raumtemperatur 15 min inkubieren.
- Fügen Sie hinzu, um die Reaktion zu stoppen, 100 µL 1 N HCl. In einem Reader Mikrotestplatte messen die Extinktion bei 450 nm.
- Messung der Intensität der Fluoreszenz-reporter
- Verwenden Sie die virale Aussetzung, um eine 10-divisibel serielle Verdünnung (von 10-1 bis 10-5) mit 1 X PBS-Puffer zu erhalten.
- 5 x 105 HEK-293T Zellen in jede Vertiefung eine 6-Well-Platte-Platte. 10 µL der einzelnen viralen Verdünnung auf die Zellen anwenden und Inkubation bei 37 ° C mit 5 % CO2 für 48 h.
- Fahren Sie mit der Fluoreszenz-aktivierte Zelle sortieren (FACS) Analyse wie folgt: Zellen durch Zugabe von 200 µL 0,05 % Trypsin-EDTA-Lösung zu lösen. Inkubieren Sie die Zellen bei 37 ° C für 5 min und Aufschwemmen Sie ihnen in 2 mL DMEM Medium (mit Serum). Sammeln Sie die Proben in ein 15 mL konische Rohr und Zentrifuge bei 400 X g bei 4 ° c Das Pellet in 500 µL PBS kalt 1 X aufzuwirbeln.
- Befestigen Sie die Zellen durch Zugabe von 500 µL 4 % Paraformaldehyd (PFA) und 10 min bei Raumtemperatur inkubieren.
- Zentrifugieren bei 400 X g bei 4 ° C und das Pellet in 1 mL 1 X PBS aufzuwirbeln. Analysieren des GFP-Ausdrucks mit einem FACS-Instrument, wie in Ortinski Et Al.30beschrieben. Verwenden Sie die folgende Formel, um festzustellen, die funktionelle Virustiter.

Hier,
TG = Anzahl der GFP-positiven Zellen;
TN = Gesamtzahl der Zellen;
N = Gesamtzahl der Zellen ausgestrahlt;
V = Volumen für die Transduktion (in Mikroliter) verwendet.
- GFP-positiven Zellen zählen
Hinweis: Bestimmen Sie die Multiplizität der Infektion (MOI), die für die Transduktion eingesetzt wird. Testen Sie eine breite Palette von MOIs (von MOI = 1 Moi = 10).- Samen 3 x 105 4 x 105 HEK-293T Zellen pro jede Vertiefung einer 6-Well-Platte.
- Wenn die Zellen erreichen > 80 % Zusammenströmen, transduzieren ihnen mit der Vektor in der MOI von Interesse.
- Inkubation bei 37 ° C mit 5 % CO2und überwachen das GFP-Signal in den Zellen für 1 – 7 Tage.
- Die Anzahl der GFP-positiven Zellen. Eine Fluoreszenz-Mikroskop zu beschäftigen (Plan Objektiv 4 X 0,1 N.A., 40 X Vergrößerung) mit einem GLP-Filter (Erregung Wellenlänge = 470 nm, Emissionswellenlänge = 525 nm). Verwenden Sie untransduced Zellen, um die Kontrollgruppe von GFP-negativen Zellen festgelegt.
- Verwenden Sie die folgende Formel zur Bestimmung von der funktionalen Titer des Virus.
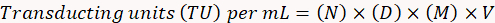
Hier,
N = Anzahl der GFP-positiven Zellen;
D = Verdünnungsfaktor;
M = Vergrößerungsfaktor;
V = Volumen des Virus für die Transduktion verwendet.
Hinweis: Berechnen Sie die Ergebnisse nach diesem Vorbild: für 10 GFP-positiven Zellen (N) bei einer Verdünnung (D) von 10-4 (01:10, 000) bei 20 X Vergrößerung (M) in einer 10 µL Probe (V) gezählt, werden die TU pro Milliliter (10 x 104) x (20) x (10) x (100) = 2 x 108 Vu/mL.
(2) Differenzierung der dopaminergen neurale Vorläuferzellen
- Kultivierung von hiPSCs
Hinweis: HiPSCs eines Patienten mit der Verdreifachung der SNCA Locus, ND34391, stammen aus dem National Institute of Neurological Disorders und Schlaganfall (NINDS) Katalog (siehe Tabelle der Materialien).- Kultur HiPSCs Feeder-unabhängige Bedingungen im Feeder-freie ESC-iPSC Kulturmedium (siehe Tabelle der Materialien) auf hESC-qualifizierte Grundmatrix Membrane (BMM)-beschichtete Platten (siehe Tabelle der Materialien). Konfluierende Kolonien mit 1 mL DMEM/F12 zu waschen, fügen Sie 1 mL Reagenz Dissoziation (siehe Tabelle der Materialien), und 3 min bei Raumtemperatur inkubieren.
- Aspirieren Sie der Dissoziation-Reagenz und 1 mL Feeder-freie ESC-iPSC Kulturmedium.
- Kratzen Sie die Platte mit einer Zelle Heber und Aufschwemmen der Kolonien in 11 mL Feeder-freie ESC-iPSC Kulturmedium durch Pipettieren 4 x-5 x mit Borosilikatglas Pipetten.
- Platte 2 mL der Suspension der Kolonie auf BMM-beschichteten Platten und platzieren Sie die Platte bei 37 ° C mit 5 % CO2. Vornehmen Sie eine tägliche mittlere Änderung und teilen Sie die Zellen, alle 5 – 7 Tage.
- Differenzierung in dopaminergen neurale Vorläuferzellen
Hinweis: Die Differenzierung des HiPSCs in dopaminergen neurale Vorläuferzellen (MD NPCs) erfolgt über einen handelsüblichen neurale Induktion mittlere Protokoll pro Anweisungen des Herstellers, mit leichten Modifikationen31,32 ( siehe Tabelle der Materialien). Der erste Tag der Unterscheidung gilt als Tag 0. Qualitativ hochwertigen HiPSCs sind für effiziente neuronale Differenzierung erforderlich. Die Induktion der MD NPCs ist Performedusing ein Embryoid Körper (EB)-Protokoll basiert.- Vor Beginn der Differenzierung der HiPSCs, vorbereiten eine Microwell-Kultur-Platte (siehe Tabelle der Materialien) nach den Angaben des Herstellers.
- Nach der Vorbereitung der Microwell-Kultur-Platte, fügen Sie 1 mL der neuralen Induktion Medium (NIM; siehe Tabelle of Materials) ergänzt mit 10 µM Y-27632.
- Die Platte erst unmittelbar vor Gebrauch beiseite.
- HiPSCs mit DMEM/F12 zu waschen, fügen Sie 1 mL der Zelle Ablösung Lösung (siehe Tabelle der Materialien), und 5 min bei 37 ° C mit 5 % CO2inkubieren.
- Einzelzellen in DMEM/F12 Aufschwemmen und sie bei 300 X g für 5 min zentrifugieren.
- Vorsichtig Aspirieren überstand und Aufschwemmen der Zellen in NIM + 10 µM Y-27632, eine Endkonzentration von 3 x 106 Zellen/mL zu erhalten.
- Fügen Sie 1 mL der Suspension einzellige zu einem einzigen Brunnen der Microwell-Kultur-Platte und Zentrifugieren Sie die Platte 100 X g für 3 min.
- Untersuchen Sie die Platte unter die Lupe genommen, um eine gleichmäßige Verteilung der Zellen unter der Microwell sicherzustellen, und inkubieren Sie die Zellen bei 37 ° C mit 5 % CO2.
- Führen Sie an Tag 1 – 4eine tägliche partielle mittlere Veränderung.
- Mit Hilfe einer 1 mL Mikropipette, 1,5 mL des Mediums zu entfernen und entsorgen. 1,5 mL frische NIM ohne Y-27632 langsam hinzugeben.
- Wiederholen Sie Schritt 2.2.10 bis Tag 4.
- Am Tag 5Mantel einen Brunnen von einem 6-Well-Platte mit BMM.
- Legen Sie eine reversible 37 µm-Sieb (siehe Tabelle der Materialien) auf ein 50 mL konische Rohr (Abfall). Zeigen Sie den Pfeil in die reversible Sieb nach oben.
- Entfernen Sie das Medium aus Microwell-Kultur-Platte ohne zu stören die gebildeten EBs.
- Fügen Sie 1 mL DMEM/F12 umgehend die EBs mit der Pipette Borosilikat und sammeln filtern sie durch das Sieb.
- Wiederholen Sie Schritt 2.2.15, bis alle EBs aus Microwell-Kultur-Platte entfernt werden.
- Invertieren Sie das Sieb über eine neue 50 mL konische Rohr und 2 mL NIM der EBs zu sammeln.
- Platte 2 mL der Suspension EB in einen einzigen Brunnen der BMM-beschichtete Platte mit einer Pipette Borosilikatglas. Inkubieren Sie die EBs bei 37 ° C mit 5 % CO2.
- Am 6. Tag, 2 mL NIM + 200 ng/mL SHH vorzubereiten (siehe Tabelle der Materialien) und eine tägliche mittlere Änderung vornehmen.
- Am 8. Taguntersuchen Sie den Prozentsatz der neuronalen Induktion.
- Zählen Sie alle angehängten EBs und bestimmen Sie speziell, die Anzahl der jedes einzelne EB, die mit neuronalen Rosetten gefüllt ist. Die neuronale Rosette Induktion mithilfe der folgenden Formel zu quantifizieren.
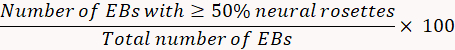
Hinweis: Wenn die neurale Induktion ist < 75 %, neuronale Rosette Auswahl möglicherweise ineffizient. - Am 12. Tagbereiten Sie 250 mL des N2B27 Mediums mit 119 mL Neurobasal Medium, 119 mL DMEM/F12 Medium, 2,5 mL GlutaMAX, 2,5 mL NEAA, 2,5 mL N2 Ergänzung, 5 mL B27 ohne Vitamin A, 250 μL von Gentamicin (50 mg/mL) , und 19. 66 μL der BSA (7 mg/mL).
- Zur Vorbereitung 50 mL des kompletten N2B27 Medium hinzufügen 3 μM CHIR99021, 2 μM SB431542, 20 ng/mL bFGF, 20 ng/mL EGF und 200 ng/mL SHH.
Hinweis: Es ist wichtig, das ausgefüllte vorzubereiten mittlere direkt vor dem Gebrauch. - Aspirieren Medium aus den Brunnen mit der neuronalen Rosetten und waschen mit 1 mL DMEM/F12.
- Ad 1 mL der neuronalen Rosette Auswahl Reagenz (siehe Tabelle der Materialien) und Inkubation bei 37 ° C mit 5 % CO2 für 1 h.
- Entfernen Sie das Auswahl-Reagenz und Zielen Sie mit Hilfe einer 1 mL-Pipette, direkt an der Rosette Cluster.
- Eine 15 mL konische Rohr die Aussetzung hinzu, und wiederholen Sie die Schritte 2.2.25 und 2.2.26 bis die Mehrheit der neuronalen Rosette Cluster gesammelt worden sind.
Hinweis: Zur Vermeidung von Kontamination mit nonneuronal Zelltypen nicht overselect. - Zentrifugieren Sie der Rosette Aussetzung bei 350 X g für 5 min. Aspirat überstand und Aufschwemmen der neuronalen Rosetten in N2B27 + 200 ng/mL SHH. Geben Sie die neuronale Rosette Suspension in eine BMM-beschichtet und inkubieren Sie die Platte bei 37 ° C mit 5 % CO2.
- Am Tage 13-17führen Sie eine tägliche mittlere Änderung mit abgeschlossenen N2B27 Medium. Durchgang der Zellen, wenn die Kulturen 80 – 90 % Zusammenfluss sind.
- Um die Zellen zu teilen, bereiten Sie eine BMM-beschichtete Platte.
- Waschen Sie die Zellen mit 1 mL DMEM/F12, aspirieren Sie das Medium, und fügen Sie 1 mL Reagenz Dissoziation (siehe Tabelle der Materialien).
- 5 min bei 37 ° C inkubieren Sie, fügen Sie 1 mL DMEM/F12 und verdrängen Sie angeschlossene Zellen durch Pipettieren rauf und runter zu. Die NPC-Suspension in einem 15 mL konische Röhrchen zu sammeln. Zentrifuge bei 300 X g für 5 Minuten.
- Den Überstand abgesaugt und Aufschwemmen der Zellen in 1 mL der komplette N2B27 + 200 ng/mL SHH.
- Zählen der Zellen und ihnen bei einer Dichte von 1,25 x 105 Zellen/cm2Teller und inkubieren Sie die Zellen bei 37 ° C mit 5 % CO2.
- Verändern Sie das Medium jeden zweiten Tag, mit kompletten N2B27 + 200 ng/mL SHH.
Hinweis: An dieser Stelle werden NPCs Durchgang (P) als 0. SHH kann das N2B27 Medium bei P2 entzogen werden. - Die Passage der Zellen Zusammenfluss von 80 – 90 % erreicht.
- Bestätigen Sie zu diesem Zeitpunkt, dass die Zellen Nestin und FoxA2 Marker express mithilfe von Immunocytochemistry und qPCR. Dieses Protokoll führt zu der Generation von 85 % doppelt-positiven Zellen für die Nestin und FoxA2 Marker.
- Wiederholen Sie für Passagierung Zellen die Schritte 2.2.31–2.2.36.
- Frieren Sie die Zellen, Durchgang P2 ab. Für das Einfrieren von Zellen, wiederholen Sie die Schritte 2.2.31–2.2.36 und Aufschwemmen der Zelle Pellet bei 2 x 106 4 x 106 Zellen/ml mit kalten neuronalen Vorläuferzellen Einfrieren Medium (siehe Tabelle der Materialien).
- 1 mL Zellsuspension in jeder Cryoröhrchen und Einfrieren der Zellen mit einem standard langsam-Rate-kontrollierten Kühlsystem. Halten Sie für die langfristige Lagerung die Zellen in flüssigem Stickstoff.
- MD-NPCs Auftauen
- Bereiten Sie eine BMM-beschichtete Platte und komplette N2B27 warm. Eine 15 mL konische Rohr 10 mL warmen DMEM/F12 hinzufügen. Platzieren Sie ein Cryoröhrchen in einem 37 ° C Hitze Block 2 min. lang.
- Übertragen Sie die Zellen von den Cryoröhrchen auf das Röhrchen mit DMEM/F12. Zentrifuge bei 300 X g für 5 Minuten.
- Aspirieren des Überstands, Aufschwemmen der Zellen in 2 mL N2B27, und geben Sie die Zellsuspension in einem der BMM-beschichtete Platte. Inkubation der Zellen bei 37 ° C mit 5 % CO2.
- Bereiten Sie eine BMM-beschichtete Platte und komplette N2B27 warm. Eine 15 mL konische Rohr 10 mL warmen DMEM/F12 hinzufügen. Platzieren Sie ein Cryoröhrchen in einem 37 ° C Hitze Block 2 min. lang.
(3) Transduktion von MD NPCs und die Analyse der Methylierung Veränderungen
-
Transduktion von MD NPCs
- Transduzieren MD NPCs mit 70 % Zusammenfluss mit LV-gRNA/dCas9-DNMT3A Vektoren auf MOI = 2. Ersetzen Sie die N2B27 mittlere 16 h Posttransduction.
- N2B27 mit 5 µg/mL Puromycin, 48 h nach der Transduktion hinzufügen. Kulturzellen Sie für 3 Wochen in N2B27 plus Puromycin, stabile MD NPC-Linien zu erhalten. Zellen sind bereit für downstream-Anwendungen (DNA, RNA, Protein-Analysen, phänotypischen Charakterisierung23, Einfrieren und Passagierung).
-
Charakterisierung des Profils Methylierung der SNCA Intron 1
- Von jeder stabil transduced Zelle DNA extrahieren Linie mit einem DNA-Extraktion kit (siehe Tabelle der Materialien).
- Verwendung 800 ng DNA ein Bisulfit Konvertierung, mit einem handelsüblichen kit (siehe Tabelle der Materialien). Eluieren Sie nach der Konvertierung Bisulfit Bisulfit umgewandelt DNA zu 20 ng/µL.
-
PCR für die Pyrosequenzierung Analyse
- Bereiten Sie die PCR-master-Mix in der Nuklease-free Tube. Verwenden Sie für jede Reaktion 0.4 µL des rückwärts-Primer (10 µM), 0.4 µL forward Primer (10 µM), 1,6 µL MgCl2 (25 mM), 2 µL 10 x CoralLoad konzentrieren, 10 µL 2 X PCR-master-Mix, 4 µL 5 x Q-Lösung 1 µL DNA , und 0,6 µL Nuklease-freies Wasser.
- Die Reaktion-Platte auf einem Thermocycler übertragen und PCR unter Verwendung der folgenden Bedingungen erfüllen: 95 ° C für 15 min, 50 Hz 94 ° c für 30 s, 56 ° C für 30 s und 72 ° C für 30 s, mit einem abschließenden 10 Minuten Verlängerung Schritt bei 72 ° C. Primer verwendet für die Pyrosequenzierung SNCA Intron 1 sind in Tabelle 1, Abbildung 7Aund ergänzende Abbildung1aufgeführt.
- Visualisieren Sie nach Verstärkung die Amplifikate mit 2 µL des PCR-Produkt mit Interkalation Bromid Färbung, folgende Agarose-Gelelektrophorese.
- Pyrosequenzierung Assays werden durch Mischungen von unmethylated (U) validiert und methyliert (M) Bisulfit-konvertiert DNAs in den folgenden Verhältnissen, nämlich 100U:0M, 75U:25M, 50U:50M, 25U:75M und 0U:100M (siehe Tabelle der Materialien).
- Conduct Pyrosequenzierung mit Pyrosequenzierung Reagenzien (siehe Tabelle der Materialien) und die Methylierung-Werte für jede CpG-Website mittels Pyrosequenzierung Software berechnen. Für eine detaillierte Pyrosequenzierung Protokoll beziehen sich auf Bassil Et Al.37.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Validierung der Produktion Titer der LV-dCas9-DNMT3A-GLP/Puro Vektoren im Vergleich zu den naiven GFP Gegenstück
Wir führten p24gag ELISA zwischen physischen Titer von LV-dCas9-DNMT3A-GLP/Puro mit den naiven GFP/Puro Kollegen zu vergleichen. Repräsentative Ergebnisse in Abbildung 5A, zeigen, dass körperliche Erträge der Vektoren, erzeugt unter Verwendung des Protokol...
Access restricted. Please log in or start a trial to view this content.
Diskussion
LVs haben damit begonnen, als das Fahrzeug der Wahl für das Epigenom bearbeiten, vor allem im Zusammenhang mit genetischen Erkrankungen entstehen, vor allem wegen ihrer Fähigkeit, (i) Platz für große DNA-Nutzlasten und (Ii) effizient transduzieren eine Vielzahl von Teilen und nondividing Zellen. Die große Verpackung Wirksamkeit der LVs ist besonders vorteilhaft für die Anwendungen, bei denen Verpackung der CRISPR/dCas9-Systeme, die übergroß sind. Aus dieser Perspektive stellen LVs die Plattform der Wahl für die ...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Duke University reichte eine provisorische Patentanmeldung im Zusammenhang mit dieser Studie.
Danksagungen
Diese Arbeit wurde teilweise von den Kahn Neurotechnologie Development Award (für OC) und die nationalen Institute der Gesundheit/National Institute of Neurological Disorders und Schlaganfall (NIH/NINDS) finanziert (R01 NS085011, OC).
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| 0.22 μM filter unit, 1 L | Corning | 430513 | |
| 0.45-μm filter unit, 500 mL | Corning | 430773 | |
| 100 mm TC-Treated Culture Dish | Corning | 430167 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 50 mL conical centrifuge tubes | Corning | 430291 | |
| 6-well plates | Corning | 3516 | |
| Aggrewell 800 | StemCell Technologies | 34811 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| BD FACS | Becton Dickinson | 338960 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| Eppendorf Cell Imaging Slides | Eppendorf | 30742060 | |
| High-binding 96-well plates | Corning | 3366 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Reversible Strainer | StemCell Technologies | 27215 | |
| SW32Ti rotor | Beckman Coulter | 369650 | |
| VWR® Disposable Serological Pipets, Glass, Nonpyrogenic | VWR | 93000-694 | |
| VWR® Vacuum Filtration Systems | VWR | 89220-694 | |
| xMark™ Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| Accutase | StemCell Technologies | 7920 | |
| Anti-Adherence Rinsing Solution | StemCell Technologies | 7010 | |
| Anti-FOXA2 Antibody | Abcam | Ab60721 | |
| Anti-Nestin Antibody | Abcam | Ab18102 | |
| Antibiotic-antimycotic solution, 100x | Sigma Aldrich | A5955-100ML | |
| B-27 Supplement (50x), minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| BES | Sigma Aldrich | B9879 - BES | |
| Bovine Albumin Fraction V (7.5% solution) | Thermo Fisher Scientific | 15260037 | |
| CHIR99021 | StemCell Technologies | 72052 | |
| Corning Matrigel hESC-Qualified Matrix | Corning | 08-774-552 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| DMEM-F12 | Lonza | 12-719 | |
| DMEM, high glucose media | Gibco | 11965 | |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | |
| EpiTect PCR Control DNA Set | Qiagen | 596945 | |
| EZ DNA Methylation Kit | Zymo Research | D5001 | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Gentamicin | Thermo Fisher Scientific | 15750078 | |
| Gentle Cell Dissociation Reagent | stemCell Technologies | 7174 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Human Recombinant bFGF | StemCell Technologies | 78003 | |
| Human Recombinant EGF | StemCell Technologies | 78006 | |
| Human Recombinant Shh (C24II) | StemCell Technologies | 78065 | |
| MEM Non-Essential Amino Acids Solution (100x) | Thermo Fisher Scientific | 11140050 | |
| mTeSR1 | StemCell Technologies | 85850 | |
| N-2 Supplement (100x) | Thermo Fisher Scientific | 17502001 | |
| Neurobasal Medium | Thermo Fisher Scientific | 21103049 | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| PyroMark PCR Kit | Qiagen | 978703 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| SB431542 | StemCell Technologies | 72232 | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| STEMdiff Neural Induction Medium | StemCell Technologies | 5835 | |
| STEMdiff Neural Progenitor Freezing Medium | StemCell Technologies | 5838 | |
| TaqMan Assay FOXA2 | Thermo Fisher Scientific | Hs00232764 | |
| TaqMan Assay GAPDH | Thermo Fisher Scientific | Hs99999905 | |
| TaqMan Assay Nestin | Thermo Fisher Scientific | Hs04187831 | |
| TaqMan Assay OCT4 | Thermo Fisher Scientific | Hs04260367 | |
| TaqMan Assay PPIA | Thermo Fisher Scientific | Hs99999904 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| Y27632 | StemCell Technologies | 72302 | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Goat serum, Sterile, 10 mL | Sigma | G9023 | Working concentration 1:1000 |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Normal mouse serum, Sterile, 500 mL | Equitech-Bio | SM30-0500 | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| pMD2.G | Addgene | 12253 | |
| pRSV-Rev | Addgene | 52961 | |
| psPAX2 | Addgene | 12259 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsmBI | New England Biolabs | R0580S | |
| BsrGI | New England Biolabs | R0575S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
Referenzen
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Gaj, T., Gersbach, C. A., Barbas, C. F. 3rd ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology. 31 (7), 397-405 (2013).
- Thakore, P. I., Black, J. B., Hilton, I. B., Gersbach, C. A. Editing the epigenome: technologies for programmable transcription and epigenetic modulation. Nature Methods. 13 (2), 127-137 (2016).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Gilbert, L. A., et al. Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. Cell. 159 (3), 647-661 (2014).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Chavez, A., et al. Highly efficient Cas9-mediated transcriptional programming. Nature Methods. 12 (4), 326-328 (2015).
- Horlbeck, M. A., et al. Nucleosomes impede Cas9 access to DNA in vivo and in vitro. eLife. 5, (2016).
- Chavez, A., et al. Comparison of Cas9 activators in multiple species. Nature Methods. 13 (7), 563-567 (2016).
- Zhou, H., et al. In vivo simultaneous transcriptional activation of multiple genes in the brain using CRISPR-dCas9-activator transgenic mice. Nature Neuroscience. 21 (3), 440-446 (2018).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell. 159 (3), 635-646 (2014).
- Konermann, S., et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517 (7536), 583-588 (2015).
- Holtzman, L., Gersbach, C. A. Editing the Epigenome: Reshaping the Genomic Landscape. Annual Review of Genomics and Human Genetics. , (2018).
- Perez-Pinera, P., et al. Synergistic and tunable human gene activation by combinations of synthetic transcription factors. Nature Methods. 10 (3), 239-242 (2013).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nature Methods. 12 (12), 1143-1149 (2015).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- McDonald, J. I., et al. Reprogrammable CRISPR/Cas9-based system for inducing site-specific DNA methylation. Biology Open. 5 (6), 866-874 (2016).
- Huang, Y. H., et al. DNA epigenome editing using CRISPR-Cas SunTag-directed DNMT3A. Genome Biology. 18 (1), 176(2017).
- Vojta, A., et al. Repurposing the CRISPR-Cas9 system for targeted DNA methylation. Nucleic Acids Research. 44 (12), 5615-5628 (2016).
- Razin, A., Kantor, B. DNA methylation in epigenetic control of gene expression. Progress in Molecular and Subcellular Biology. 38, 151-167 (2005).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18786-18791 (2009).
- Kantor, B., et al. Downregulation of SNCA Expression by Targeted Editing of DNA Methylation: A Potential Strategy for Precision Therapy in PD. Molecular Therapy. , (2018).
- Jowaed, A., Schmitt, I., Kaut, O., Wullner, U. Methylation regulates alpha-synuclein expression and is decreased in Parkinson's disease patients' brains. Journal of Neuroscience. 30 (18), 6355-6359 (2010).
- Wang, Y., et al. A DNA methyltransferase inhibitor, 5-aza-2'-deoxycytidine, exacerbates neurotoxicity and upregulates Parkinson's disease-related genes in dopaminergic neurons. CNS Neuroscience & Therapeutics. 19 (3), 183-190 (2013).
- Matsumoto, L., et al. CpG demethylation enhances alpha-synuclein expression and affects the pathogenesis of Parkinson's disease. PLOS One. 5 (11), e15522(2010).
- Desplats, P., et al. Alpha-synuclein sequesters Dnmt1 from the nucleus: a novel mechanism for epigenetic alterations in Lewy body diseases. Journal of Biological Chemistry. 286 (11), 9031-9037 (2011).
- Ai, S. X., et al. Hypomethylation of SNCA in blood of patients with sporadic Parkinson's disease. Journal of the Neurological Sciences. 337 (1-2), 123-128 (2014).
- Tagliafierro, L., Chiba-Falek, O. Up-regulation of SNCA gene expression: implications to synucleinopathies. Neurogenetics. 17 (3), 145-157 (2016).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Molecular Therapy - Methods & Clinical Development. 5, 153-164 (2017).
- Tagliafierro, L., et al. Genetic analysis of alpha-synuclein 3' untranslated region and its corresponding microRNAs in relation to Parkinson's disease compared to dementia with Lewy bodies. Alzheimer’s & Dementia. 13 (11), 1237-1250 (2017).
- Tagliafierro, L., Zamora, M. E., Chiba-Falek, O. Multiplication of the SNCA locus exacerbates neuronal nuclear aging. Human Molecular Genetics. , (2018).
- Kantor, B., McCown, T., Leone, P., Gray, S. J. Clinical applications involving CNS gene transfer. Advances in Genetics. 87, 71-124 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Advances in Genetics. 87, 125-197 (2014).
- Vijayraghavan, S., Kantor, B. A Protocol for the Production of Integrase-deficient Lentiviral Vectors for CRISPR/Cas9-mediated Gene Knockout in Dividing Cells. Journal of Visualized Experiments. (130), e56915(2017).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Molecular Therapy. 16 (12), 1968-1976 (2008).
- Bassil, C. F., Huang, Z., Murphy, S. K. Bisulfite pyrosequencing. Methods in Molecular Biology. 1049, 95-107 (2013).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Research. 43 (13), 6450-6458 (2015).
- Nishimasu, H., et al. Crystal Structure of Staphylococcus aureus Cas9. Cell. 162 (5), 1113-1126 (2015).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. Journal of Virology. 68 (4), 2632-2648 (1994).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. Journal of Virology. 71 (8), 6113-6127 (1997).
- Goffin, V., et al. Transcription factor binding sites in the pol gene intragenic regulatory region of HIV-1 are important for virus infectivity. Nucleic Acids Research. 33 (13), 4285-4310 (2005).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. Journal of Biological Chemistry. 280 (22), 21545-21552 (2005).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Molecular Therapy. 19 (3), 547-556 (2011).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten