È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Piattaforma di vettore lentivirale per la consegna efficiente di strumenti di editing Epigenome nelle umane indotte da modelli di malattia derivati da cellule staminali pluripotenti
In questo articolo
Riepilogo
Mirati epigenome DNA modifica rappresenta un approccio terapeutico potente. Questo protocollo descrive la produzione, purificazione e concentrazione di vettori lentivirali all-in-one che harboring il transgene CRISPR-dCas9-DNMT3A per applicazioni di modifica epigenome in cellule staminali pluripotenti indotte umane (hiPSC)-derivato di neuroni.
Abstract
L'uso di cellule derivate da hiPSC rappresenta un valido approccio per lo studio di malattie neurodegenerative umane. Qui, descriviamo un protocollo ottimizzato per la differenziazione di hiPSCs derivata da un paziente con la triplicazione del luogo del gene (SNCA) dell'alfa-synuclein nel morbo di Parkinson (PD)-popolazioni neuronali dopaminergici pertinenti. Raccogliendo la prova ha dimostrato che alti livelli di SNCA sono causativi per lo sviluppo del PD. riconoscere l'insoddisfatte necessario stabilire nuovi approcci terapeutici per PD, specialmente quelli targeting per la regolazione dell'espressione di SNCA , abbiamo recentemente ha sviluppato un sistema CRISPR/dCas9-DNA-metilazione-based per epigeneticamente modulano la trascrizione di SNCA arricchendo i livelli di metilazione presso la regione regolatrice di SNCA introne 1. A consegnare il sistema, costituito da un morto (disattivato) versione di Cas9 (dCas9) fuso con il dominio catalitico del 3A enzima DNA metiltransferasi (DNMT3A), viene utilizzato un vettore lentivirale. Questo sistema viene applicato alle celle con la triplicazione del locus SNCA e riduce la SNCA-mRNA e livelli della proteina di circa il 30% attraverso la metilazione del DNA mirato di SNCA introne 1. Il fine-tuned downregulation dei livelli SNCA Salva fenotipi cellulari correlati alla malattia. Il protocollo attuale, ci proponiamo di descrivere una procedura dettagliata per la differenziazione hiPSCs nella cellule progenitrici neurali (NPC) l'istituzione e la convalida di pyrosequencing saggi per la valutazione del profilo di metilazione in SNCA introne 1. Per strutturare in modo più dettagliato il sistema di lentivirus-CRISPR/dCas9 utilizzato in questi esperimenti, questo protocollo descrive come produrre, purificare e concentrare vettori lentivirali e per evidenziare la loro idoneità per applicazioni epigenome - e -editing genomico utilizzando hiPSCs e NPC. Il protocollo è facilmente adattabile e può essere usato per produrre lentivirus di alto titolo per applicazioni in vitro e in vivo.
Introduzione
Più piattaforme di editing-epigenome sono stati recentemente sviluppati per indirizzare eventuali sequenze di DNA nelle regioni che controllano l'espressione genica1,2. Gli strumenti di editing epigenome creati sono progettati per (i) regolare la trascrizione, (ii) modificare le modifiche post-traduzionali degli istoni, (iii) modificare la metilazione del DNA e (iv) modulano interazioni elemento regolatore. L'approccio di ancorare i modificatori di trascrizione/cromatina a un Cas9 (morto) disattivato (dCas9) generato da precedentemente sviluppati piattaforme epigenome di editing, come lo zinco dito proteine (ZFPs) e gli effettori di simil-attivatore di trascrizione (racconti), che harboring un dominio effector trascrizionale potente (ED) fuso a progettato dominio legante il DNA (DBD)3. I risultati del fenotipo desiderato come l'attivazione o la repressione è definito dalla molecola effettore ancorata ai loci endogene (Figura 1). Per creare attivatori trascrizionali programmabili, dCas9/gRNA moduli sono collegati a VP164,5,6 (Figura 1A), un dominio di attivazione virale che reclute Pol II ed il macchinario della trascrizione generale. La modifica di questo sistema ha incluso VP64, un tetramero di VP16 domini, fornendo un ancora più robusto attivazione tasso5,6. Il sistema è stato impiegato con successo per attivare le regioni codificanti e non codificanti mirando ai promotori ed elementi regolatori. Cosa importante, anche se VP64 molecole non modificano direttamente la struttura della cromatina della regione di destinazione, recluta i modificatori di cromatina che legano i risultati nella deposizione dei marchi attivo (l'eucromatina), tra cui come H3/H4 acetilazione e H3-K4 di / Tri-metilazione5,6. Oltre a VP64, la subunità p65 del complesso NF-κB umano ha stata legata alla dCas9/gRNA modulo7. È interessante notare che, il tethering di questi effettori per le regioni a Monte dei siti di inizio di trascrizione (TSSs) e all'interno di promotori si traduce in un'induzione genica forte. Tuttavia, VP64 e p65 effettori possono anche esercitare gli effetti attivatori pur essendo legata alle regioni situate a valle di TSSs e a distale rinforzatori7,8. Per suscitare una risposta trascrizionale più robusta, più fusioni dCas9-VP64 o dCas9-p65 devono essere assunti per un bersaglio singolo locus9,10. Come tale, il recente sviluppo di attivatori di prossima generazione, che reclutano domini effettrici multipli da un singolo dCas9-gRNA complesso, ad esempio SunTag, ha provocato una più forte capacità di attivazione confronto tra al dCas9-VP64 fusione controparti11 , 12. un'attivazione trascrizionale migliore è stato ottenuto mediante la fusione di VP64, p65 e Rta (VPR), un dominio di transattivazione da gamma-herpesvirus, fino al C-terminale di dCas913 (Figura 1A). Simili sistemi di CRISPR/dCas9 sono stati sviluppati per la repressione di target-specifici (Figura 1B).
Repressione del gene endogeno può essere raggiunto con fusioni derivati dal repressore attraverso una varietà di meccanismi (Figura 1B). È stato dimostrato che CRISPR/dCas9 sistemi, collegati al repressore DBD (anche senza un dominio effector/s), in modo efficiente possono tacere l'espressione genica mentre legato ad un promotore o a Monte/a valle-TSS regioni3,6 ,14. Gli effetti sulla trascrizione è causato dall'interferenza sterica del legame del fattore di trascrizione ed elaborazione di RNA polimerasi. Tuttavia, sono necessari approcci più completi, come repressione genica di sterico da solo spesso non è sufficiente per il silenziamento robusto. Il recente sviluppo della prossima generazione di silenziatori basati su sistemi CRISPR/dCas9 che trasportano domini di repressore trascrizionale (TRDs), i modificatori dell'istone (H3-K9 di-/ tri-metilazione, H3-K27 di-/ tri-metilazione; H3-K36 di-/ tri-metilazione, H3/H4 deacetilazione) e portato alla costruzione di strumenti epigenetici che permette più robusto silenziamento effetti4,5,15,16, la metilazione del DNA (CpG) 17,18,19,20. È stato dimostrato che l'assunzione di questi modificatori epigenetici al DNA può condurre alla formazione di più cromatina condensata e chiusa, che genera tipicamente un più potente silenziamento risultato21,22. Il dominio più comunemente silenziamento utilizzato con DBDs è la casella associata Krüppel (KRAB)4,5. L'assunzione del fattore è stata dimostrata per la corrispondenza con le modifiche della cromatina; Tuttavia, i meccanismi di queste modifiche sono ancora di essere chiarito16,17,18. Recentemente, è stato dimostrato che la localizzazione di KRAB al DNA possa promuovere l'Assemblea dell'istone metiltransferasi SETDB1 e i complessi di NuRD istone deacetilazione (HDAC), suggerendo la possibilità che queste interazioni mediano la formazione di condensazione della cromatina e trascrizione silenziante3,13. Come approccio alternativo, domini dell'effettore possono essere fusa a DBDs per creare una proteina di silenziamento epigenetica personalizzata. Questo sistema catalizza direttamente repressiva DNA marchi o modificazioni istoniche.
Recentemente, l'uso di sistemi CRISPR/dCas9 sintetici legato all'enzima di DNMT3A è stato reimpiegato per inattivazione trascrizionale. DNMT3a catalizza la metilazione del DNA che esercita la repressione trascrizionale durante la formazione dell'eterocromatina su promotori di geni endogeni e altre regioni regolarici (Figura 1B)18,20. McDonald et al.18 e Vojta et al.20 sono stati i primi autori a riferire che la metilazione del DNA può essere utilizzata per epigenome-genico o repressione, dimostrando che il sistema di fusione di plasmide-consegnato dCas9-DNMT3A potentemente può migliorare metilazione della citosina intorno il TSS18,20. McDonald e colleghe hanno dimostrato che l'occupazione della strategia può provocare una riduzione significativa (circa il 40%) in un gene oncosoppressore, CDKN2A mRNA livelli18. Allo stesso modo, targeting della regione non metilato promotore dei geni BACH o IL6ST Mostra aumentata metilazione di CpG che è stata correlata con una riduzione di duplice nell' espressione genica20. Il nostro laboratorio ha recentemente riproposto l'uso di metilazione del DNA per l'attenuazione dei risultati patologici di SNCA sovraespressione (Figura 2)23. La strategia si basa sul potenziamento selettivo nella metilazione del DNA all'interno della regione di introne 1 SNCA , come precedentemente è stato segnalato per essere ipometilato nel PD e demenza con Lewy (DLB) corpi cervelli24,25, 26. Questo hypomethylation è stato collegato alla sovraespressione di SNCA , offrendo così un obiettivo attraente per intervento terapeutico24,27,28. Recentemente abbiamo mostrato un basso livello di metilazione del DNA nel SNCA introne 1 regione in dopaminergici hiPSC-derivati NPC ottenute da un paziente del Palladio con la triplicazione di SNCA 23. Il vantaggio di questo modello sperimentale è che gli NPC possono essere robustamente propagati nella cultura o ulteriormente differenziati nei neuroni maturi, che consente uno screening efficace identificare i fattori genetici che mediano fenotipi cellulari, tra cui ossidativo lo stress e l'apoptosi29. Inoltre, questo sistema modello permette agli scienziati di ricapitolare gli eventi inerenti allo sviluppo che si sono verificati prima dell'insorgenza dei sintomi nei pazienti. Inoltre, hiPSC-derivati NPC rappresentano un ottimo strumento per testare le vie cellulari e molecolari connesse con l'espressione genica. D'importanza, hiPSC-derivati NPC abbinato alla tecnologia state-of-the-art CRISPR/Cas9-epigenome può facilitare notevolmente lo sviluppo di "farmaci di nuova generazione" per molte malattie neurodegenerative.
Per ridurre i livelli patologici di SNCA espressione, abbiamo recentemente sviluppato un sistema basato su lentivirus che trasportano una proteina di fusione dCas9-DNMT3A e gRNA specificamente metilazione di CpG di destinazione all'interno del SNCA introne 1 (Figura 2A)23. Questo protocollo descriverà vettore lentivirale (LV) progettazione e produzione in dettaglio. LVs rappresentano un mezzo efficace di fornire componenti di CRISPR/dCas9 per diversi motivi, vale a dire (i) la loro capacità di trasportare ingombrante DNA inserti, (ii) un'alta efficienza di trasduzione un'ampia gamma di cellule, compreso le cellule di divisione sia nondividing30 e (iii) loro capacità di indurre risposte citotossiche e immunogeniche minimale. Recentemente, abbiamo applicato il sistema di LV a neuroni dopaminergici hiPSC-derivato da un paziente con la triplicazione del locus SNCA e dimostrato il potenziale terapeutico di LVs per la consegna di editing epigenome metilazione23 ( degli strumenti Figura 2B). Infatti, un sistema di LV-gRNA/dCas9-DNMT3A provoca un significativo aumento nella metilazione del DNA alla regione dell'introne 1 SNCA . Questo aumento corrisponde con la riduzione dei livelli di mRNA e proteina SNCA 23. Inoltre, SNCA downregulation Salva fenotipi PD-relative nel SNCA triplicazione/hiPSC-derivato dopaminergico neuroni (ad esempio, mitocondriale ROS produzione e cella vitalità)23. Cosa importante, abbiamo dimostrato che la riduzione dell'espressione di SNCA dal sistema LV-gRNA-dCas9-DMNT3A è in grado di invertire i fenotipi che sono caratteristici per i neuroni dopaminergici hiPSC-derivato da un paziente di PD che ha trasportato il SNCA triplicazione, quali mitocondriale ROS produzione e cellula attuabilità23. L'obiettivo del presente protocollo è 1) per delineare il protocollo di produzione e la concentrazione di una piattaforma ottimizzata di LV per la generazione di alta-risero preparati virali e 2) per descrivere la differenziazione di hiPSCs in NPC modellato per diventare maturo dopaminergici neuroni31,32 e la caratterizzazione dei livelli di metilazione della regione mirata all'interno di SNCA introne 1.
Lentivirali piattaforme hanno un vantaggio importante sopra la piattaforma più popolare di vettore, vale a dire adeno-associato vettori (AAVs), che è capacità l'ex di ospitare grandi inserti genetici33,34. Adenoassociati possono essere generate in rendimenti significativamente più elevati ma possiedono una capacità di confezionamento basso (< 4,8 kb), compromettendo il loro uso per la distribuzione di sistemi CRISPR/Cas9 all-in-one. Così, sembra che il LVs sarebbe la piattaforma di scelta nelle applicazioni coinvolte nella fornitura di strumenti CRISPR/dCas9. Pertanto, il protocollo descritto qui sarà uno strumento prezioso per i ricercatori desiderosi di fornire in modo efficace modifica epigenome componenti di cellule e organi. Il protocollo ulteriormente delinea la strategia per aumentare le capacità di produzione ed espressione dei vettori tramite una modifica in cis degli elementi all'interno del vettore espressione cassetta30,35. La strategia si basa sul romanzo sistema sviluppato e studiato nel nostro laboratorio ed evidenzia la sua capacità di produrre particelle virali nella gamma di 1010 unità virali (VU) / ml30,35.
Protocollo
1. sistema progettazione e produzione di Virus
- Costruzione e progettazione di plasmide
Nota: La costruzione di un vettore di LV-gRNA-dCas9-DNMT3A all-in-one viene eseguita utilizzando una produzione- e cassetta di espressione espressione-ottimizzata pubblicato da Ortinski et al.30. La cassetta di vettore trasporta una ripetizione del sito riconoscimento del fattore di trascrizione Sp1 e un'omissione di state-of-the-art all'interno il non tradotte (U3') regione di un terminale 3'-lungo Ripeti (LTR) (Figura 2A)30,36. La spina dorsale di vettore è stata trovata per essere efficace nel fornire ed esprimendo CRISPR/Cas930,35.- Ottenere la versione di disattivazione (morti) di SpCas9 (dCas9) tramite mutagenesi sito-diretta (dati non mostrati). Sostituire il clone che harboring le mutazioni D10A e H840A HNH e RuvC domini catalitici dell'enzima, rispettivamente, con l'attivo Cas9 pBK30129 scambiando tra AgeI-BamHI frammenti (Figura 3).
- Derivare il dominio catalitico di DNMT3A da pdCas9-DNMT3A-eGFP (Vedi Tabella materiali) amplificando il DNMT3A porzione BamHI-429/R 5 '- GAGCGGATCCCCCTCCCG - 3' BamHI-429/L 5' - CTCTCCACTGCCGGATCCGG - 3' (Figura 3). Per amplificare la regione contenente DNMT3A, utilizzare le seguenti condizioni: (1) 95 ° C per 60 s, (2) 95 ° C per 10 s, (3) 60 ° C per 20 s, (4) 68 ° C per 60 s. Repeat condizioni 2 a 4 x 30. Per l'estensione finale, utilizzare 68 ° C per 3 min e tenere 4 ° C.
- Clonare il frammento di DNMT3A, digerito da un enzima di restrizione BamHI, nel sito BamHI del vettore pBK301 modificate che trasportano dCas9. Verificare la clonazione di Sanger diretto sequenziamento. Si noti che il plasmide provocato porti dCas9-DNMT3A-p2a-con puromicina transgene. Il plasmide esprime gRNA impalcatura da promotore umano U6 (Figura 3).
- Sostituire il gene reporter con puromicina con la proteina fluorescente verde (GFP) per creare dCas9-DNMT3A-p2a-GFP. Digerire dCas9-DNMT3A-p2a-Puro plasmide con FseI. Purificare il frammento di vettore utilizzando un metodo di purificazione del gel. Preparare l'inserto digerendo pBK201a (pLenti-GFP) con FseI. Clonare il frammento FseI nel vettore. Il plasmide è derivato pBK539 porti dCas9-DNMT3A-p2a-GFP transgene (Figura 3).
- Coltura delle cellule HEK 293T e celle per la transfezione di placcatura
Nota: Embrionali umane del rene 293T (HEK 293T) le cellule sono coltivate in alto-glucosio completo Dulbecco s modified esente Eagle (DMEM; 10% di siero di vitello, 1 x antibiotico antimicotico, piruvato di sodio 1 x, 1 x non indispensabile dell'aminoacido, 2 mM L-Glutammina) a 37 ° C con 5% CO 2. per la riproducibilità del protocollo, si consiglia di testare il siero di vitello quando si passa ad un lotto diverso. Fino a 15cm sei piastre sono necessari per la produzione di vettori lentivirali.- Utilizzare cellule basso-passaggio per avviare una nuova cultura (inferiori al passaggio 20). Una volta che le cellule raggiungono la confluenza di 90 – 95%, i media di aspirare e lavare delicatamente con sterile 1x tampone fosfato salino (PBS).
- Aggiungere 2 mL di tripsina-EDTA (0,05%) e incubare a 37 ° C per 3 – 5 min. Per inattivare il reagente di dissociazione, aggiungere 8 mL di completa del alto-glucosio DMEM e pipetta 10 x x-15 con una pipetta sierologica di 10 mL per creare una sospensione unicellulare di 4 x 106 cellule/mL.
- Per le transfezioni, cappotto piastre 15cm con gelatina di 0,2%. Aggiungere 22,5 mL di terreno di alto-glucosio e le cellule del seme con l'aggiunta di 2,5 mL di sospensione cellulare (per un totale di 1 ~ x 107 cellule/piastra). Incubare le piastre a 37 ° C con 5% CO2 sino al 70% – 80% confluenza.
- Transfezione di cellule HEK 293T
- Preparare 2 x soluzione tamponata BES BBS e 1m CaCl2, secondo i pii e Kantor35. Filtrare le soluzioni passandoli attraverso un filtro da 0,22 µm e conservarli a 4 ° C. Il mix di transfezione deve essere chiaro prima della sua aggiunta alle cellule. Se il mix diventa torbida durante l'incubazione, preparare fresco 2 x BBS (pH = 6.95).
- Per preparare il mix di plasmide, utilizzare i quattro plasmidi come indicato (la seguente miscela è sufficiente per un piatto di 15 cm): 37,5 µ g del vettore CRISPR/dCas9-trasferimento (pBK492 [DNMT3A-Puro-NO-gRNA] o pBK539 [DNMT3A-GFP-NO-gRNA]); 25 µ g di pBK240 (psPAX2); 12,5 µ g di pMD2.G; 6,25 µ g di OSPRO-rev (Figura 4A). Calcolare il volume dei plasmidi basato sulle concentrazioni e aggiungere la quantità necessaria in una provetta conica da 15 mL. Aggiungere 312.5 µ l di 1 M CaCl2 e portare il volume finale a 1,25 mL, utilizzando sterile dd-H2O. delicatamente aggiungere 1,25 mL di 2 x BBS soluzione mentre nel Vortex il mix. Incubare per 30 min a temperatura ambiente. Le cellule sono pronte per la transfezione, una volta che sono 70% – 80% confluenti.
- Aspirare i media e sostituirlo con 22,5 mL di preparati al momento alto-glucosio DMEM senza siero. Aggiungere 2,5 mL della miscela di transfezione goccia a goccia per ogni piastra 15 cm. Le piastre di turbinio e incubare a 37 ° C con 5% CO2 per 2 – 3 h.
- Dopo 3 h, aggiungere 2,5 mL (10%) del siero per piastra e incubare per una notte a 37 ° C con 5% CO2.
- Il giorno 1 dopo la trasfezione, osservare le cellule per garantire che non vi è nessuna o minima delle cellule morte e che le cellule hanno formato una cultura confluente (100%).
- Cambiare il supporto con l'aggiunta di 25 mL di siero DMEM e 10% alto-glucosio preparato al momento per ogni piatto.
- Incubare a 37 ° C con 5% CO2 per 48 h.
- Vendemmia del virus
- Raccogliere il surnatante da tutte le cellule trasfettate e la loro piscina in provette coniche da 50 mL. Centrifuga a 400 – 450 x g per 10 min. filtrare il surnatante attraverso un'unità di aspirazione filtro 0,45 µm. Dopo la filtrazione, il supernatante può essere mantenuto a 4 ° C per la conservazione a breve termine (fino a 4 giorni). Per l'archiviazione a lungo termine, preparare aliquote e conservare a-80 ° C.
Nota: Le preparazioni nonconcentrated virale si prevedono siano ~ 2 x 107 a 3 x 107 vu/mL (vedere sezione 1.5 per la determinazione del titolo). Si consiglia di preparare aliquote monouso, poiché più cicli di gelo-disgelo si tradurrà in una perdita di 10 – 20% nei titoli funzionali.
- Raccogliere il surnatante da tutte le cellule trasfettate e la loro piscina in provette coniche da 50 mL. Centrifuga a 400 – 450 x g per 10 min. filtrare il surnatante attraverso un'unità di aspirazione filtro 0,45 µm. Dopo la filtrazione, il supernatante può essere mantenuto a 4 ° C per la conservazione a breve termine (fino a 4 giorni). Per l'archiviazione a lungo termine, preparare aliquote e conservare a-80 ° C.
- Concentrazione di particelle virali
Nota: Per la purificazione, un doppio-saccarosio metodo in due fasi che coinvolgono un passo di gradiente di saccarosio e un passo di cuscino di saccarosio è eseguita (Figura 4B).- Per creare un gradiente di saccarosio, preparare le provette coniche ultracentrifugazione nel seguente ordine: 0,5 mL di saccarosio al 70% in PBS 1X, 0,5 mL di saccarosio al 60% in DMEM, 1 mL di saccarosio al 30% in DMEM e 2 mL di saccarosio al 20% in PBS 1X.
- Aggiungere con cautela, il surnatante, raccolto conformemente al punto 1.4, alla sfumatura. Poiché il volume totale raccolto da quattro piatti di 15 cm è 100 mL, è possibile utilizzare sei ultracentrifugazione tubi per elaborare il surnatante virale.
- Distribuire equamente il surnatante virale tra ciascuna provetta ultracentrifugazione. Per evitare la rottura del tubo durante la centrifugazione, riempire le provette di ultracentrifugazione di almeno tre quarti della loro capacità di volume totale. Bilanciare i tubi con PBS 1X. Centrifugare i campioni a 70.000 x g per 2 h a 17 ° C.
Nota: Per mantenere il livello di saccarosio durante le fasi di accelerazione e decelerazione, consentire l'ultracentrifuga per lentamente accelerare e decelerare il rotore da 0 a 200 x g e da 200 a 0 x g durante il primo e l'ultimo 3 min dello spin, rispettivamente. - Raccogliere delicatamente le frazioni di saccarosio 30% – 60% nelle provette pulite (Figura 4B). Aggiungere 1 x fino (freddo) PBS a 100 mL di volume totale. Mescolare pipettando più volte.
- Attentamente, stratificare la preparazione virale su un cuscino di saccarosio con l'aggiunta di 4 mL di saccarosio al 20% (in 1X PBS) al tubo. Continuare di pipettaggio ~ 20 – 25 mL della soluzione virale per ogni provetta. Se il volume del loro contenuto è meno di tre quarti per tubo, riempire le provette con PBS 1X. Valuta attentamente i tubi. Centrifuga a 70.000 x g per 2 h a 17 ° C. Svuotare il surnatante e invertire i tubi su carta assorbente per permettere il rimanente liquido.
- Rimuovere tutto il liquido con cautela aspirando il liquido rimanente. Si noti che, a questo punto, pellet contenenti il virus sono a malapena visibili come piccole macchie traslucide. Aggiungere 70 µ l di 1X PBS al primo tubo per risospendere il pellet. Accuratamente Pipettare la sospensione e trasferirlo al prossimo tubo fino a quando tutti i pellet sono risospese.
- Lavare le provette con un ulteriore 50 µ l di 1X PBS e mix come prima. Si noti che, a questo punto, il volume della sospensione finale è ~ 120 µ l e appare leggermente lattiginoso. Per ottenere una sospensione di chiara, procedere con una centrifugazione di 60 s a 10.000 x g. Trasferire il surnatante in una provetta nuova, fare 5 aliquote µ l e conservarli a-80 ° C.
Nota: Vettore lentivirale preparativi sono sensibili ai cicli ripetuti di congelamento e scongelamento. Inoltre, è suggerito che i passaggi rimanenti sono fatto nel contenimento di coltura del tessuto o in aree qualificato in termini di essere a un livello adeguato di norme di biosicurezza (Figura 4B).
- Quantificazione dei titoli virali
Nota: La stima dei titoli virali viene eseguita utilizzando il metodo di analisi (ELISA) enzima-collegata dell'immunosorbente p24 (p24gag ELISA) e secondo il programma di vaccino AIDS National Institutes of Health (NIH) protocollo per l'analisi di cattura dell'antigene HIV-1 p24 con lievi modifiche.- Utilizzare 200 µ l di PBS di 0.05% Tween 20 in 1x freddo (PBS-T) per lavare i pozzetti di una piastra a 96 pozzetti 3 x.
- Per ricoprire la piastra, utilizzare 100 µ l di anticorpo monoclonale anti-p24 coniugato diluito all'1:1,500 in PBS 1X. Incubare la piastra durante la notte a 4 ° C.
- Preparazione del reagente bloccante (1% albumina di siero bovino [BSA] in 1X PBS) e aggiungere 200 µ l in ogni pozzetto per evitare il legame non specifico. 200 µ l di PBS-T consente di lavare anche 3x per almeno 1 h a temperatura ambiente.
- Procedere con la preparazione del campione. Quando si lavora con le preparazioni concentrate vettoriale, diluire il vettore a 1: 100 con 1 µ l di campione, 89 µ l di dd-H20 e 10 µ l di Triton X-100 (a una concentrazione finale di 10%). Per le preparazioni nonconcentrated, diluire i campioni alle 01:10.
- Ottenere standard di HIV-1 utilizzando una diluizione seriale doppia (la concentrazione iniziale è di 5 ng/mL).
- Diluire i campioni concentrati (preparati al punto 1.6.4) in RPMI 1640 completati con 0,2% Tween 20 e 1% BSA per ottenere 01:10, 000, 01:50, 000 e 1:250,000 diluizioni. Allo stesso modo, diluire i campioni nonconcentrated (preparati al punto 1.6.4) in RPMI 1640 completati con 0,2% Tween 20 e 1% BSA per stabilire 1: 500, 1:2, 500 e 1:12,500 diluizioni.
- Aggiungere i campioni e gli standard sulla piastra in triplici copie. Incubare per una notte a 4 ° C.
- Il giorno dopo, lavare i pozzetti 6 x.
- Aggiungere 100 µ l di anticorpo anti-p24 policlonale del coniglio, diluita al 1:1,000 in RPMI 1640, 10% siero bovino fetale (FBS), 0,25% BSA e siero di topo normale 2% (NMS) e incubare a 37 ° C per 4 ore.
- Lavare i pozzetti 6 x. Aggiungere perossidasi goat anti-rabbit IgG diluito a 1: 10.000 in RPMI 1640 completato con siero di capra normale 5%, 2% NMS, BSA 0,25% e 0,01% Tween 20. Incubare a 37 ° C per 1 h.
- Lavare i pozzetti 6 x. Aggiungere substrato perossidasi TMB e incubare a temperatura ambiente per 15 min.
- Per interrompere la reazione, aggiungere 100 µ l di 1 N HCl. In un lettore di micropiastre, misurare l'assorbanza a 450 nm.
- Misura dell'intensità di fluorescenza reporter
- Utilizzare la sospensione virale per ottenere una diluizione seriale 10 volte (da 10-1 a 10-5) in PBS 1X.
- 5 x 105 HEK 293T cellule in ciascun pozzetto di una piastra a 6 pozzetti della piastra. Applicare 10 µ l di ciascuna diluizione virale alle cellule e incubare a 37 ° C con 5% CO2 per 48 h.
- Procedere alla cella di fluorescenza-attivato l'ordinamento analisi (FACS) come segue: staccare le cellule con l'aggiunta di 200 µ l di soluzione di tripsina-EDTA 0,05%. Incubare le cellule a 37 ° C per 5 min e li risospendere in 2 mL di terreno DMEM (con siero). Raccogliere i campioni in una provetta conica da 15 mL e centrifugare a 400 x g a 4 ° C. Risospendere il pellet in 500 µ l di PBS 1X freddo.
- Fissare le cellule aggiungendo 500 µ l di paraformaldeide al 4% (PFA) e incubare per 10 minuti a temperatura ambiente.
- Centrifugare a 400 x g a 4 ° C e risospendere il pellet in 1 mL di PBS 1X. Analizzare l'espressione di GFP utilizzando uno strumento di FACS, come descritto in Ortinski et al.30. Per determinare il titolo funzionale del virus, utilizzare la seguente formula.

Qui,
TG = numero di cellule GFP-positive;
TN = numero totale delle cellule;
N = numero totale di cellule trasdotte;
V = volume usato per la trasduzione (in microlitri).
- Conteggio di cellule GFP-positive
Nota: Determinare la molteplicità di infezione (MOI) che viene impiegata per la trasduzione. Testare una vasta gamma di MOIs (da MOI = 1 a MOI = 10).- 3 x 105 a 4 x 105 HEK 293T cellule per ogni pozzetto di una piastra a 6 pozzetti del seme.
- Quando le cellule raggiungono > 80% confluenza, trasduce li con il vettore presso il MOI di interesse.
- Incubare a 37 ° C con 5% CO2e monitorare il segnale GFP nelle cellule per 1 – 7 giorni.
- Contare il numero di cellule GFP-positive. Impiegare un microscopio a fluorescenza (piano obiettivo 4x, 0,1 N.A., ingrandimento 40x) utilizzando un filtro GFP (lunghezza d'onda di eccitazione = 470 nm, lunghezza d'onda di emissione = 525 nm). Utilizzare cellule untransduced per impostare la popolazione di controllo delle cellule GFP-negative.
- Utilizzare la seguente formula per determinare il titolo funzionale del virus.
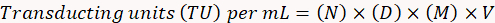
Qui,
N = numero di cellule GFP-positive;
D = fattore di diluizione;
M = fattore di ingrandimento;
V = volume del virus utilizzati per la trasduzione.
Nota: Calcolare i risultati seguendo questo esempio: per 10 cellule GFP-positive (N) ha conteggiate a una diluizione (D) di 10-4 (01:10, 000) a 20 ingrandimenti (M) in un campione di 10 µ l (V), la TU per millilitro sarà (10 x 104) x (20) x (10) x (100) = 2 x 108 vu/mL.
2. la differenziazione delle cellule progenitrici neurali dopaminergiche
- Coltura hiPSCs
Nota: hiPSCs da un paziente con la triplicazione del locus SNCA, ND34391, sono stati ottenuti dall'Istituto nazionale dei disordini neurologici e colpo (NINDS) Catalogo (Vedi tabella materiali).- Cultura hiPSCs condizioni alimentatore indipendente nel medium di coltura ESC-iPSC senza alimentatore (Vedi Tabella materiali) sulla membrana di matrice di base qualificato hESC (BMM)-rivestito di piastre (Vedi Tabella materiali). Lavare colonie confluenti con 1 mL di DMEM/F12, aggiungere 1 mL di reagente di dissociazione (Vedi Tabella materiali) e incubare per 3 min a temperatura ambiente.
- Aspirare il reagente di dissociazione e aggiungere 1 mL di medium di coltura ESC-iPSC senza alimentatore.
- Raschiare la piastra utilizzando un sollevatore di cella e risospendere le colonie in 11 mL di terreno di coltura privo di alimentatore ESC-iPSC di pipettaggio x-5 4 x utilizzando pipette borosilicato.
- Piastra 2 mL di sospensione di Colonia sulle piastre rivestite su BMM e posizionare la piastra a 37 ° C con 5% CO2. Eseguire un cambio medio giornaliero e dividere le celle ogni 5 – 7 giorni.
- Differenziazione in cellule progenitrici neurali dopaminergiche
Nota: La differenziazione di hiPSCs nelle cellule progenitrici neurali dopaminergiche (MD NPC) viene eseguita utilizzando un protocollo di induzione neurale commercialmente disponibile medio secondo le istruzioni dei produttori, con lievi modifiche31,32 ( Vedi Tabella materiali). Il primo giorno della differenziazione è considerato come giorno 0. Alta qualità hiPSCs sono necessari per efficiente differenziazione neurale. L'induzione di MD NPC è performedusing un corpo embryoid (EB)-protocollo basato su.- Prima di iniziare la differenziazione di hiPSCs, preparare una micropiastra di cultura (Vedi Tabella materiali) secondo le istruzioni del produttore.
- Dopo aver preparato la piastra microtiter di coltura, aggiungere 1 mL di medium di induzione neurale (NIM; Vedi Tabella materiali) supplementato con 10 µM Y-27632.
- Mettere da parte la piastra fino all'uso.
- Lavare hiPSCs con DMEM/F12, aggiungere 1 mL di soluzione di distacco delle cellule (Vedi Tabella materiali) e incubare per 5 min a 37 ° C con 5% CO2.
- Risospendere le cellule singole in DMEM/F12 e li Centrifugare a 300 x g per 5 min.
- Attentamente aspirare il supernatante e risospendere le cellule in NIM + 10 µM Y-27632 per ottenere una concentrazione finale di 3 x 106 cellule/mL.
- Aggiungere 1 mL della sospensione unicellulare a un singolo pozzo dei micropozzetti di cultura e centrifugare la piastra a 100 x g per 3 min.
- Esaminare la piastra sotto il microscopio per garantire una distribuzione uniforme delle cellule fra i pozzetti e incubare le cellule a 37 ° C con 5% CO2.
- Nei giorni 1 – 4, eseguire un parziale cambio medio giornaliero.
- Utilizzando una micropipetta da 1 mL, 1,5 mL di terreno di rimuovere e scartare. Lentamente, aggiungere 1,5 mL di NIM fresco senza Y-27632.
- Ripetere il passaggio 2.2.10 fino al giorno 4.
- Il giorno 5, rivestire un pozzetto di una piastra a 6 pozzetti con BMM.
- Posizionare un colino reversibile 37 µm (Vedi Tabella materiali) sulla cima di una provetta conica da 50 mL (rifiuti). Puntate la freccia del setaccio reversibile verso l'alto.
- Rimuovere il supporto dalla piastra microtiter cultura senza disturbare l'EBs formata.
- Aggiungere 1 mL di DMEM/F12 e prontamente raccogliere l'EBs con la pipetta di borosilicato e filtrarli attraverso il filtro.
- Ripetere il passaggio 2.2.15 fino a quando tutte le EBs vengono rimossi dalla piastra microtiter cultura.
- Invertire il colino sopra una nuova provetta conica 50 mL e aggiungere 2 mL di NIM per raccogliere tutti i EBs.
- Piastra 2 mL della sospensione EB in un unico pozzetto della piastra rivestite su BMM usando una pipetta di borosilicato. Incubare la EBs a 37 ° C con 5% CO2.
- Il giorno 6, preparare 2 mL di NIM + 200 ng/mL SHH (Vedi Tabella materiali) ed eseguire un cambio medio giornaliero.
- Il giorno 8, esaminare la percentuale di induzione neuronale.
- Contare EBs collegati e, in particolare, determinare il numero di ogni EB individuale che viene riempito con rosette neurale. Quantificare l'induzione neurale Rosetta utilizzando la seguente formula.
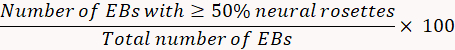
Nota: Se l'induzione neurale è < 75%, selezione di Rosetta neurale potrebbe essere inefficiente. - Il giorno 12, preparare 250 mL di terreno di N2B27 con 119 mL di medium neurobasal, 119 mL di terreno DMEM/F12, 2,5 mL di GlutaMAX, 2,5 mL di NEAA, 2,5 mL di supplemento di N2, 5 mL di B27 senza vitamina A, 250 μL di gentamicina (50 mg/mL) e 19. 66 μL di BSA (7 mg/mL).
- Per preparare 50 mL di terreno completo di N2B27, aggiungere 3 μM CHIR99021, 2 μM SB431542, 20 ng/mL bFGF, 20 ng/mL EGF e 200 ng/mL SHH.
Nota: È importante preparare il completato medio bene prima dell'uso. - Media dai pozzetti contenenti le rosette neurale di aspirare e lavare con 1 mL di DMEM/F12.
- Annuncio 1 mL di reagente di selezione neurale Rosetta (Vedi Tabella materiali) e incubare a 37 ° C con 5% CO2 per 1 h.
- Rimuovere il reagente di selezione e, utilizzando una pipetta da 1 mL, mirano direttamente ai cluster di Rosetta.
- Aggiungere la sospensione in una provetta conica da 15 mL e ripetere i passaggi 2.2.25 e 2.2.26 fino a quando la maggior parte dei glomeruli Rosetta neurali sono stati raccolti.
Nota: Per evitare contaminazioni con tipi cellulari nonneuronal, non overselect. - Centrifugare la sospensione di Rosetta a 350 x g per 5 min. aspirare il supernatante e risospendere le rosette neurale in N2B27 + 200 ng/mL SHH. Aggiungere la sospensione di Rosetta neurale in un pozzetto rivestite su BMM e incubare la piastra a 37 ° C con 5% CO2.
- Nei giorni 13 – 17, eseguire un cambio medio giornaliero usando completato N2B27 mezzo. Quando le culture sono 80% – 90% confluenti del passaggio delle cellule.
- Per dividere le celle, preparare una piastra rivestita con BMM.
- Lavare le cellule con 1 mL di DMEM/F12, aspirare il mezzo e aggiungere 1 mL di reagente di dissociazione (Vedi Tabella materiali).
- Incubare per 5 min a 37 ° C, aggiungere 1 mL di DMEM/F12 e sloggiare cellule allegate pipettando su e giù. Raccogliere la sospensione NPC in una provetta conica da 15 mL. Centrifugare a 300 x g per 5 min.
- Aspirare il supernatante e risospendere le cellule in 1 mL di N2B27 completa + 200 ng/mL SHH.
- Contare le celle e impiattatelo ad una densità di 1,25 x 105 cellule/cm2, incubare le cellule a 37 ° C con 5% CO2.
- Modificare il mezzo ogni altro giorno, utilizzando N2B27 completa + 200 ng/mL SHH.
Nota: In questo passaggio, i PNG sono considerati come passaggio (P) 0. SHH può essere ritirato dal mezzo N2B27 a P2. - Passaggio delle cellule una volta che raggiungono la confluenza di 80 – 90%.
- In questa fase, confermare che le cellule esprimono marcatori Nestin e FoxA2 mediante immunocitochimica e qPCR. Questo protocollo consente la generazione di cellule doppio-positive 85% per i marcatori Nestin e FoxA2.
- Per il passaggio di cellule, ripetere i passaggi 2.2.31–2.2.36.
- Congelare le cellule, a partire da passaggio P2. Per il congelamento delle cellule, ripetere i passaggi 2.2.31–2.2.36 e risospendere il pellet cellulare a 2 x 106 a 4 x 106 cellule/mL utilizzando freddo progenitrici neurali congelamento medio (Vedi Tabella materiali).
- Trasferire 1 mL di sospensione cellulare in ogni cryovial e congelare le celle utilizzando un sistema di raffreddamento standard lento-tasso-controllato. Per l'archiviazione a lungo termine, è necessario mantenere le cellule in azoto liquido.
- Lo scongelamento MD NPC
- Preparare un piatto di BMM-rivestito e caldo N2B27 completa. Aggiungere 10 mL di caldo DMEM/F12 una provetta conica da 15 mL. Posto un cryovial in un blocco di calore di 37 ° C per 2 min.
- Trasferire le cellule dal cryovial nella provetta contenente DMEM/F12. Centrifugare a 300 x g per 5 min.
- Aspirare il supernatante e risospendere le cellule in 2 mL di N2B27 aggiungere la sospensione cellulare in un pozzetto della piastra rivestite su BMM. Incubare le cellule a 37 ° C con 5% CO2.
- Preparare un piatto di BMM-rivestito e caldo N2B27 completa. Aggiungere 10 mL di caldo DMEM/F12 una provetta conica da 15 mL. Posto un cryovial in un blocco di calore di 37 ° C per 2 min.
3. trasduzione di MD NPC e l'analisi dei cambiamenti di metilazione
-
Trasduzione di MD NPC
- Trasdurre gli NPC MD 70% confluenza con vettori LV-gRNA/dCas9-DNMT3A a MOI = 2. Sostituire il posttransduction di media 16 h N2B27.
- Aggiungi N2B27 con 5 con puromicina µ g/mL, 48 h dopo la trasduzione. Le cellule della coltura per 3 settimane in N2B27 plus con puromicina per ottenere le righe MD NPC stabile. Cellule sono pronte per le applicazioni a valle (DNA, RNA, proteine analisi, caratterizzazione fenotipica23, congelamento e passaggio).
-
Caratterizzazione del profilo di metilazione dell'introne SNCA 1
- Estrarre il DNA da ogni cella stabilmente trasdotte riga utilizzando un'estrazione di DNA kit (Vedi Tabella materiali).
- Uso 800 ng di DNA per eseguire una conversione di bisolfito, utilizzando un disponibili in commercio kit (Vedi Tabella materiali). Dopo la conversione di bisolfito, eluire il DNA bisolfuro-convertito a 20 ng / µ l.
-
PCR per l'analisi di pyrosequencing
- Preparare il mix master di PCR in un tubo privo di nucleasi. Per ogni reazione, utilizzare 0,4 µ l di primer reverse (10 µM), 0,4 µ l di primer forward (10 µM), 1,6 µ l di MgCl2 (25 mM), 2 µ l di 10 x CoralLoad concentrato, 10 µ l di mix master di 2 x PCR, 4 µ l di 5 x Q-soluzione, 1 µ l di DNA e 0,6 µ l di acqua priva di nucleasi.
- Trasferire la piastra di reazione a un termociclatore ed eseguire PCR utilizzando le seguenti condizioni: 95 ° C per 15 min, 50 cicli di 94 ° C per 30 s, 56 ° C per 30 s e 72 ° C per 30 s, con un passo di estensione finale 10 min a 72 ° C. Primer usati per il pyrosequencing dell'introne SNCA 1 sono elencati in tabella 1e figura 7A supplementari nella figura 1.
- Dopo l'amplificazione, visualizzare gli amplificati utilizzando 2 µ l di prodotto PCR con bromuro di etidio macchiatura, seguente l'elettroforesi del gel dell'agarosi.
- Pyrosequencing saggi vengono convalidati utilizzando miscele di unmethylated (U) e metilato DNAs di bisolfito-convertito (M) nel modo seguente, vale a dire 100U:0M, 75U:25M, 50U:50M, 25U:75M e 0U:100M (Vedi Tabella materiali).
- Condurre pyrosequencing utilizzando reagenti pyrosequencing (Vedi Tabella materiali) e calcolare i valori di metilazione per ogni sito CpG utilizzando software pirosequenziamento. Per un protocollo dettagliato pyrosequencing, consultare Bassil et al.37.
Risultati
Convalida dei titoli di produzione dei vettori LV-dCas9-DNMT3A-GFP/Puro rispetto alla controparte GFP ingenuo
Abbiamo effettuato p24gag ELISA da confrontare tra titoli fisici di LV-dCas9-DNMT3A-GFP/Puro con le controparti GFP/Puro ingenuo. Risultati rappresentativi, presentati in Figura 5A, dimostrano che rese fisiche dei vettori, generati utilizzando il protocollo descri...
Discussione
LVs hanno cominciato ad emergere come il veicolo della scelta per l'editing di epigenoma, soprattutto nel contesto delle malattie genetiche, principalmente dovuto la loro capacità di (i) di ospitare grandi carichi utili di DNA e (ii) trasducono efficientemente una vasta gamma di divisione e cellule nondividing. L'efficacia di grande imballaggio del LVs è particolarmente utile per le applicazioni che coinvolgono imballaggio dei sistemi CRISPR/dCas9 che sono di grandi dimensioni. Da questa prospettiva, LVs rappresentano ...
Divulgazioni
Duke University depositato una domanda di brevetto provvisorio relazionata a questo studio.
Riconoscimenti
Questo lavoro è stato finanziato in parte dal Kahn neurotecnologie Development Award (di O.C.) e istituti di salute nazionale Istituto nazionale dei disordini neurologici e colpo (NINDS/NIH) (R01 NS085011 di O.C.).
Materiali
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| 0.22 μM filter unit, 1 L | Corning | 430513 | |
| 0.45-μm filter unit, 500 mL | Corning | 430773 | |
| 100 mm TC-Treated Culture Dish | Corning | 430167 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 50 mL conical centrifuge tubes | Corning | 430291 | |
| 6-well plates | Corning | 3516 | |
| Aggrewell 800 | StemCell Technologies | 34811 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| BD FACS | Becton Dickinson | 338960 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| Eppendorf Cell Imaging Slides | Eppendorf | 30742060 | |
| High-binding 96-well plates | Corning | 3366 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Reversible Strainer | StemCell Technologies | 27215 | |
| SW32Ti rotor | Beckman Coulter | 369650 | |
| VWR® Disposable Serological Pipets, Glass, Nonpyrogenic | VWR | 93000-694 | |
| VWR® Vacuum Filtration Systems | VWR | 89220-694 | |
| xMark™ Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| Accutase | StemCell Technologies | 7920 | |
| Anti-Adherence Rinsing Solution | StemCell Technologies | 7010 | |
| Anti-FOXA2 Antibody | Abcam | Ab60721 | |
| Anti-Nestin Antibody | Abcam | Ab18102 | |
| Antibiotic-antimycotic solution, 100x | Sigma Aldrich | A5955-100ML | |
| B-27 Supplement (50x), minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| BES | Sigma Aldrich | B9879 - BES | |
| Bovine Albumin Fraction V (7.5% solution) | Thermo Fisher Scientific | 15260037 | |
| CHIR99021 | StemCell Technologies | 72052 | |
| Corning Matrigel hESC-Qualified Matrix | Corning | 08-774-552 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| DMEM-F12 | Lonza | 12-719 | |
| DMEM, high glucose media | Gibco | 11965 | |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | |
| EpiTect PCR Control DNA Set | Qiagen | 596945 | |
| EZ DNA Methylation Kit | Zymo Research | D5001 | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Gentamicin | Thermo Fisher Scientific | 15750078 | |
| Gentle Cell Dissociation Reagent | stemCell Technologies | 7174 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Human Recombinant bFGF | StemCell Technologies | 78003 | |
| Human Recombinant EGF | StemCell Technologies | 78006 | |
| Human Recombinant Shh (C24II) | StemCell Technologies | 78065 | |
| MEM Non-Essential Amino Acids Solution (100x) | Thermo Fisher Scientific | 11140050 | |
| mTeSR1 | StemCell Technologies | 85850 | |
| N-2 Supplement (100x) | Thermo Fisher Scientific | 17502001 | |
| Neurobasal Medium | Thermo Fisher Scientific | 21103049 | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| PyroMark PCR Kit | Qiagen | 978703 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| SB431542 | StemCell Technologies | 72232 | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| STEMdiff Neural Induction Medium | StemCell Technologies | 5835 | |
| STEMdiff Neural Progenitor Freezing Medium | StemCell Technologies | 5838 | |
| TaqMan Assay FOXA2 | Thermo Fisher Scientific | Hs00232764 | |
| TaqMan Assay GAPDH | Thermo Fisher Scientific | Hs99999905 | |
| TaqMan Assay Nestin | Thermo Fisher Scientific | Hs04187831 | |
| TaqMan Assay OCT4 | Thermo Fisher Scientific | Hs04260367 | |
| TaqMan Assay PPIA | Thermo Fisher Scientific | Hs99999904 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| Y27632 | StemCell Technologies | 72302 | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Goat serum, Sterile, 10 mL | Sigma | G9023 | Working concentration 1:1000 |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Normal mouse serum, Sterile, 500 mL | Equitech-Bio | SM30-0500 | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| pMD2.G | Addgene | 12253 | |
| pRSV-Rev | Addgene | 52961 | |
| psPAX2 | Addgene | 12259 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsmBI | New England Biolabs | R0580S | |
| BsrGI | New England Biolabs | R0575S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
Riferimenti
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Gaj, T., Gersbach, C. A., Barbas, C. F. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology. 31 (7), 397-405 (2013).
- Thakore, P. I., Black, J. B., Hilton, I. B., Gersbach, C. A. Editing the epigenome: technologies for programmable transcription and epigenetic modulation. Nature Methods. 13 (2), 127-137 (2016).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Gilbert, L. A., et al. Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. Cell. 159 (3), 647-661 (2014).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Chavez, A., et al. Highly efficient Cas9-mediated transcriptional programming. Nature Methods. 12 (4), 326-328 (2015).
- Horlbeck, M. A., et al. Nucleosomes impede Cas9 access to DNA in vivo and in vitro. eLife. 5, (2016).
- Chavez, A., et al. Comparison of Cas9 activators in multiple species. Nature Methods. 13 (7), 563-567 (2016).
- Zhou, H., et al. In vivo simultaneous transcriptional activation of multiple genes in the brain using CRISPR-dCas9-activator transgenic mice. Nature Neuroscience. 21 (3), 440-446 (2018).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell. 159 (3), 635-646 (2014).
- Konermann, S., et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517 (7536), 583-588 (2015).
- Holtzman, L., Gersbach, C. A. Editing the Epigenome: Reshaping the Genomic Landscape. Annual Review of Genomics and Human Genetics. , (2018).
- Perez-Pinera, P., et al. Synergistic and tunable human gene activation by combinations of synthetic transcription factors. Nature Methods. 10 (3), 239-242 (2013).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nature Methods. 12 (12), 1143-1149 (2015).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- McDonald, J. I., et al. Reprogrammable CRISPR/Cas9-based system for inducing site-specific DNA methylation. Biology Open. 5 (6), 866-874 (2016).
- Huang, Y. H., et al. DNA epigenome editing using CRISPR-Cas SunTag-directed DNMT3A. Genome Biology. 18 (1), 176 (2017).
- Vojta, A., et al. Repurposing the CRISPR-Cas9 system for targeted DNA methylation. Nucleic Acids Research. 44 (12), 5615-5628 (2016).
- Razin, A., Kantor, B. DNA methylation in epigenetic control of gene expression. Progress in Molecular and Subcellular Biology. 38, 151-167 (2005).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18786-18791 (2009).
- Kantor, B., et al. Downregulation of SNCA Expression by Targeted Editing of DNA Methylation: A Potential Strategy for Precision Therapy in PD. Molecular Therapy. , (2018).
- Jowaed, A., Schmitt, I., Kaut, O., Wullner, U. Methylation regulates alpha-synuclein expression and is decreased in Parkinson's disease patients' brains. Journal of Neuroscience. 30 (18), 6355-6359 (2010).
- Wang, Y., et al. A DNA methyltransferase inhibitor, 5-aza-2'-deoxycytidine, exacerbates neurotoxicity and upregulates Parkinson's disease-related genes in dopaminergic neurons. CNS Neuroscience & Therapeutics. 19 (3), 183-190 (2013).
- Matsumoto, L., et al. CpG demethylation enhances alpha-synuclein expression and affects the pathogenesis of Parkinson's disease. PLOS One. 5 (11), e15522 (2010).
- Desplats, P., et al. Alpha-synuclein sequesters Dnmt1 from the nucleus: a novel mechanism for epigenetic alterations in Lewy body diseases. Journal of Biological Chemistry. 286 (11), 9031-9037 (2011).
- Ai, S. X., et al. Hypomethylation of SNCA in blood of patients with sporadic Parkinson's disease. Journal of the Neurological Sciences. 337 (1-2), 123-128 (2014).
- Tagliafierro, L., Chiba-Falek, O. Up-regulation of SNCA gene expression: implications to synucleinopathies. Neurogenetics. 17 (3), 145-157 (2016).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Molecular Therapy - Methods & Clinical Development. 5, 153-164 (2017).
- Tagliafierro, L., et al. Genetic analysis of alpha-synuclein 3' untranslated region and its corresponding microRNAs in relation to Parkinson's disease compared to dementia with Lewy bodies. Alzheimer’s & Dementia. 13 (11), 1237-1250 (2017).
- Tagliafierro, L., Zamora, M. E., Chiba-Falek, O. Multiplication of the SNCA locus exacerbates neuronal nuclear aging. Human Molecular Genetics. , (2018).
- Kantor, B., McCown, T., Leone, P., Gray, S. J. Clinical applications involving CNS gene transfer. Advances in Genetics. 87, 71-124 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Advances in Genetics. 87, 125-197 (2014).
- Vijayraghavan, S., Kantor, B. A Protocol for the Production of Integrase-deficient Lentiviral Vectors for CRISPR/Cas9-mediated Gene Knockout in Dividing Cells. Journal of Visualized Experiments. (130), e56915 (2017).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Molecular Therapy. 16 (12), 1968-1976 (2008).
- Bassil, C. F., Huang, Z., Murphy, S. K. Bisulfite pyrosequencing. Methods in Molecular Biology. 1049, 95-107 (2013).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Research. 43 (13), 6450-6458 (2015).
- Nishimasu, H., et al. Crystal Structure of Staphylococcus aureus Cas9. Cell. 162 (5), 1113-1126 (2015).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. Journal of Virology. 68 (4), 2632-2648 (1994).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. Journal of Virology. 71 (8), 6113-6127 (1997).
- Goffin, V., et al. Transcription factor binding sites in the pol gene intragenic regulatory region of HIV-1 are important for virus infectivity. Nucleic Acids Research. 33 (13), 4285-4310 (2005).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. Journal of Biological Chemistry. 280 (22), 21545-21552 (2005).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Molecular Therapy. 19 (3), 547-556 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon