Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Vecteur lentiviral plate-forme pour la livraison efficace des outils d’édition épigénome dans l’homme induit des modèles de maladies dérivées de cellules souches pluripotentes
Dans cet article
Résumé
La cible ADN épigénome édition représente une approche thérapeutique efficace. Ce protocole décrit la production, purification et concentration des vecteurs lentiviraux all-in-one, hébergeant le transgène CRISPR-dCas9-DNMT3A pour applications d’édition épigénome dans les cellules souches humaines pluripotentes induites (hiPSC)-dérivé de neurones.
Résumé
L’utilisation de cellules dérivées d’hiPSC représente une approche utile pour étudier les maladies neurodégénératives humaines. Nous décrivons ici un protocole optimisé pour la différenciation des hiPSCs provenant d’un patient présentant la triplication du locus du gène (SNCA) d’alpha-synucléine dans la maladie de Parkinson (MP)-les populations neuronales dopaminergiques pertinentes. Accumuler des preuves a montré que des niveaux élevés de SNCA sont responsables pour le développement de PD. reconnaissant la non satisfaits ont besoin d’établir de nouvelles approches thérapeutiques pour PD, en particulier celles qui visent la régulation de l’expression SNCA , nous récemment mis au point un système CRISPR/dCas9-méthylation-basées sur l’ADN pour moduler épigénétiquement SNCA transcription en enrichissant les niveaux méthylation de la région régulatrice de SNCA intron 1. Pour fournir le système, composé d’un mort (désactivée) version de Cas9 (dCas9) a fusionné avec le domaine catalytique de l’ADN méthyltransférase enzyme 3 a (DNMT3A), un vecteur lentiviral est utilisé. Ce système est appliqué aux cellules avec la triplication du locus SNCA et réduit le SNCA-ARNm et protéines d’environ 30 % par le biais de la méthylation de l’ADN ciblée de SNCA intron 1. La diminution de l’expression affinée des niveaux SNCA sauve des phénotypes cellulaires liés à la maladie. Dans le protocole actuel, notre objectif est de décrire une procédure étape par étape afin de différencier les hiPSCs dans les cellules progénitrices neurales (PNJ) et l’établissement et la validation des tests pyrosequencing pour évaluer le profil de méthylation dans le SNCA intron 1. Pour connaître plus en détail le système de lentivirus-CRISPR/dCas9 utilisé dans ces expériences, ce protocole décrit comment produire, purifier et concentrer les vecteurs lentiviraux et de mettre en évidence leurs qualités pour les applications d’édition épigénome et génome utilisant hiPSCs et PNJ. Le protocole est facilement adaptable et peut être utilisé pour produire des lentivirus titre élevé pour les applications in vitro et in vivo.
Introduction
Plusieurs plates-formes d’édition épigénome ont été récemment mis au point pour cibler toutes les séquences d’ADN dans les régions qui contrôlent l’expression de gène1,2. Les outils de retouche épigénome créés sont conçus pour (i) règlent la transcription, (ii) modifier les modifications post-traductionnelles histone, (iii) modifier la méthylation de l’ADN et (iv) modulent les interactions élément régulateur. L’approche d’ancrer les modificateurs de transcription/chromatine à un Cas9 (morts) désactivé (dCas9) proviendraient précédemment développés plates-formes édition épigénome comme zinc finger protéines (ZFP) et transcription activator comme effecteurs (contes), héberger un domaine effecteur transcriptionnelle puissant (ED) fondue vers le domaine de liaison à l’ADN conçu (DBD)3. Les résultats du phénotype désiré tels que l’activation ou la répression est défini par la molécule effectrice ancrée aux loci endogène (Figure 1). Pour créer des activateurs transcriptional programmables, dCas9/gRNA modules sont liés à VP164,5,6 (Figure 1 a), un domaine d’activation virale qui recrute Pol II et la machinerie de transcription généraux. La modification de ce système a inclus VP64, un tétramère de domaines VP16, fournissant une encore plus robustes activation tarifaire5,6. Le système a été avec succès employé pour activer des régions codantes et non codantes en ciblant des promoteurs et des éléments de régulation. Ce qui est important, même si les molécules de VP64 ne modifient pas directement la structure de la chromatine dans la région de la cible, il recrute les modificateurs de la chromatine qui lie les résultats dans les dépôts des marques actives (euchromatine), y compris comme l’acétylation H3/H4 et H3-K4 di / tri-méthylation5,6. En plus de la VP64, la sous-unité p65 de NF-κB humain complexe a été attachée à la dCas9/gRNA module7. Fait intéressant, l’attachement de ces effecteurs dans les régions en amont des sites de début de transcription (TSSs) et au sein des promoteurs se traduit par une induction forte gène. Néanmoins, VP64 et p65 Effecteurs peuvent aussi exercer les effets activatrices tout en étant liés aux régions situées en aval du TSSs et exhausteurs distale7,8. Pour obtenir une réponse transcriptionnelle plus robuste, multiples fusions dCas9-VP64 ou dCas9-p65 doivent être recrutés à une cible unique locus9,10. Par conséquent, le développement récent des activateurs de nouvelle génération, qui recrutent plusieurs domaines effecteurs par un complexe unique dCas9-gRNA, tels que SunTag, a donné lieu à une plus forte capacité activation comparant à dCas9-VP64 fusion homologues11 , 12. une meilleure activation de la transcription a été obtenue par la fusion de VP64, p65 et Rta (VPR), un domaine de transactivation de gamma-virus de l’herpès, à l’extrémité C-terminale de la dCas913 (Figure 1 a). Des systèmes similaires de CRISPR/dCas9 ont été développés pour répression spécifique à la cible (Figure 1 b).
Répression génique endogène peut être réalisée avec les fusions de répresseur machiné par le biais de divers mécanismes (Figure 1 b). Il a été démontré que les systèmes CRISPR/dCas9, liés à la répresseur DBD (même sans un domaine effecteur/s), peuvent taire efficacement l’expression des gènes tandis qu’attachés à un promoteur ou en amont/en aval-TSS régions3,6 ,14. Les effets sur la transcription est causé par l’interférence stérique de la liaison de facteur de transcription et de la transformation de l’ARN polymérase. Néanmoins, des approches plus globales sont nécessaires, car la répression génique par stérique seul n’est souvent pas suffisante pour silencieux d’échappement robuste. Le développement récent de la prochaine génération de silencieux basées sur des systèmes CRISPR/dCas9 transportant des domaines répresseur transcriptionnel (DRT), modificateurs d’histone (H3-K9 di-/ tri-méthylation, H3-K27 di-/ tri-méthylation ; H3-K36 di-/ tri-méthylation, H3/H4 désacétylation) et la méthylation de l’ADN (CpG) a conduit à la construction d’outils épigénétiques permettant plus robustes pour faire taire les effets4,5,15,16, 17,18,19,20. Il a été démontré que le recrutement de ces modificateurs épigénétiques de l’ADN peut conduire à la formation de plus la chromatine condensée et fermée, qui génèrent habituellement un plus puissant silencieux issue21,22. Le plus souvent silencieux domaine utilisé avec DBDs est la boîte Krüppel-associés (KRAB)4,5. Le recrutement du facteur a été démontré pour correspondre avec les modifications de la chromatine ; Néanmoins, les mécanismes de ces modifications doivent encore être élucidé16,17,18. Récemment, il a été démontré que la localisation de KRAB à l’ADN peut promouvoir l’Assemblée de l’histone-méthyltransférase SETDB1 et les complexes de NuRD histone désacétylation (HDAC), ce qui suggère la possibilité que ces interactions médient la formation de condensation de la chromatine et transcriptionnelle silencieux3,13. Comme approche alternative, domaines Effecteurs peuvent être fusionnés à DBDs à créer une protéine silencieux épigénétique personnalisée. Ce système directement catalyse répressif ADN marques ou modifications d’histone.
Récemment, l’utilisation de systèmes CRISPR/dCas9 synthétiques attaché à l’enzyme DNMT3A a été réaffectée transcriptionnelle de désactivation. DNMT3A catalyse la méthylation de l’ADN qui exerce une répression transcriptionnelle tout au long de la formation de l’hétérochromatine sur les promoteurs de gènes endogènes et autres régions régulatrices (Figure 1 b)18,20. McDonald et al.,18 et20 de la Vojta et coll. ont été les premiers auteurs de déclarer que la méthylation de l’ADN peut être utilisée pour épigénome-gene silencing ou de répression, ce qui démontre que le système de fusion de plasmide-prononcé dCas9-DNMT3A peut renforcer puissamment méthylation de la cytosine autour les TSS18,20. McDonald et ses collaborateurs ont démontré que l’emploi de la stratégie peut-être aboutir à une réduction significative (environ 40 %) un gène suppresseur de la tumeur, CDKN2A ARNm du niveau18. De même, ciblant la région non méthylé promoteur des gènes BACH ou IL6ST montre méthylation accrue de CpG qui a été corrélée avec une diminution de double dans l' expression de gène20. Notre laboratoire a récemment réaffecté l’utilisation de méthylation de l’ADN pour atténuer les résultats pathologiques du SNCA surexpression (Figure 2)23. La stratégie repose sur l’amélioration sélective dans la méthylation de l’ADN dans la région d’intron 1 SNCA , comme il a été signalé auparavant comme hypométhylés en PD et démence avec Lewy organes (DLB) cerveaux24,25, 26. Cette hypométhylation a été liée à la surexpression SNCA , offrant ainsi une cible attractive pour une intervention thérapeutique24,27,28. Nous avons récemment montré un faible niveau de méthylation de l’ADN dans l’intron SNCA 1 région hiPSC dérivés dopaminergiques PNJs obtenus chez un patient de PD avec le SNCA triplication23. L’avantage de ce modèle expérimental est que les PNJ peuvent être robuste propagées en culture ou encore se différencient en neurones matures, ce qui permet un dépistage efficace identifier les facteurs génétiques qui véhiculent des phénotypes cellulaires, y compris oxydant stress et apoptose29. En outre, ce système modèle permet aux scientifiques de récapituler les événements du développement qui a eu lieu avant l’apparition des symptômes chez les patients. En outre, les dérivés hiPSC PNJ représente un excellent outil pour tester les voies moléculaires et cellulaires associées à l’expression des gènes. Ce qui est important, dérivés hiPSC PNJ combinée à la technologie CRISPR/Cas9-épigénome state-of-the-art peut faciliter considérablement le développement de « médicaments de nouvelle génération » pour beaucoup de maladies neurodégénératives.
Pour réduire les niveaux pathologiques d’expression SNCA, nous avons récemment mis au point un système de lentivirus portant une protéine de fusion dCas9-DNMT3A et gRNA à spécifiquement la méthylation CpG cible au sein de l’intron SNCA 1 (Figure 2 a)23. Ce protocole décrit vecteur lentiviral (LV) conception et réalisation en détail. LVs représentent un moyen efficace de prestation des modules CRISPR/dCas9 pour plusieurs raisons, à savoir (i) leur capacité à effectuer de volumineux ADN insère, (ii) un rendement élevé de transduction d’un large éventail de cellules, y compris les cellules ne se divisant pas tant démarcation30 et (iii) leur capacité à induire des réponses cytotoxiques et immunogènes minimales. Récemment, nous avons appliqué le système de LV de neurones dopaminergiques hiPSC dérivé d’un patient ayant la triplication du locus SNCA et démontré le potentiel thérapeutique de LVs pour la livraison de l’épigénome-édition méthylation outils23 ( Figure 2 b). En effet, un système de LV-gRNA/dCas9-DNMT3A provoque une augmentation significative dans la méthylation de l’ADN à la région d’intron 1 SNCA . Cette augmentation correspond à la réduction des niveaux d’ARNm et protéines SNCA 23. En outre, SNCA downregulation sauve des phénotypes liés PD dans les SNCA triplication/hiPSC-dérivés dopaminergiques neurones (c'est-à-dire mitochondrial ROS production et cellulaires la viabilité)23. Ce qui est important, nous avons démontré que la réduction SNCA expression par le système de LV-gRNA-dCas9-DMNT3A est capable d’inverser les phénotypes qui sont caractéristiques des neurones dopaminergiques dérivés hiPSC provenant d’un patient de PD qui portaient la SNCA triples emplois, tels que mitochondries ROS production et cellule viabilité23. Le but du présent protocole est 1) pour décrire le protocole de production et de la concentration d’une plate-forme de LV optimisée pour générer des préparations virales haute-titrés et 2) pour décrire la différenciation des hiPSCs en PNJ modelé pour devenir mature dopaminergique neurones31,32 et la caractérisation des niveaux méthylation de la région ciblée au sein de l’intron SNCA 1.
Plates-formes des gènes ont un avantage majeur sur la plus populaire plateforme de vecteur, à savoir virus adeno-associé vecteurs (AAVs), qui est la capacité de ce dernier pour accueillir les plus grands inserts génétique33,34. AAVs peuvent être générés à des rendements significativement plus élevés mais possèdent une capacité d’empaquetage faible (< 4,8 kb), compromettre leur utilisation pour fournir des systèmes CRISPR/Cas9 all-in-one. Il semble donc que la VL serait la plateforme de choix dans les applications impliquées dans la fourniture d’outils CRISPR/dCas9. Par conséquent, le protocole décrit ici sera un outil précieux pour les chercheurs souhaitant dispenser efficacement l’épigénome-montage de composants aux organes et cellules. De plus, le protocole décrit la stratégie visant à accroître les capacités de production et d’expression des vecteurs via une modification en cis des éléments dans le vecteur expression cassette30,35. La stratégie est basée sur le roman système mis au point dans notre laboratoire et études met en évidence sa capacité à produire des particules virales dans la gamme de 1010 unités virale (VU) / ml30,,35.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. conception et Production de Virus
- Construction et conception de plasmide
Remarque : La construction d’un vecteur de LV-gRNA-dCas9-DNMT3A all-in-one est effectuée au moyen d’une production- et cassette d’expression expression-optimisée publiés par Ortinski et al.,30. La cassette de vector porte une répétition du site reconnaissance du facteur de transcription Sp1 et une suppression de l’état-of-the-art dans le non traduites (U3') région d’un terminal 3' long répète (LTR) (Figure 2 a)30,36. L’épine dorsale vector s’est avéré pour être efficace dans la prestation et exprimant CRISPR/Cas930,35.- Obtenir la version de désactiver (mortes) de SpCas9 (dCas9) par l’intermédiaire de mutagenèse (données non présentées). Remplacer le clone abritent de mutations D10A et H840A HNH et RuvC domaines catalytiques de l’enzyme, respectivement, de l’actif Cas9 pBK30129 en échangeant entre les fragments AgeI-BamHI (Figure 3).
- Le domaine catalytique DNMT3A dérivent de pdCas9-DNMT3A-eGFP (voir Table des matières) en amplifiant la DNMT3A portion BamHI-429/R 5 '- GAGCGGATCCCCCTCCCG - 3' 5 429/BamHI-l' - CTCTCCACTGCCGGATCCGG - 3' (Figure 3). Pour amplifier la région contenant DNMT3A, utiliser les conditions suivantes : (1) 95 ° C pendant 60 s, (2) 95 ° C pendant 10 s, (3) 60 ° C pendant 20 s, (4) 68 ° C pendant 60 s. Repeat conditions de 2 à 4 x 30. Pour l’extension finale, utilisez 68 ° C pendant 3 min et tenir à 4 ° C.
- Cloner le fragment DNMT3A, digéré par une enzyme de restriction BamHI, dans le site BamHI du vecteur mis à jour le pBK301 portant des dCas9. Vérifier le clonage par Sanger direct séquençage. Notez que le plasmide a entraîné ports dCas9-DNMT3A-p2a-puromycine transgène. Le plasmide exprime gRNA échafaudage humain promoteur U6 (Figure 3).
- Remplacer le gène rapporteur de puromycine avec la protéine fluorescente verte (GFP) pour créer des dCas9-DNMT3A-p2a-GFP. Digérer le plasmide dCas9-DNMT3A-p2a-Puro avec FseI. Purifier le fragment de vecteur à l’aide d’une méthode de purification du gel. Préparer l’insertion de digestion pBK201a (pLenti-GFP) avec FseI. Cloner le fragment de FseI dans le vecteur. Le plasmide a entraîné pBK539 ports dCas9-DNMT3A-p2a-GFP transgène (Figure 3).
- Cultivant les cellules HEK-293 t et cellules pour la transfection de placage
Remarque : Rénales embryonnaires humaines 293 t (HEK-293 t) les cellules sont cultivées en complet haute-glucose Dulbecco de l’aigle modifié (DMEM ; 10 % de sérum de veau, 1 x antibiotique-antimycosiques, pyruvate de sodium 1 x, 1 x non essentiels acides aminés, 2 mM de L-glutamine) à 37 ° C, avec 5 % CO 2. pour la reproductibilité du protocole, il est recommandé de tester le sérum de veau lors du passage d’un lot/lot différent. Jusqu'à six 15 cm plaques sont nécessaires pour la production des gènes.- Basse-passage des cellules permet de commencer une nouvelle culture (inférieur à passage 20). Une fois les cellules atteignent la confluence de 90 à 95 %, aspirer les médias et le laver doucement avec stérile 1 x solution saline tamponnée au phosphate (PBS).
- Ajouter 2 mL de trypsine-EDTA (0,05 %) et il incuber à 37 ° C pendant 3 à 5 min. Pour désactiver le réactif de dissociation, ajoutez 8 mL de DMEM complet de haut-glucose et pipette 10 x x-15 avec une pipette sérologique de 10 mL pour créer une suspension monocellulaire de 4 x 106 cellules/mL.
- Pour les transfections, enduire plaques de 15 cm avec de la gélatine de 0,2 %. Ajouter 22,5 mL du milieu de la haute-glucose et les cellules des graines en ajoutant 2,5 mL de suspension cellulaire (total ~ 1 x 107 cellules/plaque). Incuber les plaques à 37 ° C, avec 5 % de CO2 jusqu’au confluent de 70 à 80 % est atteint.
- Transfection des cellules HEK-293 t
- Préparer 2 x solution tamponnée BES BBS et 1 M CaCl2, selon doudou et Kantor35. Filtrer les solutions en les passant à travers un filtre de 0,22 µm et les stocker à 4 ° C. Le mélange de transfection doit être clair avant son ajout aux cellules. Si le mélange devient trouble durant l’incubation, préparer les frais 2 x BBS (pH = 6,95).
- Pour préparer le mélange de plasmide, utilisez les quatre plasmides figurant (le mélange suivant est suffisant pour une plaque de 15 cm) : 37,5 µg du vecteur CRISPR/dCas9-transfert (pBK492 [DNMT3A-Puro-NO-gRNA] ou pBK539 [DNMT3A-GFP-NO-gRNA]) ; 25 µg de pBK240 (psPAX2) ; 12,5 µg de pMD2.G ; 6,25 µg de pré-rinçage-rev (Figure 4 a). Calculer le volume des plasmides selon les concentrations et ajouter la quantité requise dans un tube conique de 15 mL. Ajouter 312,5 µL de 1 M CaCl2 et porter le volume final à 1,25 mL, stérile JJ-H2O. doucement ajouter 1,25 mL de 2 x BBS solution, tandis que de l’agitation du mélange. Incuber 30 min à température ambiante. Les cellules sont prêtes pour la transfection dès qu’ils sont 70 – 80 % anastomosé.
- Aspirer les médias et remplacez-le par 22,5 mL de DMEM de haut-glucose fraîchement préparée sans sérum. Ajouter 2,5 mL du mélange transfection goutte à chaque plaque de 15 cm. Agiter les plaques et incuber à 37 ° C, avec 5 % de CO2 pendant 2 – 3 h.
- Après 3 h, ajouter 2,5 mL (10 %) du sérum par plaque et incuber une nuit à 37 ° C, avec 5 % de CO2.
- Jour 1 après la transfection, observer les cellules pour s’assurer qu’il n’y aucune ou mort cellulaire minime et que les cellules forment une culture anastomosée (100 %).
- Modifier les médias en ajoutant 25 mL de sérum fraîchement préparée de haut-glucose DMEM et 10 % pour chaque plaque.
- Incuber à 37 ° C, avec 5 % de CO2 pendant 48 h.
- Récolte du virus
- Recueillir le liquide surnageant de toutes les cellules transfectées et leur piscine en tubes conique de 50 mL. Centrifuger à 400 – 450 x g pendant 10 min. filtrer le liquide surnageant à travers une unité de sous vide filtre 0,45 µm. Après filtration, le liquide surnageant peut être conservé à 4 ° C pour le stockage à court terme (jusqu'à 4 jours). Pour le stockage à long terme, préparer des aliquotes et les stocker à-80 ° C.
Remarque : Les préparations virales nonconcentrated sont censées être ~ 2 x 107 à 3 x 107 vu/mL (voir la section 1.5 pour la détermination du titre). Il est fortement recommandé de préparer des aliquotes à usage unique, étant donné que plusieurs cycles de gel-dégel seront traduira par une perte de 10 % à 20 % dans des titres fonctionnels.
- Recueillir le liquide surnageant de toutes les cellules transfectées et leur piscine en tubes conique de 50 mL. Centrifuger à 400 – 450 x g pendant 10 min. filtrer le liquide surnageant à travers une unité de sous vide filtre 0,45 µm. Après filtration, le liquide surnageant peut être conservé à 4 ° C pour le stockage à court terme (jusqu'à 4 jours). Pour le stockage à long terme, préparer des aliquotes et les stocker à-80 ° C.
- Concentration de particules virales
Remarque : Pour la purification, une méthode de double-saccharose en deux étapes impliquant une étapes de dégradé de saccharose et un coussin saccharose est réalisée (Figure 4 b).- Pour créer un gradient de sucrose, préparer les tubes coniques ultracentrifugation dans l’ordre suivant : 0,5 mL de 70 % de sucrose dans du PBS 1 x, 0,5 mL de 60 % de saccharose en DMEM, 1 mL de 30 % de saccharose en DMEM et 2 mL de 20 % de sucrose dans du PBS 1 x.
- Ajouter avec précaution, le surnageant, recueilli conformément à l’article 1.4, pour le dégradé. Étant donné que le volume total prélevé quatre plaques de 15 cm est de 100 mL, utiliser six tubes ultracentrifugation pour traiter le surnageant viral.
- Tout aussi distribuer le surnageant viral entre chaque tube d’ultracentrifugation. Pour éviter tout bris de tube lors de la centrifugation, remplir les tubes d’ultracentrifugation au moins trois quart de leur capacité de volume total. Équilibrer les tubes avec du PBS 1 x. Centrifuger les échantillons à 70 000 x g pendant 2 h à 17 ° C.
Remarque : Pour maintenir la couche de saccharose pendant les phases d’accélération et de décélération, permettent l’ultracentrifugeuse lentement accélérer et ralentir le rotor de 0 à 200 x g et de 200 à 0 x g pendant la première et la dernière 3 min de l’essaimage, respectivement. - Recueillir délicatement fractions de saccharose 30 % à 60 % dans des tubes propres (Figure 4 b). Ajouter 1 x vers le haut (froid) PBS dans 100 mL de volume total. Mélanger en pipettant également plusieurs fois.
- Soigneusement, stratifier la préparation virale sur un coussin de saccharose en ajoutant 4 mL de 20 % de sucrose (en solution de 1 PBS x) dans le tube. Continuer par pipetage ~ 20-25 mL de la solution virale par chaque tube. Remplissez les tubes avec du PBS 1 x si le volume de leur contenu est moins de trois quarts par tube. Équilibre judicieux entre les tubes. Centrifuger à 70 000 x g pendant 2 h à 17 ° C. Vider le liquide surnageant et inverser les tubes sur du papier absorbant afin de permettre le restant liquide s’écouler.
- Retirer tout le liquide en aspirant avec prudence le liquide restant. Notez que, à cette étape, les granulés contenant le virus sont à peine visibles sous forme de petites taches translucides. Ajouter 70 µL de solution 1 PBS x dans le premier tube à resuspendre le culot. Soigneusement la suspension, déposer et transférez-le vers le tube suivant jusqu'à ce que toutes les granules sont remises en suspension.
- Laver les tubes avec un supplémentaire 50 µL de 1 x PBS et mélanger comme indiqué précédemment. Notez que, à cette étape, le volume de la suspension définitive est ~ 120 µL et apparaît légèrement laiteux. Pour obtenir une suspension claire, procéder à une centrifugation de 60 s à 10 000 x g. Transférer le surnageant dans un nouveau tube, faire 5 µL d’extraits et les stocker à-80 ° C.
Remarque : Préparations de vecteur lentiviral sont sensibles à des cycles répétés de gel et de dégel. En outre, il est suggéré que les étapes restantes sont faits en culture tissulaire confinement ou dans des zones qualifiées en termes d’être à un niveau adéquat de normes de prévention des risques biotechnologiques (Figure 4 b) désignées.
- Quantification des titres viraux
Remarque : L’estimation des titres viraux est effectuée à l’aide de la méthode de test ELISA p24 immuno-enzymatique (p24gag ELISA) et selon le National Institutes of Health (NIH) AIDS Vaccine Program du protocole pour le test du VIH-1 p24 antigène Capture avec de légères modifications.- Utiliser 200 µL de 0,05 % de Tween 20 à froid 1 x PBS (PBS-T) pour laver les puits d’une plaque de 96 puits 3 x.
- Pour enduire la plaque, utilisez 100 µL d’anticorps monoclonaux anti-p24 dilué au 1:1,500 dans du PBS 1 x. Incuber la plaque pendant une nuit à 4 ° C.
- Préparer le réactif de blocage (1 % albumine de sérum [BSA] dans du PBS 1 x) et ajouter 200 µL à chaque puits pour éviter la liaison non spécifique. 200 µL de PBS-T permet de laver le bien 3 x pendant au moins 1 h à température ambiante.
- Procéder à la préparation de l’échantillon. Lorsque vous travaillez avec des préparations concentrées vector, diluer le vecteur au 1/100 en utilisant 1 µL de l’échantillon, 89 µL de JJ-H20 et 10 µL de Triton X-100 (à une concentration finale de 10 %). Pour les préparations nonconcentrated, diluer les échantillons à 01:10.
- Obtenir des normes de VIH-1 en utilisant une dilution en série double (la concentration initiale est de 5 ng/mL).
- Diluer les échantillons concentrés (préparés à l’étape 1.6.4) en milieu RPMI 1640 additionné de 0,2 % Tween 20 et 1 % de BSA pour obtenir 01:10, 000, 01:50, 000 et des dilutions de 1/250 000. De même, diluer les échantillons nonconcentrated (préparés à l’étape 1.6.4) en RPMI 1640 additionné de 0,2 % Tween 20 et 1 % de BSA d’établir 1/500, 1:2, 500 et des dilutions de 1:12,500.
- Ajouter les échantillons et étalons sur la plaque en géométrie. Incuber une nuit à 4 ° C.
- Le lendemain, laver les puits 6 x.
- Ajouter 100 µL de l’anticorps anti-p24 anticorps polyclonal de lapin, dilué à 1 : 1 000 RPMI 1640, 10 % sérum fœtal (SVF), 0,25 % de BSA et sérum de souris normales 2 % (NMS) et incuber à 37 ° C pendant 4 h.
- Laver les puits 6 x. Ajouter la peroxydase de raifort de anti-lapin goat IgG dilué au 1/10 000 en milieu RPMI 1640 additionné de sérum de chèvre normal de 5 %, 2 % NMS, BSA de 0,25 % et 0,01 % Tween 20. Incuber à 37 ° C pendant 1 h.
- Laver les puits 6 x. Ajoutez le substrat TMB peroxydase et incuber à température ambiante pendant 15 minutes.
- Pour arrêter la réaction, ajouter 100 µL de 1 N HCl. Dans un lecteur de microplaques, mesurer l’absorbance à 450 nm.
- Mesure de l’intensité de fluorescence journaliste
- La suspension virale permet d’obtenir une dilution en série 10 fois (à partir de 10-1 à 10-5) dans du PBS 1 x.
- Plaque de 5 x 105 HEK-293 t cellules dans chaque puits d’une plaque de 6 puits. Appliquer 10 µL de chaque dilution virale aux cellules et incuber à 37 ° C, avec 5 % de CO2 pendant 48 h.
- Passer à la cellule activée par fluorescence tri analyse (FACS) comme suit : détacher les cellules en ajoutant 200 µL de solution de trypsine-EDTA 0,05 %. Incuber les cellules à 37 ° C pendant 5 min et les remettre en suspension dans 2 mL de milieu DMEM (avec sérum). Prélever les échantillons dans un tube conique de 15 mL et centrifuger à 400 x g à 4 ° C. Resuspendre le culot dans 500 µL de PBS 1 x contre le rhume.
- Fixer les cellules en ajoutant 500 µL de paraformaldéhyde à 4 % (PFA) et incuber pendant 10 min à température ambiante.
- Centrifuger à 400 g à 4 ° C et resuspendre le culot dans 1 mL de PBS 1 x. Analyser l’expression de la GFP à l’aide d’un instrument de FACS, comme décrit dans Ortinski et al.,30. Pour déterminer le titre fonctionnel des virus, utilisez la formule suivante.

Ici,
TG = nombre de cellules de GFP-positives ;
TN = nombre total de cellules ;
N = nombre total de cellules transduits ;
V = volume utilisé pour la transduction (en microlitres).
- Comptage des cellules positives GFP
Remarque : Déterminer la multiplicité d’infection (MOI) qui est employée pour la transduction. Une large gamme de MOIs d’essai (de MOI = 1 pour MOI = 10).- Graines de 3 x 10-5 cellules5 HEK-293 t 4 x 10 / chaque puits d’une plaque de 6 puits.
- Quand les cellules atteignent > confluence de 80 %, leur transmettre avec le vecteur à la MOI d’intérêt.
- Incuber à 37 ° C, avec 5 % de CO2et contrôler le signal GFP dans les cellules pour 1 à 7 jours.
- Compter le nombre de cellules positives à GFP. Utiliser un microscope à fluorescence (objectif Plan 4 x, 0,1 N.A., un grossissement de 40 x) en utilisant un filtre GFP (longueur d’onde excitation = 470 nm, longueur d’onde d’émission = 525 nm). Untransduced cellules permet de définir la population témoin de cellules négatives GFP.
- Employer la formule suivante pour déterminer le titre fonctionnel du virus.
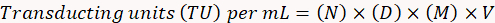
Ici,
N = nombre de cellules de GFP-positives ;
D = facteur de dilution ;
M = facteur de grossissement ;
V = volume du virus utilisé pour la transduction.
Remarque : Calculer les résultats suivant cet exemple : pour 10 cellules de GFP-positif (N) dénombrées à une dilution (D) de 10-4 (01:10, 000) à un grossissement de 20 x (M) dans un échantillon de 10 µL (V), la TU par millilitre sera (10 x 104) x (20) x (10) x (100) = 2 x 108 vu/mL.
2. différenciation des cellules progénitrices neurales dopaminergique
- Culture hiPSCs
Remarque : hiPSCs d’un patient ayant la triplication du locus SNCA, ND34391, ont été extraites de la National Institute of Neurological Disorders et catalogue Stroke (NINDS) (voir la Table des matières).- Culture hiPSCs dans des conditions d’alimentation indépendante dans un milieu culture ESC-iPSC sans chargeur (voir Table des matières) sur membrane de matrice de base complet CSEh (BMM)-revêtement des plaques (voir Table des matières). Laver les colonies confluentes avec 1 mL de DMEM/F12, ajouter 1 mL de réactif de dissociation (voir Table des matières) et incuber pendant 3 min à température ambiante.
- Aspirer le réactif de dissociation et ajouter 1 mL de milieu de culture exempt d’engraissement ESC-iPSC.
- Gratter la plaque à l’aide d’un poussoir de cellule et remettre en suspension les colonies en 11 mL de milieu de culture exempt d’engraissement ESC-iPSC de pipetage x-5 4 x à l’aide de pipettes de borosilicate.
- Plaque de 2 mL de suspension de colonie sur des plaques de revêtement BMM et placer la plaque à 37 ° C, avec 5 % de CO2. Effectuer un changement de moyen quotidien et fractionner les cellules tous les 5 – 7 jours.
- Différenciation des cellules progénitrices neurales dopaminergique
Remarque : La différenciation des hiPSCs dans des cellules progénitrices neurales dopaminergique (MD PNJs) est effectuée en utilisant un protocole moyen induction neurale disponibles dans le commerce par les instructions du fabricant, avec de légères modifications (de32 31, voir Table des matières). Le premier jour de la différenciation est considéré comme jour 0. HiPSCs de haute qualité sont nécessaires pour la différenciation neurale efficace. L’induction de PNJ MD est performedusing un corps embryoïdes (EB)-protocole basé.- Avant de commencer la différenciation des hiPSCs, préparer une plaque de microtitration culture (voir Table des matières) selon les instructions du fabricant.
- Après avoir préparé la plaque de microtitration culture, ajouter 1 mL de milieu d’induction neurale (NIM ; voir Table des matières) additionné de 10 µM Y-27632.
- Mettre la plaque de côté jusqu’au moment de l’utiliser.
- Laver hiPSCs avec DMEM/F12, ajouter 1 mL de solution de détachement cellulaire (voir Table des matières) et incuber pendant 5 min à 37 ° C, avec 5 % de CO2.
- Remettre en suspension les cellules individuelles en DMEM/F12 et eux centrifuger à 300 x g pendant 5 min.
- Soigneusement aspirer le surnageant et remettre en suspension les cellules de NIM + 10 µM Y-27632 pour obtenir une concentration finale de 3 x 106 cellules/mL.
- Ajouter 1 mL de la suspension de cellules individuelles dans un seul puits de la plaque de microtitration culture et centrifuger la plaque à 100 x g pendant 3 min.
- Examiner la plaque au microscope afin d’assurer une répartition égale des cellules entre les puits et incuber les cellules à 37 ° C, avec 5 % de CO2.
- Jours 1 à 4, effectuer un changement de moyen partiel quotidiens.
- À l’aide d’une micropipette de 1 mL, 1,5 mL du milieu de retirer et jeter. Lentement, ajouter 1,5 mL de NIM frais sans Y-27632.
- Répétez l’étape 2.2.10 jusqu’au jour 4.
- Jour 5, enduire un puits d’une plaque de 6 puits avec BMM.
- Placez une passoire réversible 37 µm (voir Table des matières) au sommet d’un tube conique de 50 mL (déchets). Pointer la flèche de la crépine réversible vers le haut.
- Retirez le support de la plaque de microtitration culture sans déranger l’EBs formé.
- Ajouter 1 mL de DMEM/F12 et rapidement percevoir l’EBs avec la pipette de borosilicate et filtrez-les à travers la passoire.
- Répétez l’étape 2.2.15 jusqu'à ce que tous les EBs sont retirés de la plaque de microtitration culture.
- Inverser la crépine sur un nouveau tube conique de 50 mL et ajouter 2 mL de NIM pour recueillir toute l’EBs.
- Plaque de 2 mL de la suspension EB dans un seul puits de la plaque de BMM-enduit à l’aide d’une pipette de borosilicate. Incuber l’EBs à 37 ° C, avec 5 % de CO2.
- Jour 6, préparer 2 mL de NIM + 200 ng/mL SHH (voir Table des matières) et effectuer un changement de moyen quotidien.
- Jour 8, examinez le pourcentage d’induction neuronal.
- Compter tout attaché EBs et, plus précisément, déterminer le nombre de chaque EB individuel qui est rempli de rosettes neuronales. Quantifier l’induction neurale rosette à l’aide de la formule suivante.
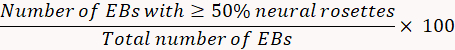
Remarque : Si l’induction neurale est < 75 %, sélection de rosette neuronaux peut-être être inefficace. - Jour 12, préparer 250 mL de milieu de N2B27 119 ml de neurobasal milieu, 119 mL de milieu DMEM/F12, 2,5 mL de GlutaMAX, 2,5 mL de NEAA, 2,5 mL de supplément de N2, 5 mL de B27 sans vitamine A, 250 μL de gentamicine (50 mg/mL) et 19. 66 μL de BSA (7 mg/mL).
- 50 mL de milieu N2B27 complet, ajoutez 3 μM CHIR99021, 2 μM SB431542, 20 ng/mL bFGF, 20 ng/mL EGF et 200 ng/mL SHH.
Remarque : Il est important de préparer le moyen juste avant utilisation. - Aspirer la moyenne provenant des puits contenant les rosettes neurones et laver avec 1 mL de DMEM/F12.
- Ad 1 mL de réactif de sélection rosette neuronal (voir Table des matières) et incuber à 37 ° C, avec 5 % de CO2 pendant 1 h.
- Retirer le réactif de sélection et, à l’aide d’une pipette de 1 mL, visent directement à des grappes de rosette.
- Ajouter la suspension d’un tube conique de 15 mL et répétez les étapes 2.2.25 et 2.2.26 jusqu'à la majorité des groupes neuronaux rosette ont été collectés.
Remarque : Pour éviter toute contamination avec des types cellulaires neuronaux, ne pas overselect. - Centrifuger la suspension de rosette à 350 x g pendant 5 min. aspirer le surnageant et remettre en suspension les rosettes neurales dans N2B27 + 200 ng/mL SHH. Ajouter la suspension neural rosette dans un puits BMM-enduit et incuber les plaques à 37 ° C, avec 5 % de CO2.
- Jours 13-17, effectuer un changement de moyen quotidien utilisant le milieu rempli de N2B27. Le passage des cellules lorsque les cultures sont confluents de 80 à 90 %.
- Pour fractionner les cellules, préparer une plaque BMM-enduit.
- Laver les cellules avec 1 mL de DMEM/F12, aspirer le milieu et ajouter 1 mL de réactif de dissociation (voir Table des matières).
- Incuber 5 min à 37 ° C, ajouter 1 mL de DMEM/F12 et déloger les cellules attachées par pipetage de haut en bas. Recueillir la suspension NPC dans un tube conique de 15 mL. Centrifuger à 300 x g pendant 5 min.
- Aspirer le surnageant et remettre en suspension les cellules dans 1 mL de N2B27 complet + 200 ng/mL SHH.
- Compter les cellules et leur plaque à une densité de 1,25 x 105 cellules/cm2et incuber les cellules à 37 ° C, avec 5 % de CO2.
- Modifier le milieu tous les jours, à l’aide de N2B27 complet + 200 ng/mL SHH.
Remarque : À ce passage, les PNJ est considérés comme passage (P) 0. SHH peut être retiré du milieu N2B27 à P2. - Le passage des cellules lorsqu’elles atteignent la confluence de 80 à 90 %.
- À ce stade, confirment que les cellules expriment des marqueurs de Nestin et FoxA2 en utilisant l’immunocytochimie et qPCR. Ce protocole permet la génération de 85 % des cellules double-positifs pour les marqueurs de Nestin et FoxA2.
- Pour le passage des cellules, répétez les étapes 2.2.31–2.2.36.
- Geler les cellules, à partir de passage P2. Pour la congélation des cellules, répétez les étapes 2.2.31–2.2.36 et resuspendre le culot cellulaire à 2 x 106 à 4 x 106 cellules/mL à l’aide de froid progénitrices neurales congélation moyen (voir Table des matières).
- Transférer 1 mL de la suspension cellulaire dans chaque cryovial et geler les cellules à l’aide d’un système de refroidissement lent-taux-controlled standard. Pour le stockage à long terme, garder les cellules dans l’azote liquide.
- Dégel des PNJs MD
- Préparer une plaque de revêtement BMM et chaleureux N2B27 complet. Ajouter 10 mL de DMEM/F12 chaudes dans un tube conique de 15 mL. Placez un cryovial dans un bloc chauffant de 37 ° C pendant 2 min.
- Les cellules de transfert de la cryovial dans le tube contenant DMEM/F12. Centrifuger à 300 x g pendant 5 min.
- Aspirer le surnageant et remettre en suspension les cellules dans 2 mL de N2B27 ajouter la suspension cellulaire à un puits de la plaque de revêtement BMM. Incuber les cellules à 37 ° C, avec 5 % de CO2.
- Préparer une plaque de revêtement BMM et chaleureux N2B27 complet. Ajouter 10 mL de DMEM/F12 chaudes dans un tube conique de 15 mL. Placez un cryovial dans un bloc chauffant de 37 ° C pendant 2 min.
3. transduction de PNJs MD et l’analyse des changements de méthylation
-
Transduction de PNJ MD
- Transduce MD PNJs à 70 % confluence avec LV-gRNA/dCas9-DNMT3A vecteurs en MOI = 2. Remplacer le posttransduction du milieu 16 h N2B27.
- Ajouter N2B27 avec 5 puromycine µg/mL, 48 h après la transduction. Culture des cellules pour 3 semaines en N2B27 plus de puromycine pour obtenir les lignes MD NPC stables. Les cellules sont prêtes pour des applications en aval (ADN, ARN, les analyses de protéines, caractérisation phénotypique23, congélation et passage).
-
Caractérisation du profil de méthylation de l’intron SNCA 1
- Extraire l’ADN de chaque cellule stablement transduite kit de la ligne à l’aide d’une extraction de l’ADN (voir Table des matières).
- Utilisation 800 ng d’ADN pour effectuer une conversion de bisulfite, utilisant un commercialement disponible nécessaire (voir la Table des matières). Après la conversion de bisulfite, éluer l’ADN bisulfite-converti à 20 ng/µL.
-
PCR pour l’analyse de pyrosequencing
- Préparer le mélange maître PCR dans un tube exempte de nucléase. Pour chaque réaction, utilisez 0,4 µL d’apprêt inverse (10 µM), 0,4 µL d’apprêt avant (10 µM), 1,6 µL de MgCl2 (25 mM), 2 µL de 10 x CoralLoad concentrer, 10 µL de 2 x PCR master mix, 4 µL de 5 x Q-Solution, 1 µL d’ADN et 0,6 µL d’eau exempte de nucléase.
- La plaque de la réaction de transfert d’un thermocycleur et effectuer de PCR avec les conditions suivantes : 95 ° C pendant 15 min, 50 cycles de 94 ° C pendant 30 s, 56 ° C pendant 30 s et 72 ° C pendant 30 s, avec une étape d’extension finale 10 min à 72 ° C. Amorces utilisées pour le pyrosequencing de l’intron SNCA 1 sont énumérés au tableau 1et Figure 7 a supplémentaire Figure 1.
- Après amplification, visualiser les amplicons utilisant 2 µL de produit de PCR avec coloration, le bromure d’éthidium après électrophorèse de gel d’agarose.
- Pyrosequencing tests sont validés à l’aide de mélanges de non méthylé (U) et méthylés DNAs de converti du bisulfite (M) dans les proportions suivantes, à savoir 100U:0M, 75U:25M, 50U:50M, 25U:75M et 0U:100M (voir la Table des matières).
- Conduite pyrosequencing utilisant des réactifs de pyrosequencing (voir Table des matières) et calculer les valeurs de méthylation pour chaque site de CpG en utilisant le logiciel pyrosequencing. Pour un protocole détaillé pyrosequencing, voir Ben et al.,37.
Access restricted. Please log in or start a trial to view this content.
Résultats
Valider les titres de la production des vecteurs LV-dCas9-DNMT3A-GFP/Puro par rapport à l’équivalent GFP naïf
Nous avons effectué p24gag ELISA à comparer entre les titres physiques de LV-dCas9-DNMT3A-GFP/Puro avec les homologues GFP/Puro naïf. Les résultats représentatifs, présentés dans la Figure 5 a, démontrent que les rendements physiques des vecteurs, gé...
Access restricted. Please log in or start a trial to view this content.
Discussion
LVs ont commencé à émerger en tant que véhicule de choix pour l’épigénome édition, surtout dans le contexte des maladies génétiques, principalement en raison de leur capacité à (i) accueillir grandes charges utiles de l’ADN et (ii) transduce efficacement un large éventail de diviser et les cellules ne se divisant pas. L’efficacité de grand emballage de la VL est particulièrement utile pour les applications impliquant des emballages des systèmes CRISPR/dCas9 qui sont surdimensionnés. Dans cette persp...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Duke University a déposé une demande de brevet provisoire relie à cette étude.
Remerciements
Ce travail a été financé en partie par le prix de développement de neurotechnologie Kahn (au décret) et la National instituts de santé/National Institute of Neurological Disorders et Stroke (NINDS/NIH) (R01 NS085011 à O.C.).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| 0.22 μM filter unit, 1 L | Corning | 430513 | |
| 0.45-μm filter unit, 500 mL | Corning | 430773 | |
| 100 mm TC-Treated Culture Dish | Corning | 430167 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 50 mL conical centrifuge tubes | Corning | 430291 | |
| 6-well plates | Corning | 3516 | |
| Aggrewell 800 | StemCell Technologies | 34811 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| BD FACS | Becton Dickinson | 338960 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| Eppendorf Cell Imaging Slides | Eppendorf | 30742060 | |
| High-binding 96-well plates | Corning | 3366 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Reversible Strainer | StemCell Technologies | 27215 | |
| SW32Ti rotor | Beckman Coulter | 369650 | |
| VWR® Disposable Serological Pipets, Glass, Nonpyrogenic | VWR | 93000-694 | |
| VWR® Vacuum Filtration Systems | VWR | 89220-694 | |
| xMark™ Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| Accutase | StemCell Technologies | 7920 | |
| Anti-Adherence Rinsing Solution | StemCell Technologies | 7010 | |
| Anti-FOXA2 Antibody | Abcam | Ab60721 | |
| Anti-Nestin Antibody | Abcam | Ab18102 | |
| Antibiotic-antimycotic solution, 100x | Sigma Aldrich | A5955-100ML | |
| B-27 Supplement (50x), minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| BES | Sigma Aldrich | B9879 - BES | |
| Bovine Albumin Fraction V (7.5% solution) | Thermo Fisher Scientific | 15260037 | |
| CHIR99021 | StemCell Technologies | 72052 | |
| Corning Matrigel hESC-Qualified Matrix | Corning | 08-774-552 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| DMEM-F12 | Lonza | 12-719 | |
| DMEM, high glucose media | Gibco | 11965 | |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | |
| EpiTect PCR Control DNA Set | Qiagen | 596945 | |
| EZ DNA Methylation Kit | Zymo Research | D5001 | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Gentamicin | Thermo Fisher Scientific | 15750078 | |
| Gentle Cell Dissociation Reagent | stemCell Technologies | 7174 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Human Recombinant bFGF | StemCell Technologies | 78003 | |
| Human Recombinant EGF | StemCell Technologies | 78006 | |
| Human Recombinant Shh (C24II) | StemCell Technologies | 78065 | |
| MEM Non-Essential Amino Acids Solution (100x) | Thermo Fisher Scientific | 11140050 | |
| mTeSR1 | StemCell Technologies | 85850 | |
| N-2 Supplement (100x) | Thermo Fisher Scientific | 17502001 | |
| Neurobasal Medium | Thermo Fisher Scientific | 21103049 | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| PyroMark PCR Kit | Qiagen | 978703 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| SB431542 | StemCell Technologies | 72232 | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| STEMdiff Neural Induction Medium | StemCell Technologies | 5835 | |
| STEMdiff Neural Progenitor Freezing Medium | StemCell Technologies | 5838 | |
| TaqMan Assay FOXA2 | Thermo Fisher Scientific | Hs00232764 | |
| TaqMan Assay GAPDH | Thermo Fisher Scientific | Hs99999905 | |
| TaqMan Assay Nestin | Thermo Fisher Scientific | Hs04187831 | |
| TaqMan Assay OCT4 | Thermo Fisher Scientific | Hs04260367 | |
| TaqMan Assay PPIA | Thermo Fisher Scientific | Hs99999904 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| Y27632 | StemCell Technologies | 72302 | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Goat serum, Sterile, 10 mL | Sigma | G9023 | Working concentration 1:1000 |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Normal mouse serum, Sterile, 500 mL | Equitech-Bio | SM30-0500 | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| pMD2.G | Addgene | 12253 | |
| pRSV-Rev | Addgene | 52961 | |
| psPAX2 | Addgene | 12259 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsmBI | New England Biolabs | R0580S | |
| BsrGI | New England Biolabs | R0575S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
Références
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Gaj, T., Gersbach, C. A., Barbas, C. F. 3rd ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology. 31 (7), 397-405 (2013).
- Thakore, P. I., Black, J. B., Hilton, I. B., Gersbach, C. A. Editing the epigenome: technologies for programmable transcription and epigenetic modulation. Nature Methods. 13 (2), 127-137 (2016).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Gilbert, L. A., et al. Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. Cell. 159 (3), 647-661 (2014).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Chavez, A., et al. Highly efficient Cas9-mediated transcriptional programming. Nature Methods. 12 (4), 326-328 (2015).
- Horlbeck, M. A., et al. Nucleosomes impede Cas9 access to DNA in vivo and in vitro. eLife. 5, (2016).
- Chavez, A., et al. Comparison of Cas9 activators in multiple species. Nature Methods. 13 (7), 563-567 (2016).
- Zhou, H., et al. In vivo simultaneous transcriptional activation of multiple genes in the brain using CRISPR-dCas9-activator transgenic mice. Nature Neuroscience. 21 (3), 440-446 (2018).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell. 159 (3), 635-646 (2014).
- Konermann, S., et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517 (7536), 583-588 (2015).
- Holtzman, L., Gersbach, C. A. Editing the Epigenome: Reshaping the Genomic Landscape. Annual Review of Genomics and Human Genetics. , (2018).
- Perez-Pinera, P., et al. Synergistic and tunable human gene activation by combinations of synthetic transcription factors. Nature Methods. 10 (3), 239-242 (2013).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nature Methods. 12 (12), 1143-1149 (2015).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- McDonald, J. I., et al. Reprogrammable CRISPR/Cas9-based system for inducing site-specific DNA methylation. Biology Open. 5 (6), 866-874 (2016).
- Huang, Y. H., et al. DNA epigenome editing using CRISPR-Cas SunTag-directed DNMT3A. Genome Biology. 18 (1), 176(2017).
- Vojta, A., et al. Repurposing the CRISPR-Cas9 system for targeted DNA methylation. Nucleic Acids Research. 44 (12), 5615-5628 (2016).
- Razin, A., Kantor, B. DNA methylation in epigenetic control of gene expression. Progress in Molecular and Subcellular Biology. 38, 151-167 (2005).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18786-18791 (2009).
- Kantor, B., et al. Downregulation of SNCA Expression by Targeted Editing of DNA Methylation: A Potential Strategy for Precision Therapy in PD. Molecular Therapy. , (2018).
- Jowaed, A., Schmitt, I., Kaut, O., Wullner, U. Methylation regulates alpha-synuclein expression and is decreased in Parkinson's disease patients' brains. Journal of Neuroscience. 30 (18), 6355-6359 (2010).
- Wang, Y., et al. A DNA methyltransferase inhibitor, 5-aza-2'-deoxycytidine, exacerbates neurotoxicity and upregulates Parkinson's disease-related genes in dopaminergic neurons. CNS Neuroscience & Therapeutics. 19 (3), 183-190 (2013).
- Matsumoto, L., et al. CpG demethylation enhances alpha-synuclein expression and affects the pathogenesis of Parkinson's disease. PLOS One. 5 (11), e15522(2010).
- Desplats, P., et al. Alpha-synuclein sequesters Dnmt1 from the nucleus: a novel mechanism for epigenetic alterations in Lewy body diseases. Journal of Biological Chemistry. 286 (11), 9031-9037 (2011).
- Ai, S. X., et al. Hypomethylation of SNCA in blood of patients with sporadic Parkinson's disease. Journal of the Neurological Sciences. 337 (1-2), 123-128 (2014).
- Tagliafierro, L., Chiba-Falek, O. Up-regulation of SNCA gene expression: implications to synucleinopathies. Neurogenetics. 17 (3), 145-157 (2016).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Molecular Therapy - Methods & Clinical Development. 5, 153-164 (2017).
- Tagliafierro, L., et al. Genetic analysis of alpha-synuclein 3' untranslated region and its corresponding microRNAs in relation to Parkinson's disease compared to dementia with Lewy bodies. Alzheimer’s & Dementia. 13 (11), 1237-1250 (2017).
- Tagliafierro, L., Zamora, M. E., Chiba-Falek, O. Multiplication of the SNCA locus exacerbates neuronal nuclear aging. Human Molecular Genetics. , (2018).
- Kantor, B., McCown, T., Leone, P., Gray, S. J. Clinical applications involving CNS gene transfer. Advances in Genetics. 87, 71-124 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Advances in Genetics. 87, 125-197 (2014).
- Vijayraghavan, S., Kantor, B. A Protocol for the Production of Integrase-deficient Lentiviral Vectors for CRISPR/Cas9-mediated Gene Knockout in Dividing Cells. Journal of Visualized Experiments. (130), e56915(2017).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Molecular Therapy. 16 (12), 1968-1976 (2008).
- Bassil, C. F., Huang, Z., Murphy, S. K. Bisulfite pyrosequencing. Methods in Molecular Biology. 1049, 95-107 (2013).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Research. 43 (13), 6450-6458 (2015).
- Nishimasu, H., et al. Crystal Structure of Staphylococcus aureus Cas9. Cell. 162 (5), 1113-1126 (2015).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. Journal of Virology. 68 (4), 2632-2648 (1994).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. Journal of Virology. 71 (8), 6113-6127 (1997).
- Goffin, V., et al. Transcription factor binding sites in the pol gene intragenic regulatory region of HIV-1 are important for virus infectivity. Nucleic Acids Research. 33 (13), 4285-4310 (2005).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. Journal of Biological Chemistry. 280 (22), 21545-21552 (2005).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Molecular Therapy. 19 (3), 547-556 (2011).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon